Regulacin de la expresin gnica Para que la
































- Slides: 69

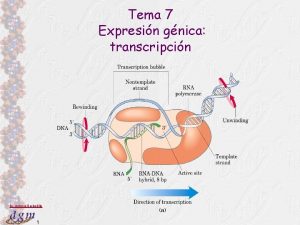
Regulación de la expresión génica Para que la información genética almacenada en el DNA sea utilizada por los organismos se requiere que sean sintetizadas cadenas de RNA complementarias al DNA por medio de un proceso conocido como transcripción (de “scribere”=escribir y “trans”= pasar de uno a otro, en este caso de una escritura a otra). Bioquímica III Prof. Humberto González M.

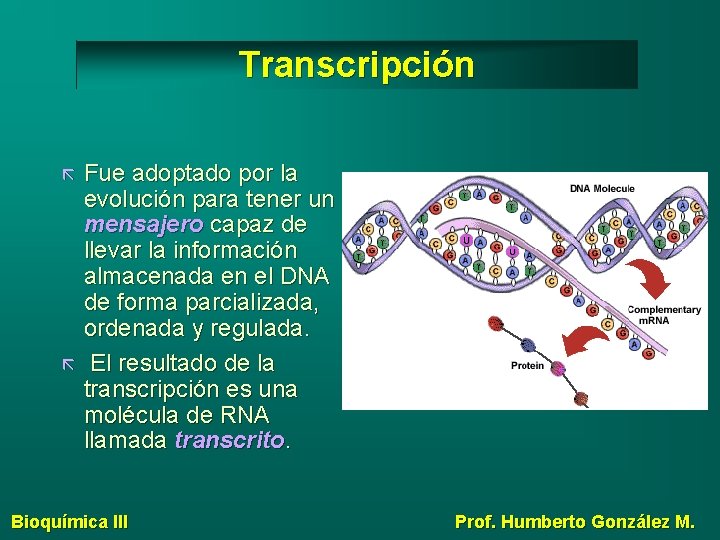
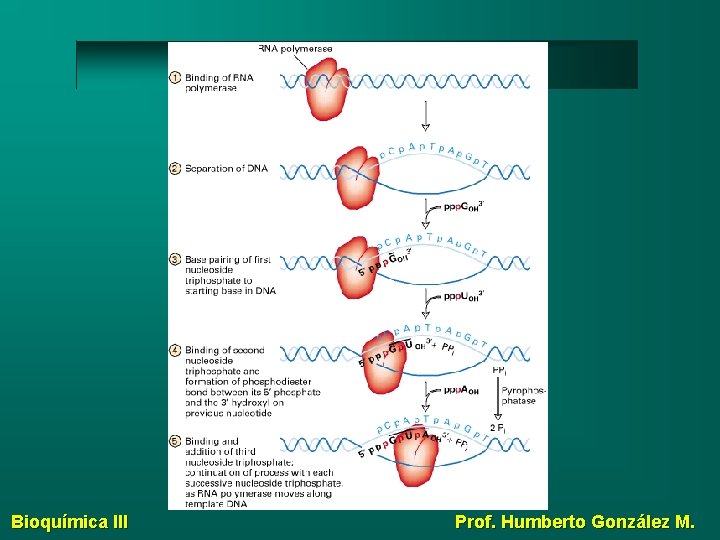
Transcripción Fue adoptado por la evolución para tener un mensajero capaz de llevar la información almacenada en el DNA de forma parcializada, ordenada y regulada. El resultado de la transcripción es una molécula de RNA llamada transcrito. Bioquímica III Prof. Humberto González M.

Inicio Bioquímica III Prof. Humberto González M.

Transcritos Cuando sirve como patrón para la síntesis de proteínas se conoce como RNA mensajero o RNAm; Cuando constituye parte de la estructura de los ribosomas, como RNA ribosomal o RNAr; Cuando participa en la síntesis de proteínas como adaptador de los aminoácidos, como RNA de transferencia o RNAt; o Puede tener varias otras actividades como enzimáticas, ribozima o actividades de regulación de la expresión genética, RNA de interferencia o RNAi. Bioquímica III Prof. Humberto González M.

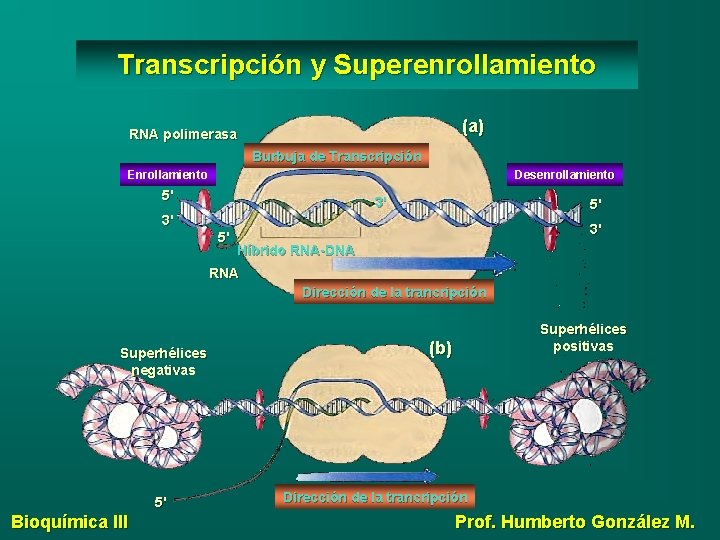
Transcripción y Superenrollamiento (a) RNA polimerasa Burbuja de Transcripción Enrollamiento Desenrollamiento 5' 3' Híbrido RNA-DNA RNA Dirección de la trancripción Superhélices negativas 5' Bioquímica III Superhélices positivas (b) Dirección de la trancripción Prof. Humberto González M.

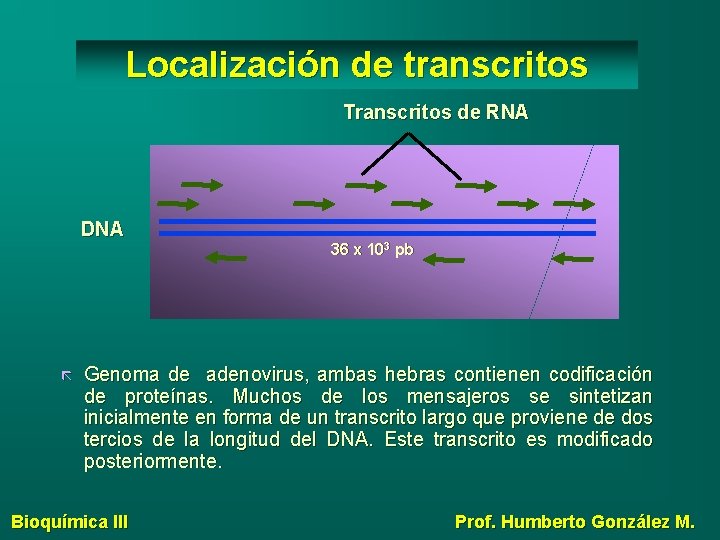
Localización de transcritos Transcritos de RNA DNA 36 x 103 pb Genoma de adenovirus, ambas hebras contienen codificación de proteínas. Muchos de los mensajeros se sintetizan inicialmente en forma de un transcrito largo que proviene de dos tercios de la longitud del DNA. Este transcrito es modificado posteriormente. Bioquímica III Prof. Humberto González M.

Nomenclatura de las hebras de DNA en la transcripción Hebra molde Hebra no molde Hebra menos (-) Hebra más (+) Hebra codante Hebra no codante Bioquímica III Prof. Humberto González M.

Ejemplo de complementariedad (5')CGCTATAGCTGTTT(3') Hebra de DNA no molde (+) (3')GCGATATCGACAAA(5') Hebra de DNA molde (-) (5')CGCUAUAGCUGUUU(3') Transcrito de RNA Bioquímica III Prof. Humberto González M.

RNA polimerasa Subunidad σ Núcleo de la enzima Bioquímica III Prof. Humberto González M.

Prokaryotic RNA polymerase structure RNA polymerase of E. coli is a multisubunit protein Subunit α β β’ σ Number 2 1 1 1 Role uncertain forms phosphodiester bonds binds DNA template recognizes promoter and facilitates initiation α 2ββ ’σ α 2ββ ’ holoenzyme core polymerase Bioquímica III + σ sigma factor Prof. Humberto González M.

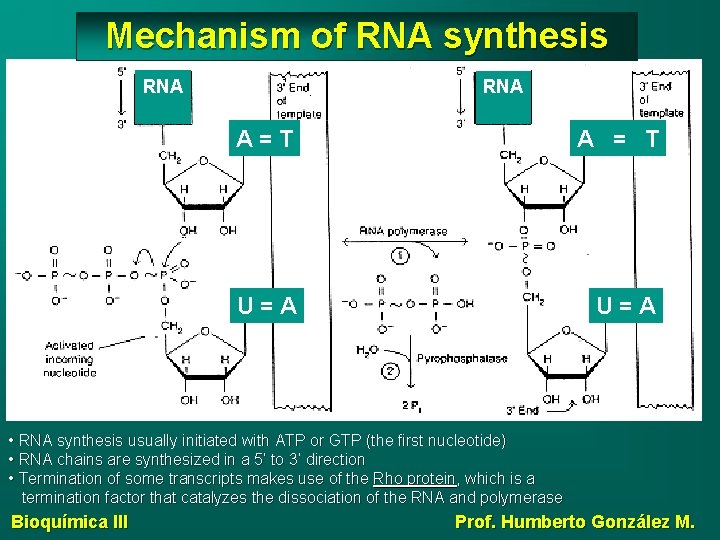
Mechanism of RNA synthesis RNA A=T A = T U=A • RNA synthesis usually initiated with ATP or GTP (the first nucleotide) • RNA chains are synthesized in a 5’ to 3’ direction • Termination of some transcripts makes use of the Rho protein, which is a termination factor that catalyzes the dissociation of the RNA and polymerase Bioquímica III Prof. Humberto González M.

The function of sigma factor the sigma subunit of RNA polymerase is an “initiation factor” there are several different sigma factors in E. coli that are specific for different sets of genes sigma factor functions to ensure that RNA polymerase binds stably to DNA only at promoters sigma destablizes nonspecific binding to non-promoter DNA sigma stabilizes specific binding to promoter DNA this accelerates the search for promoter DNA Bioquímica III Prof. Humberto González M.

The function of sigma factor Ka (M-1) Any DNA (nonspecific) Core 2 X 1011 Holo 1 X 107 Promoter DNA (specific) 1013 to 1015 • promoters vary in “strength” by ~two orders of magnitude Bioquímica III Prof. Humberto González M.

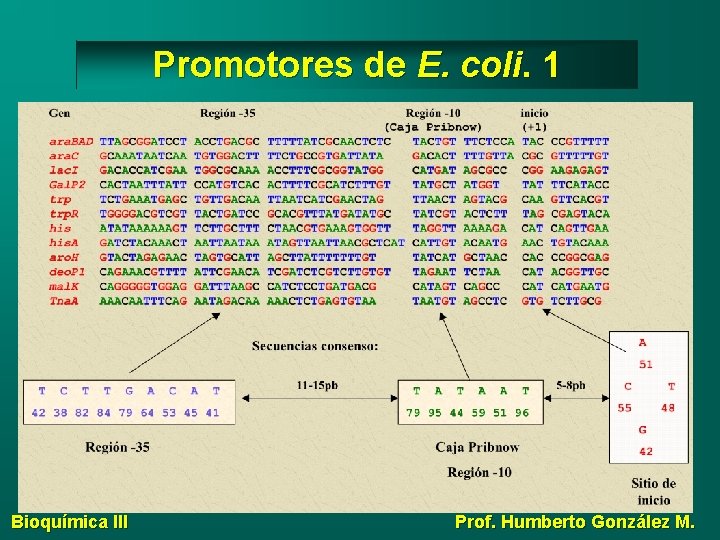
Promotores de E. coli. 1 Bioquímica III Prof. Humberto González M.

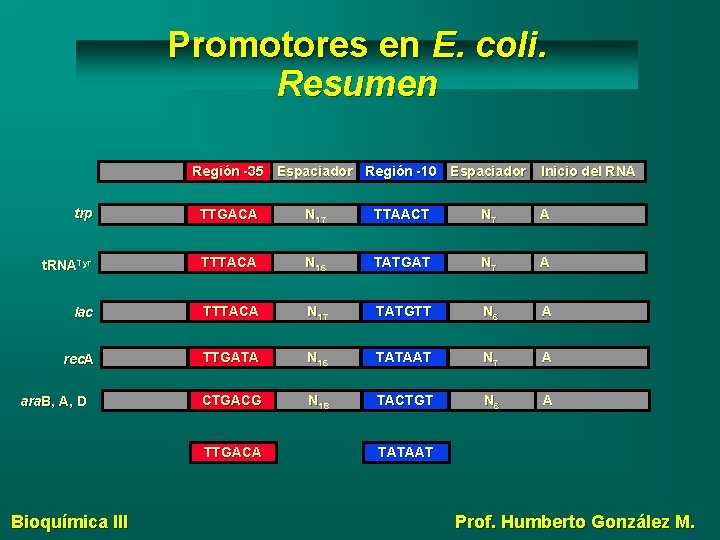
Promotores en E. coli. Resumen Región -35 Espaciador Región -10 Espaciador Inicio del RNA trp TTGACA N 17 TTAACT N 7 A t. RNATyr TTTACA N 16 TATGAT N 7 A lac TTTACA N 17 TATGTT N 6 A rec. A TTGATA N 16 TATAAT N 7 A CTGACG N 18 TACTGT N 6 A ara. B, A, D TTGACA Bioquímica III TATAAT Prof. Humberto González M.

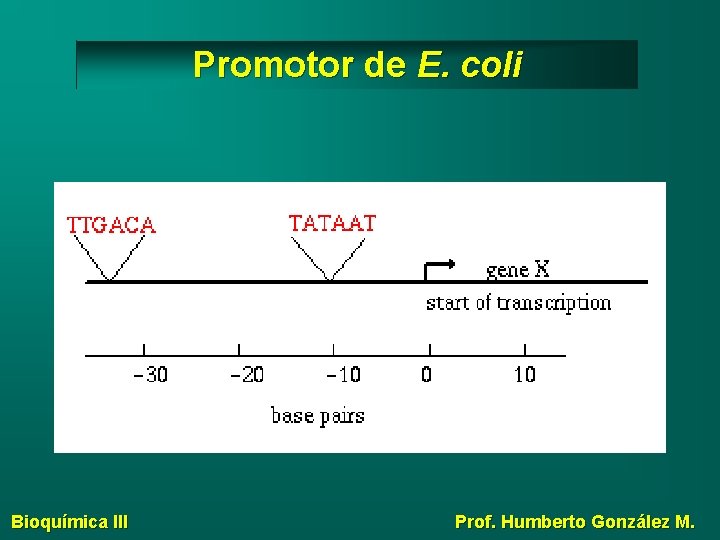
Promotor de E. coli Bioquímica III Prof. Humberto González M.

Promotores en E. coli con regulación lejana Bioquímica III Prof. Humberto González M.

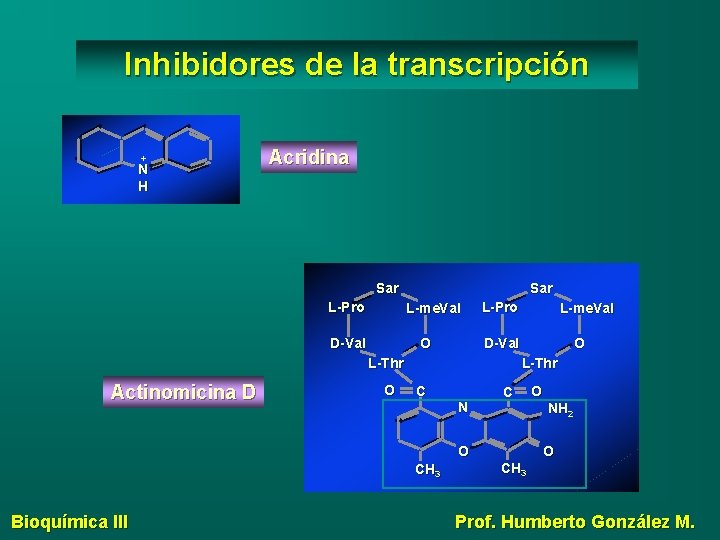
Inhibidores de la transcripción + N H Acridina Sar L-Pro Sar L-me. Val D-Val O L-Pro D-Val L-Thr Actinomicina D O L-me. Val O L-Thr C NH 2 O O CH 3 Bioquímica III O CH 3 Prof. Humberto González M.

Terminación de la transcripción Una vez que el proceso de transcripción ha comenzado la burbuja de transcripción avanza a medida que el RNA es transcrito y se continúa hasta que la polimerasa encuentra una secuencia que induce la disociación del complejo DNA-RNA y la separación de la enzima. En eucariontes esta secuencia no ha sido bien estudiada, mientras que para los procariontes como E. coli se han reconocido dos tipos de señales de terminación que se diferencian por la ausencia o presencia de un factor proteico que parece reconocer dicha señal. Bioquímica III Prof. Humberto González M.

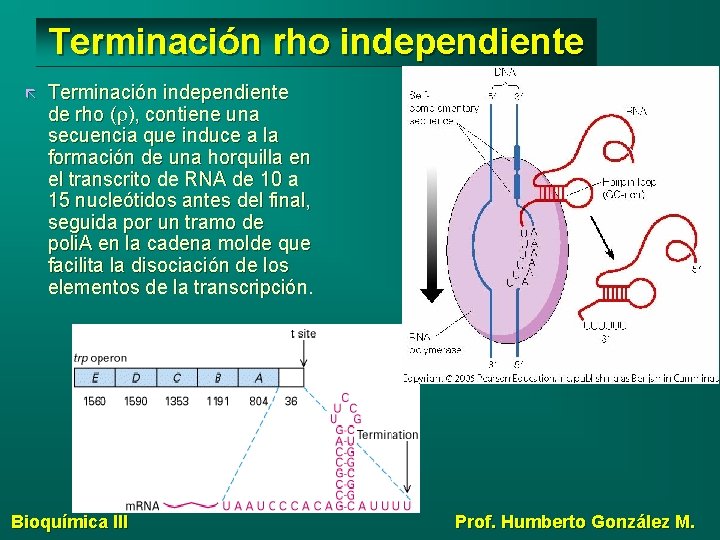
Terminación rho independiente Terminación independiente de rho (r), contiene una secuencia que induce a la formación de una horquilla en el transcrito de RNA de 10 a 15 nucleótidos antes del final, seguida por un tramo de poli. A en la cadena molde que facilita la disociación de los elementos de la transcripción. Bioquímica III Prof. Humberto González M.

Bioquímica III En la terminación dependiente de r no se presenta el tramo de poli. A, aunque sí se forma una horquilla en la que la RNA polimerasa hace una pausa y si se encuentra la proteína r presente se detiene la transcripción. No se conoce a detalle el mecanismo de disociación, pero en este se produce la hidrólisis de ATP por efecto de r. Prof. Humberto González M.

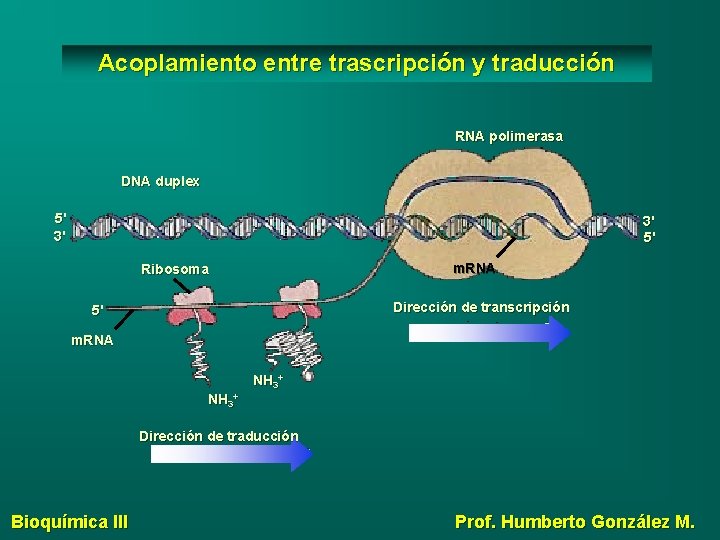
Acoplamiento entre trascripción y traducción RNA polimerasa DNA duplex 5' 3' 3' 5' m. RNA Ribosoma Dirección de transcripción 5' m. RNA NH 3+ Dirección de traducción Bioquímica III Prof. Humberto González M.

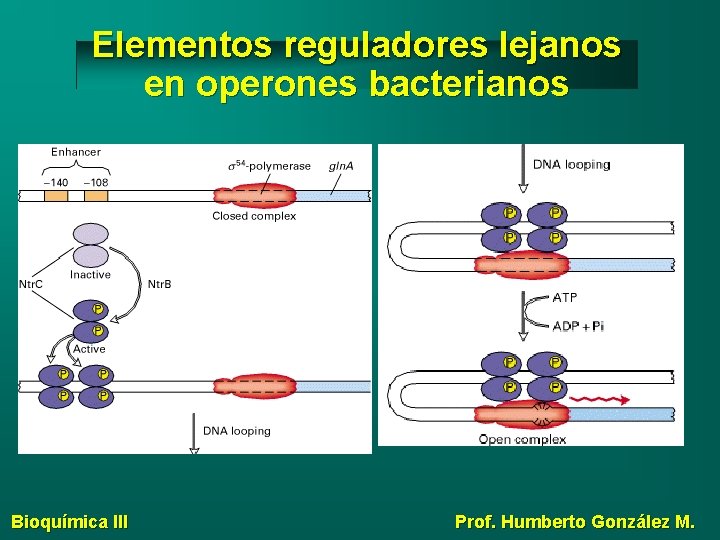
Elementos reguladores lejanos en operones bacterianos Bioquímica III Prof. Humberto González M.

Regulación de la transcripción en procariontes. Muchas proteínas bacterias son inducibles su síntesis es regulada dependiendo del estado nutricional de la célula. La expresión diferencial de los genes que codifican para esas proteínas esta regulada generalmente a nivel de la iniciación de la transcripción Bioquímica III Prof. Humberto González M.

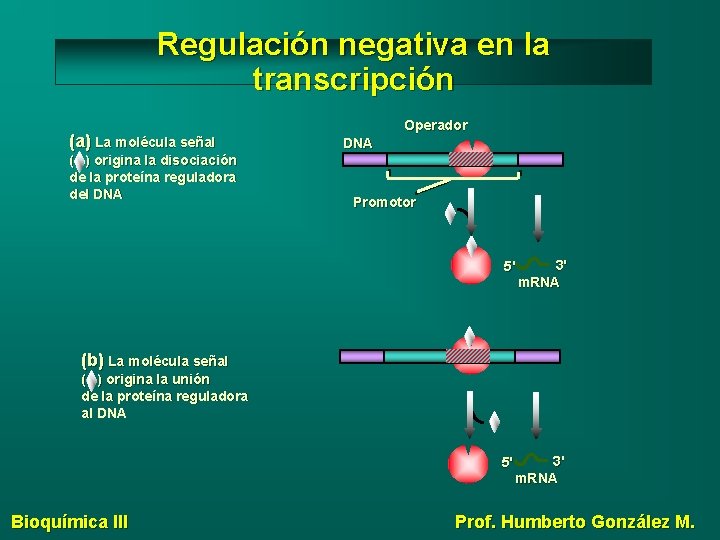
Regulación negativa en la transcripción (a) La molécula señal ( ) origina la disociación de la proteína reguladora del DNA Operador DNA Promotor 5' 3' m. RNA (b) La molécula señal ( ) origina la unión de la proteína reguladora al DNA 5' Bioquímica III 3' m. RNA Prof. Humberto González M.

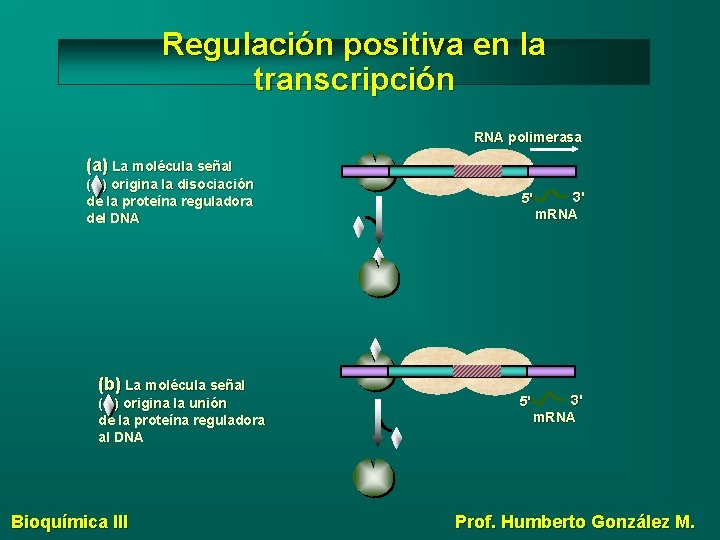
Regulación positiva en la transcripción RNA polimerasa (a) La molécula señal ( ) origina la disociación de la proteína reguladora del DNA (b) La molécula señal ( ) origina la unión de la proteína reguladora al DNA Bioquímica III 5' 5' 3' m. RNA Prof. Humberto González M.

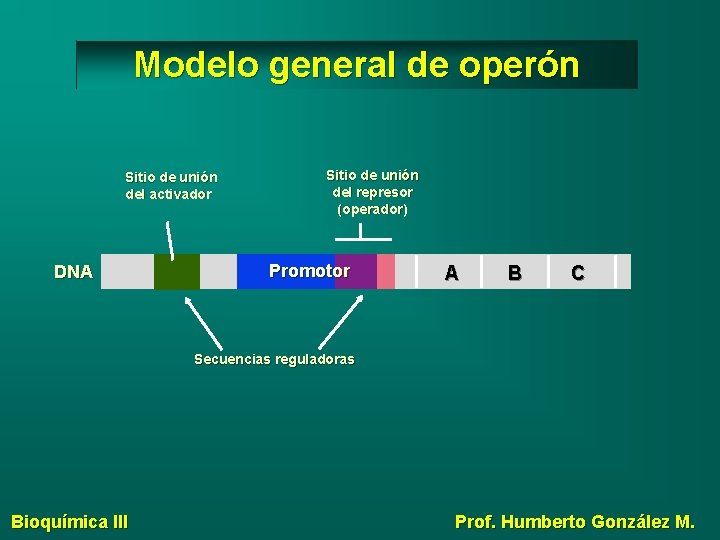
Modelo general de operón Sitio de unión del activador DNA Sitio de unión del represor (operador) Promotor A B C Secuencias reguladoras Bioquímica III Prof. Humberto González M.

Operón lac Jacob y Monod describieron el control transcripcional del operón de lactosa, que codifica 3 proteínas inducibles. Bioquímica III Prof. Humberto González M.

Operón lac Es reprimido por unión de la proteína represora de lac a la secuencia operadora. En presencia de lactosa o otros inductores, esta represión se levanta y el operón lac se transcribe. Bioquímica III Prof. Humberto González M.

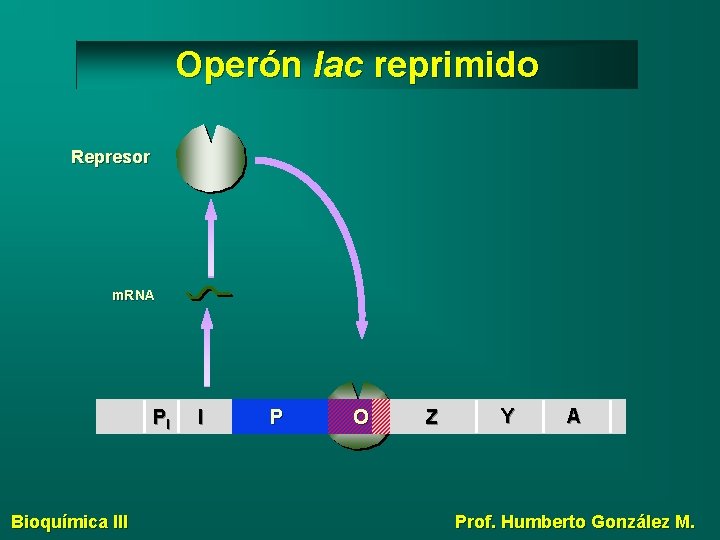
Operón lac reprimido Represor m. RNA PI Bioquímica III I P O Z Y A Prof. Humberto González M.

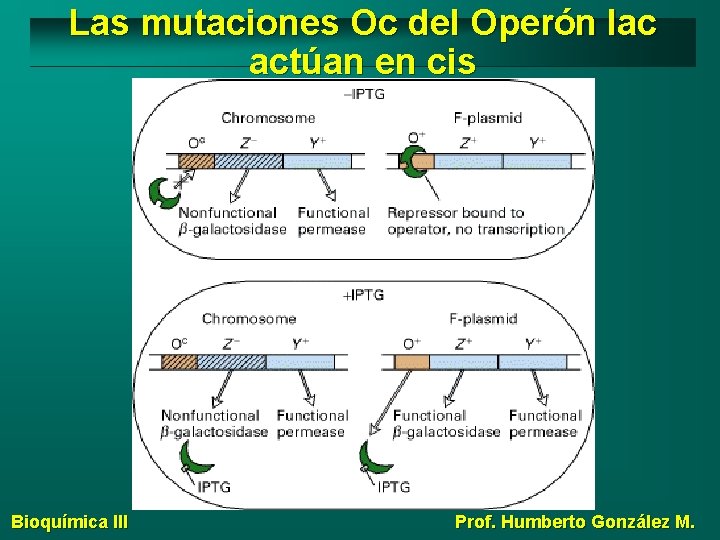
Mutaciones del promotor o el operador Las mutaciones en el promotor, o en el operador, actúan en cis; esto es sólo afectan la expresión de los genes en la misma molécula de DNA en que ocurre la mutación. Bioquímica III Prof. Humberto González M.

Las mutaciones Oc del Operón lac actúan en cis Bioquímica III Prof. Humberto González M.

Mutaciones constitutivas Las mutaciones en la secuencia de un operador que resulta en la disminución de la unión del represor, resultan en una transcripción constitutiva. Bioquímica III Prof. Humberto González M.

Mutaciones Las mutaciones en una secuencia promotora, que afectan la afinidad de la unión de la RNA polimerasa pueden disminuir (mutación negativa) o incrementar (mutación positiva) la transcripción. Bioquímica III Prof. Humberto González M.

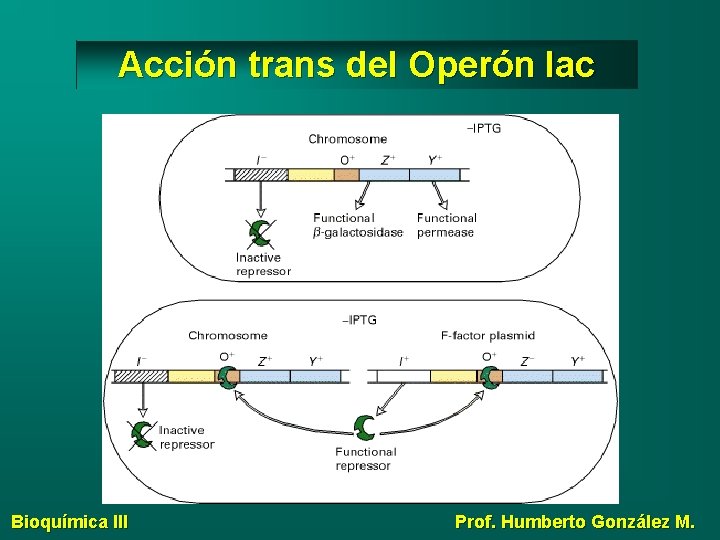
Regulación en trans Los represores y los activadores actúan en trans; esto es, afectan a la expresión de los genes que regulan sin importar en qué molécula de DNA están localizados en la célula. Bioquímica III Prof. Humberto González M.

Acción trans del Operón lac Bioquímica III Prof. Humberto González M.

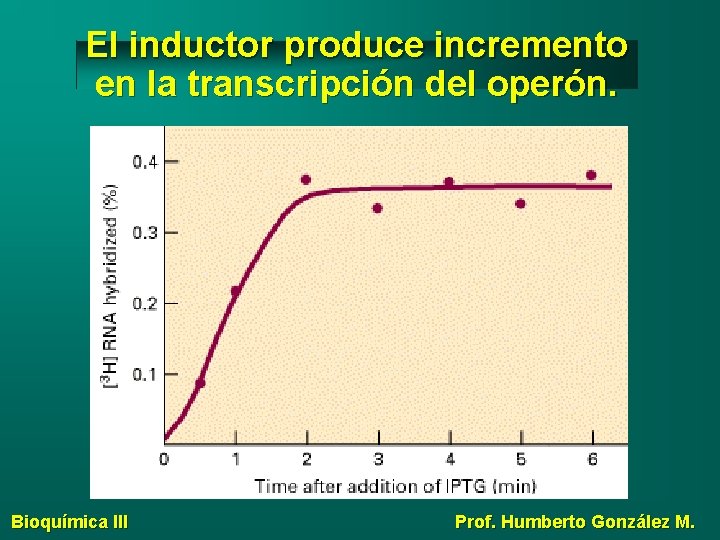
El inductor produce incremento en la transcripción del operón. Bioquímica III Prof. Humberto González M.

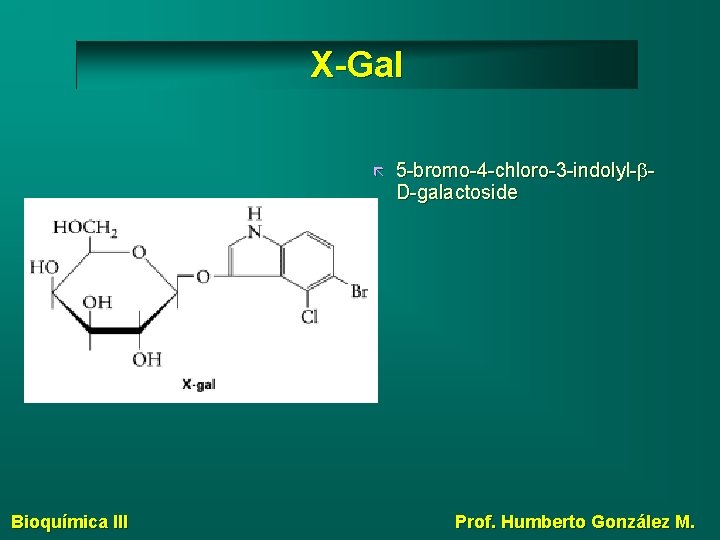
X-Gal Bioquímica III 5 -bromo-4 -chloro-3 -indolyl-b. D-galactoside Prof. Humberto González M.

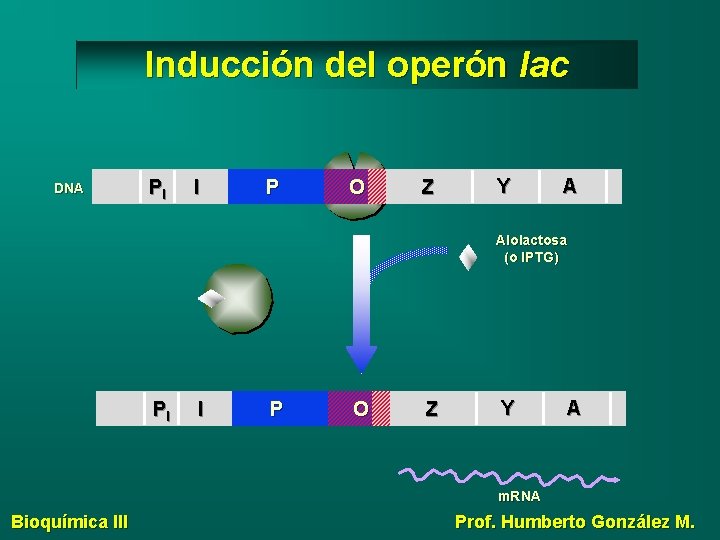
Inducción del operón lac DNA PI I P O Z Y A Alolactosa (o IPTG) PI I P O Z Y A m. RNA Bioquímica III Prof. Humberto González M.

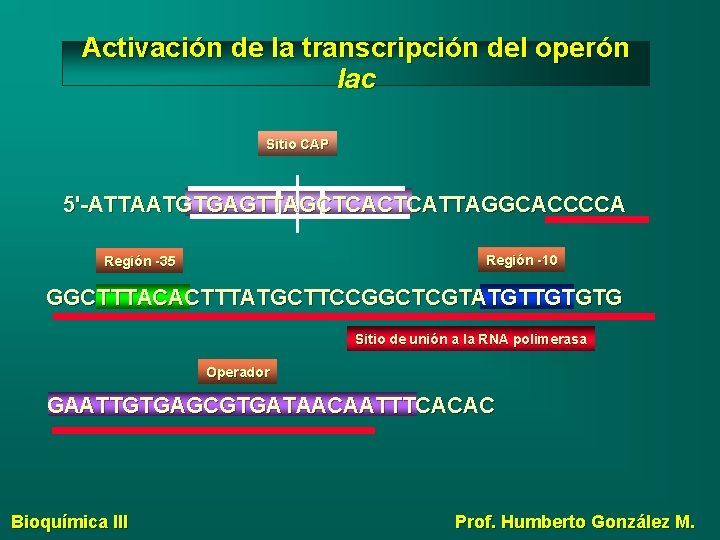
Activación de la transcripción del operón lac Sitio CAP 5'-ATTAATGTGAGTTAGCTCATTAGGCACCCCA Región -10 Región -35 GGCTTTACACTTTATGCTTCCGGCTCGTATGTTGTGTG Sitio de unión a la RNA polimerasa Operador GAATTGTGAGCGTGATAACAATTTCACAC Bioquímica III Prof. Humberto González M.

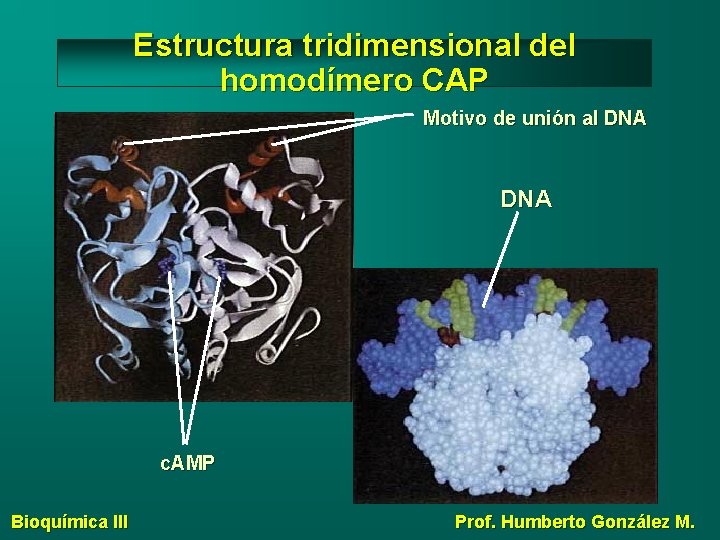
Estructura tridimensional del homodímero CAP Motivo de unión al DNA c. AMP Bioquímica III Prof. Humberto González M.

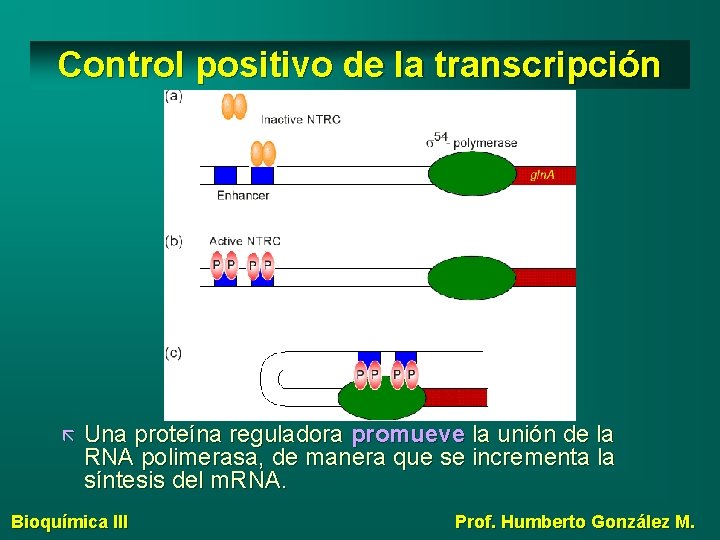
Control positivo de la transcripción Una proteína reguladora promueve la unión de la RNA polimerasa, de manera que se incrementa la síntesis del m. RNA. Bioquímica III Prof. Humberto González M.

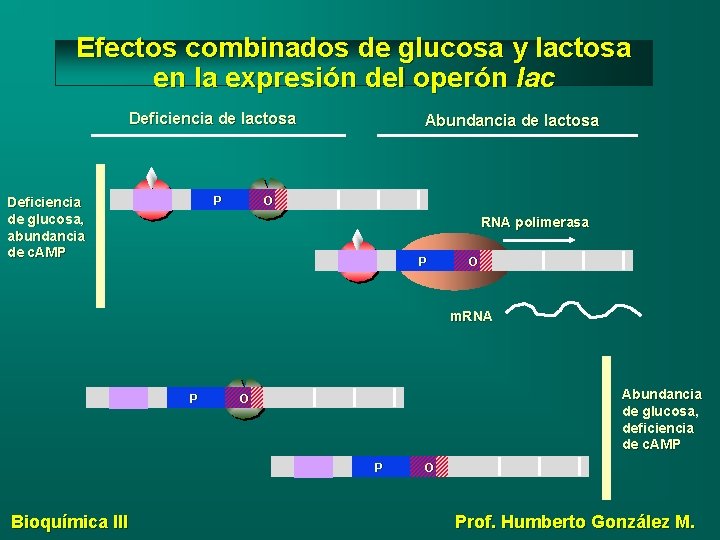
Efectos combinados de glucosa y lactosa en la expresión del operón lac Deficiencia de lactosa P Deficiencia de glucosa, abundancia de c. AMP Abundancia de lactosa O RNA polimerasa P O m. RNA P Abundancia de glucosa, deficiencia de c. AMP O P Bioquímica III O Prof. Humberto González M.

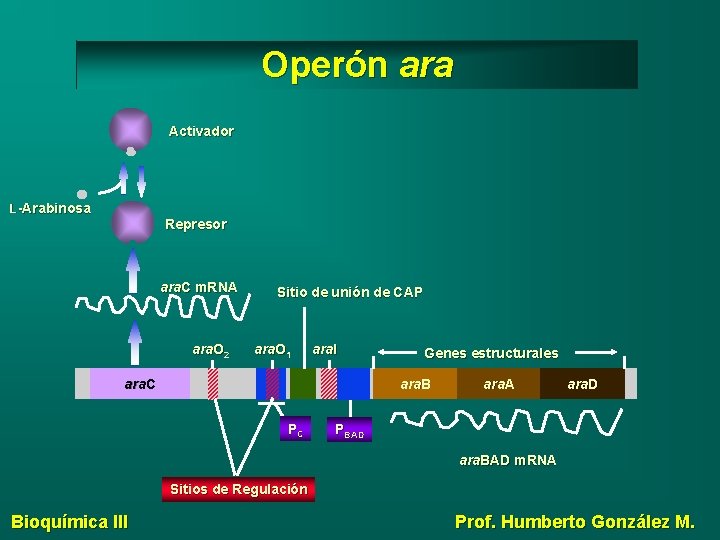
Operón ara Activador L-Arabinosa Represor ara. C m. RNA ara. O 2 Sitio de unión de CAP ara. O 1 ara. I ara. C Genes estructurales ara. B PC ara. A ara. D PBAD ara. BAD m. RNA Sitios de Regulación Bioquímica III Prof. Humberto González M.

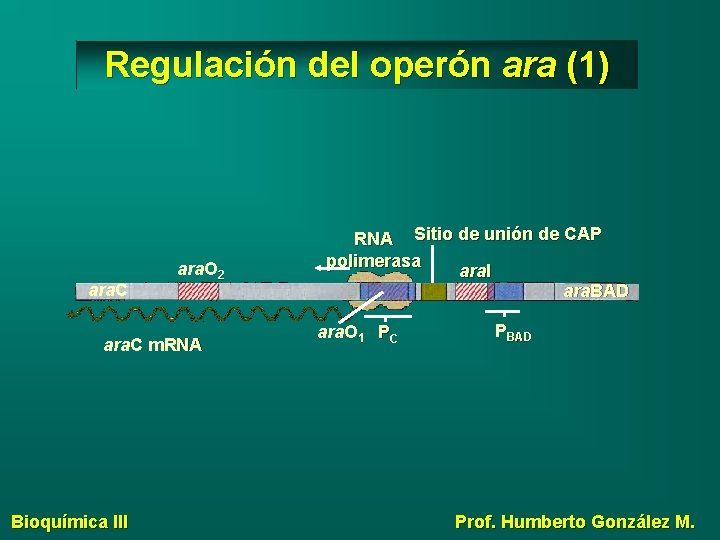
Regulación del operón ara (1) ara. C ara. O 2 ara. C m. RNA Bioquímica III RNA Sitio de unión de CAP polimerasa ara. I ara. BAD ara. O 1 PC PBAD Prof. Humberto González M.

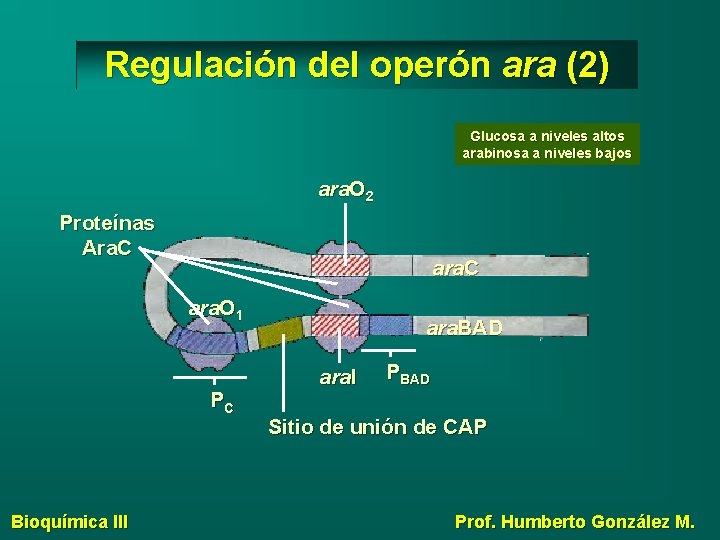
Regulación del operón ara (2) Glucosa a niveles altos arabinosa a niveles bajos ara. O 2 Proteínas Ara. C ara. O 1 PC Bioquímica III ara. BAD ara. I PBAD Sitio de unión de CAP Prof. Humberto González M.

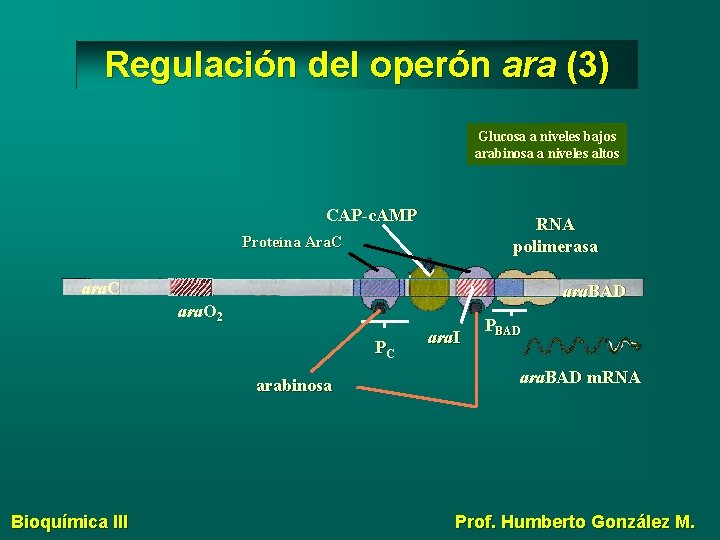
Regulación del operón ara (3) Glucosa a niveles bajos arabinosa a niveles altos CAP-c. AMP RNA polimerasa Proteína Ara. C ara. BAD ara. O 2 PC arabinosa Bioquímica III ara. I PBAD ara. BAD m. RNA Prof. Humberto González M.

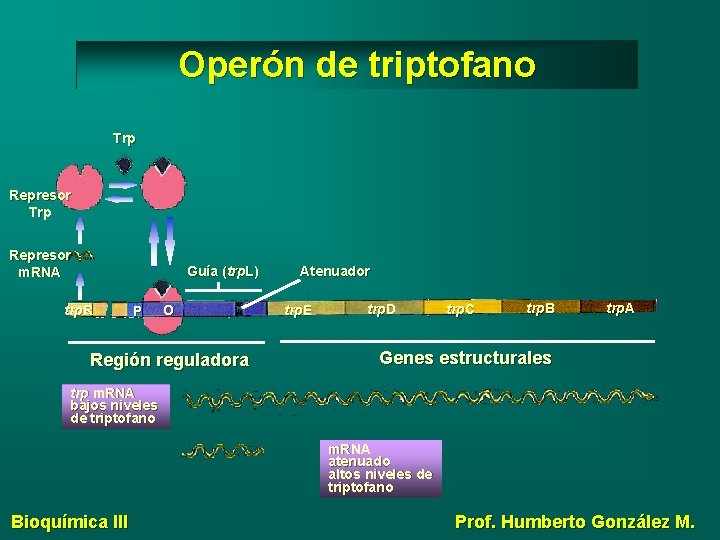
Operón de triptofano Trp Represor m. RNA Guía (trp. L) trp. R P O Región reguladora Atenuador trp. E trp. D trp. C trp. B trp. A Genes estructurales trp m. RNA bajos niveles de triptofano m. RNA atenuado altos niveles de triptofano Bioquímica III Prof. Humberto González M.

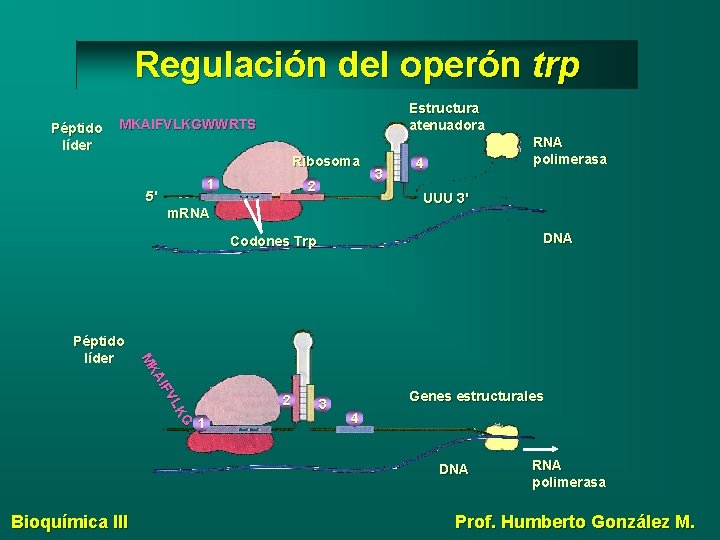
Regulación del operón trp Péptido líder Estructura atenuadora MKAIFVLKGWWRTS Ribosoma 1 5' 2 3 RNA polimerasa 4 UUU 3' m. RNA DNA Codones Trp G LK FV AI MK Péptido líder 2 1 3 Genes estructurales 4 DNA Bioquímica III RNA polimerasa Prof. Humberto González M.

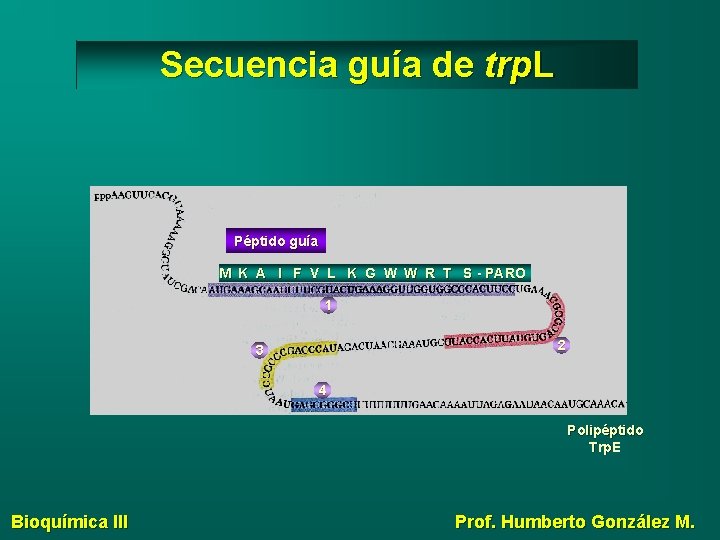
Secuencia guía de trp. L Péptido guía M K A I F V L K G W W R T S - PARO 1 2 3 4 Polipéptido Trp. E Bioquímica III Prof. Humberto González M.

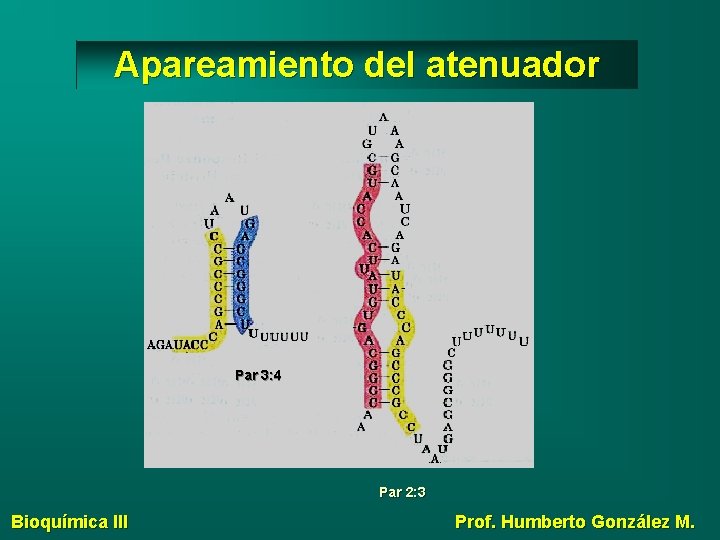
Apareamiento del atenuador Par 3: 4 Par 2: 3 Bioquímica III Prof. Humberto González M.

Otros mecanismos de atenuación Las Bacteria Gram positivas (B. subtilis) utilizan la atenuación. El mecanismo es independiente de la traducción. Utiliza una proteína que se une al RNA cuando existe un efector. El efector puede ser el aminoácido regulado pero también su aminoacil-t. RNA. La proteína se llama proteína trp de atenuación (en el caso del triptofano). Bioquímica III Prof. Humberto González M.

Otros sistemas de atenuación Operones de la biosíntesis de pirimidinas (E. coli traduccional y B. subtilis con proteína de atenuación). Atenuación traduccional. La traducción del péptido líder previene la traducción del siguiente gen en un mensajero policistrónico. Genes de resistencia a antibióticos en Gram positivas. Bioquímica III Prof. Humberto González M.

Redes de control global Sistemas que regulan muchos genes simultáneamente en respuesta a una señal ambiental específica. Los sistemas de control global pueden incluir más de un regulón. Modulón es un grupo de genes que pueden responder a una proteína reguladora común incluso cuando son de diferentes regulones. Estimulón es un grupo de genes que responden a la misma señal ambiental Bioquímica III Prof. Humberto González M.

Factores sigma alternativos Factor sigma es la subunidad de la RNA polimerasa que reconoce al promotor. La mayoría de genes de E. coli responden a s 70. La cantidad del factor sigma alternativo regula la respuesta del E. coli sistema global. En E. coli hay 7 factores s diferentes. B. subtilis tiene un factor s más. Bioquímica III Prof. Humberto González M.

Affinity of lac repressor for DNA (M-1) DNA lac operator Repressor + Inducer 2 X 1013 2 X 1010 All other DNA 2 X 106 __________________________ Specificity 1 107 104 __________________________ 1 Specificity is the ratio of (Ka for binding to operator DNA) / (Ka for binding to random DNA) Bioquímica III Prof. Humberto González M.

Algunos sistemas de control global en Escherichia coli. Bioquímica III Prof. Humberto González M.

Quorum sensing (percepción del quorum) Señal regulatoria controlada por la densidad de población. Cada bacteria sintetiza una lactona de homoserina acilada (AHL). La AHL difunde hacia el exterior de la célula sin problema. Se alcanza un concentración intracelular alta de AHL cuando hay alta densidad celular. AHL se une a una proteína activadora y enciende genes. Sistema luciferasa bacteriana, genes de producción de biofilms (películas) en P. aeruginosa, en Staphylococcus aureus produce varias proteínas virulentas. Bioquímica III Prof. Humberto González M.

Transducción de señales y sistemas reguladores de dos componentes La señal externa no es transmitida directamente a la proteína reguladora. La señal es detectada primero por un sensor La señal se transmite en una forma diferente hacia el sistema regulador en un proceso llamado transducción de señales. Bioquímica III Prof. Humberto González M.

Componentes del sistema regulador Proteína detectora específica localizada en la membrana. Generalmente una quinasa detectora. Proteína reguladora de respuesta específica. Bioquímica III Prof. Humberto González M.

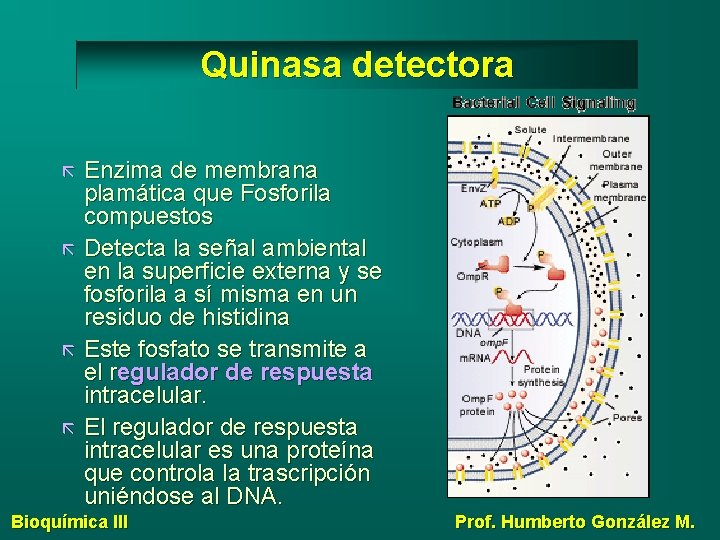
Quinasa detectora Enzima de membrana plamática que Fosforila compuestos Detecta la señal ambiental en la superficie externa y se fosforila a sí misma en un residuo de histidina Este fosfato se transmite a el regulador de respuesta intracelular. El regulador de respuesta intracelular es una proteína que controla la trascripción uniéndose al DNA. Bioquímica III Prof. Humberto González M.

Sistemas reguladores de dos componentes en la regulación de la transcripción de E. coli Bioquímica III Prof. Humberto González M.

fin Bioquímica III Prof. Humberto González M.

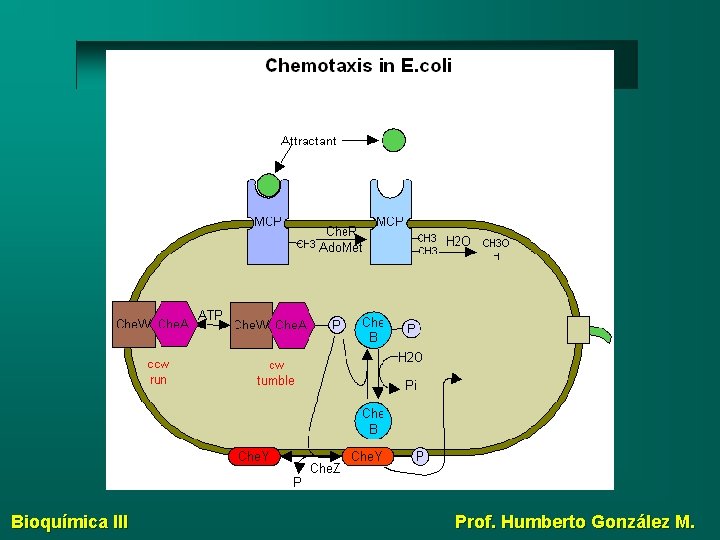
Quimiotaxis Movimiento de acercamiento o alejamiento bacteriano de un compuesto químico particular No es una respuesta a gradientes espaciales de una sustancia sino gradientes temporales Sienten el cambio en concentración de un compuesto fuera de la célula a intervalos Sistema de dos componentes para sentir este cambio y regulan el movimiento flagelar. Bioquímica III Prof. Humberto González M.

Proteínas involucradas en quimiotaxis Proteínas sensoras: Aceptoras de grupos metilo (MCPs) En E. coli se han identificado cinco MCPs Todas son transmemebranales Las MCPs se unen directamente a un compuesto o indirectamente a una proteína periplasmática Se disparan una serie de reacciones que afectan la rotación del flagelo. Rotación antihorario, la célula continua, rotación horario, la bacteria da un viraje. Bioquímica III Prof. Humberto González M.

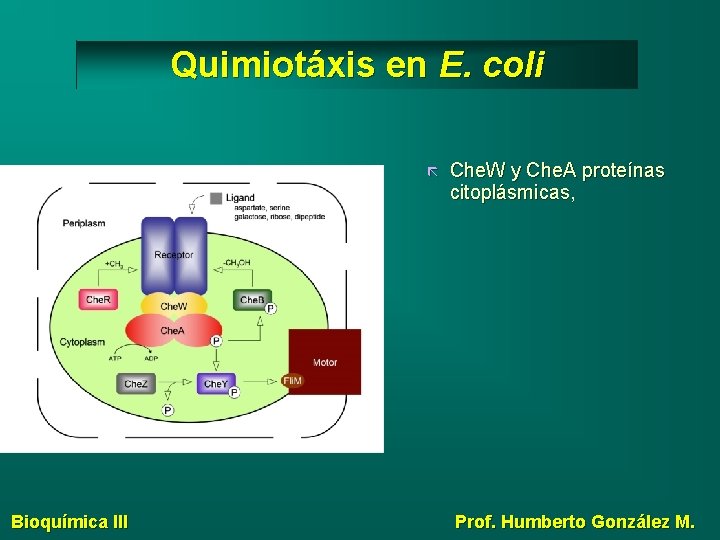
Quimiotáxis en E. coli Bioquímica III Che. W y Che. A proteínas citoplásmicas, Prof. Humberto González M.

La mayoría de sistemas de regulación controlan la transcripción mediante proteínas reguladoras. Existen RNAs reguladores (antisentido) Bioquímica III Prof. Humberto González M.

< Bioquímica III Prof. Humberto González M.

Resumen de la transcripción Bioquímica III Prof. Humberto González M.
 Cubo de un binomio
Cubo de un binomio Sintaxis logica articulo sustantivo adjetivo calificativo
Sintaxis logica articulo sustantivo adjetivo calificativo Topic 19 animation and expression techniques
Topic 19 animation and expression techniques Expresin
Expresin Gnica
Gnica Gnica
Gnica Gnica
Gnica Catabolica
Catabolica Gnica
Gnica Gnica
Gnica Arn polimerasa
Arn polimerasa Gnica
Gnica Gnica
Gnica Gnica
Gnica Gnica
Gnica Gnica
Gnica Gnica
Gnica Gnica
Gnica Que es polea
Que es polea El sumillado consiste en...
El sumillado consiste en... Felipe nos dijo que no hacía buen tiempo para que tomates.
Felipe nos dijo que no hacía buen tiempo para que tomates. Ventajas de rotafolio
Ventajas de rotafolio Yo soy
Yo soy Runa gibur
Runa gibur Qué debo hacer para que
Qué debo hacer para que Porque tuvo dios que mostrarse para que sepamos como es
Porque tuvo dios que mostrarse para que sepamos como es Para que o mal triunfe basta
Para que o mal triunfe basta Receita instrucional
Receita instrucional Verde por fuera verde por dentro
Verde por fuera verde por dentro Por donde sale el sol
Por donde sale el sol O que é publicidade
O que é publicidade Para que sirve evernote
Para que sirve evernote Quien invento el antecesor del ebook
Quien invento el antecesor del ebook Tratamos de ser felices no perfectos
Tratamos de ser felices no perfectos Em uma obra para permitir o transporte de objetos para cima
Em uma obra para permitir o transporte de objetos para cima Consumir para viver ou viver para consumir
Consumir para viver ou viver para consumir Subtrair para si ou para outrem coisa alheia móvel
Subtrair para si ou para outrem coisa alheia móvel Trabaja para vivir no vivas para trabajar
Trabaja para vivir no vivas para trabajar Reglas para clases virtuales para niños camara encendida
Reglas para clases virtuales para niños camara encendida Reglas para clases virtuales para niños camara encendida
Reglas para clases virtuales para niños camara encendida Todo para todos nada para nosotros
Todo para todos nada para nosotros Modelo bidimensional para evaluar el riesgo para la salud
Modelo bidimensional para evaluar el riesgo para la salud Yo soy de mi amado y mi amado es mio
Yo soy de mi amado y mi amado es mio Injecao ventro glútea
Injecao ventro glútea La leyenda de los temblores
La leyenda de los temblores Frases para cuidar el agua para niños
Frases para cuidar el agua para niños 10 medicamentos del sistema respiratorio
10 medicamentos del sistema respiratorio Mensajes por el dia del maestro boliviano
Mensajes por el dia del maestro boliviano Tono grave y agudo
Tono grave y agudo La gente que me gusta mario benedetti pdf
La gente que me gusta mario benedetti pdf Moins aussi plus
Moins aussi plus Quien dice lo que no debe oye lo que no quiere
Quien dice lo que no debe oye lo que no quiere Qqqqu
Qqqqu No trote
No trote Me gusta la gente rara
Me gusta la gente rara Youtube para que sirve
Youtube para que sirve Estaciones vialucis
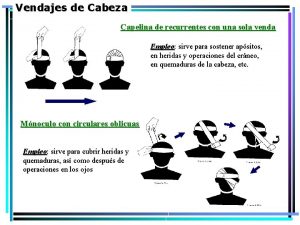
Estaciones vialucis Capelina vendaje
Capelina vendaje Vendaje en ocho o tortuga
Vendaje en ocho o tortuga Unidad de aprendizaje curricular
Unidad de aprendizaje curricular El que no provee para su casa estudio
El que no provee para su casa estudio Carbamazepina para que sirve
Carbamazepina para que sirve Tipos de limas
Tipos de limas Texto informativo estructura
Texto informativo estructura Caracteristicas de los textos laborales
Caracteristicas de los textos laborales Para que sirven las toberas
Para que sirven las toberas Ejemplo del tercer mandamiento
Ejemplo del tercer mandamiento Suturas no absorbibles de origen vegetal
Suturas no absorbibles de origen vegetal Sonda de malecot
Sonda de malecot Oraciones cordinadas
Oraciones cordinadas