M 13 Molekulare Physiologie Prof Dr Thorsten Mascher










- Slides: 27

M 13 Molekulare Physiologie Prof. Dr. Thorsten Mascher Dr. Diana Wolf Jara Radeck Philipp Popp Susanne Krause

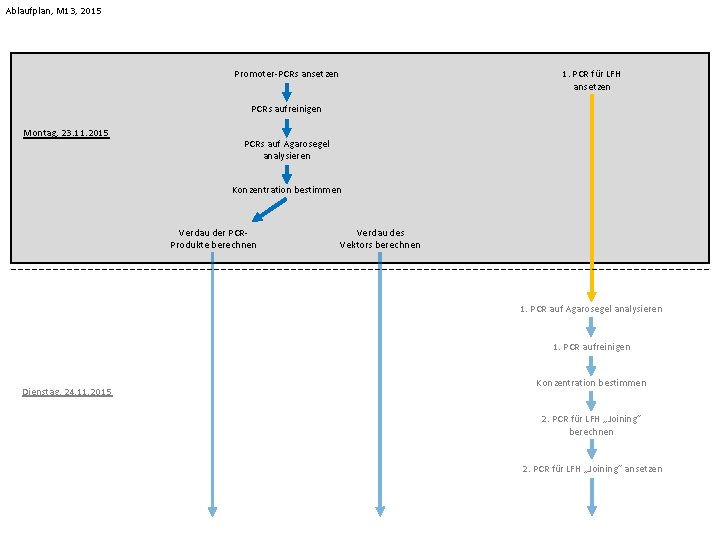
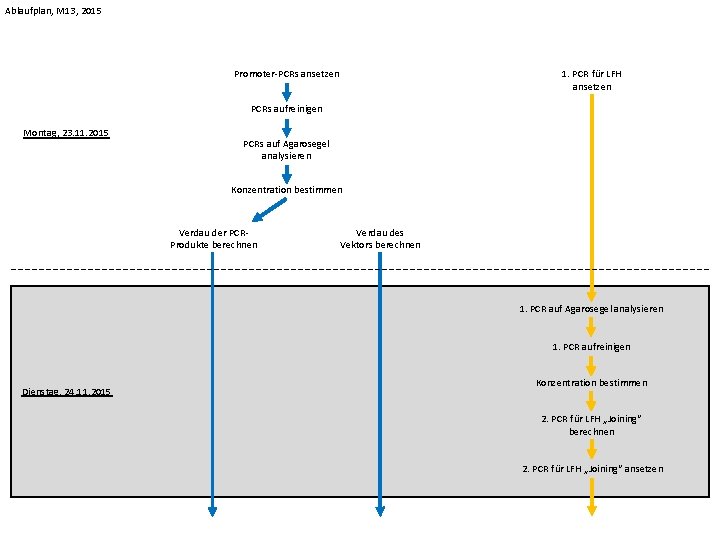
Ablaufplan, M 13, 2015 Promoter-PCRs ansetzen 1. PCR für LFH ansetzen PCRs aufreinigen Montag, 23. 11. 2015 PCRs auf Agarosegel analysieren Konzentration bestimmen Verdau der PCRProdukte berechnen Verdau des Vektors berechnen 1. PCR auf Agarosegel analysieren 1. PCR aufreinigen Dienstag, 24. 11. 2015 Konzentration bestimmen 2. PCR für LFH „Joining“ berechnen 2. PCR für LFH „Joining“ ansetzen

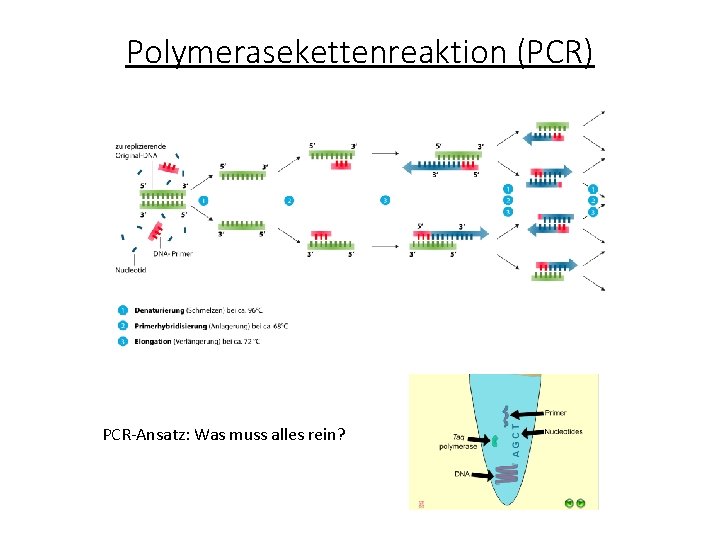
Polymerasekettenreaktion (PCR) PCR-Ansatz: Was muss alles rein?

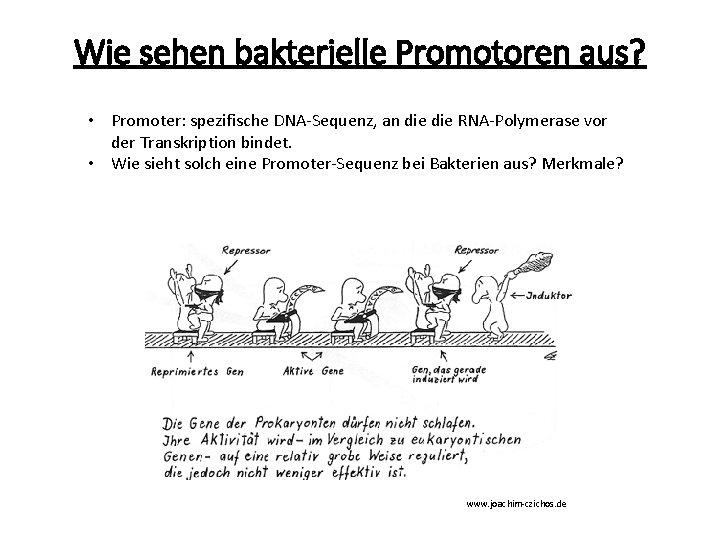
Wie sehen bakterielle Promotoren aus? • Promoter: spezifische DNA-Sequenz, an die RNA-Polymerase vor der Transkription bindet. • Wie sieht solch eine Promoter-Sequenz bei Bakterien aus? Merkmale? www. joachim-czichos. de

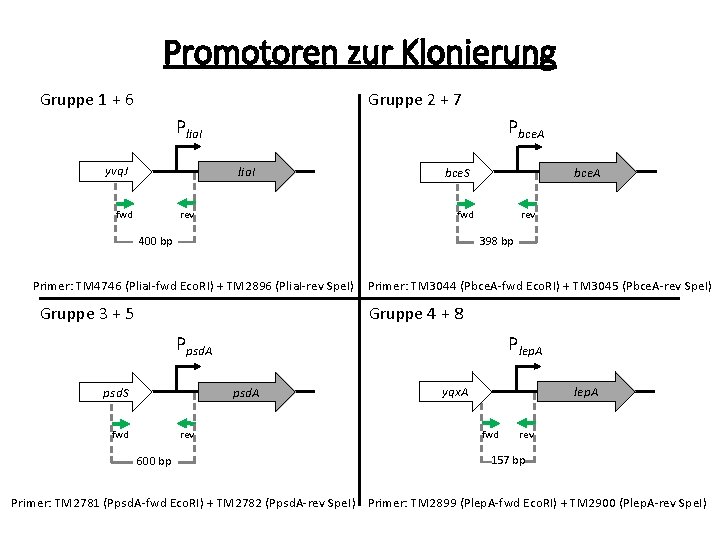
Promotoren zur Klonierung Gruppe 1 + 6 Gruppe 2 + 7 Plia. I yvq. J Pbce. A lia. I rev fwd yvq. J bce. S bce. A rev fwd 400 bp 398 bp Primer: TM 4746 (Plia. I-fwd Eco. RI) + TM 2896 (Plia. I-rev Spe. I) Primer: TM 3044 (Pbce. A-fwd Eco. RI) + TM 3045 (Pbce. A-rev Spe. I) Gruppe 3 + 5 Gruppe 4 + 8 Ppsd. A yvq. J psd. S Plep. A psd. A rev fwd 600 bp yvq. J yqx. A lep. A fwd rev 157 bp Primer: TM 2781 (Ppsd. A-fwd Eco. RI) + TM 2782 (Ppsd. A-rev Spe. I) Primer: TM 2899 (Plep. A-fwd Eco. RI) + TM 2900 (Plep. A-rev Spe. I)

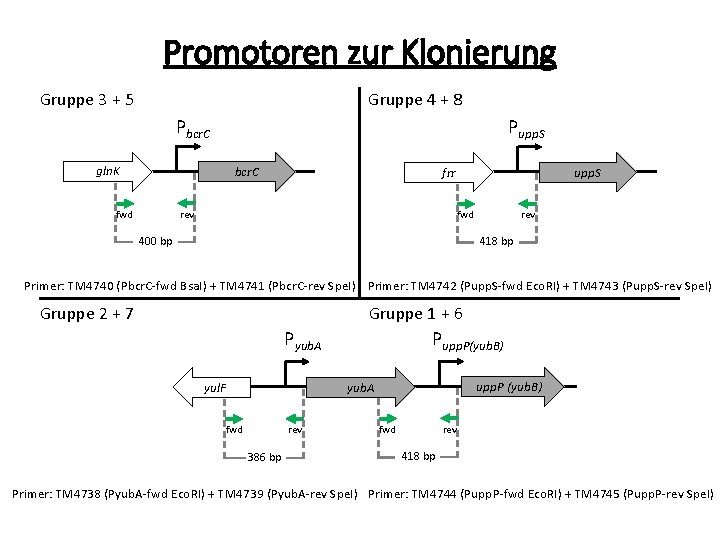
Promotoren zur Klonierung Gruppe 3 + 5 Gruppe 4 + 8 Pbcr. C gln. K Pupp. S bcr. C yvq. J frr rev fwd upp. S rev fwd 400 bp 418 bp Primer: TM 4740 (Pbcr. C-fwd Bsa. I) + TM 4741 (Pbcr. C-rev Spe. I) Primer: TM 4742 (Pupp. S-fwd Eco. RI) + TM 4743 (Pupp. S-rev Spe. I) Gruppe 2 + 7 Gruppe 1 + 6 Pyub. A yvq. J yul. F Pupp. P(yub. B) upp. P (yub. B) yub. A rev fwd 386 bp rev fwd 418 bp Primer: TM 4738 (Pyub. A-fwd Eco. RI) + TM 4739 (Pyub. A-rev Spe. I) Primer: TM 4744 (Pupp. P-fwd Eco. RI) + TM 4745 (Pupp. P-rev Spe. I)

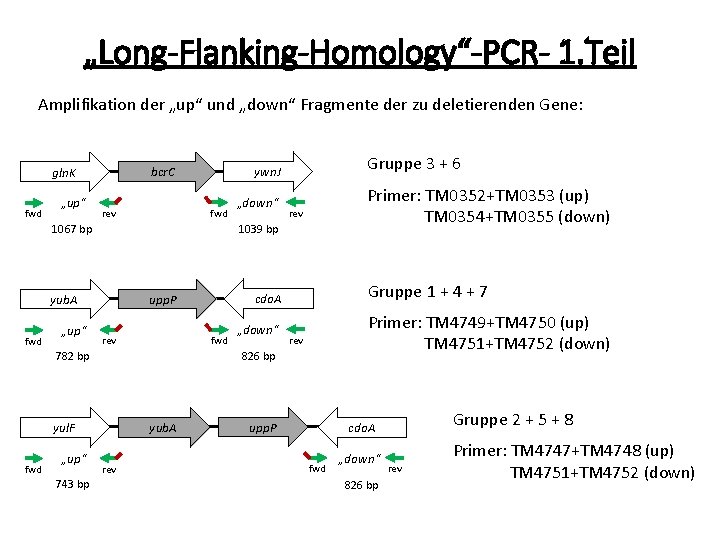
„Long-Flanking-Homology“-PCR- 1. Teil Amplifikation der „up“ und „down“ Fragmente der zu deletierenden Gene: fwd „up“ fwd rev 1067 bp fwd rev 743 bp rev Gruppe 1 + 4 + 7 „down“ Primer: TM 4749+TM 4750 (up) TM 4751+TM 4752 (down) rev 826 bp yub. A yul. F fwd Primer: TM 0352+TM 0353 (up) TM 0354+TM 0355 (down) cdo. A upp. P 782 bp „up“ „down“ 1039 bp yub. A „up“ Gruppe 3 + 6 ywn. J bcr. C gln. K rev upp. P Gruppe 2 + 5 + 8 cdo. A fwd „down“ 826 bp rev Primer: TM 4747+TM 4748 (up) TM 4751+TM 4752 (down)

„Long-Flanking-Homology“-PCR- 1. Teil Amplifikation der Kanamycin- und Macrolid-Lincosamid-Streptogramin (MLS)Resistenzkassette: • Resistenzkassetten sind spezifisch für B. subtilis etabliert (Guerout-Fleury et al. , Gene (1995)) • Resistenzkassetten sind auf Plasmiden lokalisiert Plasmide werden als DNATemplate genutzt • Gruppe 1, 2, 6, 7+8 Kanamycin-Resistenzkassette - p. DG 1513 als Template - Primer TM 0137 + TM 0138 - Größe: 1479 bp • Gruppe 3, 4, 5 (MLS)-Resistenzkassette - p. BS 1 E als Template - Primer TM 0139 + TM 0140 - Größe: 1113 bp kan rev fwd mls fwd rev

Restriktionsverdau • Prinzip des Restriktionsverdaus: gezieltes „Zerschneiden“ von DNA durch Restriktionsenzyme (Restriktionsendonukleasen) • Nomenklatur der Restriktionsendonukleasen: z. B. Eco. RI Escherichia coli RI 13, „I“ = Nummerierung • Restriktionsenzyme erkennen spezifische Sequenzabschnitte Palindrome • Die Spaltung der DNA führt zu „sticky ends“ oder „blunt ends“ „sticky ends“ Eco. RI G▼AATT C C TTAA▲G Spe. I A▼CTAG T T GATC▲A „blunt ends“ Sca. I AGT▼ACT TCA▲TGA

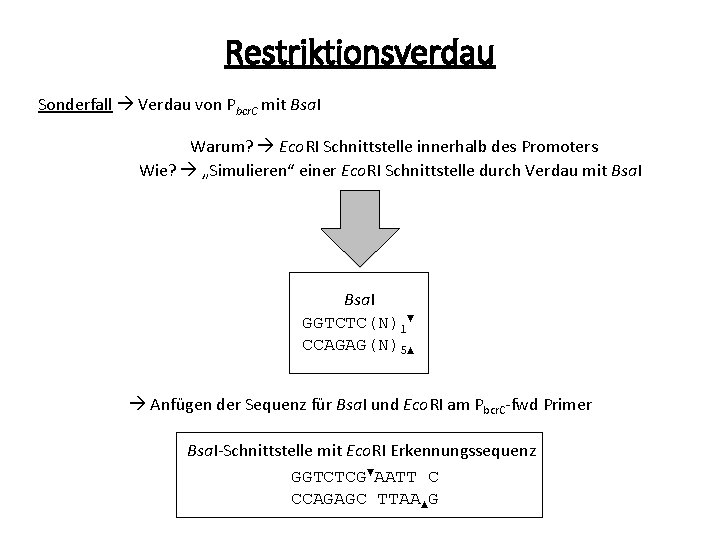
Restriktionsverdau Sonderfall Verdau von Pbcr. C mit Bsa. I Warum? Eco. RI Schnittstelle innerhalb des Promoters Wie? „Simulieren“ einer Eco. RI Schnittstelle durch Verdau mit Bsa. I GGTCTC(N)1▼ CCAGAG(N)5▲ Anfügen der Sequenz für Bsa. I und Eco. RI am Pbcr. C-fwd Primer Bsa. I-Schnittstelle mit Eco. RI Erkennungssequenz GGTCTCG▼AATT C CCAGAGC TTAA▲G

Restriktionsverdau Berechnung der Enzymmenge: MW (R) MW (x) n (R) n (x) Molekulargewicht der Referenz-DNA (Ad 2 -DNA = 35, 9 kb) Molekulargewicht der zu verdauenden DNA in kb Anzahl der Schnittstellen für das Enzym in der Referenz-DNA Anzahl der Schnittstellen in der zu verdauenden DNA Beispielrechnung für Pupp. S-Verdau (0, 5 µg) mit Eco. RI (20 U/µl): 17 U/µg DNA 8, 5 U für 500 ng DNA Doppelte Menge einsetzen: 17 U 0, 85 µl Enzym für Verdau

Restriktionsverdau • Eigenschaften der im Praktikum eingesetzten Enzyme: Eco. RI-HF (20 U/µl, Cut. Smart-Puffer) Spe. I-HF (20 U/µl, Cut. Smart-Puffer) Bsa. I (10 U/µl, Cut. Smart-Puffer) • Für Verdau werden die jeweiligen Enzyme mit der zu verdauenden DNA, Puffer und Wasser gemischt Doppelverdau (= 2 Enzyme pro Ansatz) • Jeweils einen Ansatz für die Promotoren und einen Ansatz für den Vektor p. BS 3 Clux (10649 bp)

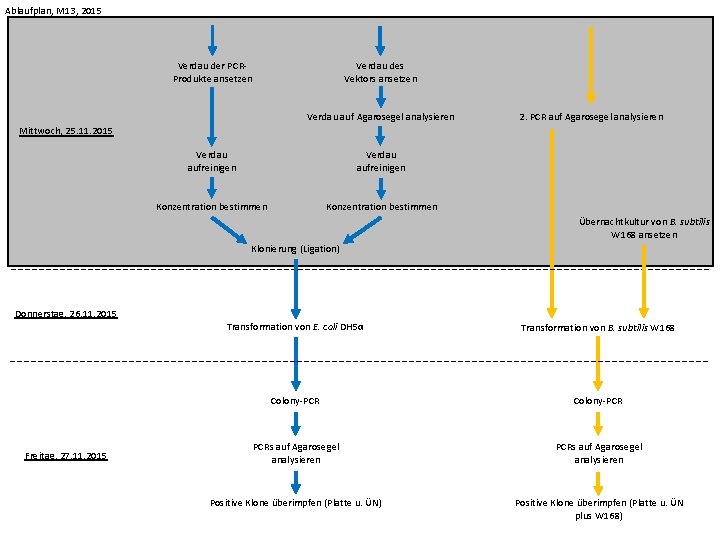
Ablaufplan, M 13, 2015 Verdau der PCRProdukte ansetzen Verdau des Vektors ansetzen Verdau auf Agarosegel analysieren 2. PCR auf Agarosegel analysieren Mittwoch, 25. 11. 2015 Verdau aufreinigen Konzentration bestimmen Übernachtkultur von B. subtilis W 168 ansetzen Klonierung (Ligation) Donnerstag, 26. 11. 2015 Freitag, 27. 11. 2015 Transformation von E. coli DH 5α Transformation von B. subtilis W 168 Colony-PCR PCRs auf Agarosegel analysieren Positive Klone überimpfen (Platte u. ÜN) Positive Klone überimpfen (Platte u. ÜN plus W 168)

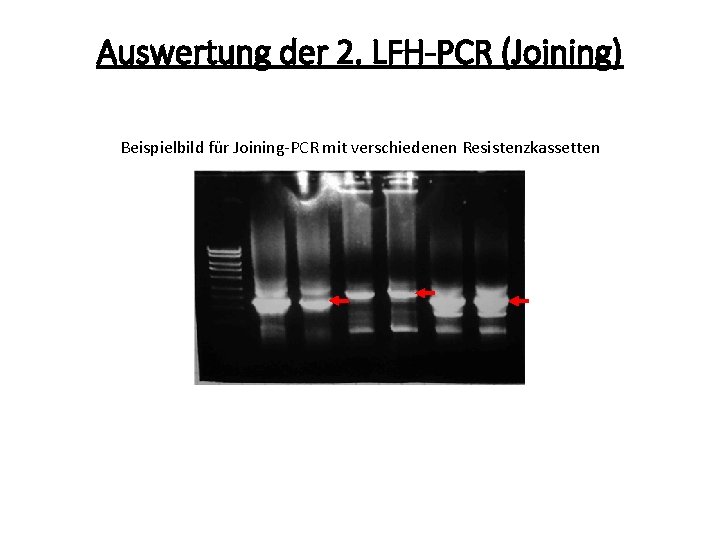
Auswertung der 2. LFH-PCR (Joining) Beispielbild für Joining-PCR mit verschiedenen Resistenzkassetten

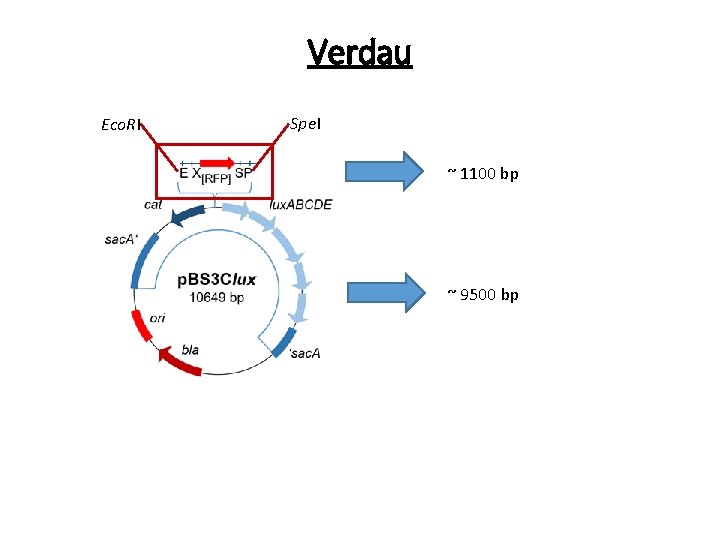
Verdau Eco. RI Spe. I ~ 1100 bp ~ 9500 bp

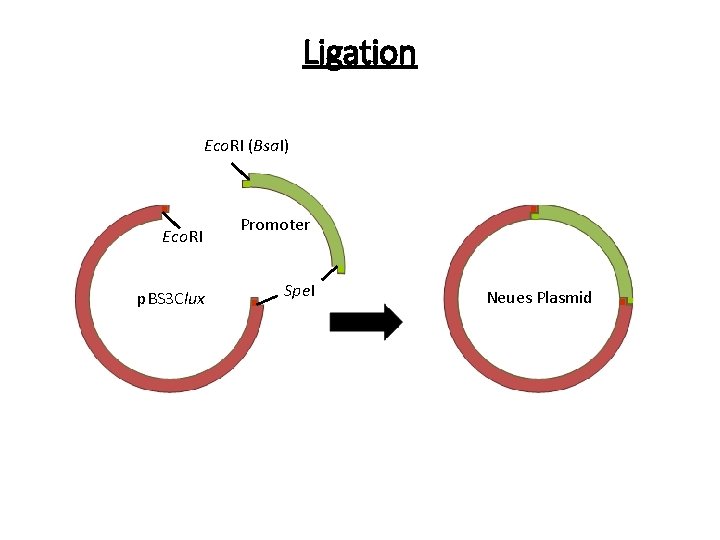
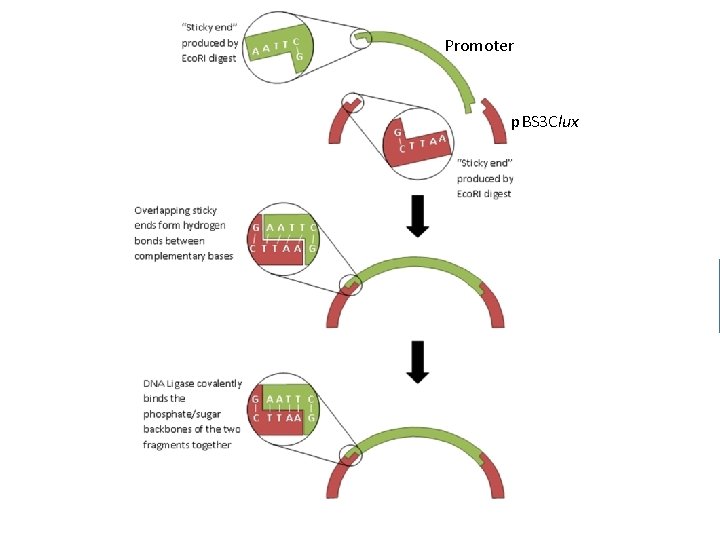
Ligation Eco. RI (Bsa. I) Eco. RI p. BS 3 Clux Promoter Spe. I Neues Plasmid

Promoter p. BS 3 Clux

Berechnen des Ligationsansatzes • Wichtig: das Volumen des Ligationsansatzes so gering wie möglich halten (~ ca. 10 µl) • Jede Gruppe setzt die Ligation mit eigenen Promotoren und Vektor an sowie mit Vektor von uns ( Warum das? ) • Material für die Ligation: • • • Verdauter p. BS 3 Clux-Vektor (200 ng) Verdautes Promoter-Fragment (3 -fach molarer Überschuss) Ligationspuffer (10 x) Ligase Wasser zum Auffüllen 3 -fach molarer Überschuss an Promoter-Fragment? ? ? X = Menge (ng) mal 3 (3 -fach mehr) = Menge an DNA, die für die Ligation eingesetzt werden muss

Ablaufplan, M 13, 2015 Promoter-PCRs ansetzen 1. PCR für LFH ansetzen PCRs aufreinigen Montag, 23. 11. 2015 PCRs auf Agarosegel analysieren Konzentration bestimmen Verdau der PCRProdukte berechnen Verdau des Vektors berechnen 1. PCR auf Agarosegel analysieren 1. PCR aufreinigen Dienstag, 24. 11. 2015 Konzentration bestimmen 2. PCR für LFH „Joining“ berechnen 2. PCR für LFH „Joining“ ansetzen

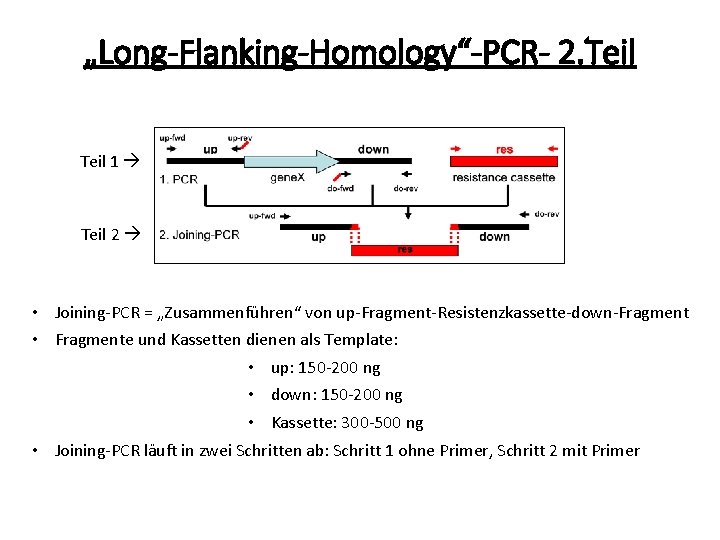
„Long-Flanking-Homology“-PCR- 2. Teil 1 Teil 2 • Joining-PCR = „Zusammenführen“ von up-Fragment-Resistenzkassette-down-Fragment • Fragmente und Kassetten dienen als Template: • up: 150 -200 ng • down: 150 -200 ng • Kassette: 300 -500 ng • Joining-PCR läuft in zwei Schritten ab: Schritt 1 ohne Primer, Schritt 2 mit Primer

The universal Bio. Brick cloning standard Xba. I Eco. RI E X S P Spe. I S P Pst. I EX Bio. Brick 1 Bio. Brick 2 A A Cut with E&S Cut with X&P EX S Bio. Brick 1 Bio. Brick 2 5‘ -- A 3‘ -- TGATC Spe. I EX S P X CTAGA -- 3‘ T -- 5‘ Xba. I -- ACTAGA –-- TGATCT -M Bio. Brick 1 Bio. Brick 2 Compatible ends, no recognition site! S P

Vectors luciferase reporter vectors

Ablaufplan, M 13, 2015 Sequenzanalyse Theoretische Planung und Vorbereitung des „Plate-Reader“-Assay Klon 1 der ÜN-Kultur animpfen TMB 2841 und W 168 animpfen Mikroskopie Mittwoch, 02. 12. 2015 Praktische Durchführung des 1. „Plate-Reader“-Assay Auswertung des 1. „Plate-Reader“- Assay Klon 2 als ÜN-Kultur animpfen TMB 2841 und W 168 als ÜN-Kultur animpfen Klon 2 der ÜN-Kultur animpfen TMB 2841 und W 168 animpfen Theoretische Planung und Vorbereitung des „Plate-Reader“-Assay Donnerstag, 03. 12. 2015 Praktische Durchführung des 2. „Plate-Reader“-Assay Auswertung am Freitag, 04. 12. 2015

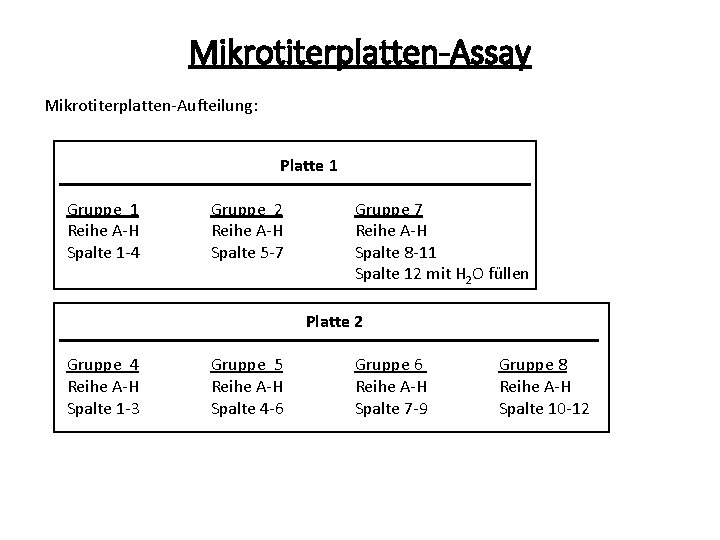
Mikrotiterplatten-Assay Mikrotiterplatten-Aufteilung: Platte 1 Gruppe 1 Reihe A-H Spalte 1 -4 Gruppe 2 Reihe A-H Spalte 5 -7 Gruppe 7 Reihe A-H Spalte 8 -11 Spalte 12 mit H 2 O füllen Platte 2 Gruppe 4 Reihe A-H Spalte 1 -3 Gruppe 5 Reihe A-H Spalte 4 -6 Gruppe 6 Reihe A-H Spalte 7 -9 Gruppe 8 Reihe A-H Spalte 10 -12

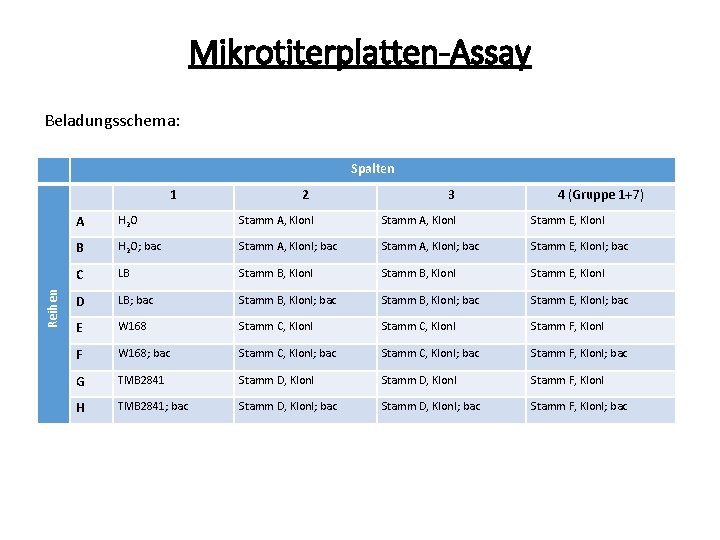
Mikrotiterplatten-Assay Beladungsschema: Spalten Reihen 1 2 3 4 (Gruppe 1+7) A H 2 O Stamm A, Klon. I Stamm E, Klon. I B H 2 O; bac Stamm A, Klon. I; bac Stamm E, Klon. I; bac C LB Stamm B, Klon. I Stamm E, Klon. I D LB; bac Stamm B, Klon. I; bac Stamm E, Klon. I; bac E W 168 Stamm C, Klon. I Stamm F, Klon. I F W 168; bac Stamm C, Klon. I; bac Stamm F, Klon. I; bac G TMB 2841 Stamm D, Klon. I Stamm F, Klon. I H TMB 2841; bac Stamm D, Klon. I; bac Stamm F, Klon. I; bac

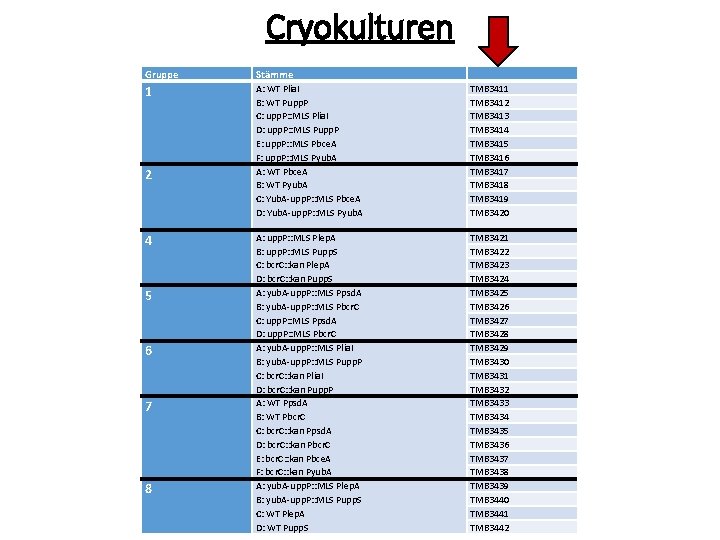
Cryokulturen Gruppe Stämme 1 A: WT Plia. I B: WT Pupp. P C: upp. P: : MLS Plia. I D: upp. P: : MLS Pupp. P E: upp. P: : MLS Pbce. A F: upp. P: : MLS Pyub. A A: WT Pbce. A B: WT Pyub. A C: Yub. A-upp. P: : MLS Pbce. A D: Yub. A-upp. P: : MLS Pyub. A TMB 3411 TMB 3412 TMB 3413 TMB 3414 TMB 3415 TMB 3416 TMB 3417 TMB 3418 TMB 3419 TMB 3420 A: upp. P: : MLS Plep. A B: upp. P: : MLS Pupp. S C: bcr. C: : kan Plep. A D: bcr. C: : kan Pupp. S A: yub. A-upp. P: : MLS Ppsd. A B: yub. A-upp. P: : MLS Pbcr. C C: upp. P: : MLS Ppsd. A D: upp. P: : MLS Pbcr. C A: yub. A-upp. P: : MLS Plia. I B: yub. A-upp. P: : MLS Pupp. P C: bcr. C: : kan Plia. I D: bcr. C: : kan Pupp. P A: WT Ppsd. A B: WT Pbcr. C C: bcr. C: : kan Ppsd. A D: bcr. C: : kan Pbcr. C E: bcr. C: : kan Pbce. A F: bcr. C: : kan Pyub. A A: yub. A-upp. P: : MLS Plep. A B: yub. A-upp. P: : MLS Pupp. S C: WT Plep. A D: WT Pupp. S TMB 3421 TMB 3422 TMB 3423 TMB 3424 TMB 3425 TMB 3426 TMB 3427 TMB 3428 TMB 3429 TMB 3430 TMB 3431 TMB 3432 TMB 3433 TMB 3434 TMB 3435 TMB 3436 TMB 3437 TMB 3438 TMB 3439 TMB 3440 TMB 3441 TMB 3442 2 4 5 6 7 8

Cryokulturen Noch mehr Cryokulturen für die folgenden Gruppen (AARGGHHH!!!): Gruppe 4: upp. P: : MLS = TMB 3408 Gruppe 6: bcr. C: : kan = TMB 3410 Gruppe 8: yub. A-upp. P: : MLS = TMB 3409 Wie wird die Cryokultur gemacht? 900µl 50% Glycerol + 900µl Kultur in 2 ml Cryoröhrchen mischen. STERIL arbeiten!!! Wie beschriften? Deckel: Nummer (ohne TMB) und „I“ für Klon I Tube: Cryo-Label mit Datum, Gruppennummer, Stammhintergrund (W 168 oder z. B. upp. P: : MLS) und Promoter (z. B. Plia. I-lux); Klon I beschriften Label auf Cryoröhrchen kleben und nochmal mit Tesafilm umkleben
 Thorsten mascher
Thorsten mascher Tina mascher
Tina mascher Renin angiotensin aldosteron system amboss
Renin angiotensin aldosteron system amboss Georges dolisi
Georges dolisi Physiologie renale
Physiologie renale Simultankontrast physiologie
Simultankontrast physiologie Glande zygomatique chien
Glande zygomatique chien Noyau gris centraux
Noyau gris centraux Tripelpunkt des wassers
Tripelpunkt des wassers Vernalisation
Vernalisation Physiologie de la vision
Physiologie de la vision Dr dina khnaba
Dr dina khnaba National geographic photo of the year
National geographic photo of the year Domain vs workgroup
Domain vs workgroup Thorsten butz
Thorsten butz Thorsten kollegger
Thorsten kollegger Gms bredstedt
Gms bredstedt Subclaviakatheter
Subclaviakatheter Thorsten joachims
Thorsten joachims Thorsten saager
Thorsten saager Thorsten reinhard
Thorsten reinhard Large language models in machine translation
Large language models in machine translation Thorsten joachims
Thorsten joachims Produktautomat
Produktautomat Heinz mandl
Heinz mandl Prof. mag.
Prof. mag. Check liste formation prof
Check liste formation prof Submetacentricni hromozomi
Submetacentricni hromozomi