ISOLAMENTO degli ACIDI NUCLEICI AVVERTENZE Utilizzare tessuti freschi


![CARATTERIZZAZIONE SPETTROFOTOMETRICA CONCENTRAZIONE: Misurazione dell’assorbimento a 260 nm A 260 = 1 OD [DNA]= CARATTERIZZAZIONE SPETTROFOTOMETRICA CONCENTRAZIONE: Misurazione dell’assorbimento a 260 nm A 260 = 1 OD [DNA]=](https://slidetodoc.com/presentation_image_h/f54aa21ec0ebba56973a49a82eb82147/image-3.jpg)














- Slides: 43

ISOLAMENTO degli ACIDI NUCLEICI AVVERTENZE: Utilizzare tessuti freschi o correttamente conservati Evitare di agitare fortemente o di pipettare La QUALITA’ del materiale di partenza influenza la qualità e la resa del DNA isolato Molecole grandi di DNA sono facilmente danneggiate dalle forze frizionali Lavorare in sterilità per prevenire contaminazioni da nucleasi Autoclavare soluzioni e strumenti Aggiungere alle soluzioni EDTA per chelare gli ioni Mg 2+ necessari per l’attività delle DNasi Trattare materiali e soluzioni con DEPC per inibire le RNasi

. . . ISOLAMENTO degli ACIDI NUCLEICI PROCEDURA: Rottura dei tessuti (mortaio e pestello, omogeneizzatori…) Trattamenti con RNasi e DNasi Digestione enzimatica della parete cellulare e lisi della membrana plasmatica Deproteinizzazione mediante soluzioni acquose di fenolo/cloroformio Le proteine denaturate rimangono all’interfaccia fra la fase organica e quella acquosa Precipitazione con etanolo o isopropanolo Conservazione a -20°C/-80°C in un tampone contenente EDTA Il DNA è soggetto a idrolisi acida se conservato in acqua
![CARATTERIZZAZIONE SPETTROFOTOMETRICA CONCENTRAZIONE Misurazione dellassorbimento a 260 nm A 260 1 OD DNA CARATTERIZZAZIONE SPETTROFOTOMETRICA CONCENTRAZIONE: Misurazione dell’assorbimento a 260 nm A 260 = 1 OD [DNA]=](https://slidetodoc.com/presentation_image_h/f54aa21ec0ebba56973a49a82eb82147/image-3.jpg)
CARATTERIZZAZIONE SPETTROFOTOMETRICA CONCENTRAZIONE: Misurazione dell’assorbimento a 260 nm A 260 = 1 OD [DNA]= 50 mg/ml A 260 = 1 OD [RNA]= 40 mg/ml PUREZZA: Rapporto A 260/A 280 = 1. 7/1. 9 (DNA) A 260/A 280 = 1. 9/2. 1 (RNA) Le misure spettrofotometriche non differenziano fra DNA ed RNA Il fenolo ha un massimo di assorbimento a 270 -275 nm

CONTROLLO INTEGRITA’ e TAGLIA Elettroforesi su gel di agarosio M 1 2 3 4 5 6 7 8 M Kb 97. 0 48. 5 M: Markers 1: Sangue 2: Cellule He. La 3: Cellule CHO 4: E. coli 5: B. subtilis 6: Coda di topo 7: Fegato 8: S� S. cerevisiae 1 2 3 28 S 26 S 4 5 25 S 23 S 18 S 16 S 18 S 1: Topo 2: Uomo 3: Lievito 4: Batterio 5: Pianta

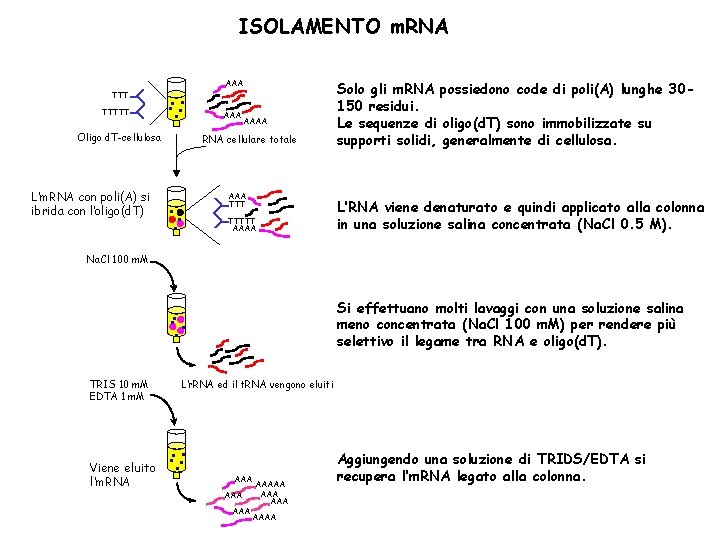
ISOLAMENTO m. RNA TTTTT . . . Oligo d. T-cellulosa L’m. RNA con poli(A) si ibrida con l’oligo(d. T) AAA AAAA RNA cellulare totale . . . AAA TTTTT AAAA Solo gli m. RNA possiedono code di poli(A) lunghe 30150 residui. Le sequenze di oligo(d. T) sono immobilizzate su supporti solidi, generalmente di cellulosa. L’RNA viene denaturato e quindi applicato alla colonna in una soluzione salina concentrata (Na. Cl 0. 5 M). Na. Cl 100 m. M Si effettuano molti lavaggi con una soluzione salina meno concentrata (Na. Cl 100 m. M) per rendere più selettivo il legame tra RNA e oligo(d. T). . . TRIS 10 m. M EDTA 1 m. M Viene eluito l’m. RNA L’r. RNA ed il t. RNA vengono eluiti . . . AAA AAAAA AAA AAAA Aggiungendo una soluzione di TRIDS/EDTA si recupera l’m. RNA legato alla colonna.

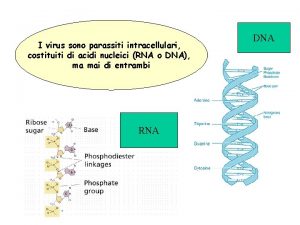
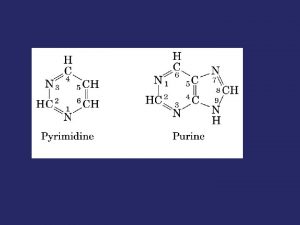
STRUTTURA degli ACIDI NUCLEICI

ANALISI FISICA Maggiore è G+C, maggiore è Tm. 1. 2 50 1. 1 1. 0 40 60 Tm 80 Temperatura (°C) 0 100 Grado di denaturazione (%) Temperatura di melting (Tm): Temperatura in corrispondenza della quale il DNA è denaturato al 50%. 1. 3 Assorbanza a 260 nm Effetto ipercromico: Aumento dell’A 260 nm durante il processo di denaturazione del DNA. Curva di fusione

CINETICA di RINATURAZIONE Curva C 0 t Effetto ipocromico: 0 Diminuzione dell’A 260 nm durante il processo di rinaturazione del DNA. 1) C 0 2) Tempo Concentrazione espressa in nucleotidi/unità di volume Rinaturazione (%) Parametri che influenzano la rinaturazione: DNA a rinaturazione rapida 50 DNA a rinaturazione lenta 100 La misura della velocità di rinaturazione può fornire utili informazioni circa la complessità del DNA -2 -1 0 1 2 Log Cot 3 4 5

TECNOLOGIA DEL DNA RICOMBINANTE Ha reso possibile il CLONAGGIO dei GENI permettendo di ISOLARE AMPLIFICARE SEQUENZIARE frammenti di DNA Perché manipolare i geni? 1) Per facilitare lo studio dell’espressione genica e della regolazione fisiologica; 2) per identificare il prodotto di un gene e/o per ottenerne la sovraespressione; 3) per studiare la relazione fra struttura e funzione delle proteine; 4) per identificare componenti cellulari che interagiscono con particolari sequenze di acidi nucleici o con particolari domini proteici

PROCEDURA di CLONAGGIO ISOLAMENTO del gene INSERZIONE del gene in un VETTORE PLASMIDICO INTRODUZIONE del vettore plasmidico IN CELLULE VIVENTI per propagarlo Le fasi più delicate sono quelle del taglio e dell’unione di sequenze di DNA in modo preciso…. …. . tutto ciò si ottiene con l’ausilio di ENZIMI

ENDONUCLEASI di RESTRIZIONE Sono enzimi che tagliano entrambi i filamenti della doppia elica del DNA in corrispondenza di specifiche sequenze. Riconoscono SEQUENZE PALINDROMICHE di 4 o 6 nucleotidi 5’-GAATTC-3’ 3’-CTTAAG-5’ Entrambe le catene hanno la stessa sequenza se lette in direzione 5’ 3’ Si distinguono in due classi: Enzimi di CLASSE I Tagliano il DNA in siti adiacenti alla sequenza riconosciuta Enzimi di CLASSE II Tagliano il DNA all’interno della sequenza riconosciuta

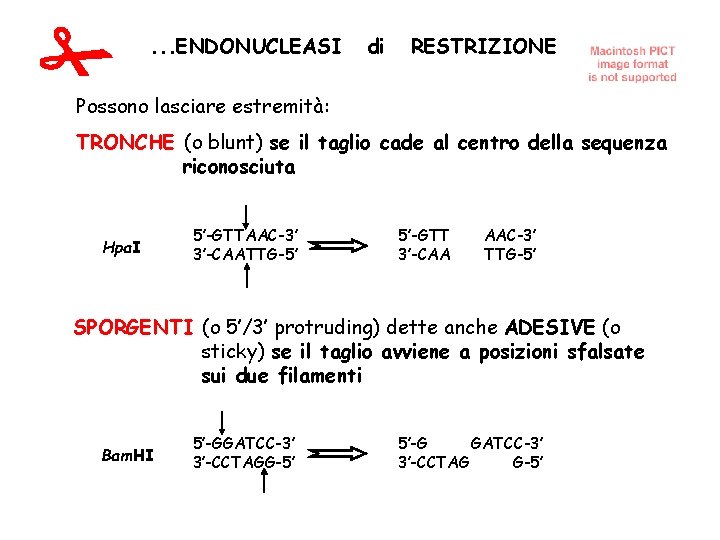
. . . ENDONUCLEASI di RESTRIZIONE Possono lasciare estremità: TRONCHE (o blunt) se il taglio cade al centro della sequenza riconosciuta Hpa. I 5’-GTTAAC-3’ 3’-CAATTG-5’ 5’-GTT 3’-CAA AAC-3’ TTG-5’ SPORGENTI (o 5’/3’ protruding) dette anche ADESIVE (o sticky) se il taglio avviene a posizioni sfalsate sui due filamenti Bam. HI 5’-GGATCC-3’ 3’-CCTAGG-5’ 5’-G GATCC-3’ 3’-CCTAG G-5’

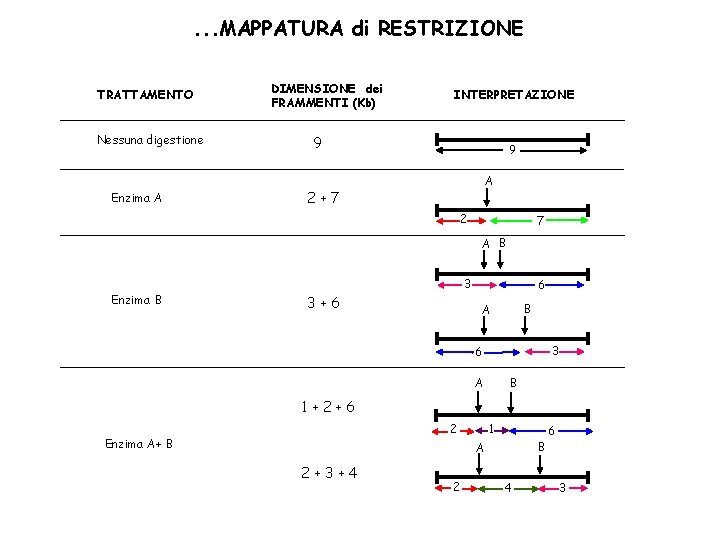
MAPPATURA di RESTRIZIONE E’ il processo di determinazione delle posizioni dei siti di taglio delle endonucleasi di restrizione all’interno di un pezzo di DNA. IMPIEGHI COMUNI: Distinzione di molecole di DNA della stessa lunghezza, ma con sequenze diverse senza doverle sequenziare. Individuazione di mutazioni geniche responsabili di malattie genetiche. Identificazione di un frammento di interesse da un digerito in base al suo peso molecolare.

. . . MAPPATURA di RESTRIZIONE TRATTAMENTO Nessuna digestione Enzima A DIMENSIONE dei FRAMMENTI (Kb) INTERPRETAZIONE 9 9 A 2+7 2 7 A B Enzima B 3 3+6 6 B A 3 6 A B 1+2+6 2 Enzima A+ B 1 B A 2+3+4 2 4 6 3

IDENTIFICAZIONE del FRAMMENTO di INTERESSE mediante Southern blotting M 1 � 2 3 4 5 1 2 3 4 5 6 7 8 9 10 11 12 M 1 2 3 4 5 6 Markers p. Blue. Star Xba. I/Sst. I Xba. I Bam. HI/Xba. I Nco. I 7 8 9 10 11 12 Sst. I/Nco. I Hin. DIII Nco. I/Hin. DIII Nco. I Nde. I/Nco. I Bam. HI/Nco. I 6 7 8 9 10 11 12

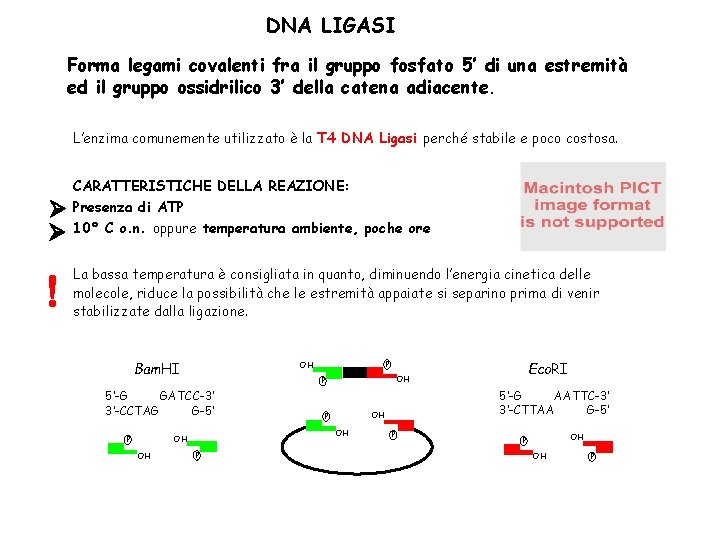
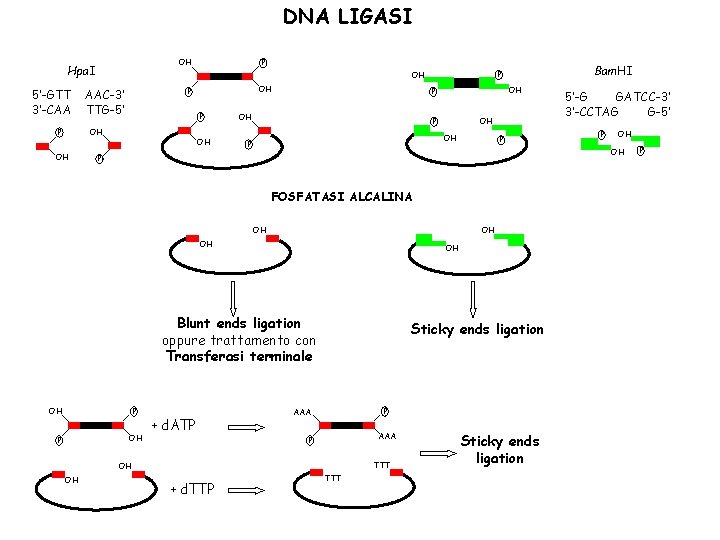
DNA LIGASI Forma legami covalenti fra il gruppo fosfato 5’ di una estremità ed il gruppo ossidrilico 3’ della catena adiacente. L’enzima comunemente utilizzato è la T 4 DNA Ligasi perché stabile e poco costosa. CARATTERISTICHE DELLA REAZIONE: Presenza di ATP 10° C o. n. oppure temperatura ambiente, poche ore La bassa temperatura è consigliata in quanto, diminuendo l’energia cinetica delle molecole, riduce la possibilità che le estremità appaiate si separino prima di venir stabilizzate dalla ligazione. Bam. HI OH P 5’-G AATTC-3’ 3’-CTTAA G-5’ OH P OH OH Eco. RI OH P 5’-G GATCC-3’ 3’-CCTAG G-5’ P P OH P

DNA LIGASI 5’-GTT 3’-CAA P OH P AAC-3’ TTG-5’ P OH OH P Bam. HI P OH OH OH P OH Hpa. I P FOSFATASI ALCALINA OH OH Blunt ends ligation oppure trattamento con Transferasi terminale P OH P + d. ATP Sticky ends ligation P AAA P TTT OH OH + d. TTP P OH OH P OH 5’-G GATCC-3’ 3’-CCTAG G-5’ TTT Sticky ends ligation P

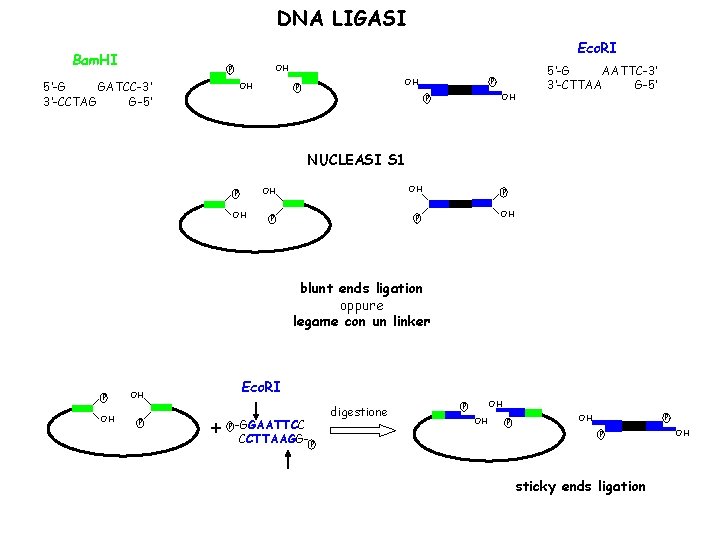
DNA LIGASI Eco. RI Bam. HI OH P 5’-G GATCC-3’ 3’-CCTAG G-5’ OH P 5’-G AATTC-3’ 3’-CTTAA G-5’ NUCLEASI S 1 P OH OH OH P P P OH blunt ends ligation oppure legame con un linker P OH Eco. RI OH P + P -GGAATTCC CCTTAAGG- P digestione OH P P OH P sticky ends ligation OH

PLASMIDI Piccole molecole di DNA batterico extracromosomico, circolare. In natura conferiscono vantaggi selettivi ai ceppi che li contengono. Ingegnerizzati per l’uso di laboratorio. Per essere un buon vettore di clonaggio, un plasmide deve avere: Dimensioni contenute Un sito di origine della replicazione di tipo batterico controllato in maniera “rilassata” o “stringente” geni che conferiscono resistenza a due Due antibiotici Siti unici di restrizione

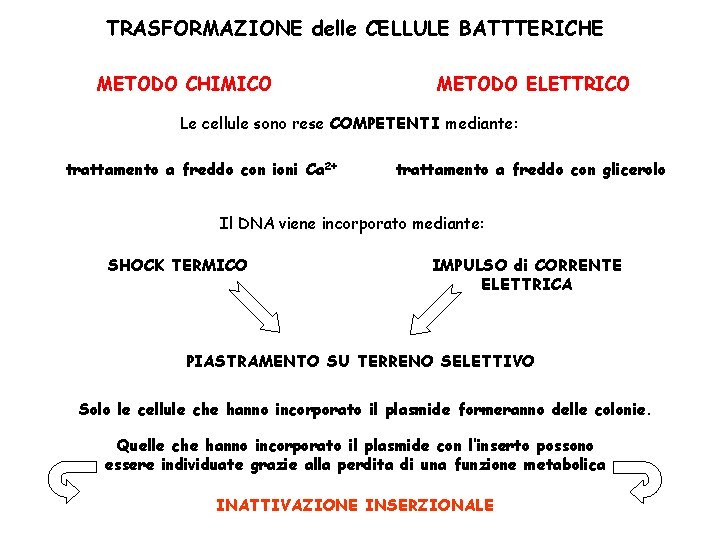
TRASFORMAZIONE delle CELLULE BATTTERICHE METODO CHIMICO METODO ELETTRICO Le cellule sono rese COMPETENTI mediante: trattamento a freddo con ioni Ca 2+ trattamento a freddo con glicerolo Il DNA viene incorporato mediante: SHOCK TERMICO IMPULSO di CORRENTE ELETTRICA PIASTRAMENTO SU TERRENO SELETTIVO Solo le cellule che hanno incorporato il plasmide formeranno delle colonie. Quelle che hanno incorporato il plasmide con l’inserto possono essere individuate grazie alla perdita di una funzione metabolica INATTIVAZIONE INSERZIONALE

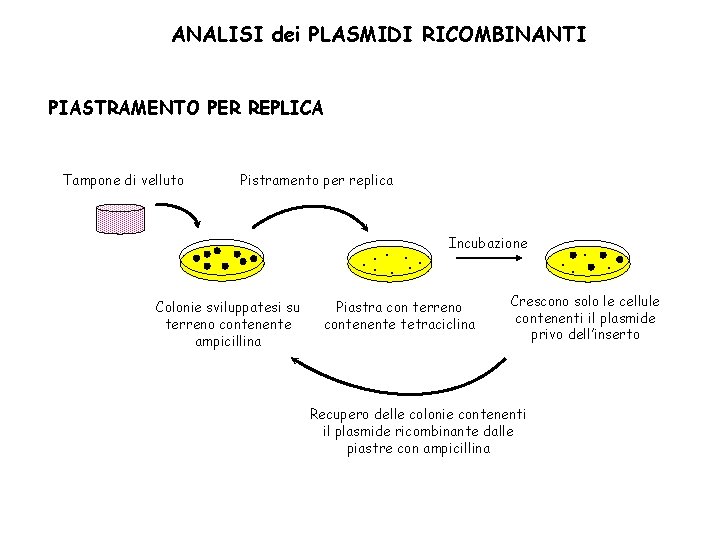
ANALISI dei PLASMIDI RICOMBINANTI PIASTRAMENTO PER REPLICA Tampone di velluto Pistramento per replica Colonie sviluppatesi su terreno contenente ampicillina Incubazione Piastra con terreno contenente tetraciclina Crescono solo le cellule contenenti il plasmide privo dell’inserto Recupero delle colonie contenenti il plasmide ricombinante dalle piastre con ampicillina

…ANALISI dei PLASMIDI RICOMBINANTI SCREENING BLU/BIANCO lac. Z gene che codifica per la b-galattosidasi interrotto dal sito di clonaggio multiplo (pcs) L’enzima b-galattosidasi è in grado di metabolizzare il substrato incolore X-Gal formando un composto di colore blu. Un ceppo batterico lac-, trasformato con il plasmide ricombinante p. UC 19, è in grado di metabolizzare il substrato X-Gal solo se il plasmide è privo dell’inserto e forma colonie di colore blu. Colonie di batteri lac+ che contengono plasmidi senza inserto Colonie di batteri lac- che contengono plasmidi ricombinanti Piastra con terreno contenente X-Gal pcs

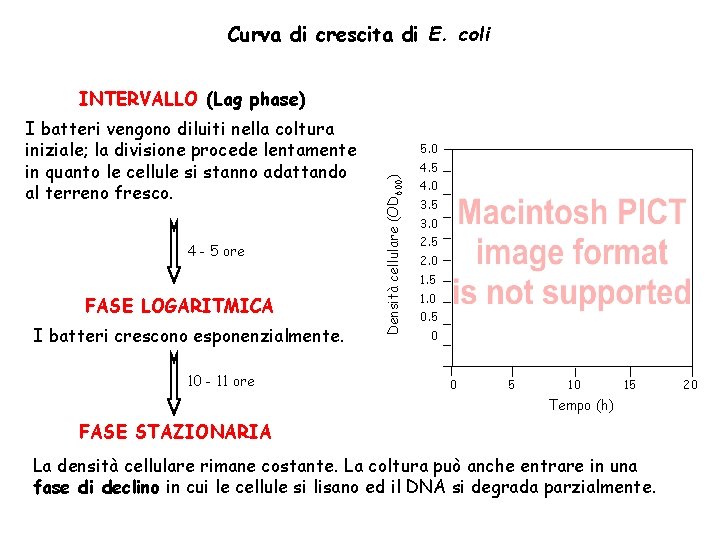
Curva di crescita di E. coli INTERVALLO (Lag phase) 4 - 5 ore FASE LOGARITMICA I batteri crescono esponenzialmente. 10 - 11 ore 5. 0 Densità cellulare (OD 600) I batteri vengono diluiti nella coltura iniziale; la divisione procede lentamente in quanto le cellule si stanno adattando al terreno fresco. 4. 5 4. 0 3. 5 3. 0 2. 5 2. 0 1. 5 1. 0 0. 5 0 0 5 10 15 Tempo (h) FASE STAZIONARIA La densità cellulare rimane costante. La coltura può anche entrare in una fase di declino in cui le cellule si lisano ed il DNA si degrada parzialmente. 20

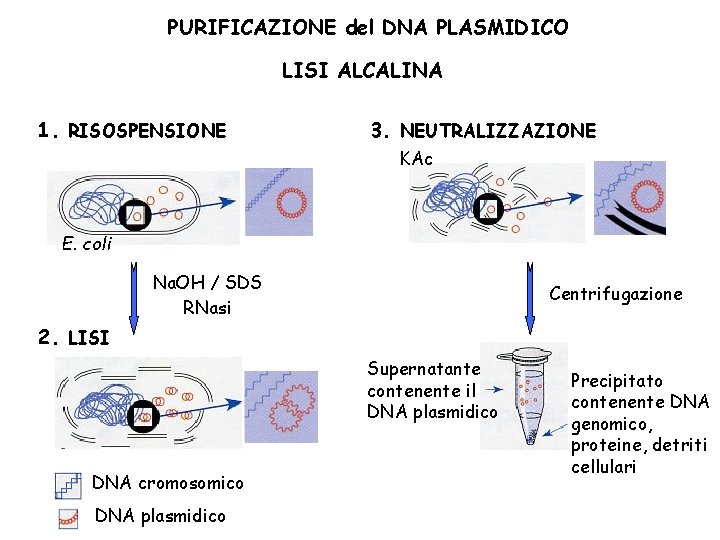
PURIFICAZIONE del DNA PLASMIDICO LISI ALCALINA 1. RISOSPENSIONE 3. NEUTRALIZZAZIONE KAc E. coli Na. OH / SDS RNasi Centrifugazione 2. LISI Supernatante contenente il DNA plasmidico DNA cromosomico DNA plasmidico Precipitato contenente DNA genomico, proteine, detriti cellulari

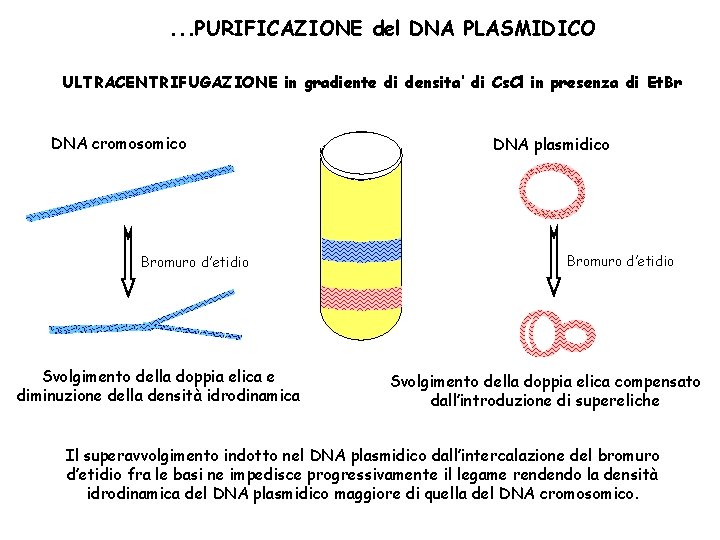
. . . PURIFICAZIONE del DNA PLASMIDICO ULTRACENTRIFUGAZIONE in gradiente di densita’ di Cs. Cl in presenza di Et. Br DNA cromosomico Bromuro d’etidio Svolgimento della doppia elica e diminuzione della densità idrodinamica DNA plasmidico Bromuro d’etidio Svolgimento della doppia elica compensato dall’introduzione di supereliche Il superavvolgimento indotto nel DNA plasmidico dall’intercalazione del bromuro d’etidio fra le basi ne impedisce progressivamente il legame rendendo la densità idrodinamica del DNA plasmidico maggiore di quella del DNA cromosomico.

VETTORI di ESPRESSIONE CLONARE OTTENERE NUMEROSE COPIE IDENTICHE di un certo frammento di DNA MA, CLONARE IN UN VETTORE DI ESPRESSIONE OTTENERE DISCRETE QUANTITÀ del PRODOTTO PROTEICO codificato dal gene di interesse INSULINA ORMONE DELLA CRESCITA UMANO INTERFERONI sono solo pochi esempi di proteine commercializzate prodotte da batteri

. . . VETTORI di ESPRESSIONE PROPRIETA’: Oltre alle normali caratteristiche di un vettore di clonaggio, un vettore per l’espressione di geni in cellule batteriche DEVE POSSEDERE: SEQUENZA PROMOTRICE SEQUENZA SHINE-DALGARNO a monte di uno o più siti di inserzione del DNA estraneo AVVERTENZE: UTILIZZARE il c. DNA CLONARE il c. DNA nella CORRETTA CORNICE di LETTURA CONSIDERAZIONI: I batteri non sono in grado di eseguire modificazioni post-traduzionali

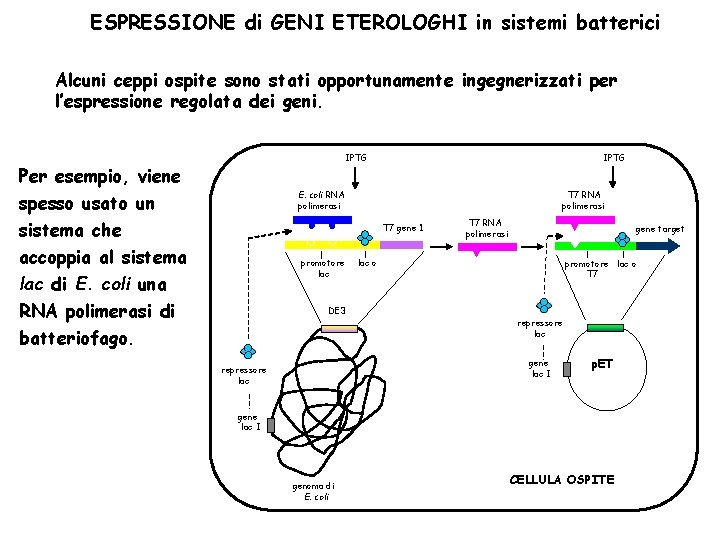
ESPRESSIONE di GENI ETEROLOGHI in sistemi batterici Alcuni ceppi ospite sono stati opportunamente ingegnerizzati per l’espressione regolata dei geni. IPTG Per esempio, viene spesso usato un IPTG E. coli RNA polimerasi sistema che accoppia al sistema T 7 RNA polimerasi T 7 gene 1 promotore lac di E. coli una RNA polimerasi di batteriofago. DE 3 T 7 RNA polimerasi gene target lac o promotore lac o T 7 repressore lac gene lac I repressore lac p. ET gene lac I genoma di E. coli CELLULA OSPITE

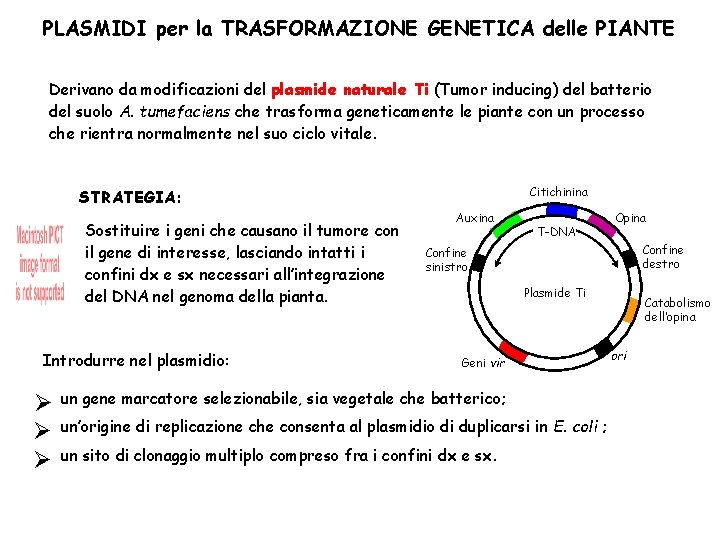
PLASMIDI per la TRASFORMAZIONE GENETICA delle PIANTE Derivano da modificazioni del plasmide naturale Ti (Tumor inducing) del batterio del suolo A. tumefaciens che trasforma geneticamente le piante con un processo che rientra normalmente nel suo ciclo vitale. Citichinina STRATEGIA: Sostituire i geni che causano il tumore con il gene di interesse, lasciando intatti i confini dx e sx necessari all’integrazione del DNA nel genoma della pianta. Introdurre nel plasmidio: Auxina T-DNA Opina Confine destro Confine sinistro Plasmide Ti Geni vir un gene marcatore selezionabile, sia vegetale che batterico; un’origine di replicazione che consenta al plasmidio di duplicarsi in E. coli ; un sito di clonaggio multiplo compreso fra i confini dx e sx. Catabolismo dell’opina ori

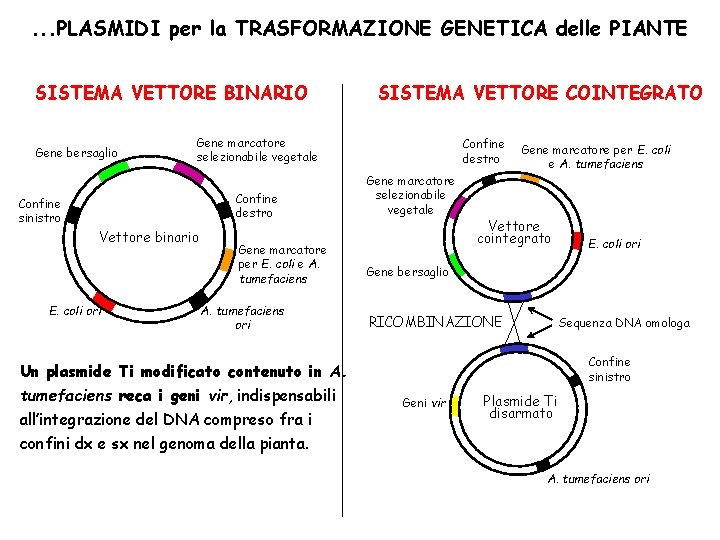
. . . PLASMIDI per la TRASFORMAZIONE GENETICA delle PIANTE SISTEMA VETTORE BINARIO Gene bersaglio Gene marcatore selezionabile vegetale Confine destro Confine sinistro Vettore binario E. coli ori SISTEMA VETTORE COINTEGRATO Gene marcatore per E. coli e A. tumefaciens ori Un plasmide Ti modificato contenuto in A. tumefaciens reca i geni vir, indispensabili all’integrazione del DNA compreso fra i confini dx e sx nel genoma della pianta. Confine destro Gene marcatore selezionabile vegetale Gene marcatore per E. coli e A. tumefaciens Vettore cointegrato E. coli ori Gene bersaglio RICOMBINAZIONE Sequenza DNA omologa Confine sinistro Geni vir Plasmide Ti disarmato A. tumefaciens ori

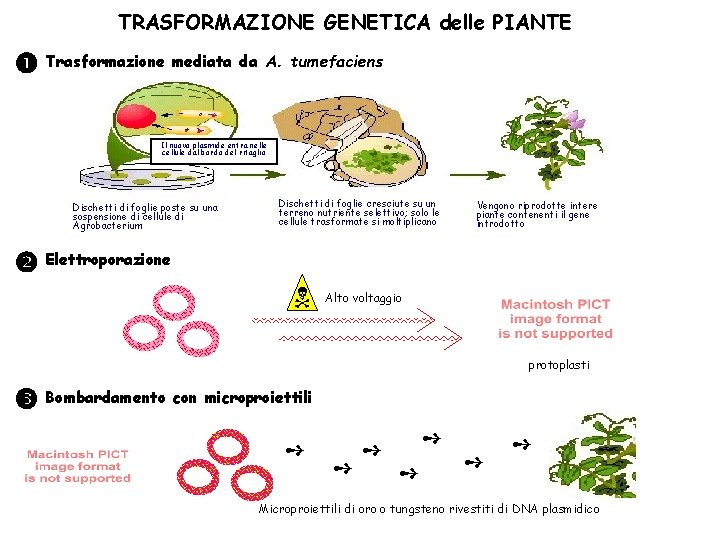
TRASFORMAZIONE GENETICA delle PIANTE Trasformazione mediata da A. tumefaciens Il nuovo plasmide entra nelle cellule dal bordo del ritaglio Dischetti di foglie poste su una sospensione di cellule di Agrobacterium Dischetti di foglie cresciute su un terreno nutriente selettivo; solo le cellule trasformate si moltiplicano Vengono riprodotte intere piante contenenti il gene introdotto Elettroporazione Alto voltaggio protoplasti Bombardamento con microproiettili ➺ ➺ ➺ ➺ Microproiettili di oro o tungsteno rivestiti di DNA plasmidico

OLTRE AI PLASMIDI. . . I plasmidi sono i vettori di elezione per clonare piccoli frammenti di DNA (5 -10 Kb). INFATTI, Plasmidi di grandi dimensioni sono instabili e possono andare incontro a delezioni spontanee. La trasformazione non avviene efficentemente. QUINDI, Per clonare frammenti più grandi si impiegano come vettori del DNA dei batteriofagi (virus batterici)che consentono di iniettare grosse molecole di DNA nelle cellule batteriche per infezione. VETTORI FAGICI VETTORI COSMIDICI

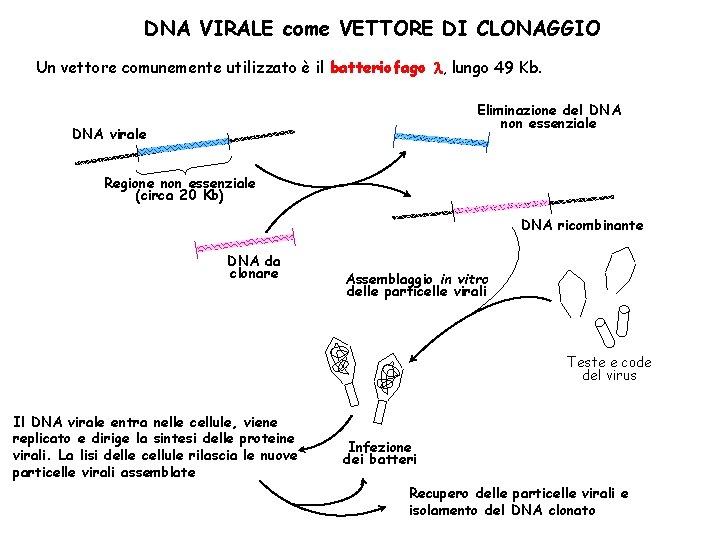
DNA VIRALE come VETTORE DI CLONAGGIO Un vettore comunemente utilizzato è il batteriofago l, lungo 49 Kb. Eliminazione del DNA non essenziale DNA virale Regione non essenziale (circa 20 Kb) DNA ricombinante DNA da clonare Assemblaggio in vitro delle particelle virali Teste e code del virus Il DNA virale entra nelle cellule, viene replicato e dirige la sintesi delle proteine virali. La lisi delle cellule rilascia le nuove particelle virali assemblate Infezione dei batteri Recupero delle particelle virali e isolamento del DNA clonato

COSMIDI Sono PLASMIDI, ma contengono, oltre alle caratteristiche essenziali di un plasmide, anche un SITO COS La presenza di questo elemento è determinante per consentire l’inserimento della molecola di DNA ricombinante nella testa del virus che, a sua volta, la inietta in una cellula batterica per infezione.

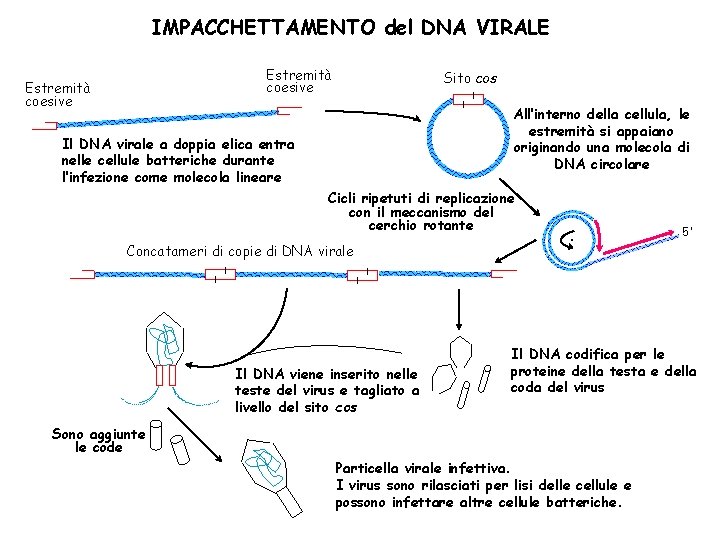
IMPACCHETTAMENTO del DNA VIRALE Estremità coesive Sito cos All’interno della cellula, le estremità si appaiano originando una molecola di DNA circolare Il DNA virale a doppia elica entra nelle cellule batteriche durante l’infezione come molecola lineare Cicli ripetuti di replicazione con il meccanismo del cerchio rotante Concatameri di copie di DNA virale Il DNA viene inserito nelle teste del virus e tagliato a livello del sito cos • 5’ Il DNA codifica per le proteine della testa e della coda del virus Sono aggiunte le code Particella virale infettiva. I virus sono rilasciati per lisi delle cellule e possono infettare altre cellule batteriche.

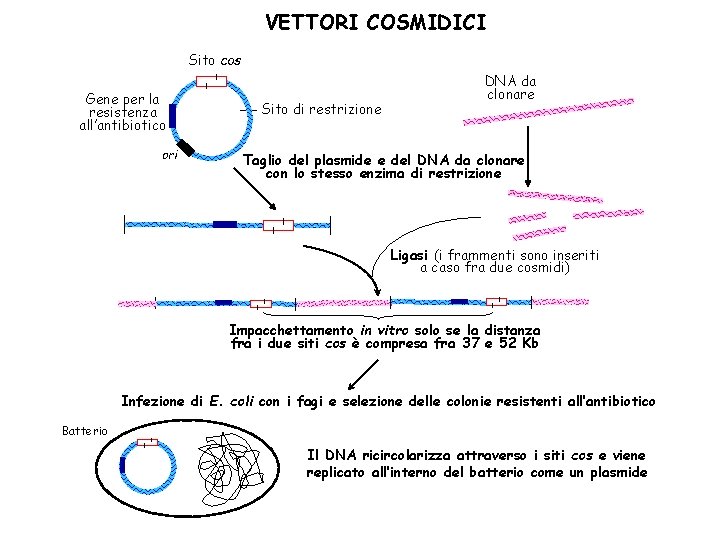
VETTORI COSMIDICI Sito cos Gene per la resistenza all’antibiotico ori Sito di restrizione DNA da clonare Taglio del plasmide e del DNA da clonare con lo stesso enzima di restrizione Ligasi (i frammenti sono inseriti a caso fra due cosmidi) Impacchettamento in vitro solo se la distanza fra i due siti cos è compresa fra 37 e 52 Kb Infezione di E. coli con i fagi e selezione delle colonie resistenti all’antibiotico Batterio Il DNA ricircolarizza attraverso i siti cos e viene replicato all’interno del batterio come un plasmide

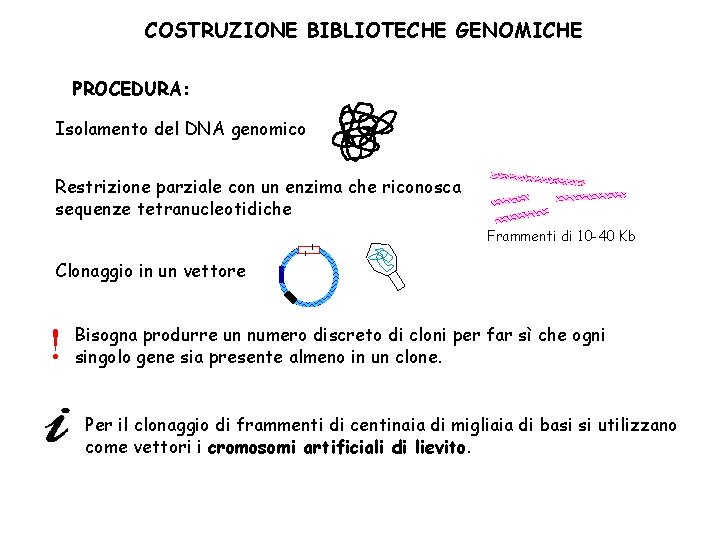
COSTRUZIONE BIBLIOTECHE GENOMICHE PROCEDURA: Isolamento del DNA genomico Restrizione parziale con un enzima che riconosca sequenze tetranucleotidiche Frammenti di 10 -40 Kb Clonaggio in un vettore Bisogna produrre un numero discreto di cloni per far sì che ogni singolo gene sia presente almeno in un clone. Per il clonaggio di frammenti di centinaia di migliaia di basi si utilizzano come vettori i cromosomi artificiali di lievito.

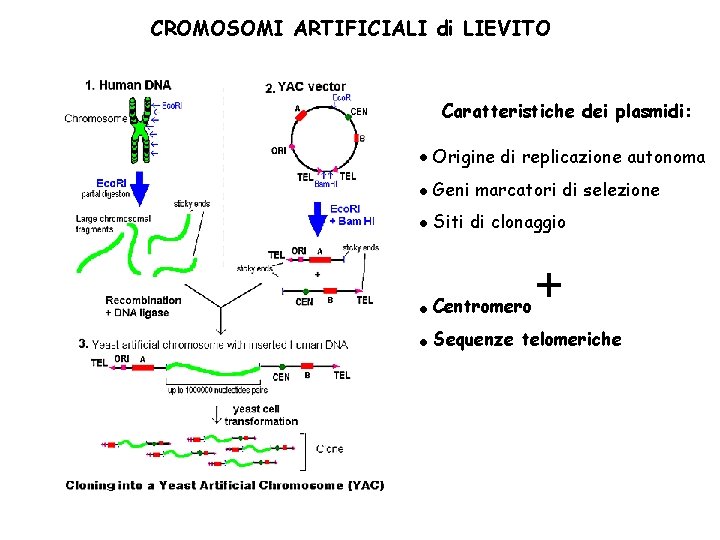
CROMOSOMI ARTIFICIALI di LIEVITO Caratteristiche dei plasmidi: Origine di replicazione autonoma Geni marcatori di selezione Siti di clonaggio Centromero Sequenze telomeriche

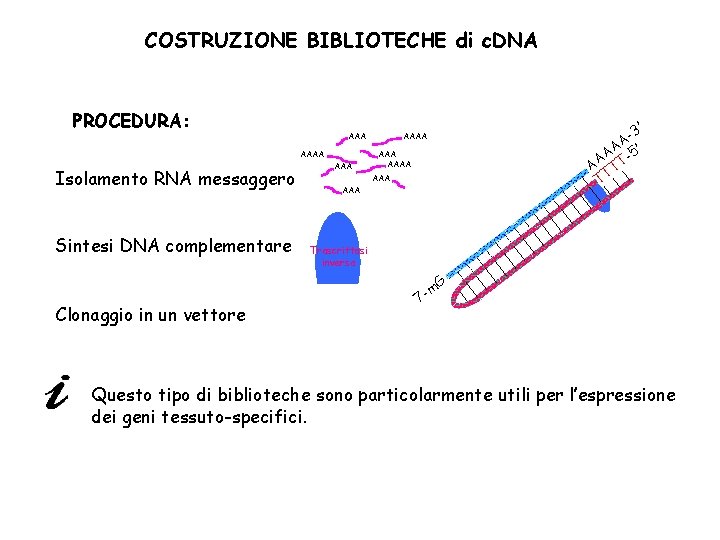
COSTRUZIONE BIBLIOTECHE di c. DNA PROCEDURA: AAAA Isolamento RNA messaggero Sintesi DNA complementare AAA 3’ AA 5’ A AA TT TT AAAA AAA AAA Trascrittasi inversa G Clonaggio in un vettore m 7 - Questo tipo di biblioteche sono particolarmente utili per l’espressione dei geni tessuto-specifici.

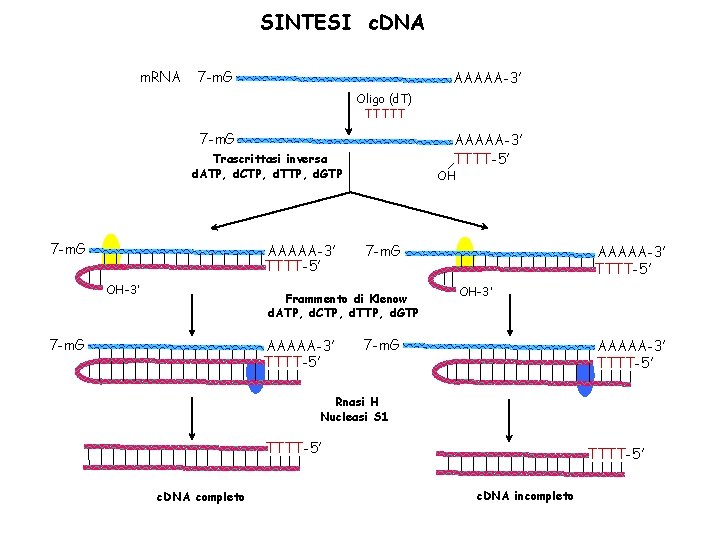
SINTESI c. DNA m. RNA 7 -m. G AAAAA-3’ Oligo (d. T) TTTTT 7 -m. G AAAAA-3’ TTTT-5’ Trascrittasi inversa d. ATP, d. CTP, d. TTP, d. GTP 7 -m. G AAAAA-3’ TTTT-5’ OH-3’ OH 7 -m. G Frammento di Klenow d. ATP, d. CTP, d. TTP, d. GTP 7 -m. G AAAAA-3’ TTTT-5’ OH-3’ 7 -m. G AAAAA-3’ TTTT-5’ Rnasi H Nucleasi S 1 TTTT-5’ c. DNA completo TTTT-5’ c. DNA incompleto

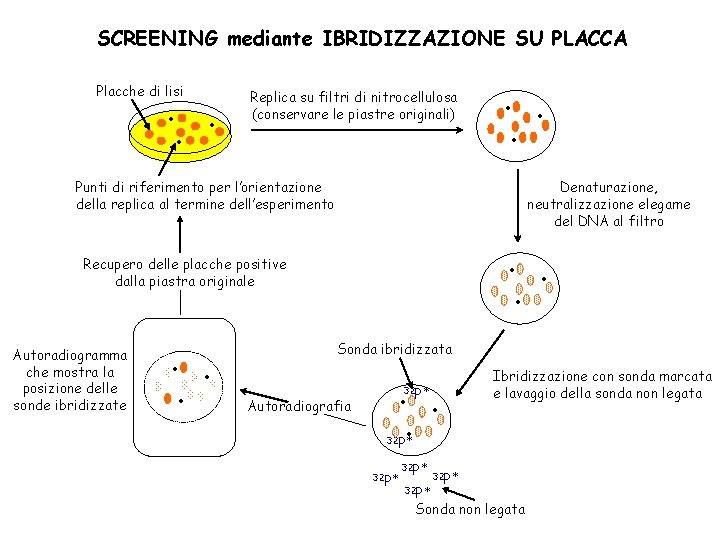
SCREENING mediante IBRIDIZZAZIONE SU PLACCA Placche di lisi • • • Replica su filtri di nitrocellulosa (conservare le piastre originali) • • Denaturazione, neutralizzazione elegame del DNA al filtro Punti di riferimento per l’orientazione della replica al termine dell’esperimento Recupero delle placche positive dalla piastra originale Autoradiogramma che mostra la posizione delle sonde ibridizzate • • • Sonda ibridizzata • 32 P • Autoradiografia • • Ibridizzazione con sonda marcata e lavaggio della sonda non legata 32 P 32 P Sonda non legata

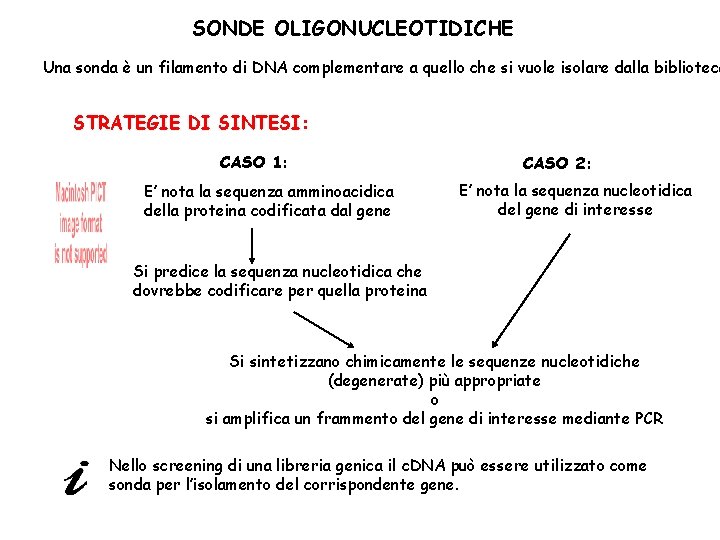
SONDE OLIGONUCLEOTIDICHE Una sonda è un filamento di DNA complementare a quello che si vuole isolare dalla biblioteca STRATEGIE DI SINTESI: CASO 1: E’ nota la sequenza amminoacidica della proteina codificata dal gene CASO 2: E’ nota la sequenza nucleotidica del gene di interesse Si predice la sequenza nucleotidica che dovrebbe codificare per quella proteina Si sintetizzano chimicamente le sequenze nucleotidiche (degenerate) più appropriate o si amplifica un frammento del gene di interesse mediante PCR Nello screening di una libreria genica il c. DNA può essere utilizzato come sonda per l’isolamento del corrispondente gene.

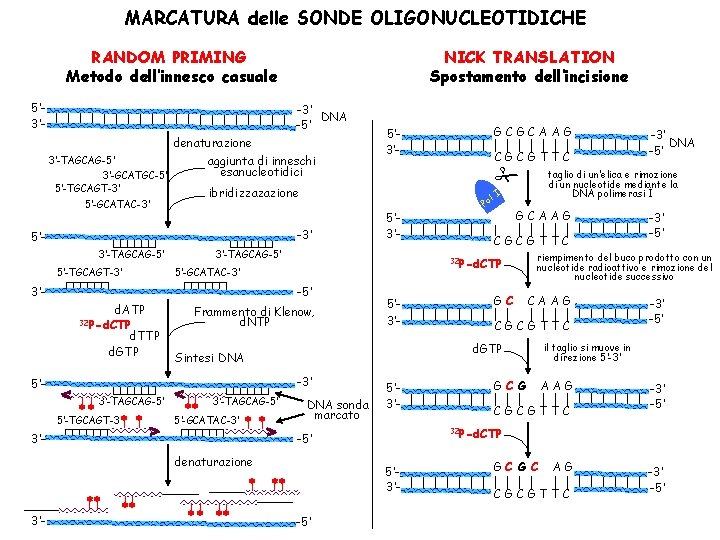
MARCATURA delle SONDE OLIGONUCLEOTIDICHE RANDOM PRIMING Metodo dell’innesco casuale 5’ 3’- NICK TRANSLATION Spostamento dell’incisione -3’ DNA -5’ denaturazione aggiunta di inneschi esanucleotidici 3’-TAGCAG-5’ 3’-GCATGC-5’ 5’-TGCAGT-3’ 3’-TAGCAG-5’ d. ATP 32 P-d. CTP d. TTP d. GTP Frammento di Klenow, d. NTP 3’-TAGCAG-5’ 5’-GCATAC-3’ 3’- DNA sonda marcato 5’ 3’- CGCGTTC GC denaturazione 5’ 3’- -5’ -3’ -5’ riempimento del buco prodotto con un nucleotide radioattivo e rimozione del nucleotide successivo CAAG CGCGTTC GC G -3’ -5’ il taglio si muove in direzione 5’-3’ AAG CGCGTTC -3’ -5’ 32 P-d. CTP -5’ GCAAG d. GTP -3’ 5’-TGCAGT-3’ 5’ 3’- Sintesi DNA 5’ 3’-TAGCAG-5’ G 32 P-d. CTP 5’-GCATAC-3’ 3’- 5’ 3’- Po -3’ DNA -5’ taglio di un’elica e rimozione di un nucleotide mediante la DNA polimerasi I l. I -3’ 5’- CGCGTTC ibridizzazazione 5’-GCATAC-3’ GCGCAAG 5’ 3’- GC G C AG CGCGTTC -3’ -5’
 Ibridazione dna
Ibridazione dna Legame fosfodiesterico
Legame fosfodiesterico Gemmazione virus
Gemmazione virus Acidi nucleici
Acidi nucleici Acidi nucleici
Acidi nucleici Polifagia paradossa
Polifagia paradossa Acidi nucleici
Acidi nucleici Acidi nucleici
Acidi nucleici Quali sono le biomolecole
Quali sono le biomolecole Glutammina
Glutammina Ghiandola acinosa composta
Ghiandola acinosa composta Prismi dello smalto
Prismi dello smalto Fibre xilari
Fibre xilari Structura primara adn
Structura primara adn Junta de isolamento elétrico
Junta de isolamento elétrico O que é microbiologia
O que é microbiologia Diffusione termica
Diffusione termica Isolamento reprodutivo
Isolamento reprodutivo L'isolamento prezigotico impedisce
L'isolamento prezigotico impedisce Isolamento prezigotico impedisce
Isolamento prezigotico impedisce Isolamento reprodutivo
Isolamento reprodutivo Speciazione parapatrica
Speciazione parapatrica Acidi e basi forti e deboli
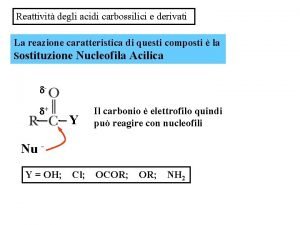
Acidi e basi forti e deboli Scala reattività derivati acidi carbossilici
Scala reattività derivati acidi carbossilici Reazione di saponificazione zanichelli
Reazione di saponificazione zanichelli Farmaceutska tehnologija s kozmetologijom
Farmaceutska tehnologija s kozmetologijom Acidi e basi forti e deboli
Acidi e basi forti e deboli Intermedi ciclo di krebs
Intermedi ciclo di krebs Acido carbossilico
Acido carbossilico Composti inorganici
Composti inorganici Sali biliari
Sali biliari Acidi basi e sali mappa concettuale
Acidi basi e sali mappa concettuale Acidi di lewis esempi
Acidi di lewis esempi Fosfatidilglicerolo
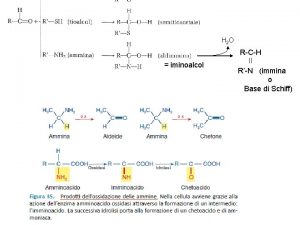
Fosfatidilglicerolo Immina
Immina Acidi e basi forti e deboli
Acidi e basi forti e deboli Perclorato rameico
Perclorato rameico Acidi glutaminici
Acidi glutaminici Mappa concettuale acidi e basi
Mappa concettuale acidi e basi Acidi e basi forti e deboli
Acidi e basi forti e deboli Ionizzazione dell'acqua zanichelli
Ionizzazione dell'acqua zanichelli Acidi di arrhenius
Acidi di arrhenius Metabolismo acidi grassi
Metabolismo acidi grassi Acidi e basi si scambiano protoni zanichelli
Acidi e basi si scambiano protoni zanichelli