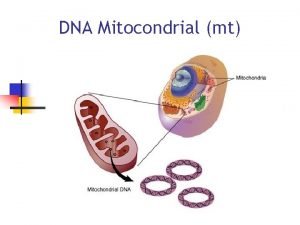
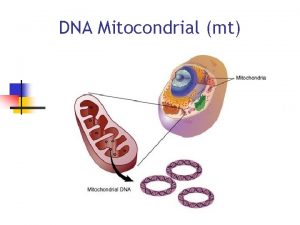
DNA mitocondrial Michelle de Saint Pierre FACSO Universidad









- Slides: 25

DNA mitocondrial Michelle de Saint Pierre FACSO, Universidad de Chile.

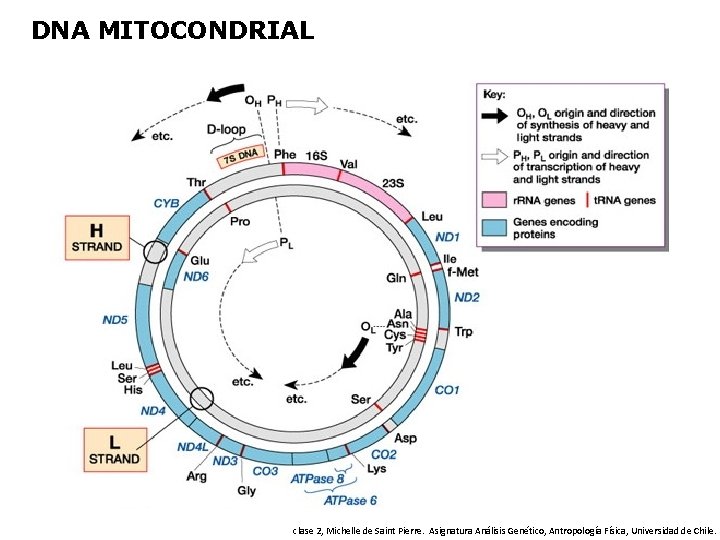
DNA MITOCONDRIAL Es una molécula bicatenaria, circular, cerrada, sin extremos Codifica para 37 genes; dos ARN ribosómicos, 22 ARN de transferencia y 13 proteínas que participan en la fosforilación oxidativa -Dos cadenas diferentes L y H -Las dos cadenas se pueden separar en gradientes de densidad de p. H clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

DNA MITOCONDRIAL clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

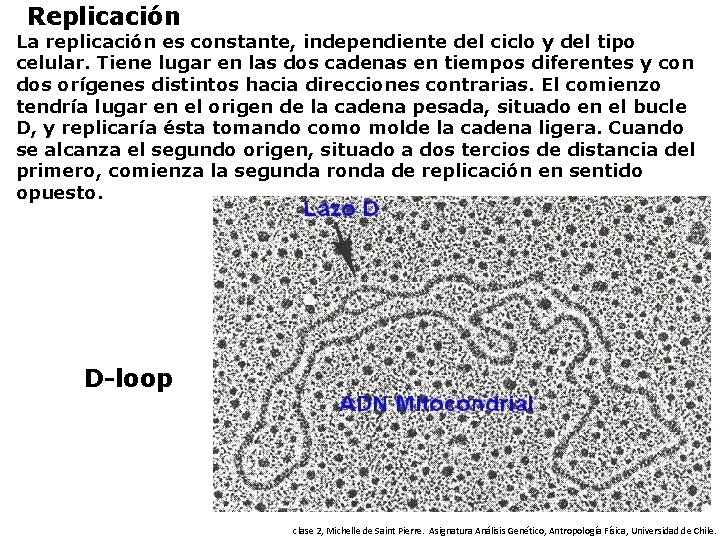
Replicación La replicación es constante, independiente del ciclo y del tipo celular. Tiene lugar en las dos cadenas en tiempos diferentes y con dos orígenes distintos hacia direcciones contrarias. El comienzo tendría lugar en el origen de la cadena pesada, situado en el bucle D, y replicaría ésta tomando como molde la cadena ligera. Cuando se alcanza el segundo origen, situado a dos tercios de distancia del primero, comienza la segunda ronda de replicación en sentido opuesto. D-loop clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

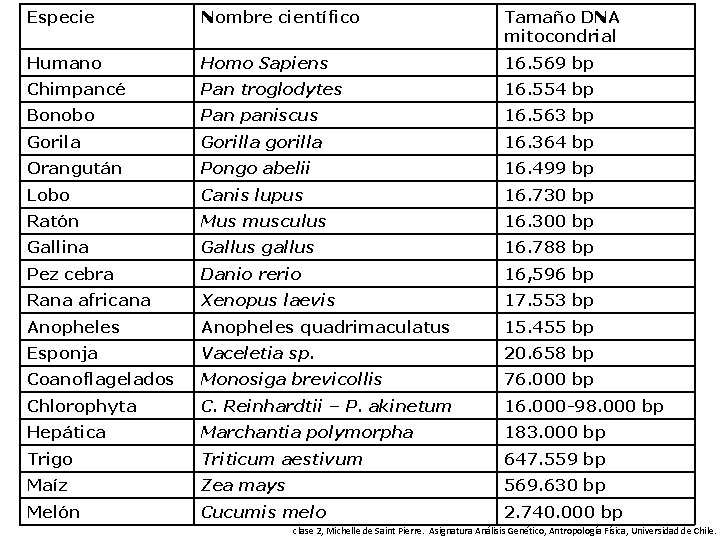
Especie Nombre científico Tamaño DNA mitocondrial Humano Homo Sapiens 16. 569 bp Chimpancé Pan troglodytes 16. 554 bp Bonobo Pan paniscus 16. 563 bp Gorila Gorilla gorilla 16. 364 bp Orangután Pongo abelii 16. 499 bp Lobo Canis lupus 16. 730 bp Ratón Mus musculus 16. 300 bp Gallina Gallus gallus 16. 788 bp Pez cebra Danio rerio 16, 596 bp Rana africana Xenopus laevis 17. 553 bp Anopheles quadrimaculatus 15. 455 bp Esponja Vaceletia sp. 20. 658 bp Coanoflagelados Monosiga brevicollis 76. 000 bp Chlorophyta C. Reinhardtii – P. akinetum 16. 000 -98. 000 bp Hepática Marchantia polymorpha 183. 000 bp Trigo Triticum aestivum 647. 559 bp Maíz Zea mays 569. 630 bp Melón Cucumis melo 2. 740. 000 bp clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

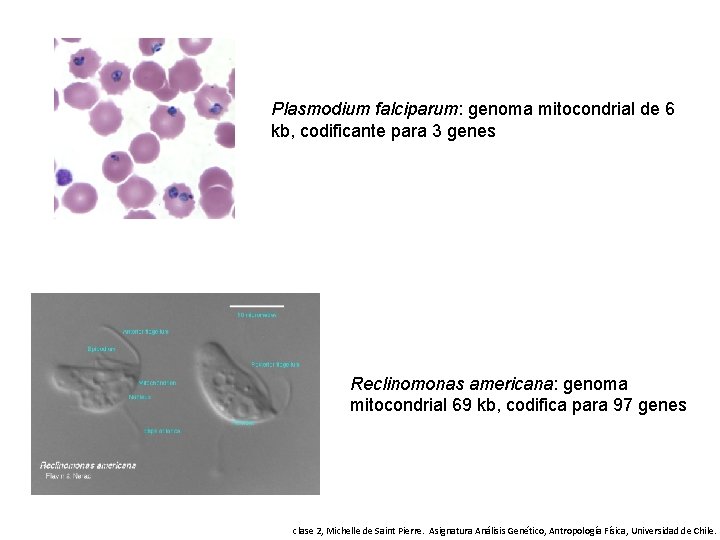
Plasmodium falciparum: genoma mitocondrial de 6 kb, codificante para 3 genes Reclinomonas americana: genoma mitocondrial 69 kb, codifica para 97 genes clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

Evolución Celular: Origen de las Mitocondrias Lynn Margulis en 1967 en su libro “symbiosis in cell evolution” propuso la teoría endosimbiotica seriada para explicar el origen de los eucariontes. Esta teoría describe el origen de los eucariontes en una serie de cuatro incorporaciones (núcleo, flagelos, mitocondrias y cloroplastos) mediante las cuales, por la unión simbiogenética de bacterias, se originaron las células que conforman a los individuos de los otros cuatro reinos (protistas, animales, hongos y plantas), hace 2. 000 millones de años atrás. Hoy se cree que la mitocondria provendría de probablemente alfa proteobacterias (ej. Rickettsias) Rickettsia rickettsii en células de garrapata Lynn Margulis 1938 -2011 clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

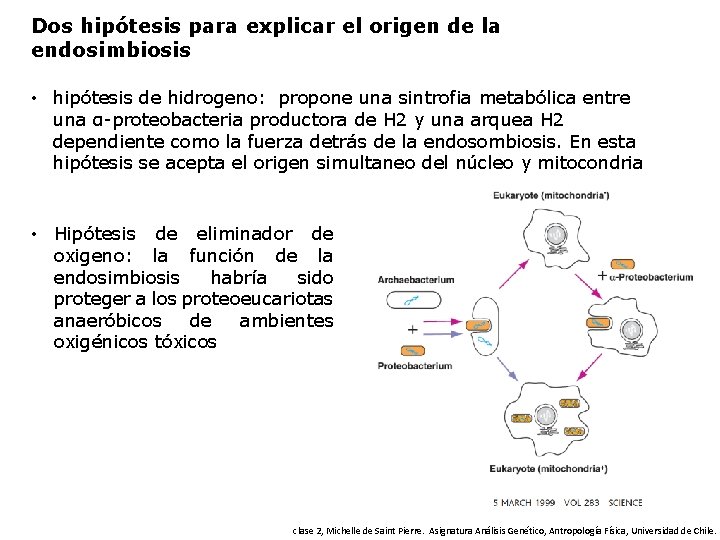
Dos hipótesis para explicar el origen de la endosimbiosis • hipótesis de hidrogeno: propone una sintrofia metabólica entre una α-proteobacteria productora de H 2 y una arquea H 2 dependiente como la fuerza detrás de la endosombiosis. En esta hipótesis se acepta el origen simultaneo del núcleo y mitocondria • Hipótesis de eliminador de oxigeno: la función de la endosimbiosis habría sido proteger a los proteoeucariotas anaeróbicos de ambientes oxigénicos tóxicos clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

ADN Mitocondrial • Multicopia • ~16. 5 Kbp • Herencia de tipo materna (en la mayoría de los casos) • En algunos casos posee heteroplasmia (formas originales y mutadas coexisten) • No sufre recombinación • Mayor tasa de cambio en región hipervariable • Más estable para análisis forenses • El mayor grado de variación se encuentra en la D-loop clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

NUCLEAR VS MITOCONDRIAL nuc. ADN mt. ADN Forma Lineal Circular Número de copias 2 por célula Miles por célula Tamaño Variable (6 x 109) ~16. 5 Kbp Tipo de herencia Biparental Uniparental Número de alelos Dep. del nivel de ploidía 1 Recombinación Si No Estabilidad Baja Alta Tasa de mutación Baja Alta Análisis Complejo Simple Transformación Clonación minipreps haplotipo Secuenciación clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

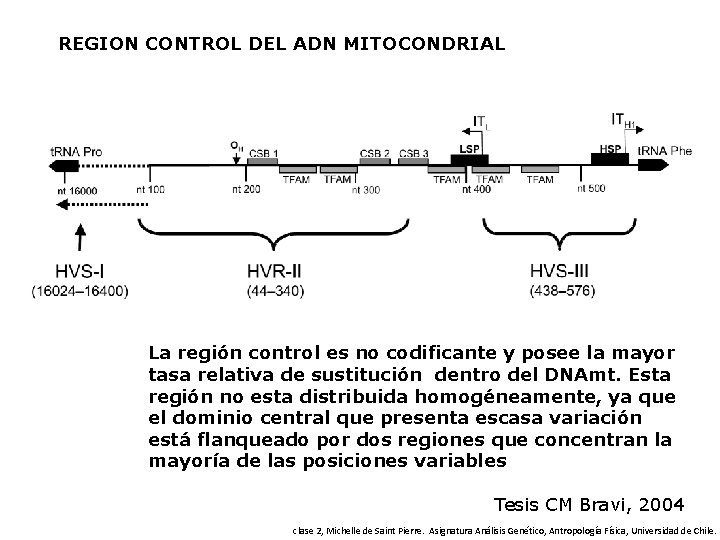
REGION CONTROL DEL ADN MITOCONDRIAL La región control es no codificante y posee la mayor tasa relativa de sustitución dentro del DNAmt. Esta región no esta distribuida homogéneamente, ya que el dominio central que presenta escasa variación está flanqueado por dos regiones que concentran la mayoría de las posiciones variables Tesis CM Bravi, 2004 clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

Tipos de mutaciones en el DNA mitocondrial ATTCGCTATCGAACGCTGCGAACGGG ATTCGTTATCGAACGCTGCGAACGGG Sustitución ATTCGCTATCGAA- - -TACGCTGCGAACGGG Deleción ATTCGCTATCGAACGCCGTACGCTGCGAACGGG Inserción clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

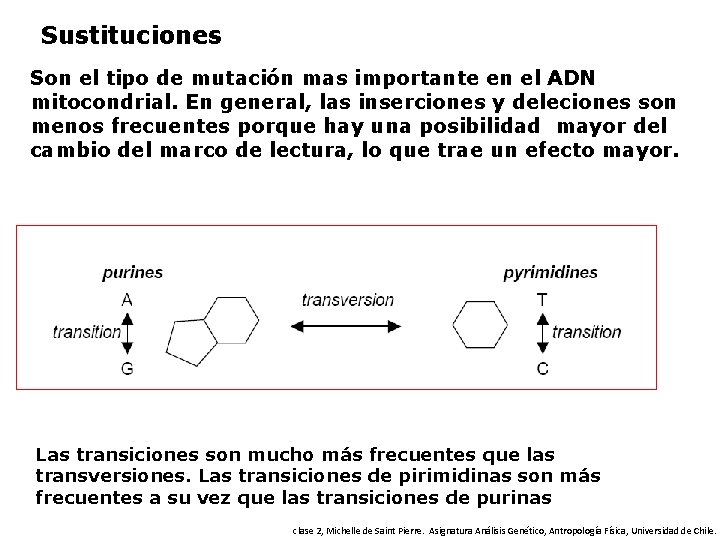
Sustituciones Son el tipo de mutación mas importante en el ADN mitocondrial. En general, las inserciones y deleciones son menos frecuentes porque hay una posibilidad mayor del cambio del marco de lectura, lo que trae un efecto mayor. Las transiciones son mucho más frecuentes que las transversiones. Las transiciones de pirimidinas son más frecuentes a su vez que las transiciones de purinas clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

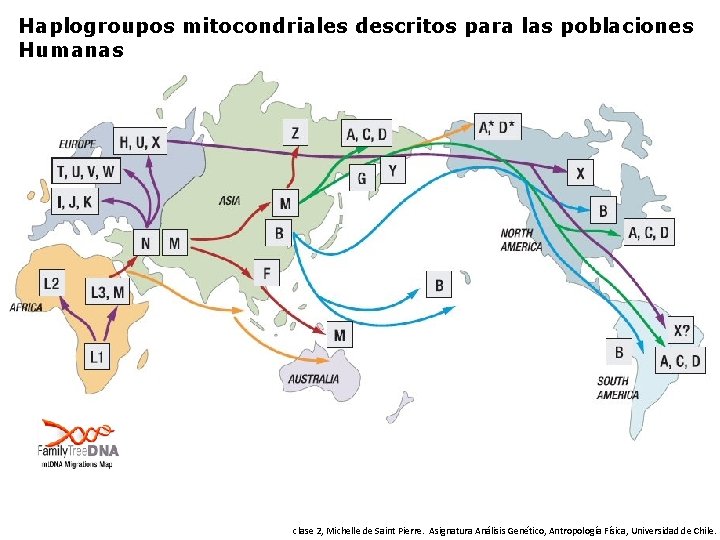
Haplogroupos mitocondriales descritos para las poblaciones Humanas clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

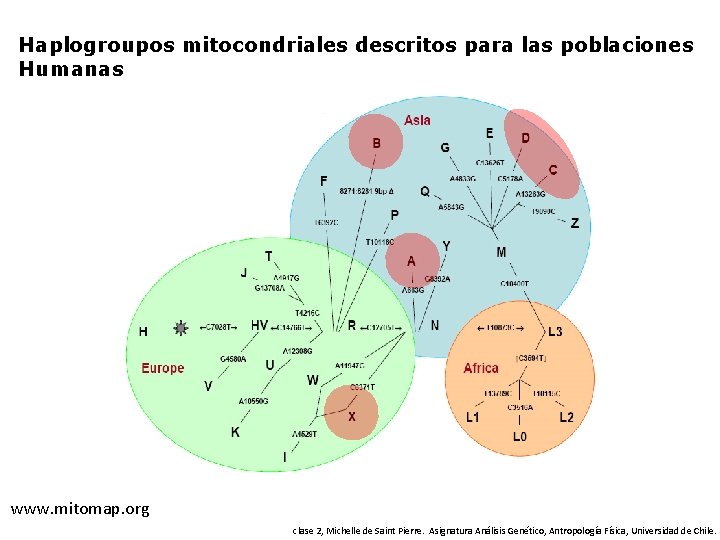
Haplogroupos mitocondriales descritos para las poblaciones Humanas www. mitomap. org clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

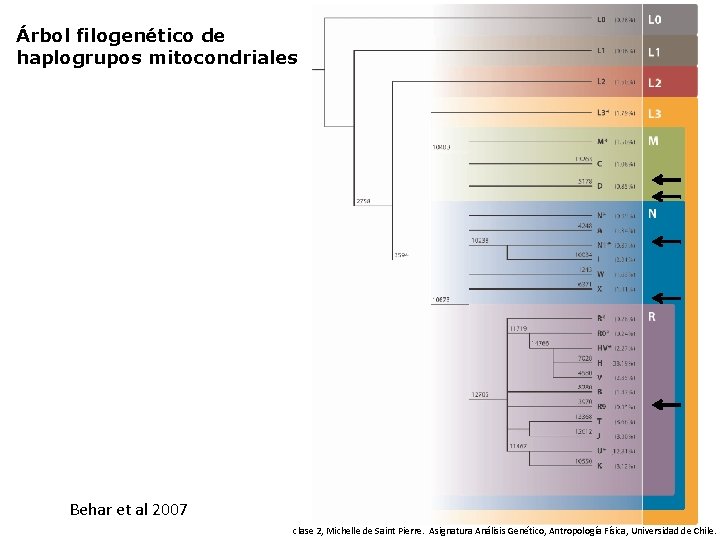
Árbol filogenético de haplogrupos mitocondriales Behar et al 2007 clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

DEFINICION HAPLOGRUPOS MITOCONDRIALES AMERINDIOS Haplogrupo Hae. III Deleción Hinc. II Alu. I 663 9 pb 13259 5176 A + − + + B − + + + C − − − + D − − + − clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

Hitos en la caracterización de haplogrupos mitocondriales Amerindios Johnson et al. (1983) analiza 30 Waraos de Venezuela 170 asiáticos, europeos y africanos, mediante RFLP Wallace et al. (1985) analiza indígenas norteamericanos, define haplogrupo C Shurr et al. (1990) confirmaron C y otros marcadores asiáticos en Pima, Maya, Ticuna, caracterización haplogrupos B y A Wallace at al. (1992) Con PCR-RFPL de alta resolución analizaron 167 nativos americanos, confirmando los marcadores descritos anteriormente y la separación de indígenas americanos en 4 grupos monofiléticos (A, B, C, D) Tesis CM Bravi, 2004 clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

Torroni et al. (1993) confirmación de resultados anteriores y secuenciación de HV 1 de 38 individuos de americanos y 11 asiáticos Horai et al. (1993) y Ginther et al. (1993) Rectificación independiente haplogrupos amerindios Baillet et al. (1994) encuentra 1 nuevo haplogrupo en navajos de Norteamérica Foster et al. (1996) denominación haplogrupo X descrito por Baillet et al, el cual solo ha sido descrito en Norteamérica Tesis CM Bravi, 2004 clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

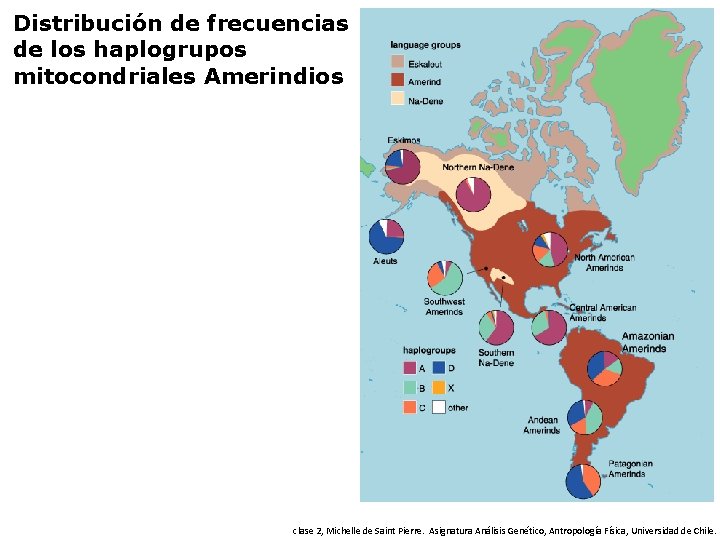
Distribución de frecuencias de los haplogrupos mitocondriales Amerindios clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

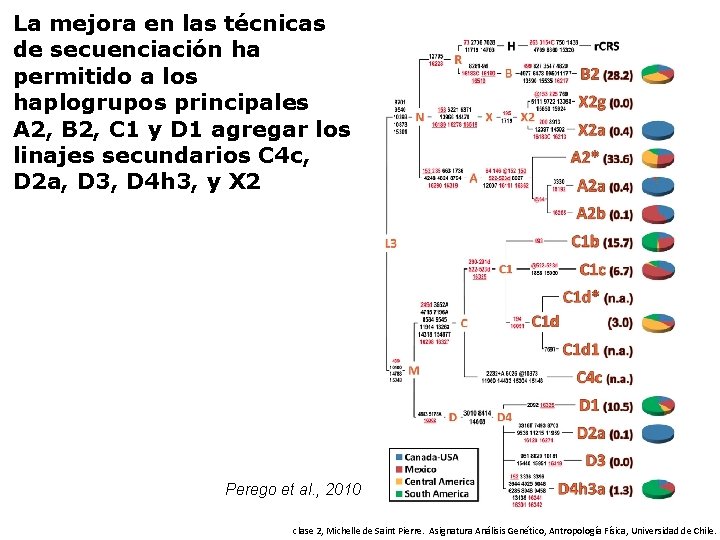
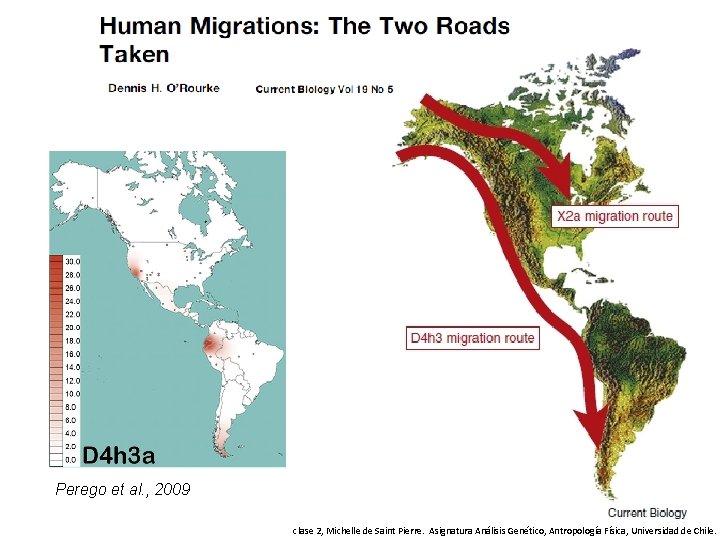
La mejora en las técnicas de secuenciación ha permitido a los haplogrupos principales A 2, B 2, C 1 y D 1 agregar los linajes secundarios C 4 c, D 2 a, D 3, D 4 h 3, y X 2 Perego et al. , 2010 clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

ARBOL BASAL QUE COMPRENDE LA RAIZ DE LOS HAPLOGRUPOS AMERICANOS Achilli et al. , 2008 clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

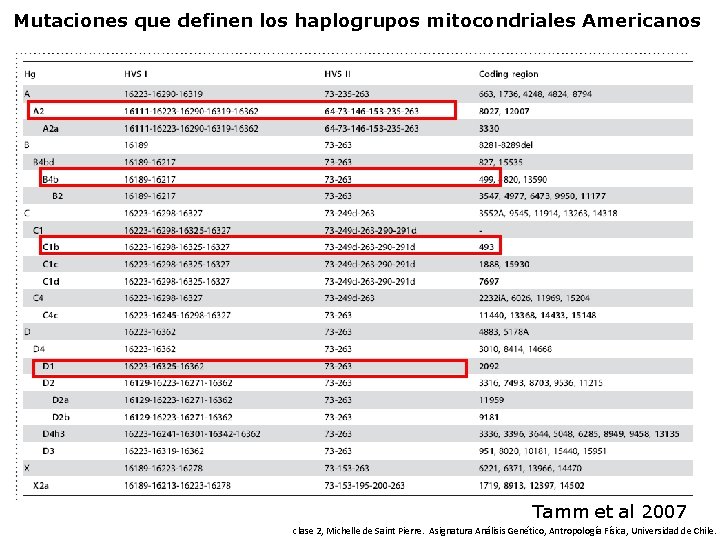
Mutaciones que definen los haplogrupos mitocondriales Americanos Tamm et al 2007 clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

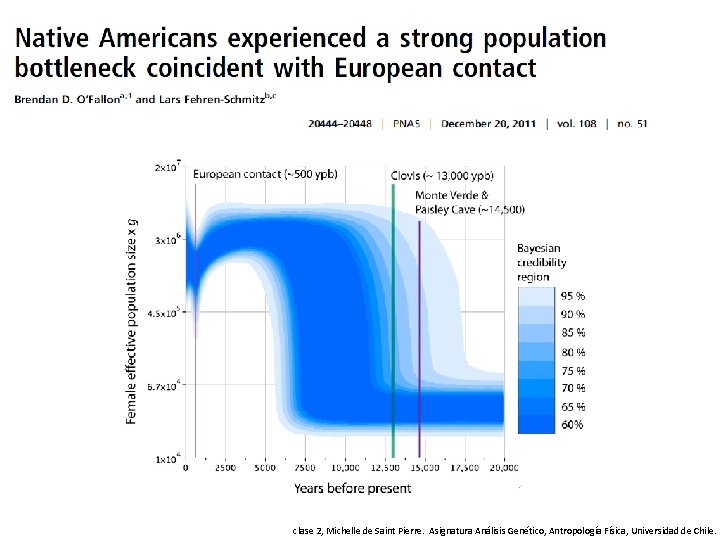
clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.

Perego et al. , 2009 clase 2, Michelle de Saint Pierre. Asignatura Análisis Genético, Antropología Física, Universidad de Chile.
 Circonscription saint pierre 1
Circonscription saint pierre 1 Eglise saint pierre aumaitre angouleme
Eglise saint pierre aumaitre angouleme Hugo grocio y bernardino de saint pierre teoria
Hugo grocio y bernardino de saint pierre teoria Trves
Trves Cloroplasto
Cloroplasto Camara externa mitocondrial
Camara externa mitocondrial Eva mitocondrial
Eva mitocondrial Adn mitocondrial
Adn mitocondrial Membrana mitocondrial
Membrana mitocondrial Mitocondrial
Mitocondrial Membrana mitocondrial
Membrana mitocondrial Lipdeos
Lipdeos St annes high school for girls
St annes high school for girls February 3 saint
February 3 saint Vous me ferez un sanctuaire
Vous me ferez un sanctuaire Universidad st john's cdmx
Universidad st john's cdmx Enzyme involved in dna replication
Enzyme involved in dna replication Replication fork
Replication fork Chapter 11 dna and genes
Chapter 11 dna and genes Bioflix activity dna replication nucleotide pairing
Bioflix activity dna replication nucleotide pairing Coding dna and non coding dna
Coding dna and non coding dna Michelle buchanan ornl
Michelle buchanan ornl Mental health policy, plans and programmes michelle funk
Mental health policy, plans and programmes michelle funk Michelle warkentin
Michelle warkentin Michelle madigan defcon
Michelle madigan defcon Orgitv
Orgitv