CORSO DI IMMUNOLOGIA per il corso di Laurea
































- Slides: 78

CORSO DI IMMUNOLOGIA per il corso di Laurea in biotecnologie a. a. 2005 -2006 II semestre DOCENTE: Dott. ssa Vladia Monsurrò Dipartimento di patologia Sezione di immunologia Universita’ degli Studi di Verona 045 8074256 vladia. monsurro@univr. it ORARIO DELLE LEZIONI: lunedì: 4: 00 -5: 30 giovedì 4: 00 -5: 30 dal 20 marzo al 25 maggio 2006 aula F Facolta’ di Scienze

DOVE TROVARE LE LEZIONI on line: http: //www. scienze. univr. it/fol/main? ent=oi&cs=6&id=20061&lang=it In “materiale didattico” Per qunato riguarda il programma e l’esame e’ come discusso in classe

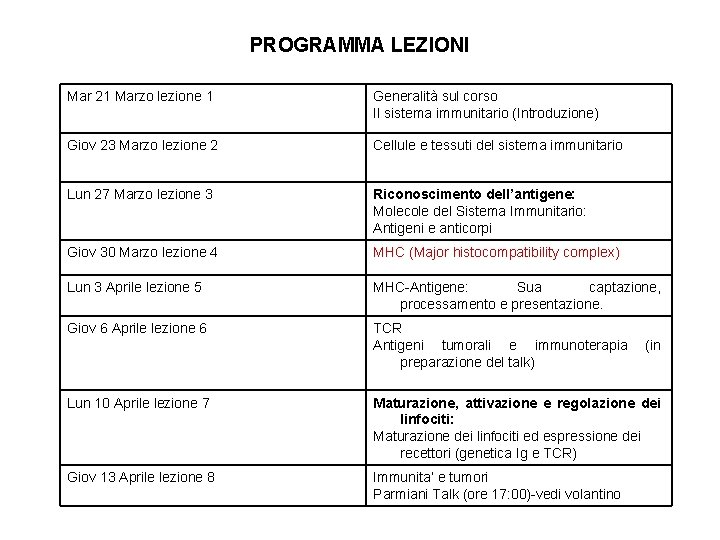
PROGRAMMA LEZIONI Mar 21 Marzo lezione 1 Generalità sul corso Il sistema immunitario (Introduzione) Giov 23 Marzo lezione 2 Cellule e tessuti del sistema immunitario Lun 27 Marzo lezione 3 Riconoscimento dell’antigene: Molecole del Sistema Immunitario: Antigeni e anticorpi Giov 30 Marzo lezione 4 MHC (Major histocompatibility complex) Lun 3 Aprile lezione 5 MHC-Antigene: Sua captazione, processamento e presentazione. Giov 6 Aprile lezione 6 TCR Antigeni tumorali e immunoterapia preparazione del talk) (in Lun 10 Aprile lezione 7 Maturazione, attivazione e regolazione dei linfociti: Maturazione dei linfociti ed espressione dei recettori (genetica Ig e TCR) Giov 13 Aprile lezione 8 Immunita’ e tumori Parmiani Talk (ore 17: 00)-vedi volantino

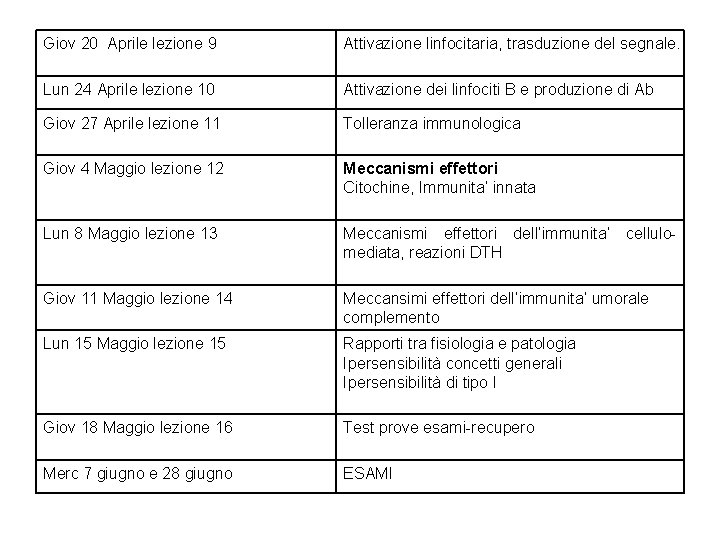
Giov 20 Aprile lezione 9 Attivazione linfocitaria, trasduzione del segnale. Lun 24 Aprile lezione 10 Attivazione dei linfociti B e produzione di Ab Giov 27 Aprile lezione 11 Tolleranza immunologica Giov 4 Maggio lezione 12 Meccanismi effettori Citochine, Immunita’ innata Lun 8 Maggio lezione 13 Meccanismi effettori dell’immunita’ cellulomediata, reazioni DTH Giov 11 Maggio lezione 14 Meccansimi effettori dell’immunita’ umorale complemento Lun 15 Maggio lezione 15 Rapporti tra fisiologia e patologia Ipersensibilità concetti generali Ipersensibilità di tipo I Giov 18 Maggio lezione 16 Test prove esami-recupero Merc 7 giugno e 28 giugno ESAMI

I RECETTORI PER GLI ANTIGENI: • IG • MHC • TCR

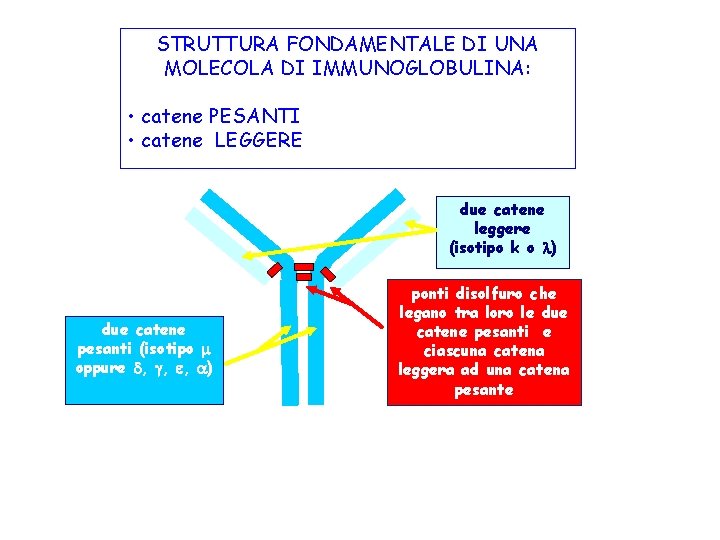
STRUTTURA FONDAMENTALE DI UNA MOLECOLA DI IMMUNOGLOBULINA: • catene PESANTI • catene LEGGERE due catene leggere (isotipo k o ) due catene pesanti (isotipo oppure , , , ) ponti disolfuro che legano tra loro le due catene pesanti e ciascuna catena leggera ad una catena pesante

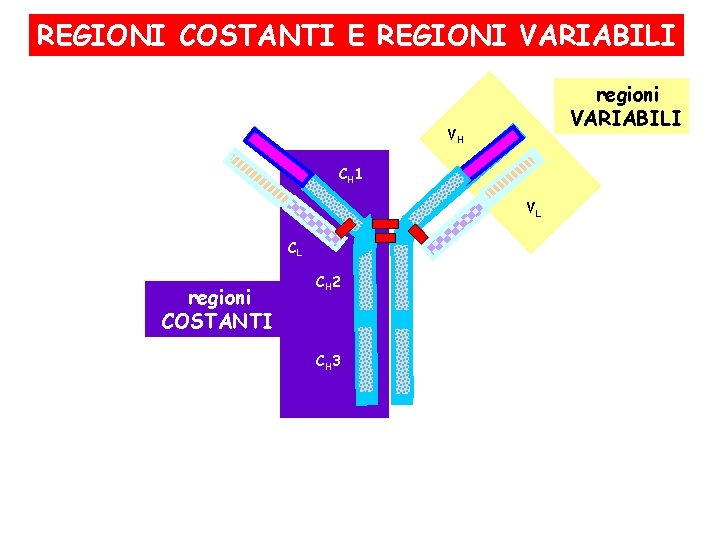
REGIONI COSTANTI E REGIONI VARIABILI regioni VARIABILI VH CH 1 VL CL regioni COSTANTI CH 2 CH 3

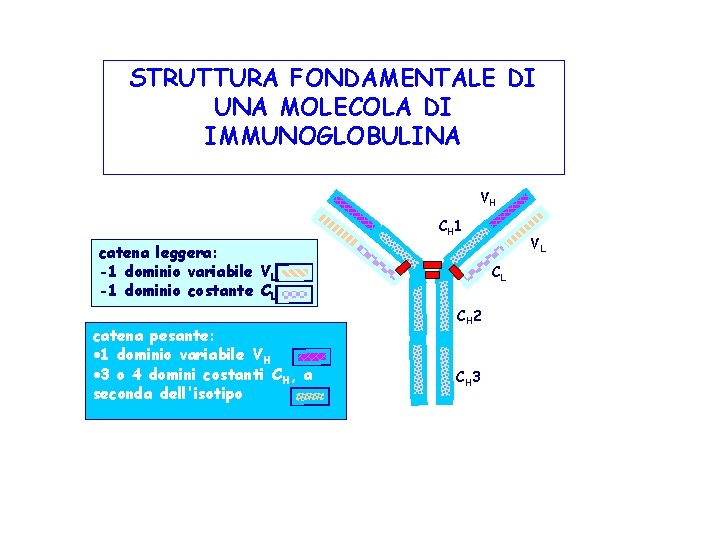
STRUTTURA FONDAMENTALE DI UNA MOLECOLA DI IMMUNOGLOBULINA VH CH 1 catena leggera: -1 dominio variabile VL -1 dominio costante CL catena pesante: · 1 dominio variabile VH · 3 o 4 domini costanti CH, a seconda dell'isotipo VL CL CH 2 CH 3

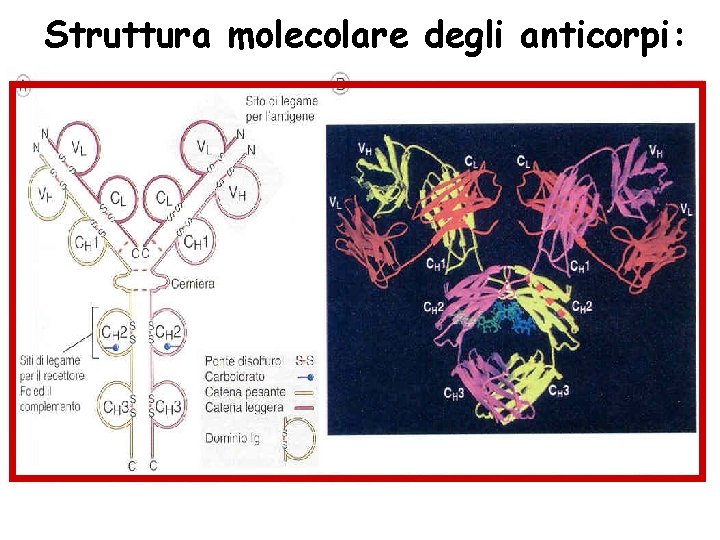
Struttura molecolare degli anticorpi:

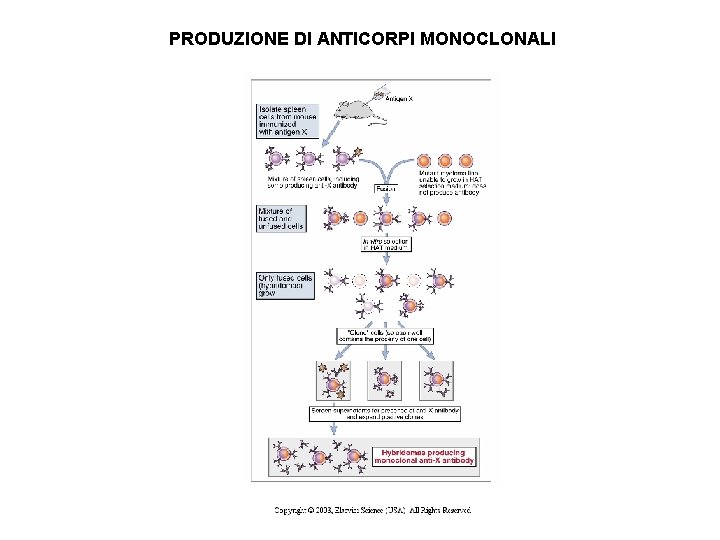
PRODUZIONE DI ANTICORPI MONOCLONALI

I RECETTORI PER GLI ANTIGENI: • IG • MHC • TCR

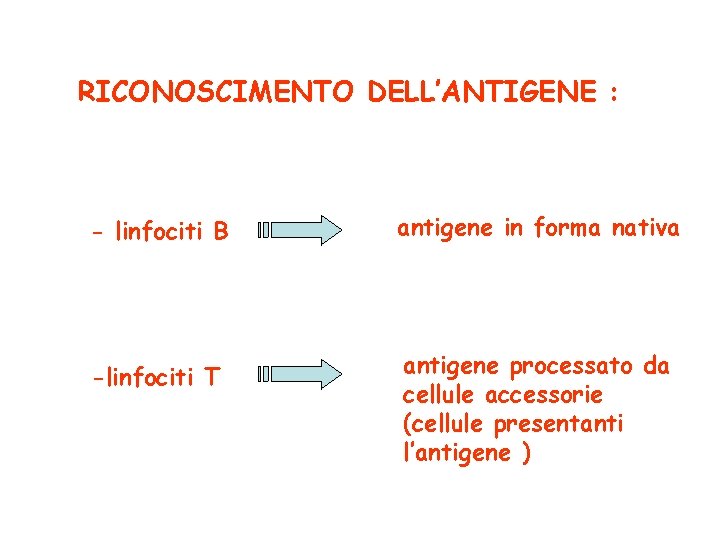
RICONOSCIMENTO DELL’ANTIGENE : - linfociti B -linfociti T antigene in forma nativa antigene processato da cellule accessorie (cellule presentanti l’antigene )

• I linfociti T contro microbi intracellulari e l’attivazione di altre cellule (macrofagi e linfociti B)

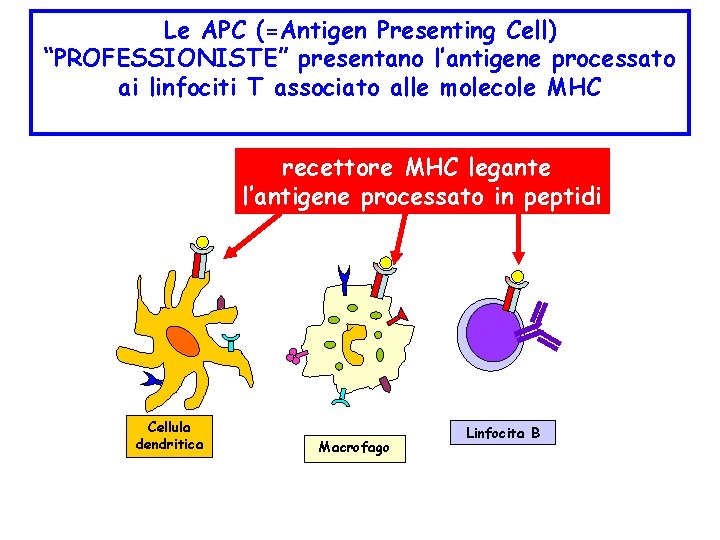
Le APC (=Antigen Presenting Cell) “PROFESSIONISTE” presentano l’antigene processato ai linfociti T associato alle molecole MHC recettore MHC legante l’antigene processato in peptidi Cellula dendritica Macrofago Linfocita B

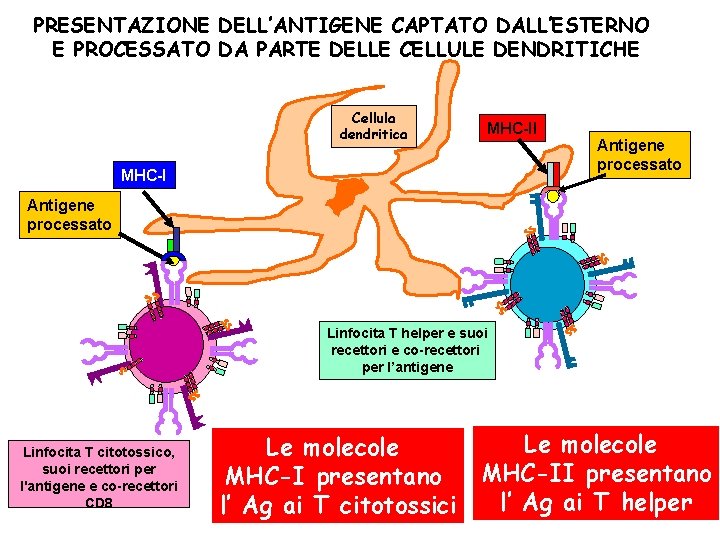
PRESENTAZIONE DELL’ANTIGENE CAPTATO DALL’ESTERNO E PROCESSATO DA PARTE DELLE CELLULE DENDRITICHE Cellula dendritica MHC-II MHC-I Antigene processato Linfocita T helper e suoi recettori e co-recettori per l’antigene Linfocita T citotossico, suoi recettori per l'antigene e co-recettori CD 8 Le molecole MHC-I presentano l’ Ag ai T citotossici Le molecole MHC-II presentano l’ Ag ai T helper

MOLECOLE MHC struttura e genetica

Cosa significa MHC? • MHC (complesso maggiore di istocompatibilità) è il locus dove mappano gli HLA • È chiamato così perché storicamente e’ stato associato ai rigetti di trapianto (altamente polimorfico e immunogeno).

• Un allele è una variante di un gene polimorfo • Un individuo è: Omozigote se ha 2 alleli uguali Eterozigote se ha 2 alleli differenti

• Topi singenici hanno tutti i geni identici • Due ceppi allogenici sono due gruppi di topi singenici differenti fra loro

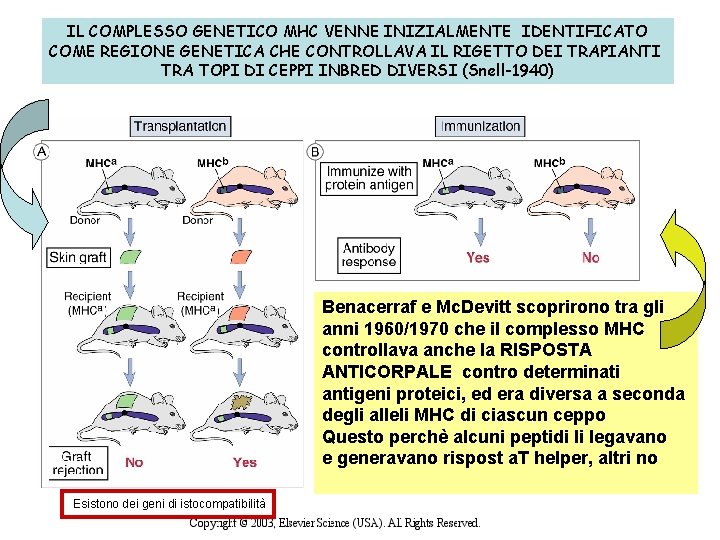
IL COMPLESSO GENETICO MHC VENNE INIZIALMENTE IDENTIFICATO COME REGIONE GENETICA CHE CONTROLLAVA IL RIGETTO DEI TRAPIANTI TRA TOPI DI CEPPI INBRED DIVERSI (Snell-1940) Benacerraf e Mc. Devitt scoprirono tra gli anni 1960/1970 che il complesso MHC controllava anche la RISPOSTA ANTICORPALE contro determinati antigeni proteici, ed era diversa a seconda degli alleli MHC di ciascun ceppo Questo perchè alcuni peptidi li legavano e generavano rispost a. T helper, altri no Esistono dei geni di istocompatibilità

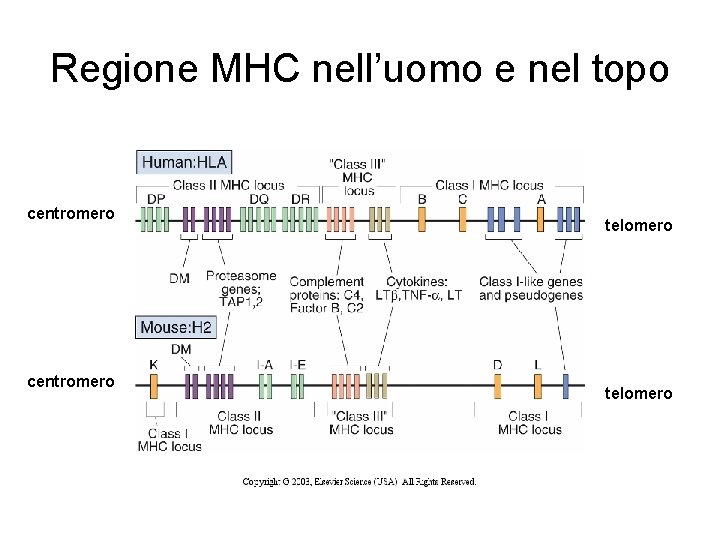
Regione MHC nell’uomo e nel topo centromero telomero

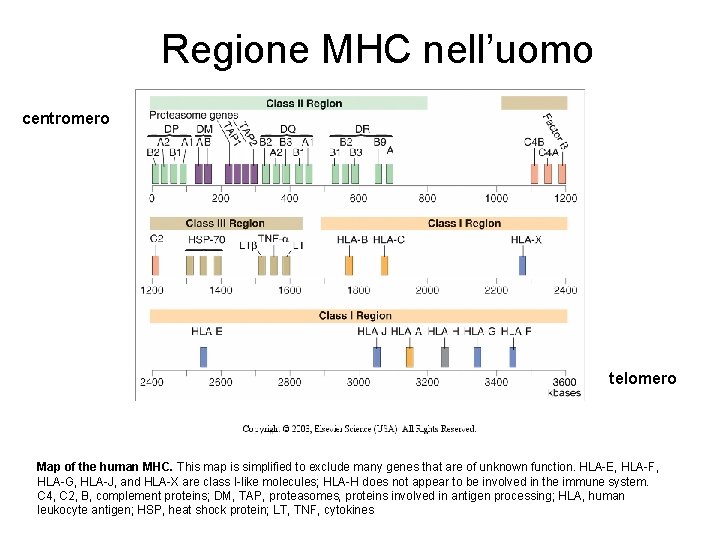
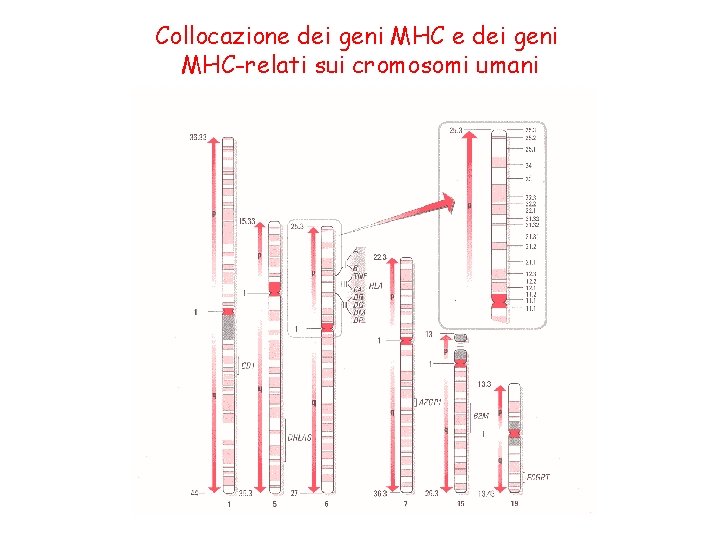
Regione MHC nell’uomo centromero telomero Map of the human MHC. This map is simplified to exclude many genes that are of unknown function. HLA-E, HLA-F, HLA-G, HLA-J, and HLA-X are class I-like molecules; HLA-H does not appear to be involved in the immune system. C 4, C 2, B, complement proteins; DM, TAP, proteasomes, proteins involved in antigen processing; HLA, human leukocyte antigen; HSP, heat shock protein; LT, TNF, cytokines

Collocazione dei geni MHC-relati sui cromosomi umani

• Gli MHC nell’uomo sono chiamati HLA (antigeni leucocitari umani) perche’ identificati per vie sierologiche mediante Ab che riconoscevano i leucociti durante le trasfusioni

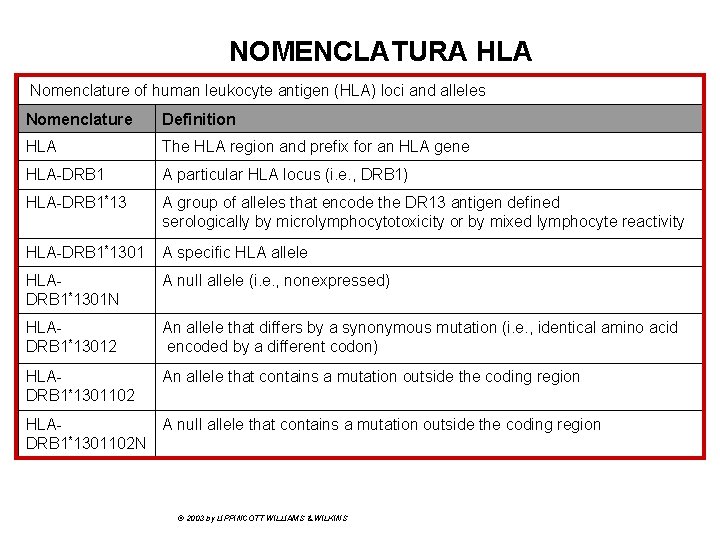
NOMENCLATURA HLA Nomenclature of human leukocyte antigen (HLA) loci and alleles Nomenclature Definition HLA The HLA region and prefix for an HLA gene HLA-DRB 1 A particular HLA locus (i. e. , DRB 1) HLA-DRB 1*13 A group of alleles that encode the DR 13 antigen defined serologically by microlymphocytotoxicity or by mixed lymphocyte reactivity HLA-DRB 1*1301 A specific HLA allele HLADRB 1*1301 N A null allele (i. e. , nonexpressed) HLADRB 1*13012 An allele that differs by a synonymous mutation (i. e. , identical amino acid encoded by a different codon) HLADRB 1*1301102 An allele that contains a mutation outside the coding region HLAA null allele that contains a mutation outside the coding region DRB 1*1301102 N © 2003 by LIPPINCOTT WILLIAMS & WILKINS

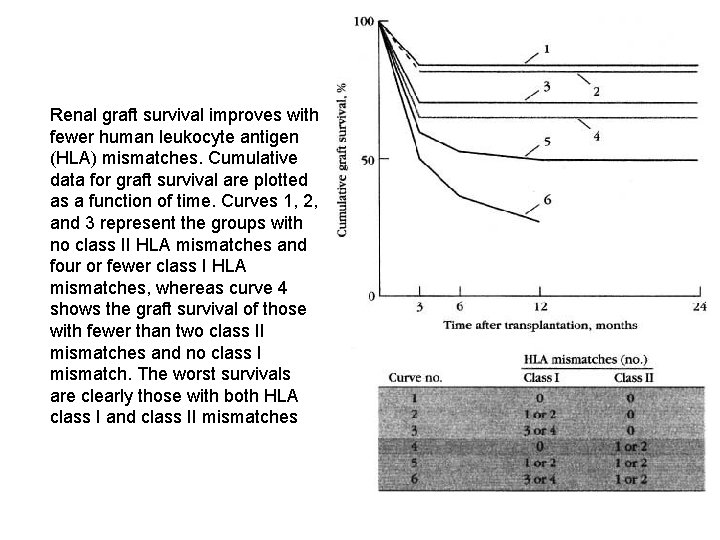
Renal graft survival improves with fewer human leukocyte antigen (HLA) mismatches. Cumulative data for graft survival are plotted as a function of time. Curves 1, 2, and 3 represent the groups with no class II HLA mismatches and four or fewer class I HLA mismatches, whereas curve 4 shows the graft survival of those with fewer than two class II mismatches and no class I mismatch. The worst survivals are clearly those with both HLA class I and class II mismatches

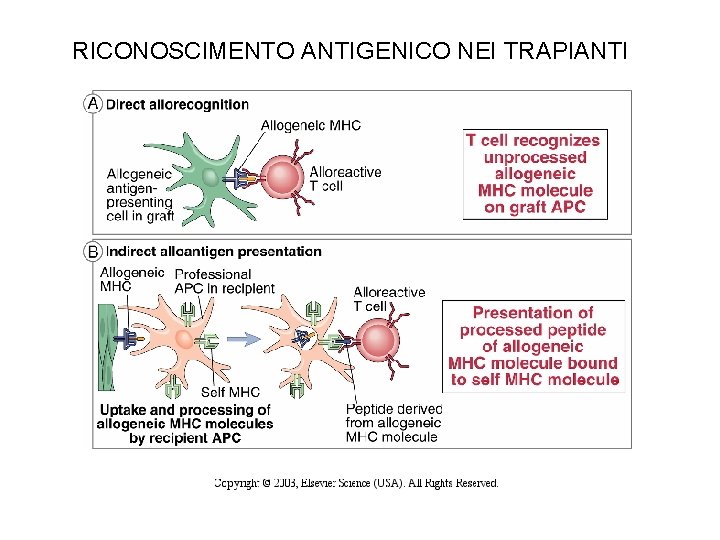
RICONOSCIMENTO ANTIGENICO NEI TRAPIANTI


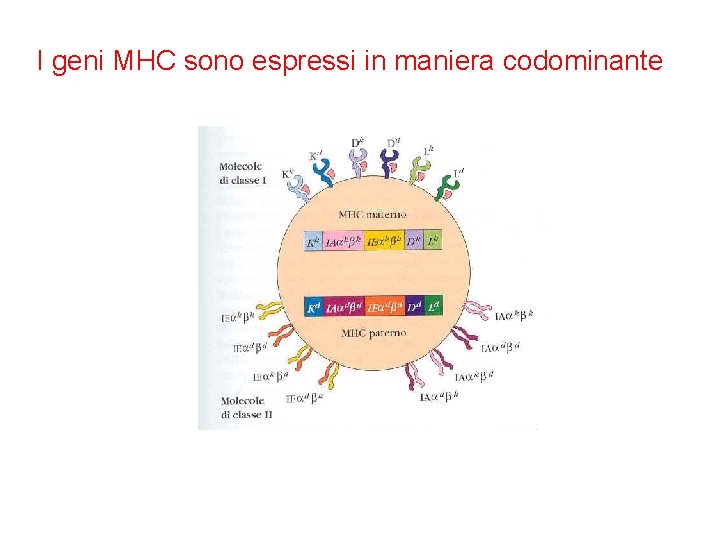
• Gli alleli dell’ MHC sono altamente polimorfici • Codominanti • Cl I e Cl II sono distinte ma omologhe

POLIMORFISMO DELLE MOLECOLE MHC • Un allele ad un locus genetico che abbia una frequenza 0. 01 è denominato “rara variante allelica”. • Un allele con una frequenza 0. 01 (ma 1) è definito come “polimorfico”. • Gli alleli ai loci MHC sono polimorfici poichè presentano sempre frequenze elevate nella popolazione. • Il polimorfismo è concentrato negli esoni che codificano per i domini che legano il peptide e il TCR.

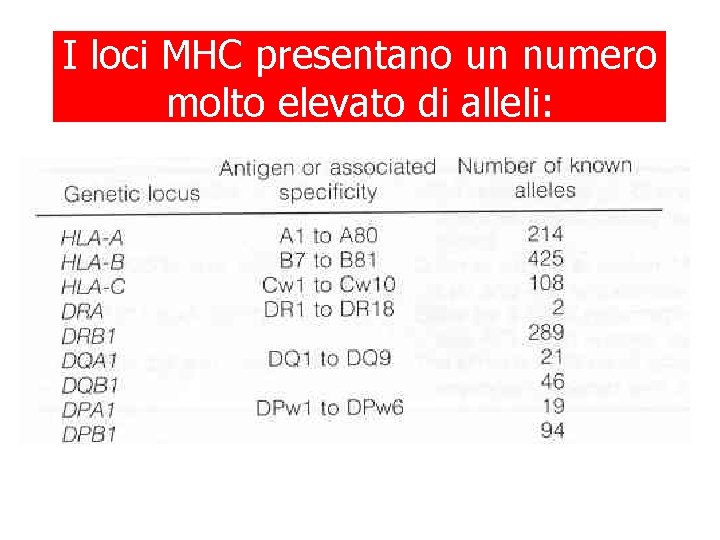
I NUMERO DI ALLELI HLA AD loci MHCALCUNI presentano un numero LOCI molto elevato di alleli:

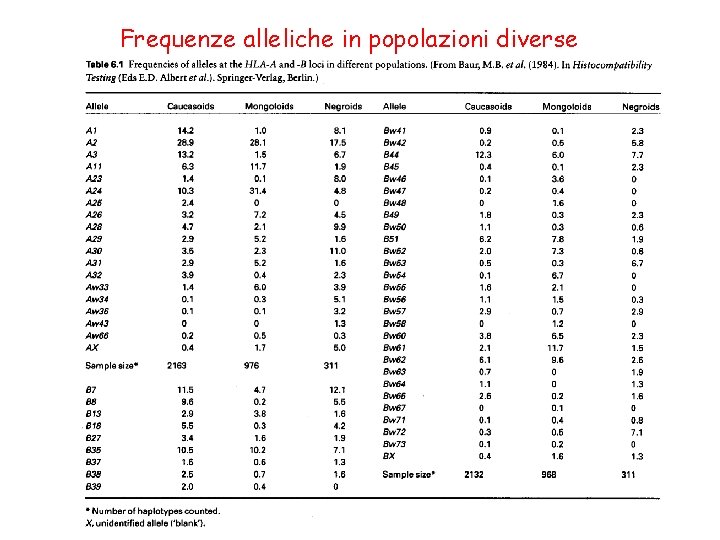
Frequenze alleliche in popolazioni diverse

I geni MHC sono espressi in maniera codominante

• Struttura dell’MHC

• Similitudini Cl I e Cl II 1. Hanno una tasca di legame per il peptide, due domini Ig, dominio transmembrana e dominio citoplasmatico 2. Il polimorfismo e’ nella tasca 3. Le zone non polimorfe legano i recettori CD 4 o CD 8

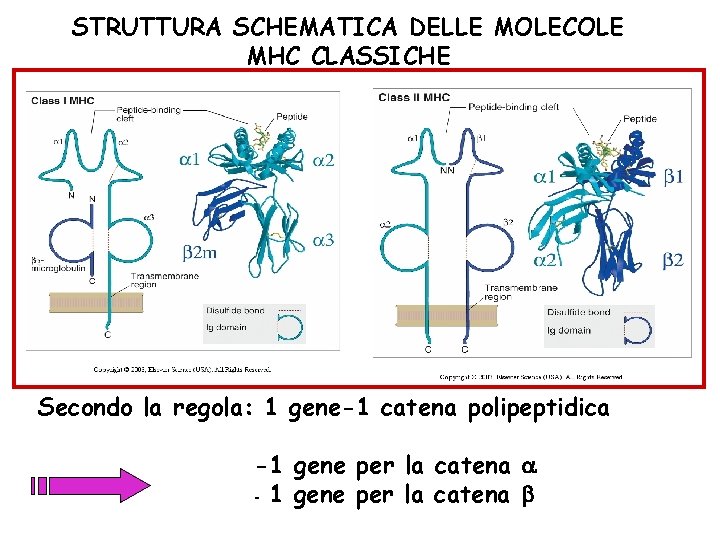
STRUTTURA SCHEMATICA DELLE MOLECOLE MHC CLASSICHE Secondo la regola: 1 gene-1 catena polipeptidica -1 gene per la catena - 1 gene per la catena

• Similitudini Cl. I e Cl II 1. Hanno una tasca di legame pe ril peptide, due domini Ig, dominio transmembrana e dominio citoplasmatico 2. Il polimorfismo e’ nella tasca 3. Le zone non polimorfe legano i recettori CD 4 o CD 8

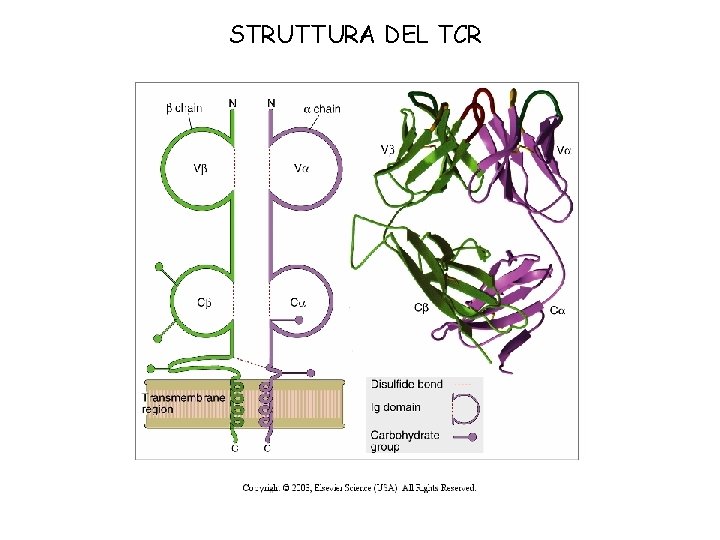
STRUTTURA DEL TCR

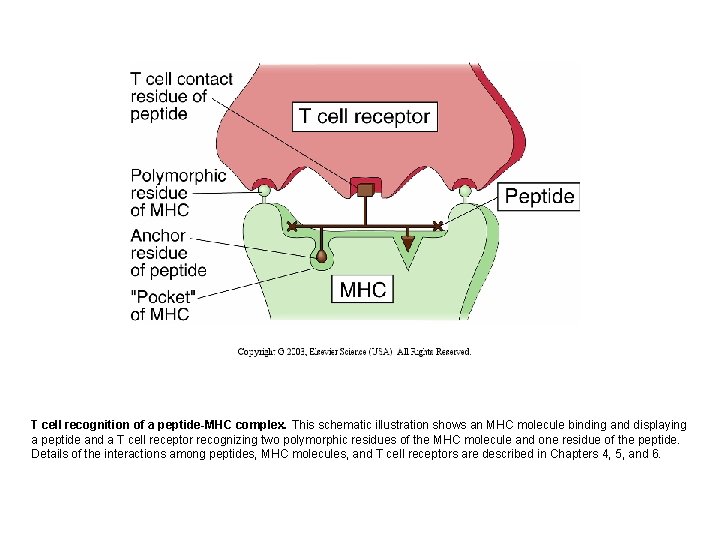
T cell recognition of a peptide-MHC complex. This schematic illustration shows an MHC molecule binding and displaying a peptide and a T cell receptor recognizing two polymorphic residues of the MHC molecule and one residue of the peptide. Details of the interactions among peptides, MHC molecules, and T cell receptors are described in Chapters 4, 5, and 6.

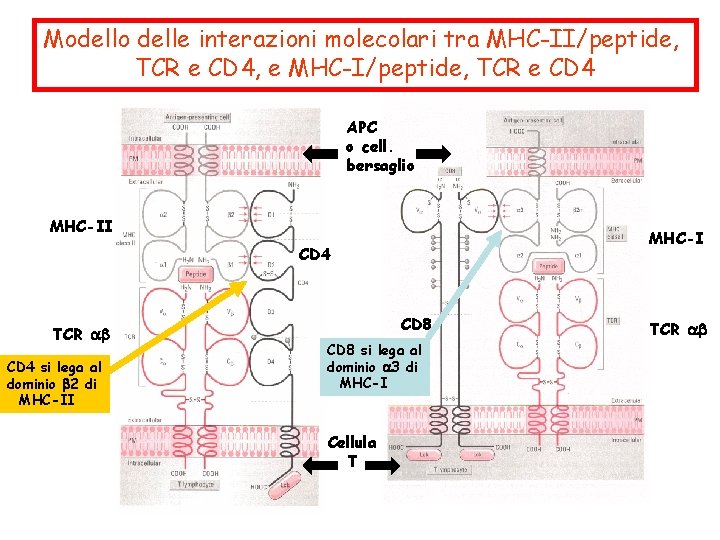
Modello delle interazioni molecolari tra MHC-II/peptide, TCR e CD 4, e MHC-I/peptide, TCR e CD 4 APC o cell. bersaglio MHC-II MHC-I CD 4 TCR CD 4 si lega al dominio 2 di MHC-II CD 8 si lega al dominio 3 di MHC-I Cellula T TCR

• MHC Cl I

2 tipi di Molecole MHC-I classiche, MHC-Ia: non classiche, MHC-Ib: • • HLA-A, -B, -C (uomo) H-2 K, -D, -L (topo) sono polimorfiche e legano peptidi antigenici che presentano ai TCR associati a CD 8 Essendo codominanti ogni cellula ne esprime 6 diverse CD 1 H 2 -M 3 MICA n. Fc. R Rae 1 Presentano polimorfismo molto limitato e spesso il solco di legame con il peptide è molto ridotto

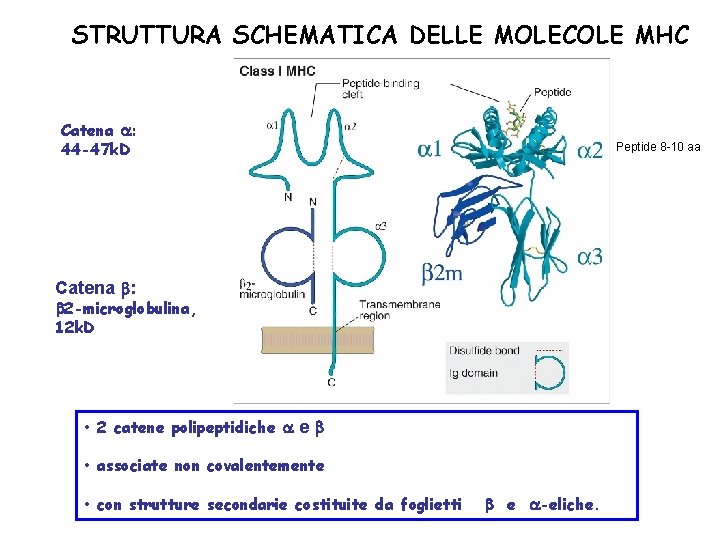
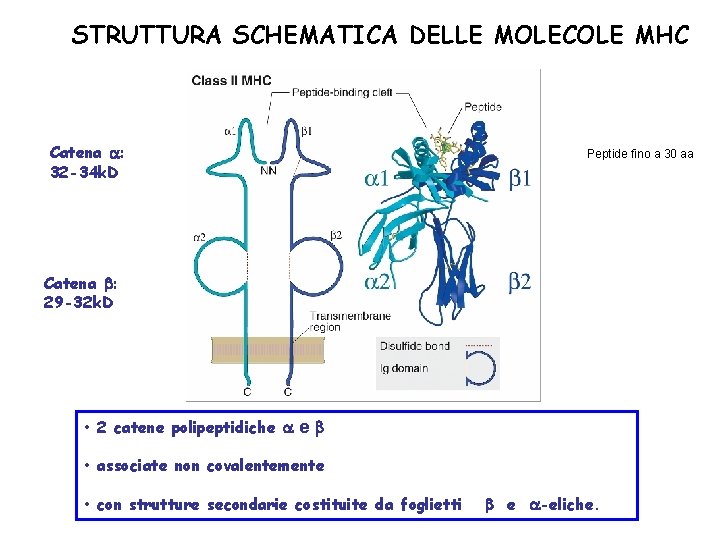
STRUTTURA SCHEMATICA DELLE MOLECOLE MHC Catena : 44 -47 k. D Peptide 8 -10 aa Catena : 2 -microglobulina, 12 k. D • 2 catene polipeptidiche e • associate non covalentemente • con strutture secondarie costituite da foglietti e -eliche.

• MHC Cl II

STRUTTURA SCHEMATICA DELLE MOLECOLE MHC Catena : 32 -34 k. D Peptide fino a 30 aa Catena : 29 -32 k. D • 2 catene polipeptidiche e • associate non covalentemente • con strutture secondarie costituite da foglietti e -eliche.

Circa 20 possibili combinazioni perche’ DQ puo’ legarsi anche a DP e DR, sia materno che paterno

Le molecole MHC-I ed MHC-II completamente assemblate sono eterotrimeri, composti da: 1. una catena ; 2. una catena ; 3. il peptide antigenico nella tasca. 4. L’espressione stabile sulla membrana cellulare 5. richiede la presenza di tutti e tre i componenti.


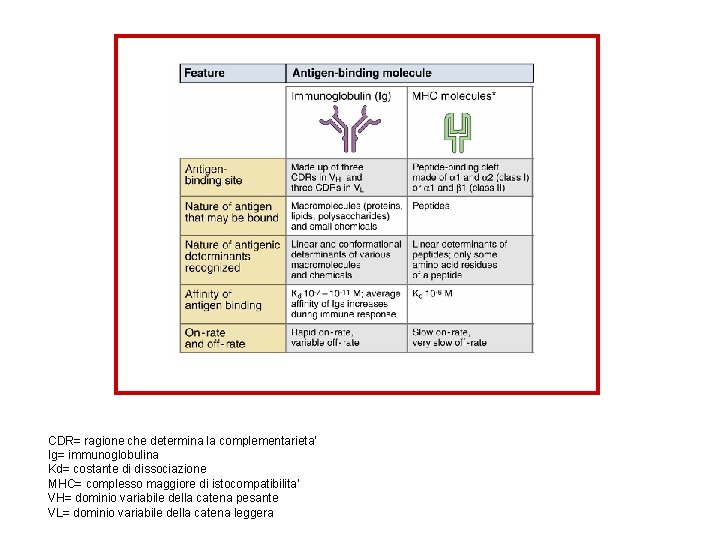
CDR= ragione che determina la complementarieta’ Ig= immunoglobulina Kd= costante di dissociazione MHC= complesso maggiore di istocompatibilita’ VH= dominio variabile della catena pesante VL= dominio variabile della catena leggera

Legame MHC-antigene

• Similitudini Cl. I e Cl II 1. Hanno una tasca di legame pe ril peptide, due domini Ig, dominio transmembrana e dominio citoplasmatico 2. Il polimorfismo e’ nella tasca 3. Le zone non polimorfe legano i recettori CD 4 o CD 8

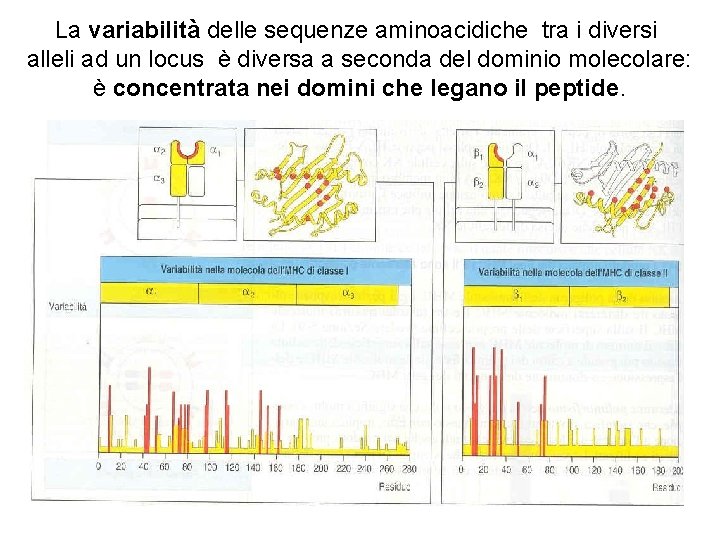
La variabilità delle sequenze aminoacidiche tra i diversi alleli ad un locus è diversa a seconda del dominio molecolare: è concentrata nei domini che legano il peptide.

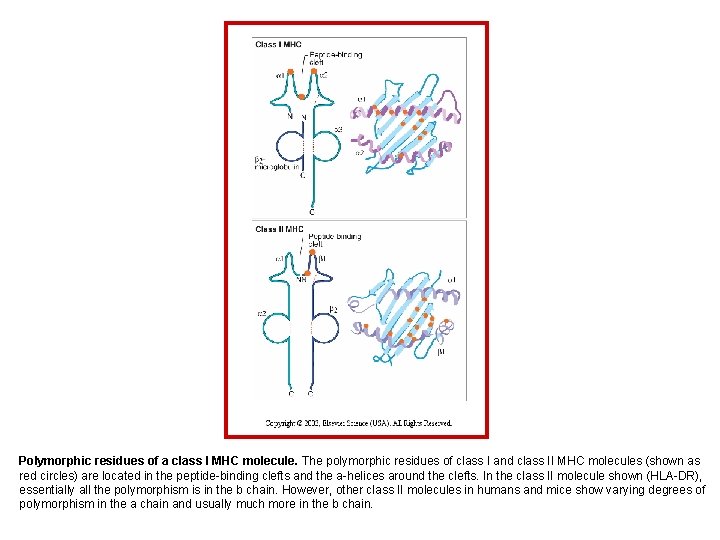
Polymorphic residues of a class I MHC molecule. The polymorphic residues of class I and class II MHC molecules (shown as red circles) are located in the peptide-binding clefts and the a-helices around the clefts. In the class II molecule shown (HLA-DR), essentially all the polymorphism is in the b chain. However, other class II molecules in humans and mice show varying degrees of polymorphism in the a chain and usually much more in the b chain.

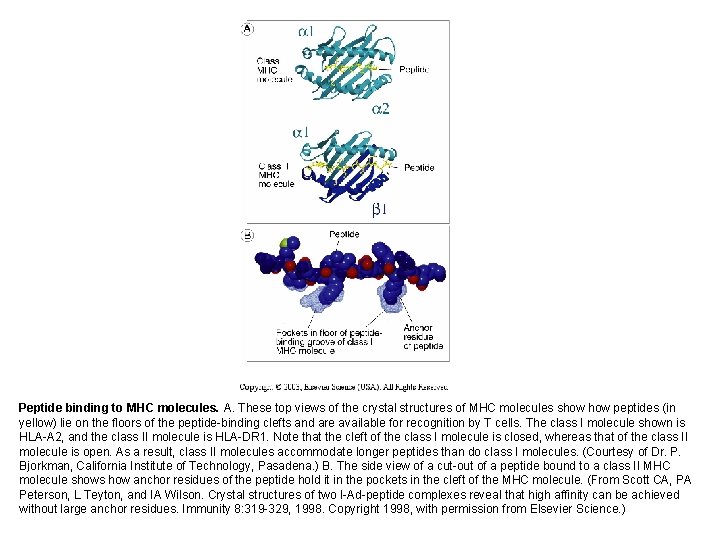
Peptide binding to MHC molecules. A. These top views of the crystal structures of MHC molecules show peptides (in yellow) lie on the floors of the peptide-binding clefts and are available for recognition by T cells. The class I molecule shown is HLA-A 2, and the class II molecule is HLA-DR 1. Note that the cleft of the class I molecule is closed, whereas that of the class II molecule is open. As a result, class II molecules accommodate longer peptides than do class I molecules. (Courtesy of Dr. P. Bjorkman, California Institute of Technology, Pasadena. ) B. The side view of a cut-out of a peptide bound to a class II MHC molecule shows how anchor residues of the peptide hold it in the pockets in the cleft of the MHC molecule. (From Scott CA, PA Peterson, L Teyton, and IA Wilson. Crystal structures of two I-Ad-peptide complexes reveal that high affinity can be achieved without large anchor residues. Immunity 8: 319 -329, 1998. Copyright 1998, with permission from Elsevier Science. )

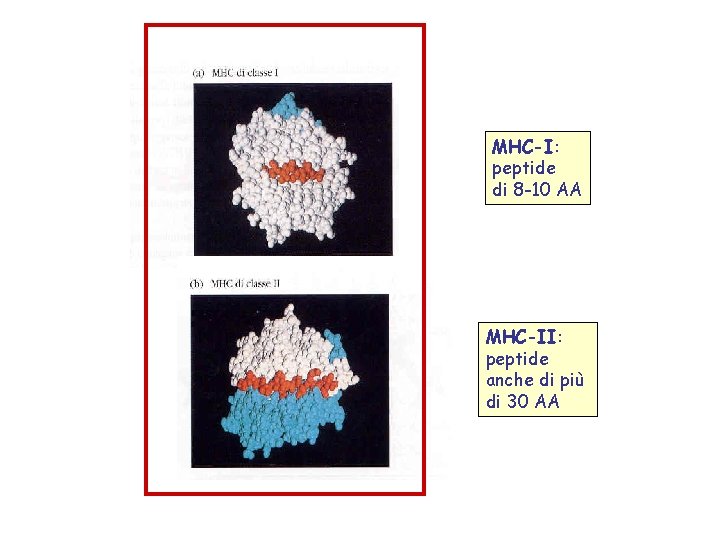
MHC-I: peptide di 8 -10 AA MHC-II: peptide anche di più di 30 AA

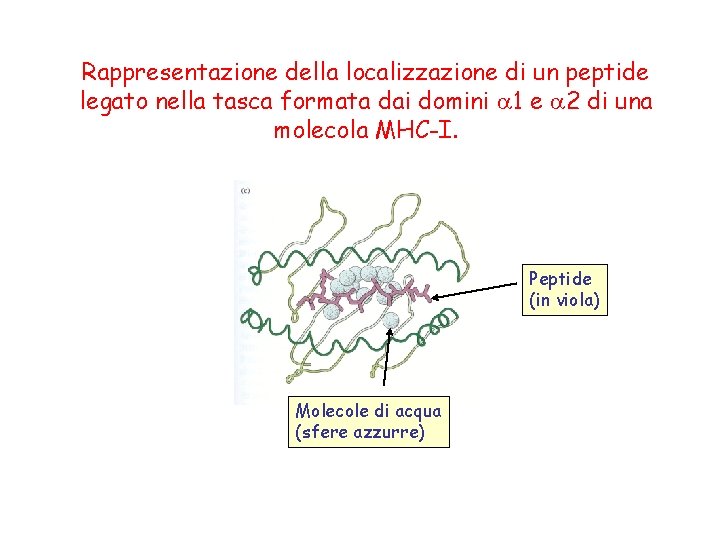
Rappresentazione della localizzazione di un peptide legato nella tasca formata dai domini 1 e 2 di una molecola MHC-I. Peptide (in viola) Molecole di acqua (sfere azzurre)

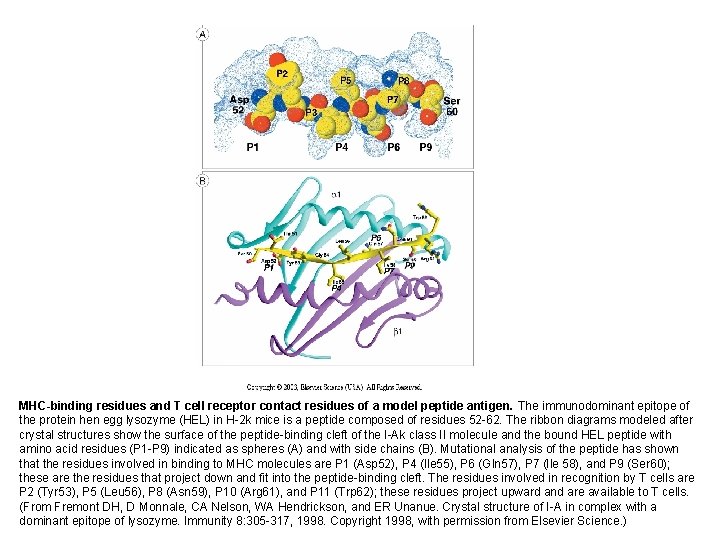
MHC-binding residues and T cell receptor contact residues of a model peptide antigen. The immunodominant epitope of the protein hen egg lysozyme (HEL) in H-2 k mice is a peptide composed of residues 52 -62. The ribbon diagrams modeled after crystal structures show the surface of the peptide-binding cleft of the I-Ak class II molecule and the bound HEL peptide with amino acid residues (P 1 -P 9) indicated as spheres (A) and with side chains (B). Mutational analysis of the peptide has shown that the residues involved in binding to MHC molecules are P 1 (Asp 52), P 4 (Ile 55), P 6 (Gln 57), P 7 (Ile 58), and P 9 (Ser 60); these are the residues that project down and fit into the peptide-binding cleft. The residues involved in recognition by T cells are P 2 (Tyr 53), P 5 (Leu 56), P 8 (Asn 59), P 10 (Arg 61), and P 11 (Trp 62); these residues project upward and are available to T cells. (From Fremont DH, D Monnale, CA Nelson, WA Hendrickson, and ER Unanue. Crystal structure of I-A in complex with a dominant epitope of lysozyme. Immunity 8: 305 -317, 1998. Copyright 1998, with permission from Elsevier Science. )

Caratteristiche delle interazioni peptide-MHC: - le molecole MHC hanno una certa promiscuità nel legame col peptide; -ogni molecola MHC-I o -II possiede una sola tasca di legame con il peptide, che può ospitare, in momenti diversi, molti peptidi differenti, ma con alcune caratteristiche strutturali in comune ; - la fine specificità del riconoscimento antigenico è dovuta prevalentemente alla selettività del TCR; - il legame dei peptidi alle molecole MHC è un’interazione non covalente tra residui AA del peptide e residui AA della tasca della molecola MHC, saturabile a bassa affinità, con bassi tassi di associazione e di dissociazione; - le molecole MHC di un individuo non distinguono tra peptidi estranei (es. da Ag microbici) e peptidi self (= da Ag derivati da proteine proprie).

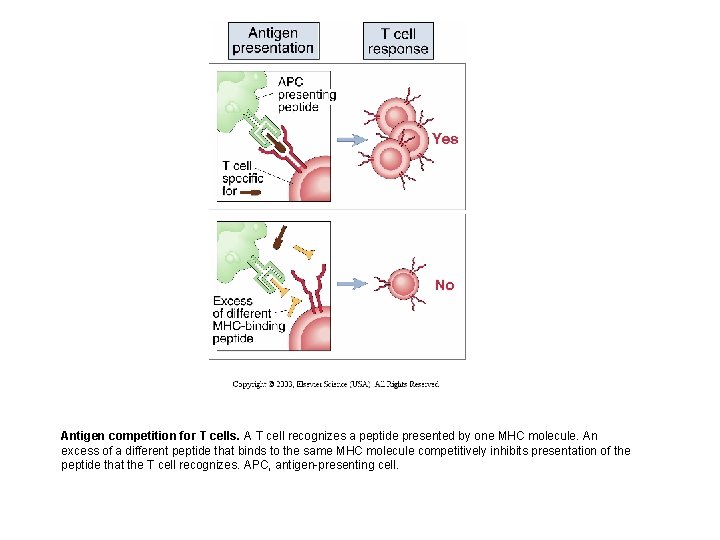
Antigen competition for T cells. A T cell recognizes a peptide presented by one MHC molecule. An excess of a different peptide that binds to the same MHC molecule competitively inhibits presentation of the peptide that the T cell recognizes. APC, antigen-presenting cell.


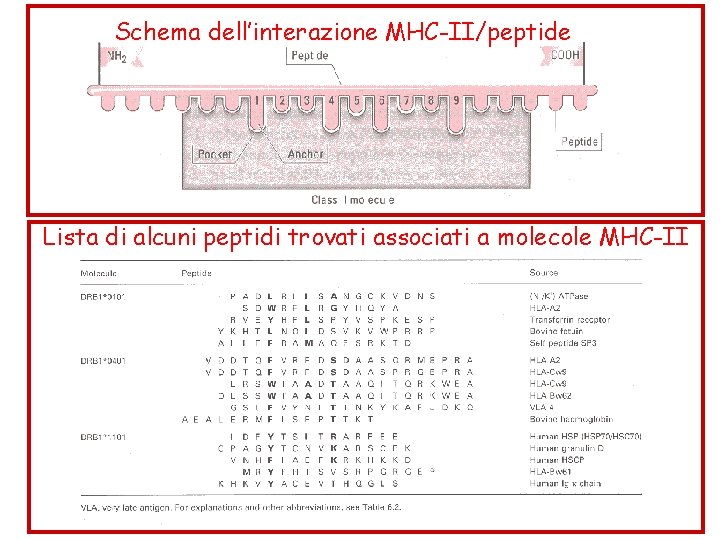
Lista di alcuni peptidi trovati associati a molecole MHC-I

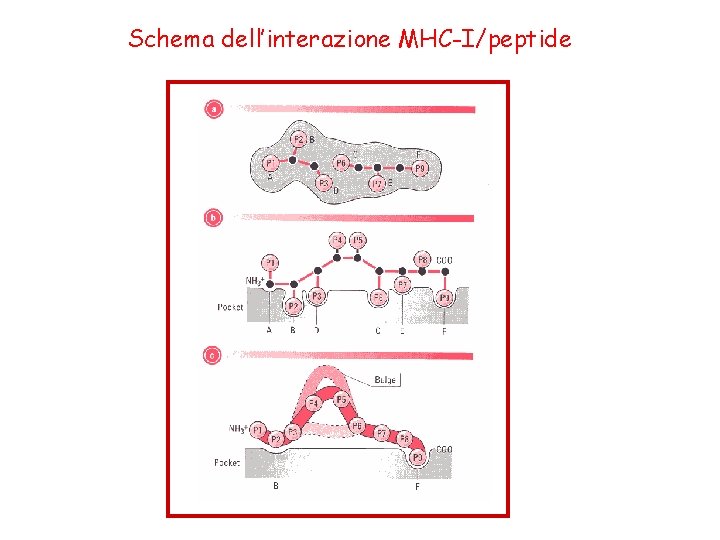
Schema dell’interazione MHC-I/peptide

Schema dell’interazione MHC-II/peptide Lista di alcuni peptidi trovati associati a molecole MHC-II

Scoperta di epitopi: i. TOPIA (filmino)

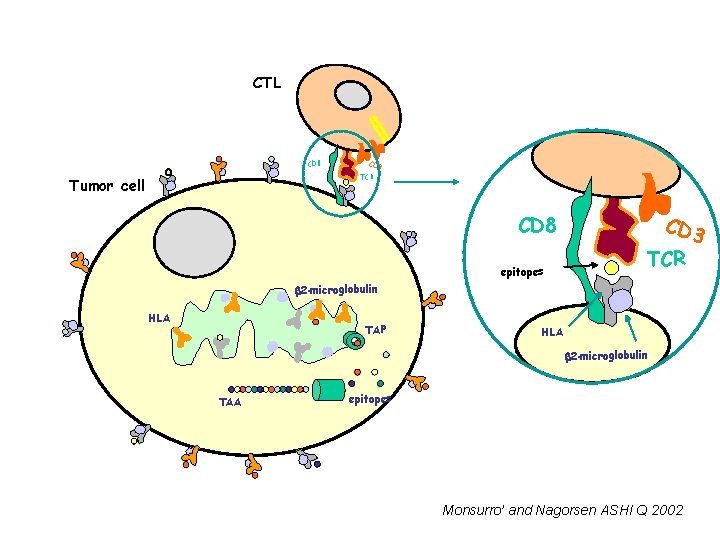
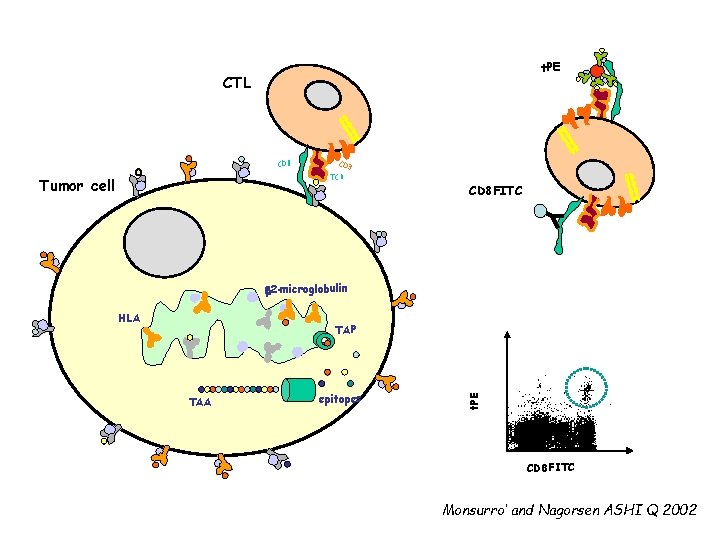
CTL CD 8 CD 3 TCR Tumor cell CD 3 CD 8 epitopes TCR 2 -microglobulin HLA TAP HLA 2 -microglobulin TAA epitopes Monsurro’ and Nagorsen ASHI Q 2002

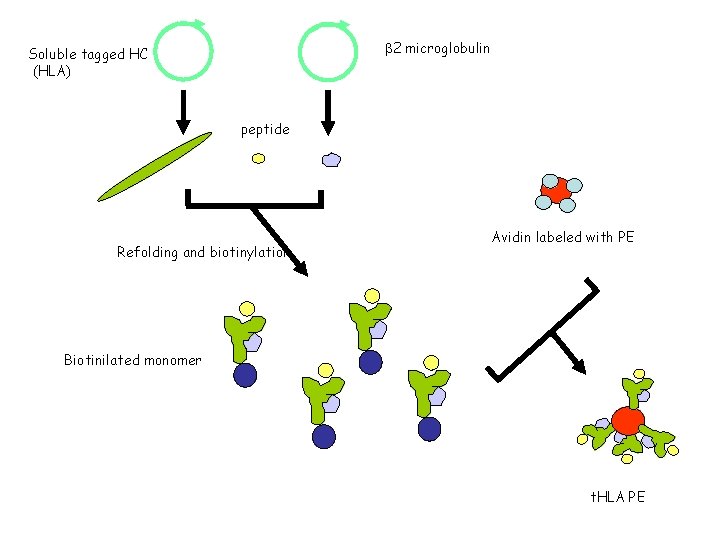
2 microglobulin Soluble tagged HC (HLA) peptide Refolding and biotinylation Avidin labeled with PE Biotinilated monomer t. HLA PE

t. PE CTL CD 8 CD 3 TCR Tumor cell CD 8 FITC 2 -microglobulin HLA TAA epitopes t. PE TAP CD 8 FITC Monsurro’ and Nagorsen ASHI Q 2002

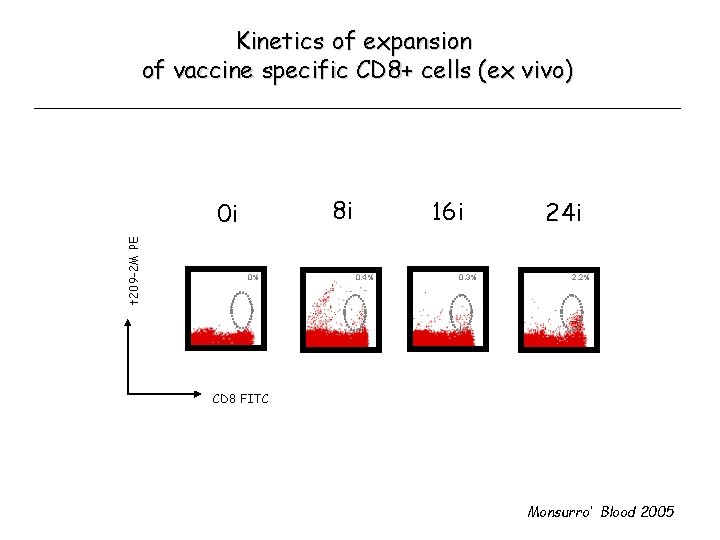
Kinetics of expansion of vaccine specific CD 8+ cells (ex vivo) 8 i t 209 -2 M PE 0 i 0% 16 i 0. 4% 0. 3% 24 i 2. 2% CD 8 FITC Monsurro’ Blood 2005

Utilizzo biotecnologico dell’MHC Tetrameri (filmino)

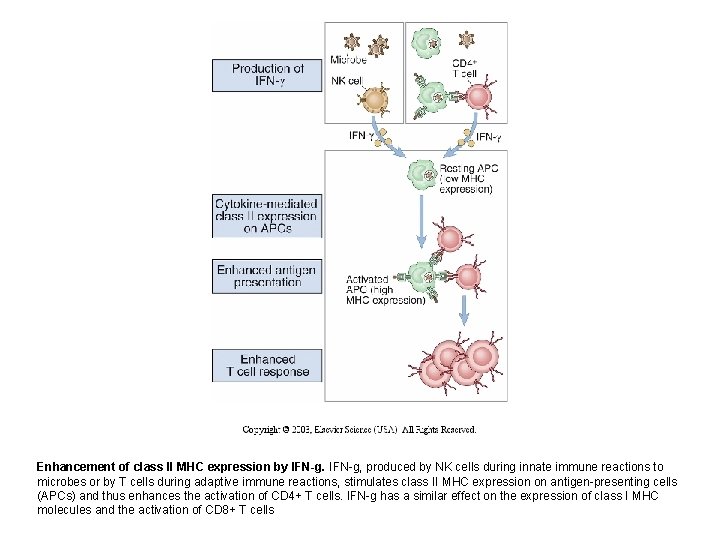
Enhancement of class II MHC expression by IFN-g, produced by NK cells during innate immune reactions to microbes or by T cells during adaptive immune reactions, stimulates class II MHC expression on antigen-presenting cells (APCs) and thus enhances the activation of CD 4+ T cells. IFN-g has a similar effect on the expression of class I MHC molecules and the activation of CD 8+ T cells

The end

Response rate after peptide-based vaccinations against melanoma Responses Patients % MART -127 -35 in IFA 0 20 0 g 209 -2 M in IFA 0 11 0 g 209 -2 M in IFA + IL-2 (720, 000 IU/Kg TID) 13 31 42 Treatment: Rosenberg et al. Nature Med, 1998

Come è possibile che le molecole MHC presentino un numero di peptidi diversi sufficiente a coprire la maggior parte delle proteine antigeniche ? Attraverso duplicazione genica ed elevato polimorfismo: • tanti loci codificanti per molecole simili • alleli polimorfici allo stesso locus • codominanza

Ricadute del polimorfismo dell’MHC sull’ immunopatologia : • Trapianti • Suscettibilità a varie patologie, spesso autoimmuni E’ importante in alcuni casi tipizzare i pazienti per il genotipo MHC

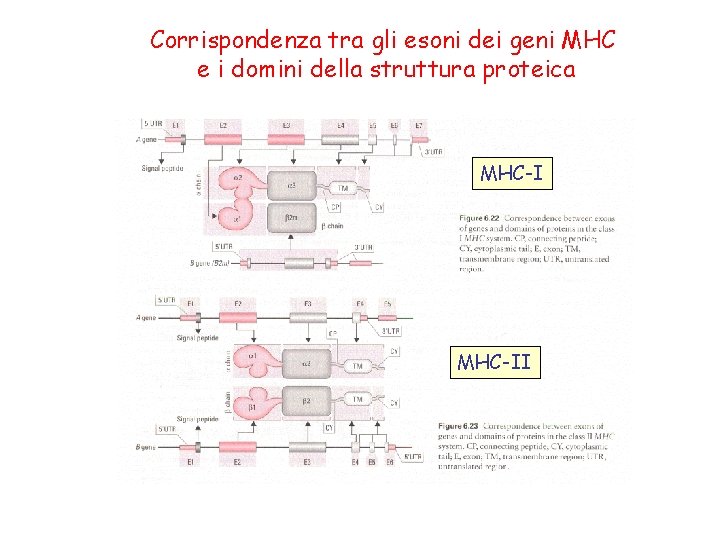
Corrispondenza tra gli esoni dei geni MHC e i domini della struttura proteica MHC-II

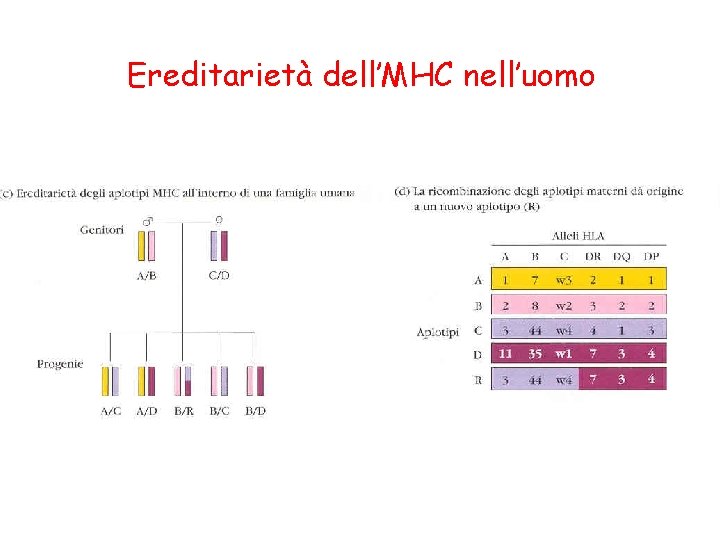
Ereditarietà dell’MHC nell’uomo

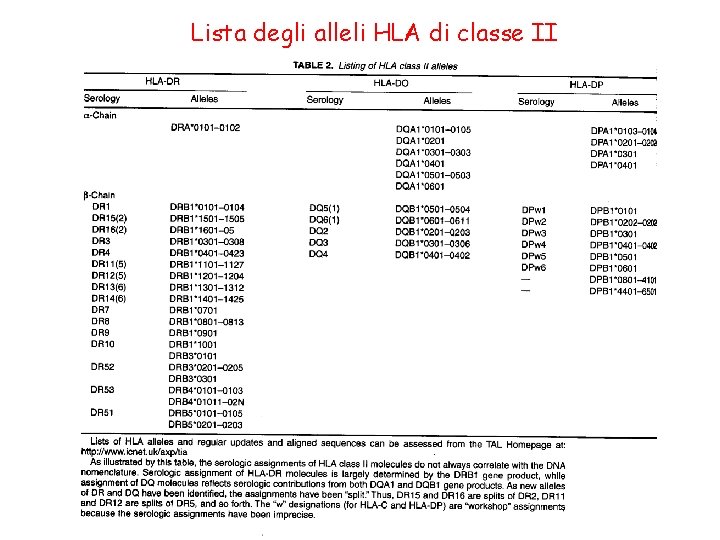
Lista degli alleli HLA di classe II

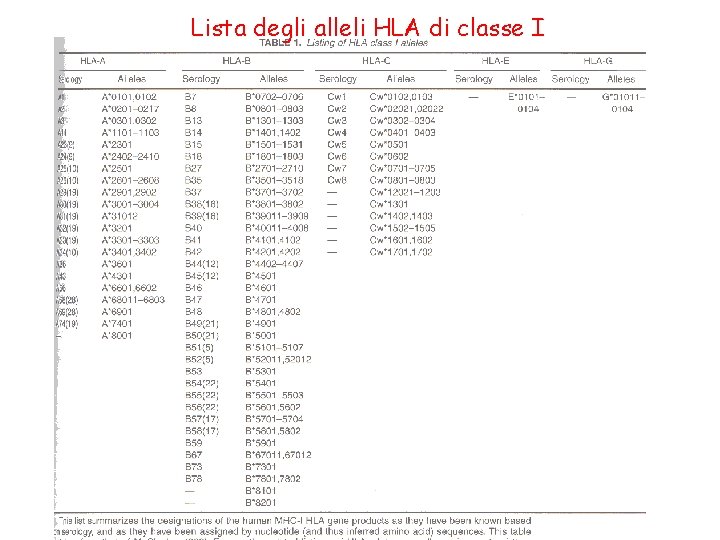
Lista degli alleli HLA di classe I
 Paraortico
Paraortico Presidente corso di laurea
Presidente corso di laurea Language acquisition and language learning
Language acquisition and language learning Immunologia pisa
Immunologia pisa Hassal testek
Hassal testek Tommasini burlo
Tommasini burlo Tcr immunologia
Tcr immunologia Gestione per conto dello stato
Gestione per conto dello stato Slide haccp ppt
Slide haccp ppt Laurea specialistica informatica
Laurea specialistica informatica Base laurea
Base laurea Tiina ranta laurea
Tiina ranta laurea Laurea amk
Laurea amk Laurea finna
Laurea finna Laurea magistrale in editoria e giornalismo
Laurea magistrale in editoria e giornalismo Unito domanda laurea
Unito domanda laurea Laurea specialistica informatica
Laurea specialistica informatica L 39 laurea
L 39 laurea Enpam riscatto laurea
Enpam riscatto laurea Pakki laurea
Pakki laurea Linguaggio di programmazione cruciverba
Linguaggio di programmazione cruciverba Laurea finna
Laurea finna Calcolo voto laurea unisalento
Calcolo voto laurea unisalento Laurea siirtohaku
Laurea siirtohaku Laurea erillishaku
Laurea erillishaku 1hr to sec
1hr to sec Cantico dei cantici il mio diletto
Cantico dei cantici il mio diletto Longum iter est per praecepta breve et efficax per exempla
Longum iter est per praecepta breve et efficax per exempla 1800 dam per jam berapa hm per menit
1800 dam per jam berapa hm per menit Nuo vandenyje gimusi vandens bijo
Nuo vandenyje gimusi vandens bijo Per capita vs per stirpes
Per capita vs per stirpes Moltiplicazioni con numeri periodici
Moltiplicazioni con numeri periodici 27 miles per gallon into kilometers per liter
27 miles per gallon into kilometers per liter Per stirpes v per capita
Per stirpes v per capita Catullus 84
Catullus 84 Coop per te
Coop per te Multas per gentes et multa per aequora vectus
Multas per gentes et multa per aequora vectus Corso tutela del consumatore
Corso tutela del consumatore Slide corso sicurezza croce rossa
Slide corso sicurezza croce rossa Slide corso ple
Slide corso ple Corso oss poliambulanza
Corso oss poliambulanza Slide corso macchine movimento terra
Slide corso macchine movimento terra Liceo linguistico correggio
Liceo linguistico correggio Formazione dirigenti sicurezza ppt
Formazione dirigenti sicurezza ppt Corso vhdl
Corso vhdl Slide corso rspp datore di lavoro rischio basso
Slide corso rspp datore di lavoro rischio basso Corso ecdl
Corso ecdl Slide corso antincendio rischio basso
Slide corso antincendio rischio basso Corso generale sicurezza sul lavoro
Corso generale sicurezza sul lavoro Corso dirigente accompagnatore basket
Corso dirigente accompagnatore basket Corso monosettimanale significato
Corso monosettimanale significato Corso valutazione investimenti
Corso valutazione investimenti Modellistica energetica
Modellistica energetica Corso primo soccorso slide
Corso primo soccorso slide Serie eluotropa
Serie eluotropa Relazione finale scuola primaria compilata
Relazione finale scuola primaria compilata Corso tatuaggi asl lecce 2021
Corso tatuaggi asl lecce 2021 Gru mobile autocarrata
Gru mobile autocarrata Corso dirigente accompagnatore calcio
Corso dirigente accompagnatore calcio Per una buona scrittura giuridica
Per una buona scrittura giuridica Ic camigliano
Ic camigliano Corso allenatore di base basket
Corso allenatore di base basket Glc cromatografia
Glc cromatografia Test verifica corso carroponte pdf
Test verifica corso carroponte pdf Corso toscana 108
Corso toscana 108 Open foam corso
Open foam corso Corso di sociologia bagnasco barbagli cavalli
Corso di sociologia bagnasco barbagli cavalli Corso linguistica computazionale
Corso linguistica computazionale Slide corso macchine movimento terra
Slide corso macchine movimento terra William paterson financial aid office
William paterson financial aid office Corso somministratori cils
Corso somministratori cils Slide corso rspp
Slide corso rspp Slide antincendio
Slide antincendio Corso icq
Corso icq Corso gascromatografia napoli
Corso gascromatografia napoli Slide corso ponteggi
Slide corso ponteggi Corso trattori agricoli ppt
Corso trattori agricoli ppt Corso toeic bassano
Corso toeic bassano Slide corso trattori agricoli
Slide corso trattori agricoli