1 La cellule et les protines cellule 1


















- Slides: 48

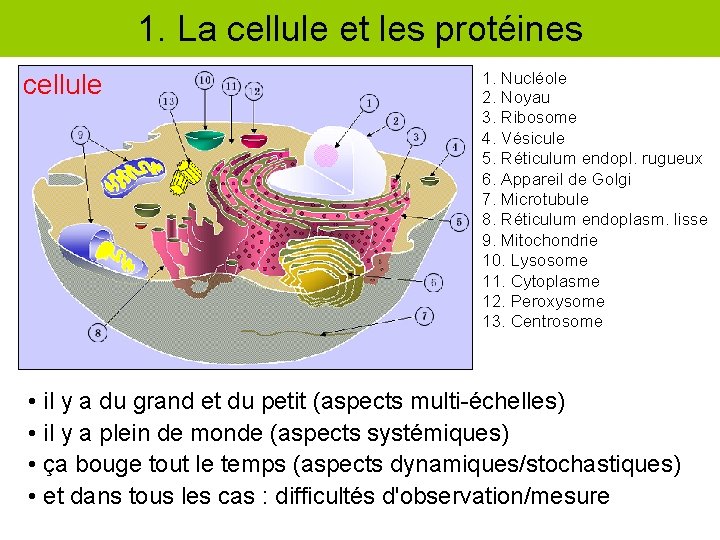
1. La cellule et les protéines cellule 1. Nucléole 2. Noyau 3. Ribosome 4. Vésicule 5. Réticulum endopl. rugueux 6. Appareil de Golgi 7. Microtubule 8. Réticulum endoplasm. lisse 9. Mitochondrie 10. Lysosome 11. Cytoplasme 12. Peroxysome 13. Centrosome • il y a du grand et du petit (aspects multi-échelles) • il y a plein de monde (aspects systémiques) • ça bouge tout le temps (aspects dynamiques/stochastiques) • et dans tous les cas : difficultés d'observation/mesure

échelles (un atome de 12 C a une masse de 12 Daltons) http: //bionumbers. hms. harvard. edu et méthodes

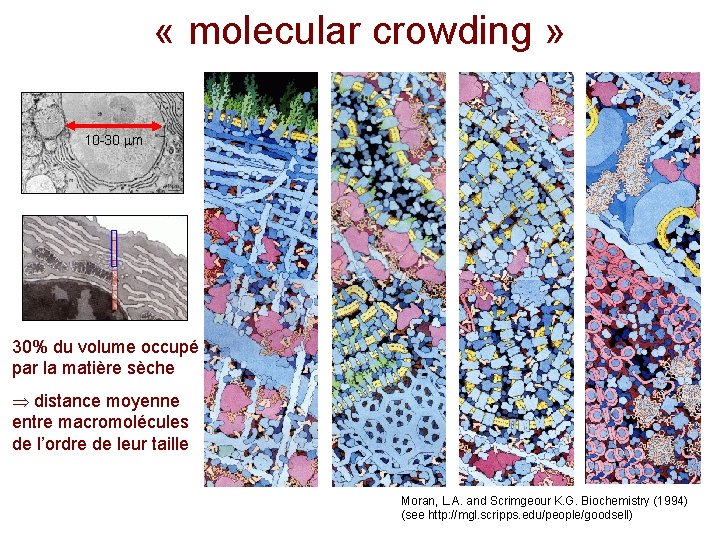
« molecular crowding » 10 -30 m 30% du volume occupé par la matière sèche distance moyenne entre macromolécules de l’ordre de leur taille Moran, L. A. and Scrimgeour K. G. Biochemistry (1994) (see http: //mgl. scripps. edu/people/goodsell)

macromolécules ADN, protéines, glucides, lipides information génétique énergie 30000 protéines différentes membranes

protéines

protéines des rôles multiples… Enzymes. catalyseurs qui accélèrent les vitesses de réaction en abaissant la barrière énergétique entre les réactifs et de produits. Protéines régulatrices. impliqués dans la communication moléculaire et le contrôle des voies biochimiques. Protéines de transport. rôle spécifique dans la liaison et le transport de molécules dans et hors de la cellule. Beaucoup sont des protéines membranaires, mais aussi globulaires. Exemple : hémoglobine. Protéines de stockage. matières premières pour d'autres réactions biochimiques. Exemple : l’ovalbumine est une source de nutriments pour l'embryon d'oiseau. Protéines mobiles et contractiles. protéines capables de mouvement et de contraction. Souvent protéines fibreuses, en raison de leur résistance à la traction. Protéines architecturales. impliquées dans l’organisation des structures cellulaires, par exemple dans l’organisation de l’ADN à l’intérieur du noyau (chromatine) Protéines structurales. une grande partie de la «colle» et «fibres» qui vous permet de rester ensemble implique des protéines fibreuses. Tendons, cartilages, cheveux, ongles, peau. . . Protéines d'échafaudage. sur lesquelles molécules impliquées dans le transfert des signaux viennent se fixer simultanément avec l’orientation qui leur permet d’interagir. Moteurs moléculaires. chargés des actions mécaniques impliqués dans différents phénomènes et du transport.

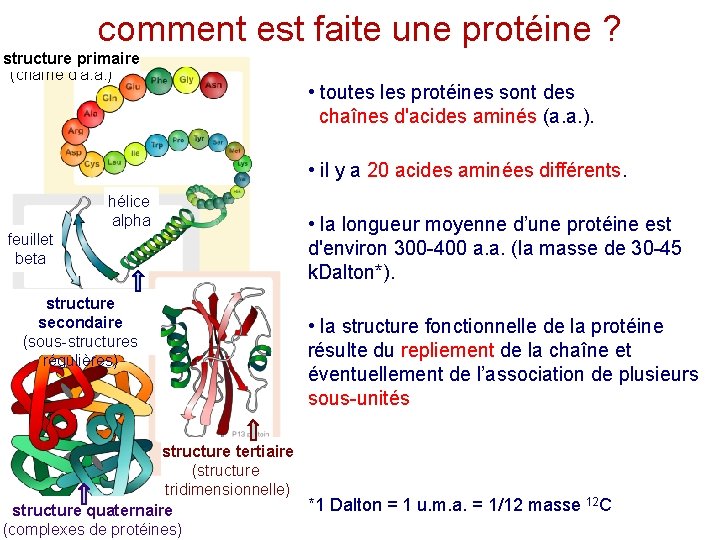
comment est faite une protéine ? structure primaire (chaîne d’a. a. ) • toutes les protéines sont des chaînes d'acides aminés (a. a. ). • il y a 20 acides aminées différents. hélice alpha feuillet beta structure secondaire (sous-structures régulières) • la longueur moyenne d’une protéine est d'environ 300 -400 a. a. (la masse de 30 -45 k. Dalton*). • la structure fonctionnelle de la protéine résulte du repliement de la chaîne et éventuellement de l’association de plusieurs sous-unités structure tertiaire (structure tridimensionnelle) *1 Dalton = 1 u. m. a. = 1/12 masse 12 C structure quaternaire (complexes de protéines)

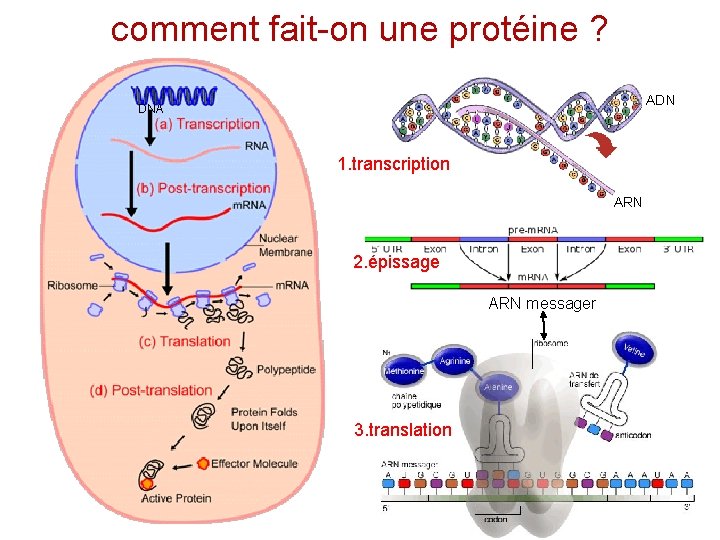
comment fait-on une protéine ? ADN DNA 1. transcription ARN 2. épissage ARN messager 3. translation

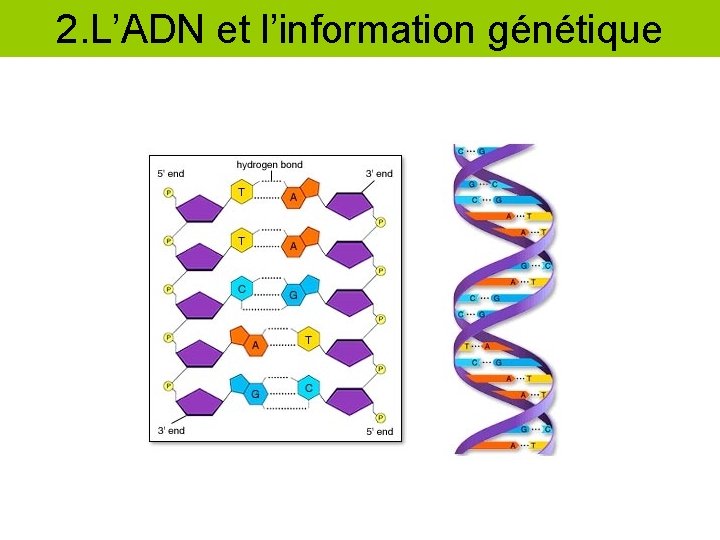
2. L’ADN et l’information génétique

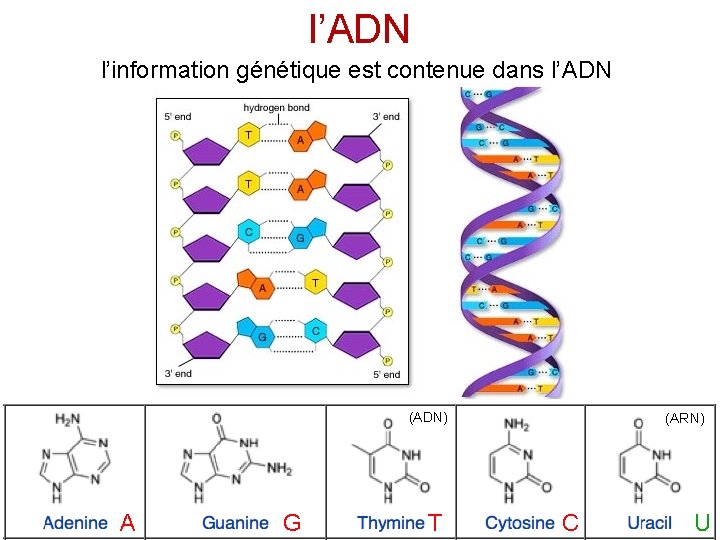
l’ADN l’information génétique est contenue dans l’ADN (ADN) (ARN) A G T C U

transcription l’information génétique est organisée par triplets (codons)

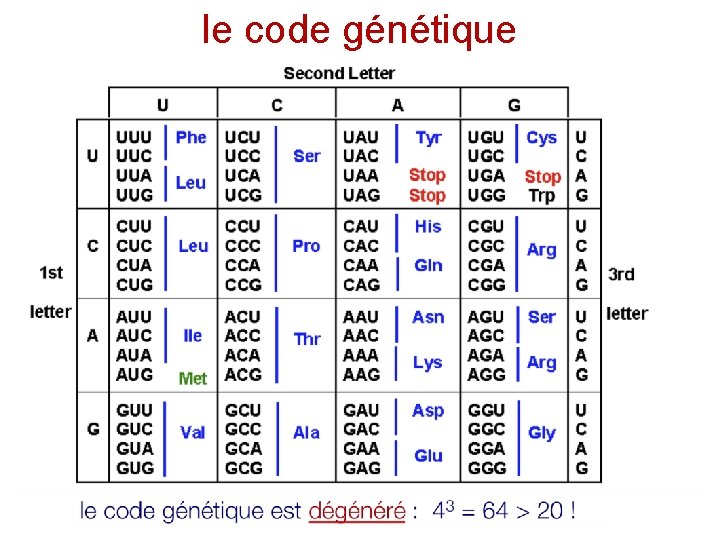
le code génétique 1 triplet = 1 codon = 3 lettres = 1 acide aminé

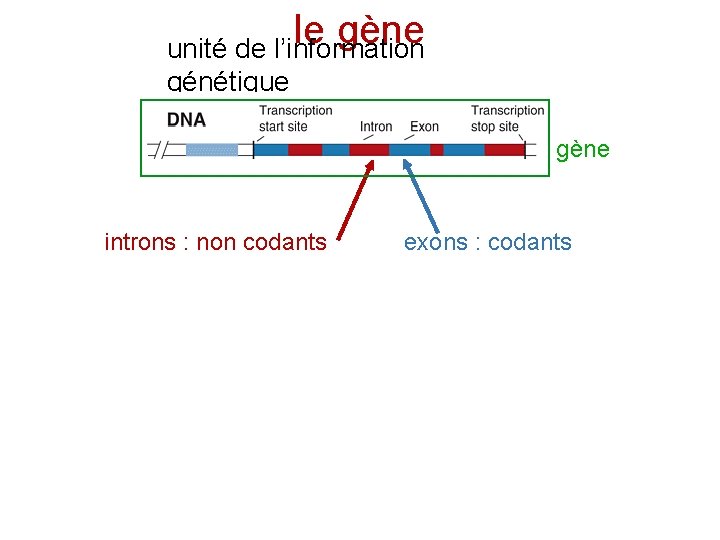
le gène unité de l’information génétique gène introns : non codants exons : codants

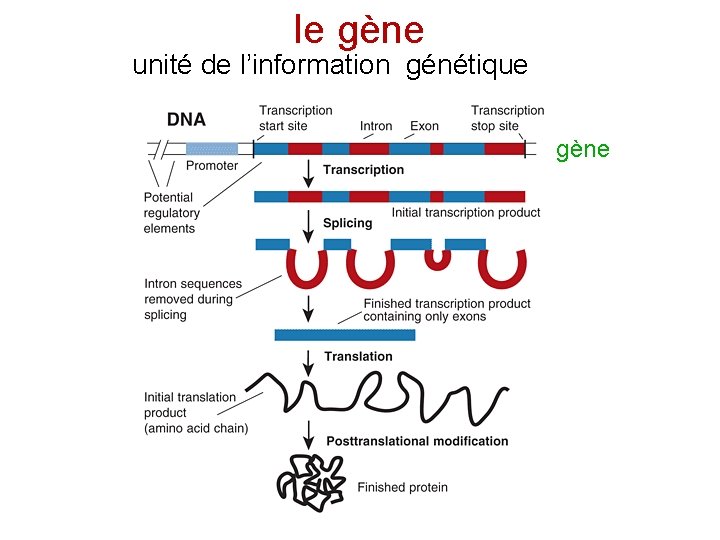
le gène unité de l’information génétique gène

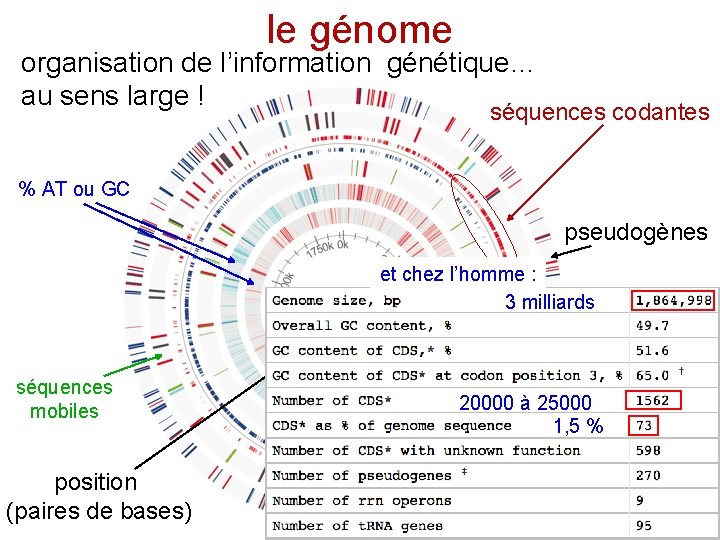
le génome organisation de l’information génétique… au sens large ! séquences codantes % AT ou GC pseudogènes et chez l’homme : 3 milliards séquences mobiles position (paires de bases) 20000 à 25000 gènes pour 1, 5 % t. ARN et r. ARN

ADN codant et non codant en résumé : ADN « poubelle » exons introns pourquoi autant d’ADN non codant ? peut-on en comprendre le rôle ?

3. étudier les séquences d’ADN approche « déterministe » : comparaison entre séquences / alignement de séquences : • recherche de gènes • recherche de similarités entre espèces - évolution • recherches de motifs répétés - régulation, organisation approche statistique : à la recherche de caractéristiques « globales » : • différencier les régions codantes et non codantes • recher un « ordre » dans le désordre apparent…

analyse statistiques des séquences qu’est-ce qui différencie les séquences d’ADN de simples séquences aléatoires ? mesurer l’ordre dans l’ADN 1. information mutuelle 2. fonction de corrélation / densité spectrale de puissance 3. techniques basées sur la « marche ADN » 4. …

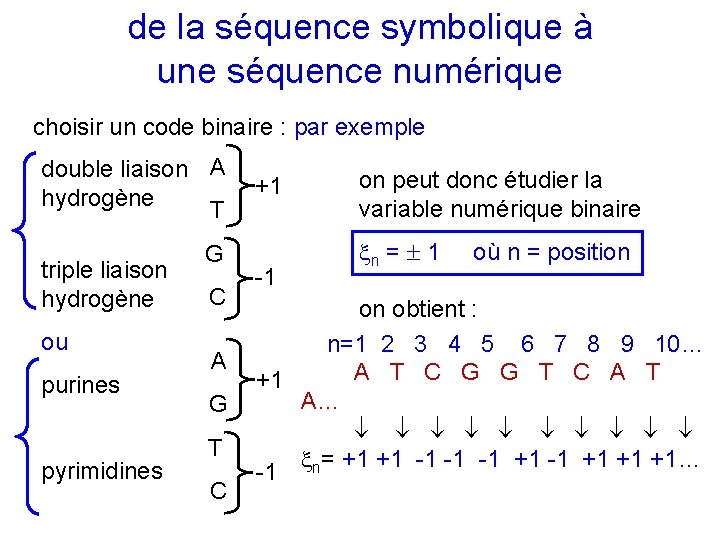
de la séquence symbolique à une séquence numérique choisir un code binaire : par exemple double liaison A hydrogène T triple liaison hydrogène ou purines pyrimidines G C A G T C +1 -1 on peut donc étudier la variable numérique binaire n = 1 où n = position on obtient : n=1 2 3 4 5 6 7 8 9 10… +1 A T C G G T C A T A… = +1 +1 -1 -1 -1 +1 +1 +1… -1 n

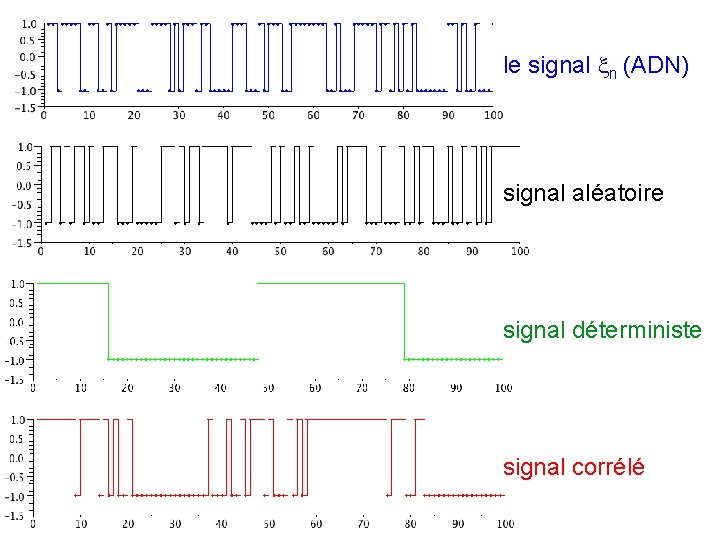
le signal n (ADN) signal aléatoire signal déterministe signal corrélé

fonction de corrélation et densité spectrale de puissance (DSP) soit (t) signal aléatoire fonction du temps t, stationnaire : 1. on peut définir la fonction de corrélation de (t) 2. si « ergodique » , on peut remplacer la moyenne d’ensemble par une moyenne sur le temps : chaque t 0 initial considéré comme une nouvelle réalisation

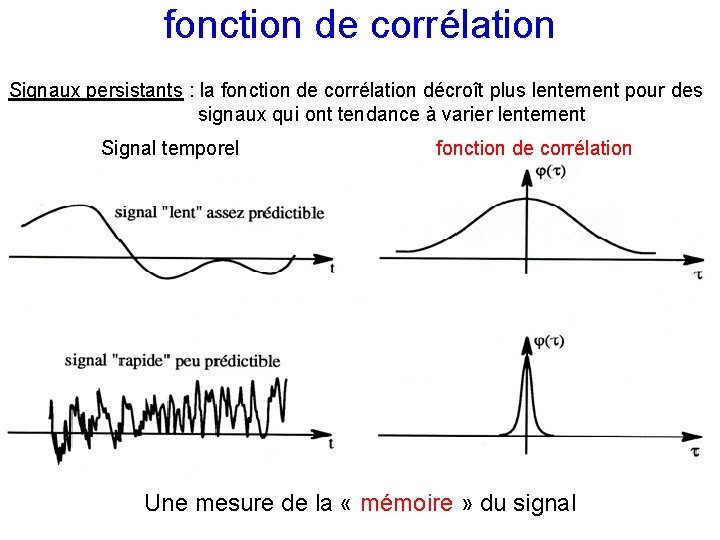
fonction de corrélation Signaux persistants : la fonction de corrélation décroît plus lentement pour des signaux qui ont tendance à varier lentement Signal temporel fonction de corrélation Une mesure de la « mémoire » du signal

bruit blanc et mouvement brownien

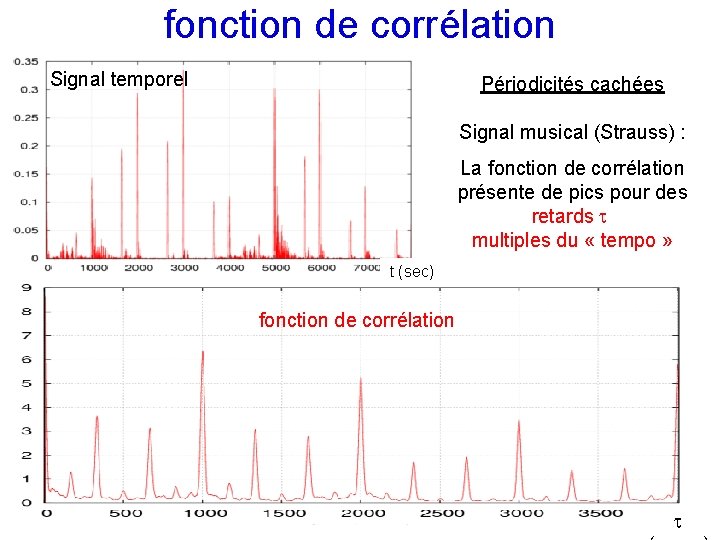
fonction de corrélation Signal temporel Périodicités cachées Signal musical (Strauss) : La fonction de corrélation présente de pics pour des retards multiples du « tempo » t (sec) fonction de corrélation

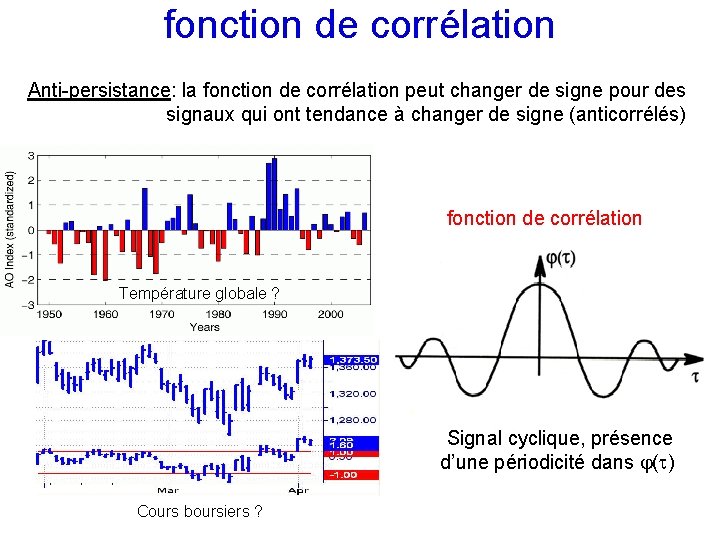
fonction de corrélation Anti-persistance: la fonction de corrélation peut changer de signe pour des signaux qui ont tendance à changer de signe (anticorrélés) fonction de corrélation Température globale ? Signal cyclique, présence d’une périodicité dans ( ) Cours boursiers ?

densité spectrale de puissance (DSP) 3. on peut passer à la représentation en fréquence par transformée de Fourier (TF) : on obtient la densité spectrale de puissance 4. Théorème de Wiener-Khintchine : où limitée à l’intérvalle [0, T] 5. On peut alors évaluer S ( f ) directement à partir du signal : estimateur de la DSP d’un signal réel :

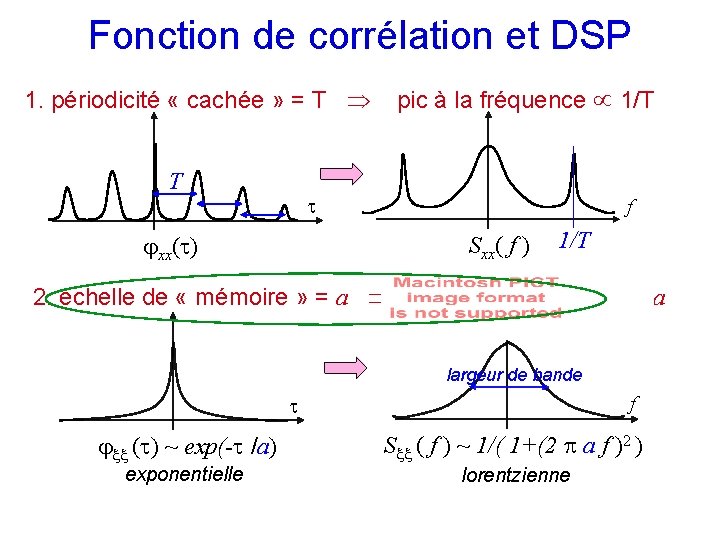
Fonction de corrélation et DSP 1. périodicité « cachée » = T pic à la fréquence 1/T T xx( ) f Sxx( f ) 1/T 2. échelle de « mémoire » = a largeur de bande 1/a largeur de bande f ( ) ~ exp(- /a) exponentielle S ( f ) ~ 1/( 1+(2 a f )2 ) lorentzienne

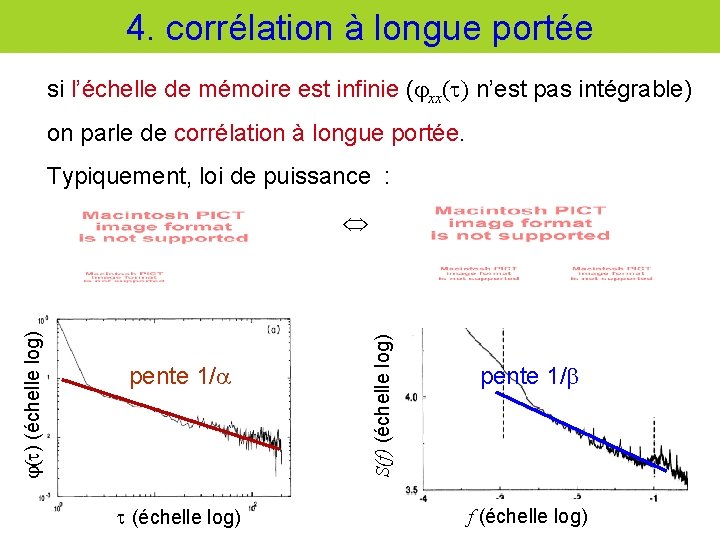
4. corrélation à longue portée si l’échelle de mémoire est infinie ( xx( ) n’est pas intégrable) on parle de corrélation à longue portée. Typiquement, loi de puissance : pente 1/ (échelle log) S(f) (échelle log) ( ) (échelle log) pente 1/ f (échelle log)

corrélation à longue portée pour la marche aléatoire x(t) associée on peut définir un troisième coefficient, le coefficient de Hurst : METTERE INSIEME CON SEGUENTE +fig

corrélation à longue portée si est à moyenne zéro et stationnaire, on montre pour la variance de x : avec exposant de Hurst : Si H =1/2 : séquence décorrélée, diffusion normale Si 0 < H < 1/2 : séquence anti-corrélée, « sub-diffusion » Si 1/2 < H < 1 : séquence corrélée à longue portée, « super-diffusion » diffusion x(t) propriétés de corrélation longue portée

corrélation longue portée : phénomène universel généralités : c’est courant en physique (faire) http: //www. scholarpedia. org/arti cle/1/f_noise

5. corrélation et ADN

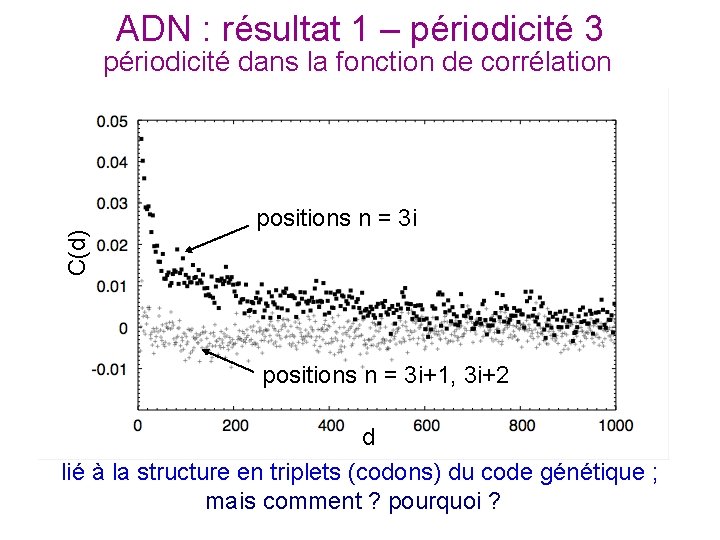
ADN : résultat 1 – périodicité 3 C(d) périodicité dans la fonction de corrélation positions n = 3 i+1, 3 i+2 d lié à la structure en triplets (codons) du code génétique ; mais comment ? pourquoi ?

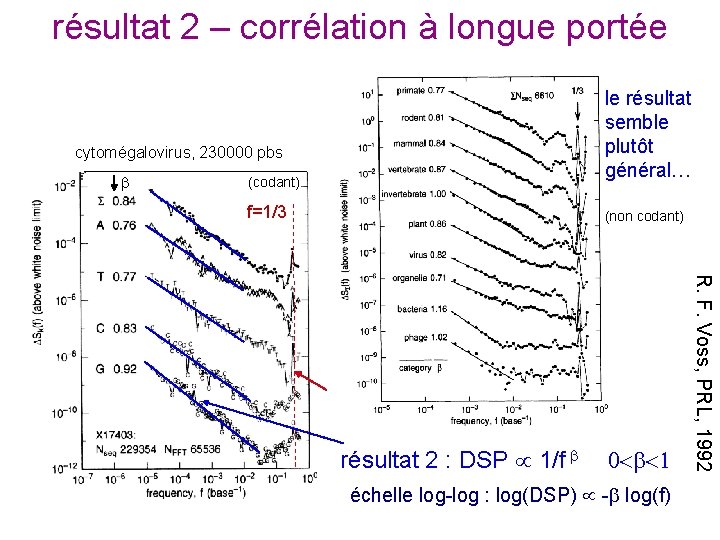
résultat 2 – corrélation à longue portée cytomégalovirus, 230000 pbs (codant) f=1/3 le résultat semble (variation de la méthode : plutôt construction de 4 sous-séquences général… 0/1 pour A, T, C, G) (non codant) résultat 2 : DSP 1/f échelle log-log : log(DSP) - log(f) R. F. Voss, PRL, 1992 résultat 1 : pic à f = 1/3

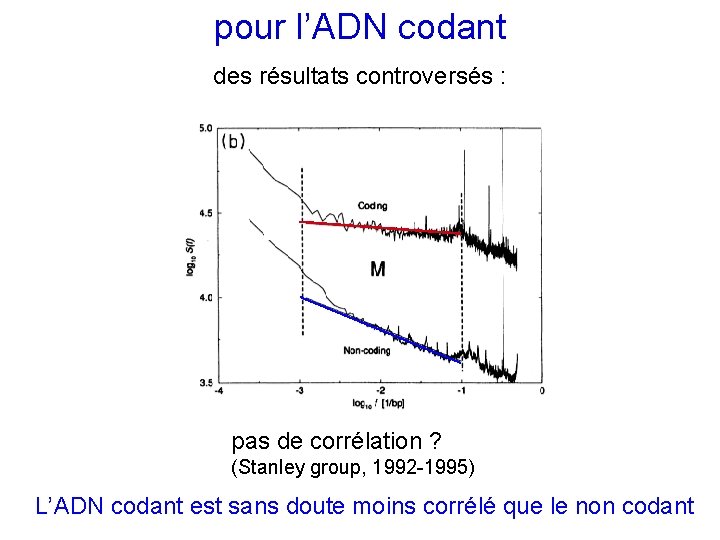
pour l’ADN codant des résultats controversés : pas de corrélation ? (Stanley group, 1992 -1995) L’ADN codant est sans doute moins corrélé que le non codant

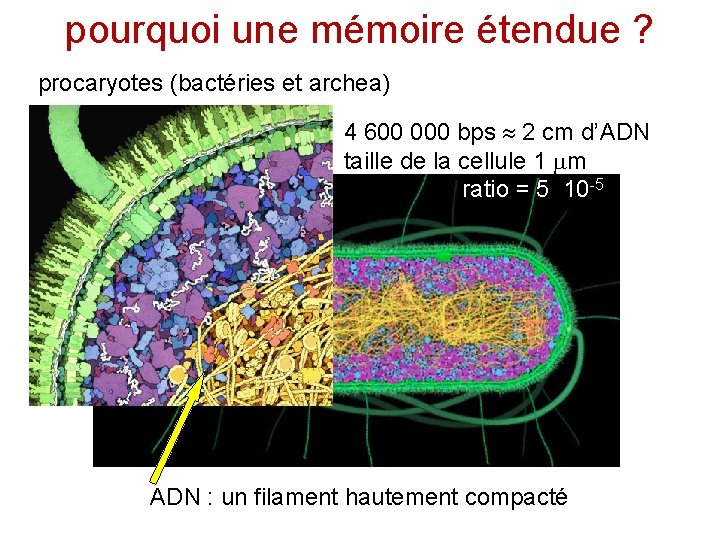
pourquoi une mémoire étendue ? procaryotes (bactéries et archea) 4 600 000 bps 2 cm d’ADN taille de la cellule 1 m ratio = 5 10 -5 ADN : un filament hautement compacté

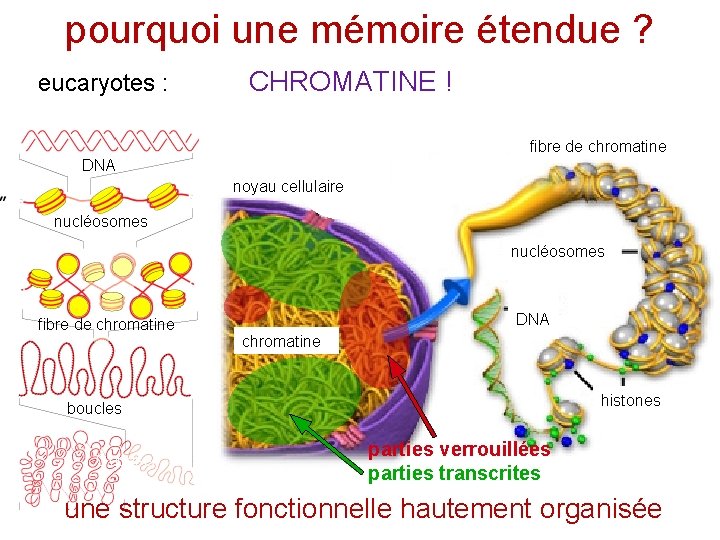
pourquoi une mémoire étendue ? eucaryotes : CHROMATINE ! fibre de chromatine DNA noyau cellulaire nucléosomes fibre de chromatine DNA chromatine histones boucles Goodsell parties verrouillées parties transcrites une structure fonctionnelle hautement organisée

d’autres images

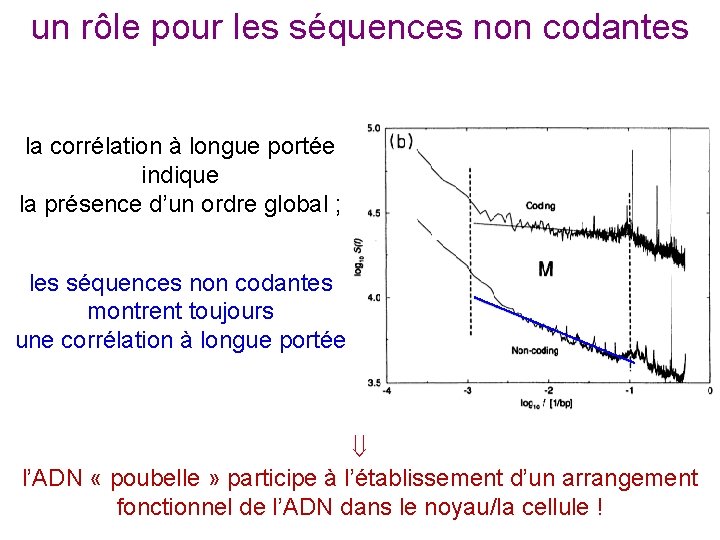
un rôle pour les séquences non codantes la corrélation à longue portée indique la présence d’un ordre global ; les séquences non codantes montrent toujours une corrélation à longue portée l’ADN « poubelle » participe à l’établissement d’un arrangement fonctionnel de l’ADN dans le noyau/la cellule !

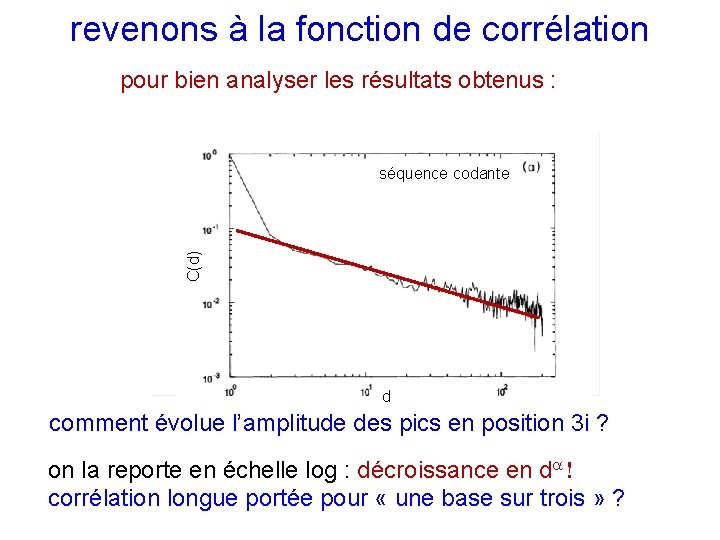
revenons à la fonction de corrélation pour bien analyser les résultats obtenus : C(d) séquence codante positions n = 3 i+1, 3 i+2 d d comment évolue l’amplitude des pics en position 3 i ? on la reporte en échelle log : décroissance en d corrélation longue portée pour « une base sur trois » ?

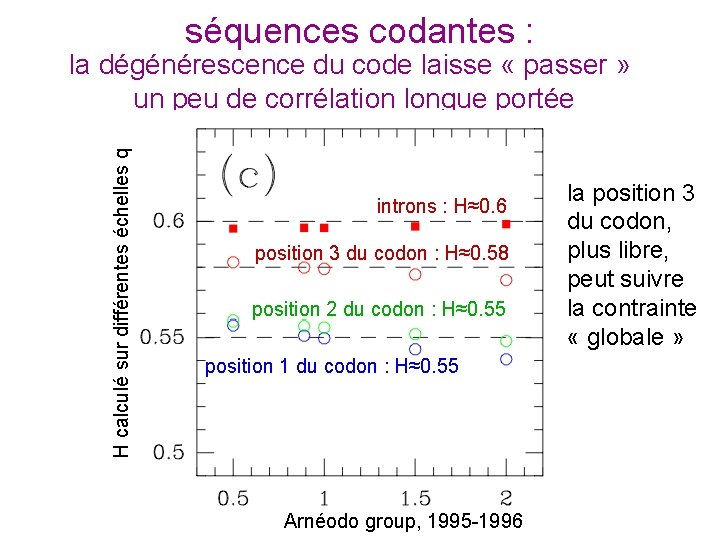
séquences codantes : H calculé sur différentes échelles q la dégénérescence du code laisse « passer » un peu de corrélation longue portée dégénérescence la position 3 introns : H≈0. 6 sur la troisième du codon, plus libre, position 3 du codon : H≈0. 58 lettre du codon peut suivre la contrainte position 2 du codon : H≈0. 55 « globale » position 1 du codon : H≈0. 55 Arnéodo group, 1995 -1996

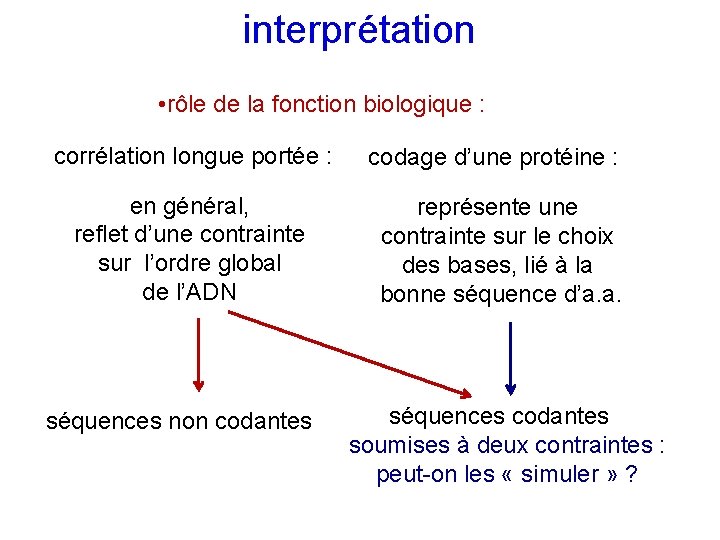
interprétation • rôle de la fonction biologique : corrélation longue portée : en général, reflet d’une contrainte sur l’ordre global de l’ADN séquences non codantes codage d’une protéine : représente une contrainte sur le choix des bases, lié à la bonne séquence d’a. a. séquences codantes soumises à deux contraintes : peut-on les « simuler » ?

signal discret fonction de corrélation de n n = 1 2 3 4 5 6 7 8 9 10 11… A T C G G T C A T A C… n= +1 +1 -1 -1 -1 +1 +1 +1 -1… n signal aléatoire discret fonction de la position n plutôt que du temps t : si < n>=0 (autrement, on soustrait la moyenne) alors la fonction de corrélation s’écrit d = distance entre 2 sites le long de la séquence moyenne d’ensemble moyenne sur n

fonction de corrélation de n en pratique : sur un ordinateur, le signal est toujours discret ! z(t) (z 1, z 2, z 3, z 4, … z. N) = z pour nous, le signal est intrinsèquement discret (pas=1), car c’est la séquence. sous scilab, il donc d’utiliser la fonction : corr : corr(z, dmax) = fonction de corrélation de x, en fonction de la distance d, pour d = 0, 1, 2, … dmax-1 sous scilab, corr soustrait automatiquement la moyenne de z

DSP de n rajouter un slide fft (cours du TP)

DSP de n Wiener-Khintchine : estimateur DSP = signal discret de pas = 1 et N points : - la fréquence max « observable » est fe = 1/pas = 1 - le spectre contient N points, séparés de ∆f = 1/durée = 1/N FFT = transformée de Fourier Rapide sous scilab : fft : fft(z, -1) = fft(z) = TF de z pour f = 0, ∆f, 2∆f, … fe-1 d’où |fft(z)|2 / N ≈ DSP de z sous scilab, commencer par soustraire la moyenne de z

fin

1. information mutuelle probabilité jointe d’avoir les symboles i et j à distance d probabilités d’avoir les symboles i et j (densités) zéro si indépendants car Pij(d)=Pi. Pj remarque : en thermo, S = -k. B∑pilnpi … c’est une mesure d’entropie! voir « entropie de Shannon »