Zytologie 3 4 Dr Attila Magyar 20 09












- Slides: 35

Zytologie 3 -4. Dr. Attila Magyar 20. 09. 2013 27. 09. 2013

Endoplasmatisches Retikulum und Ribosomen

Endoplasmatisches Retikulum 1. • Miteinander verbundener Netzwerk in der Zelle aus Zysternen und Tubuli. Alle Raume des ERs in einer Zelle sind gemeinsam. ER ist durch ER-Membran bedeckt. • An einigen Stellen die äußere Oberfläche der Membran Ribosomenfrei ist (glattes ER, smooth ER, s. ER), an anderesn Stellen sitzen daran Ribosomen: raues ER (r. ER). • ER-Zysternen kommunizieren nur mit perinukleären Zysternen. • Durch Transportvesikeln ist ER-Lumen mit dem des Golgi. Apparats verknüpft.

Endoplasmatisches Retikulum 2. • Funktion des ERs r. ER: Synthese von Proteinen für Golgi, Lysosomen, Sekretionsvesikeln, Zellmembran. Proteine, die gelangen in r. ER-Zysternen, bekommen nach vollendeter Synthese Oligosaccharid-Nebenketten (sog. N-Glykosylierung, generell und gleich für alle Proteine). s. ER: Phospholipid-Synthese, Steroidsynthese, Entgiftung, Ca. Speicher

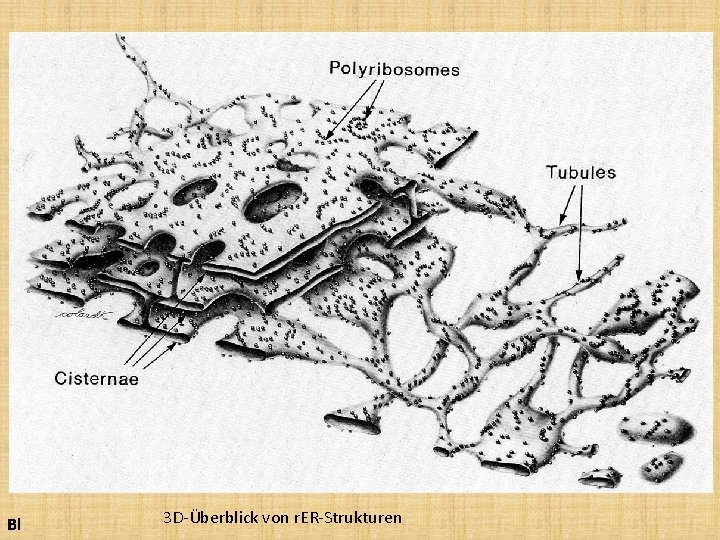
Bl 3 D-Überblick von r. ER-Strukturen

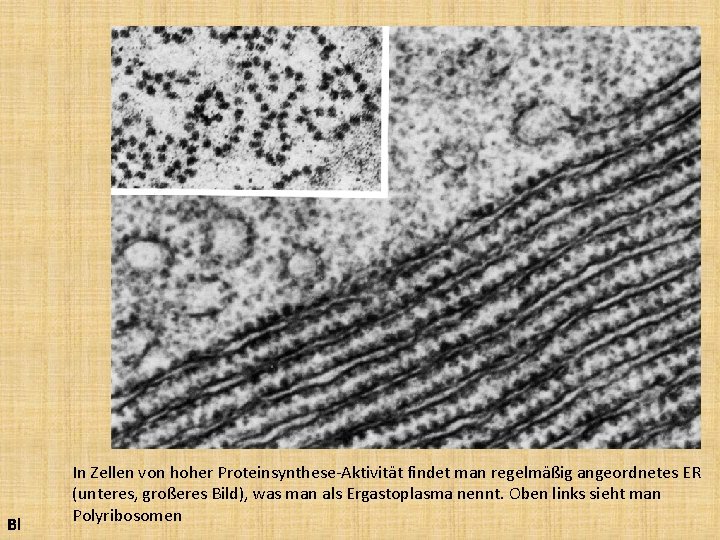
Bl In Zellen von hoher Proteinsynthese-Aktivität findet man regelmäßig angeordnetes ER (unteres, großeres Bild), was man als Ergastoplasma nennt. Oben links sieht man Polyribosomen

Ribosomen

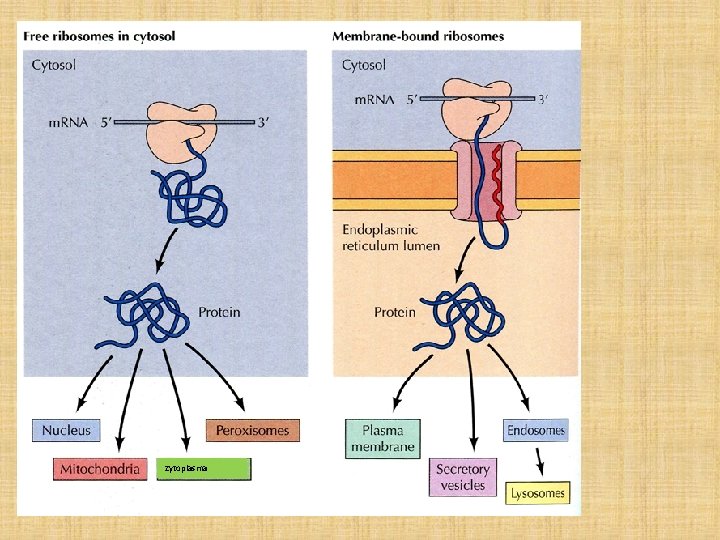
Ribosomen • Proteinsynthese passiert ausschließlich an den Ribosomen. • Ribosomen sind in die kleine und große Einheiten zerfallen, falls sie kein m. RNS binden. (m. RNS induziert Ribosome-Zusammenbau, ribosome assembly). • Alle Proteinsynthese beginnt immer an freien Ribosomen in der Zytoplasma. • Kurz nach dem Anfang der Proteinsynthese wird es entscheiden: wo passiert die weitere ribosomale Synthese? • Wenn das Protein (beim N-terminalen Ende) kein Sequenz für r. ER besitzt, bleibt das Ribosom im Zytoplasma. • Wenn das Protein (beim N-terminalen Ende) ein Sequenz (sog. Signalsequenz) für r. ER besitzt, wird die Synthese an demselben Ribosom, aber schon an der Oberfläche der r. ER fortgesetzt.

An einer m. RNS (waagerecht ziehendes Fädchen, roter Pfeil) angeordnete Ribosomen (kugelförmige Strukturen; insgesamt: Polyribosome): gleichzeitig passiert mehrfache Proteinsynthese (ausstülpende Proteinkette hängen nach oben und unten) von derselben m. RNS (natürlich in zeitlicher Verschiebung) Bl

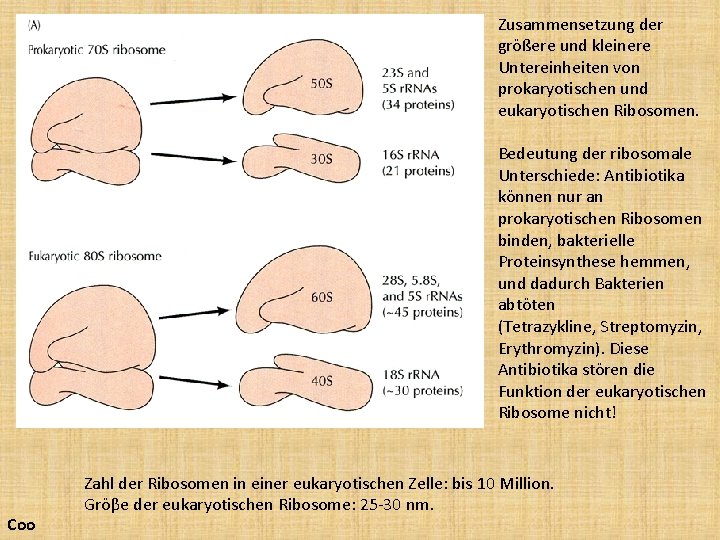
Zusammensetzung der größere und kleinere Untereinheiten von prokaryotischen und eukaryotischen Ribosomen. Bedeutung der ribosomale Unterschiede: Antibiotika können nur an prokaryotischen Ribosomen binden, bakterielle Proteinsynthese hemmen, und dadurch Bakterien abtöten (Tetrazykline, Streptomyzin, Erythromyzin). Diese Antibiotika stören die Funktion der eukaryotischen Ribosome nicht! Coo Zahl der Ribosomen in einer eukaryotischen Zelle: bis 10 Million. Gröβe der eukaryotischen Ribosome: 25 -30 nm.

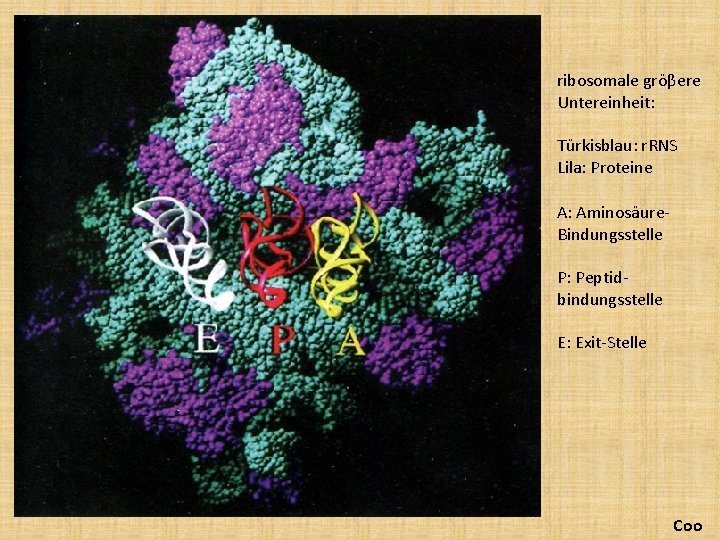
ribosomale gröβere Untereinheit: Türkisblau: r. RNS Lila: Proteine A: Aminosäure. Bindungsstelle P: Peptidbindungsstelle E: Exit-Stelle Coo

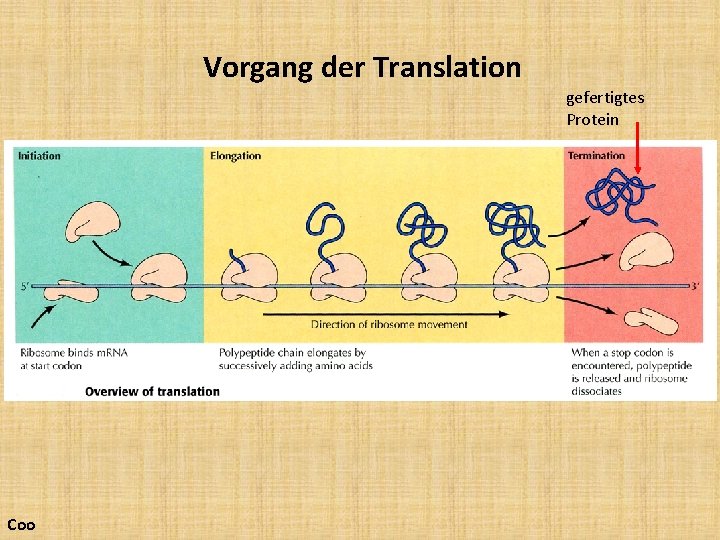
Vorgang der Translation gefertigtes Protein Coo

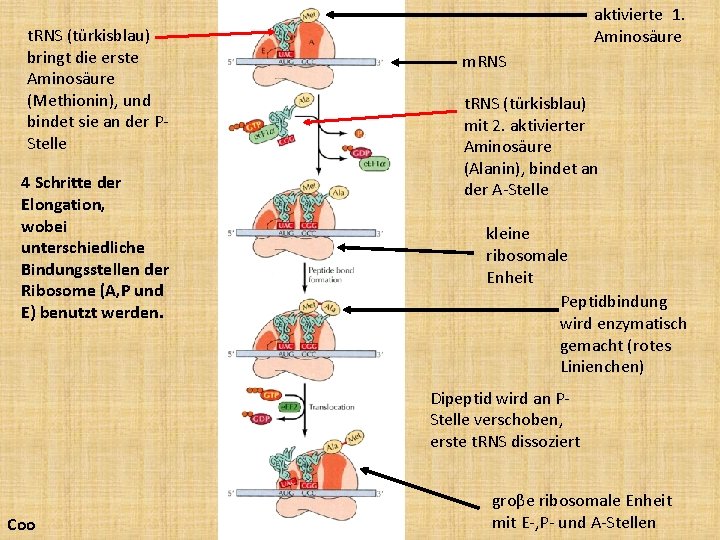
t. RNS (türkisblau) bringt die erste Aminosäure (Methionin), und bindet sie an der PStelle 4 Schritte der Elongation, wobei unterschiedliche Bindungsstellen der Ribosome (A, P und E) benutzt werden. aktivierte 1. Aminosäure m. RNS t. RNS (türkisblau) mit 2. aktivierter Aminosäure (Alanin), bindet an der A-Stelle kleine ribosomale Enheit Peptidbindung wird enzymatisch gemacht (rotes Linienchen) Dipeptid wird an PStelle verschoben, erste t. RNS dissoziert Coo groβe ribosomale Enheit mit E-, P- und A-Stellen

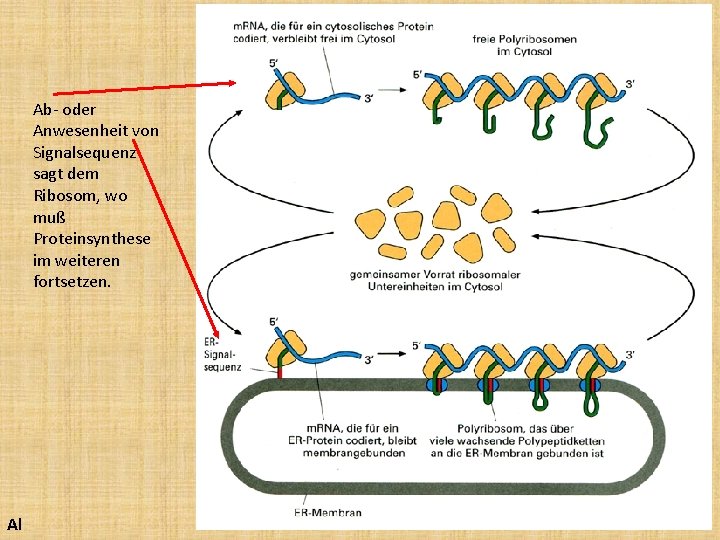
Ab- oder Anwesenheit von Signalsequenz sagt dem Ribosom, wo muß Proteinsynthese im weiteren fortsetzen. Al

Zytoplasma

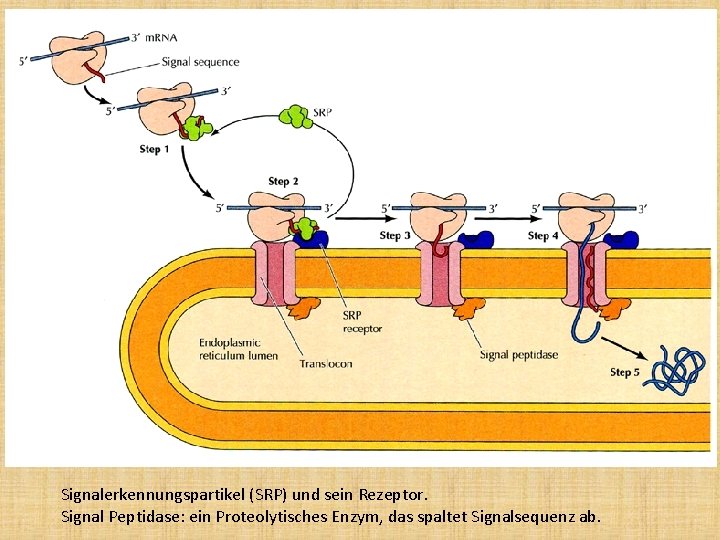
Signalerkennungspartikel (SRP) und sein Rezeptor. Signal Peptidase: ein Proteolytisches Enzym, das spaltet Signalsequenz ab.

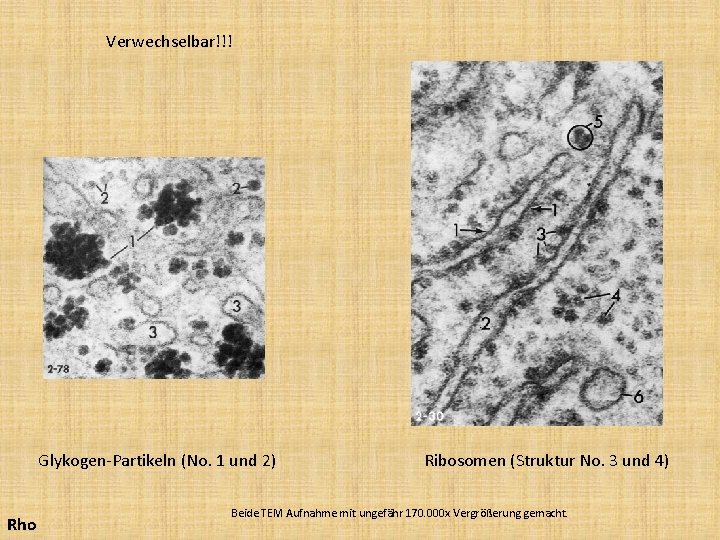
Verwechselbar!!! Glykogen-Partikeln (No. 1 und 2) Rho Ribosomen (Struktur No. 3 und 4) Beide TEM Aufnahme mit ungefähr 170. 000 x Vergrößerung gemacht.

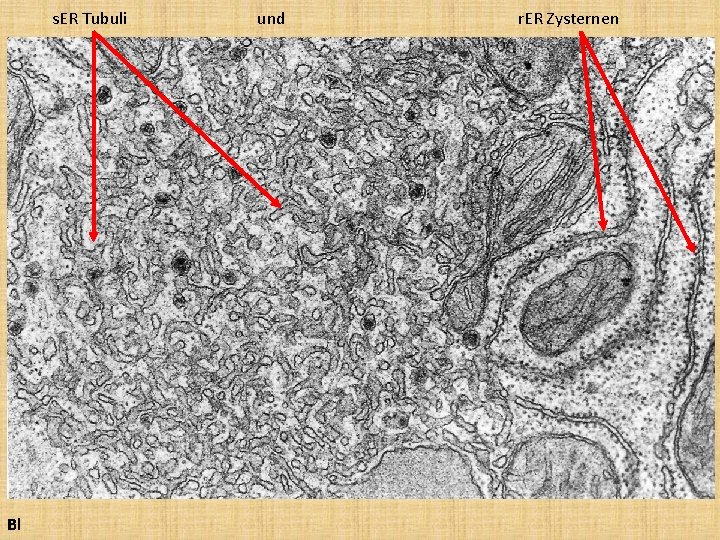
s. ER Tubuli Bl und r. ER Zysternen

Golgi-Apparat

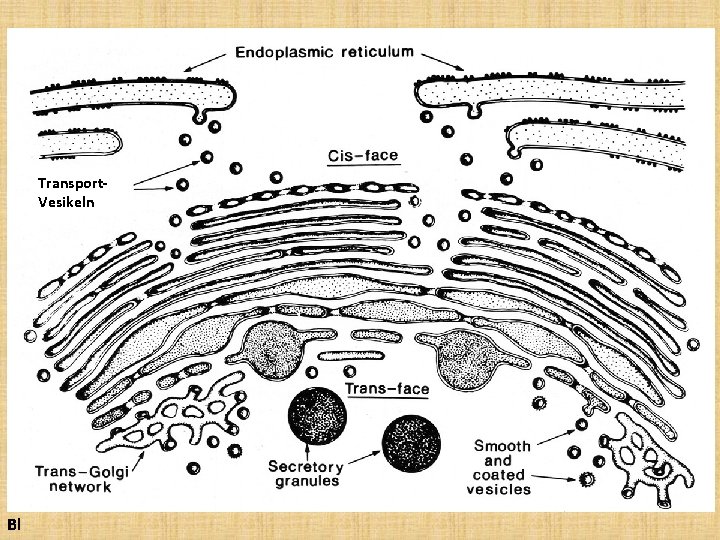
Golgi Apparat 1. • Geschloßene Zysternensystem. Hier werden Proteine posttranslational modifiziert. • Modifizierungen: die schon vorhandene Oligosaccharid Nebenketten (N-Glykosylation) werden weitermodifiziert oder neue Nebenketen werden kovalent gebunden (OGlykosylation). • Golgi Apparat besteht aus: cis-Zysterne mittlere Zysterne(n) trans-Zysterne • Jede Zysterne enthält eine bestimmte Enzymgarnitur für bestimmte Oligisaccharid-Modifizierungen.

Golgi Apparat 2. • Kommunikation zwischen r. ER und Golgi, beziehungsweise zwischen den Golgi-Zysternen passiert mit Transportvesikeln. • Reihenfolge: r. ER→cis Golgi→mittlere Golgi→trans-Golgi→sortierte Vesikel (Lysosom, Sekretion) • Die Modifizierungen hängen von bestimmten Sequenzen in der Proteinkette. Z. B. : bestimmte Sequenz, der sog. signal patch (wenn kommt in einem Protein vor), sagt dem enzymatischen Apparat der Golgi, Monosaccharide zu Phosphorylieren (dadurch entstehen Mannose-6 -Phosphate Einheiten in der Oligosaccharid Nebenkette). M-6 -P enthaltene Proteine werden zu den Lysosomen durch spezifische M-6 -P-Rezeptoren sortiert.

Transport. Vesikeln Bl

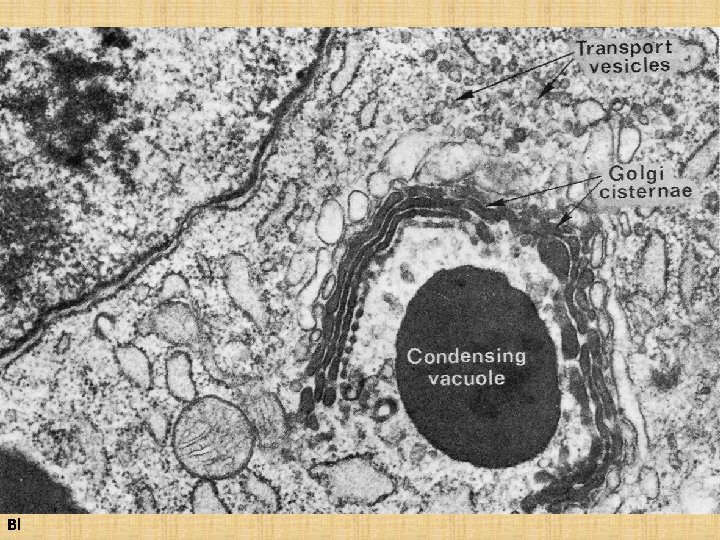
Bl

Bl

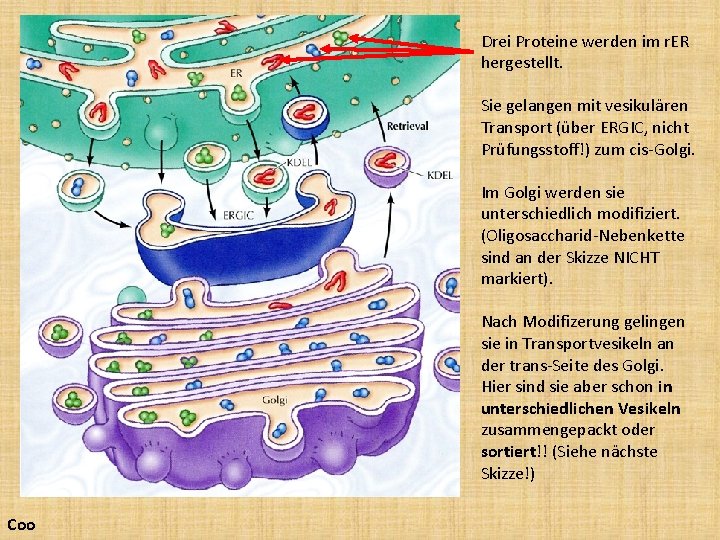
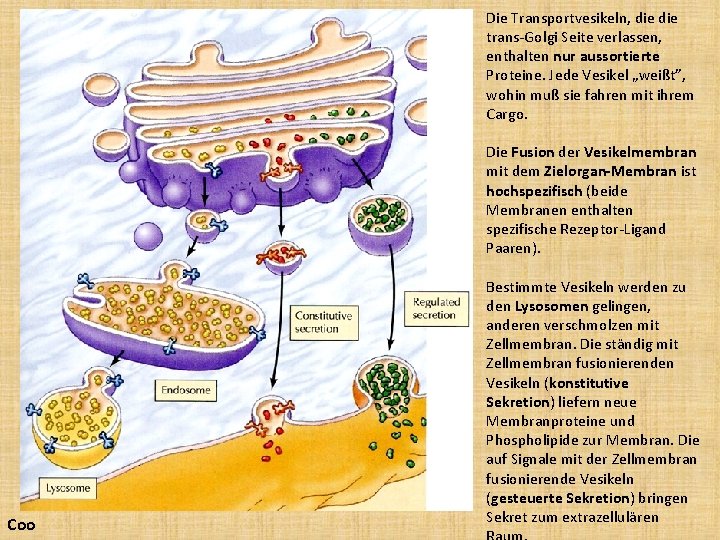
Drei Proteine werden im r. ER hergestellt. Sie gelangen mit vesikulären Transport (über ERGIC, nicht Prüfungsstoff!) zum cis-Golgi. Im Golgi werden sie unterschiedlich modifiziert. (Oligosaccharid-Nebenkette sind an der Skizze NICHT markiert). Nach Modifizerung gelingen sie in Transportvesikeln an der trans-Seite des Golgi. Hier sind sie aber schon in unterschiedlichen Vesikeln zusammengepackt oder sortiert!! (Siehe nächste Skizze!) Coo

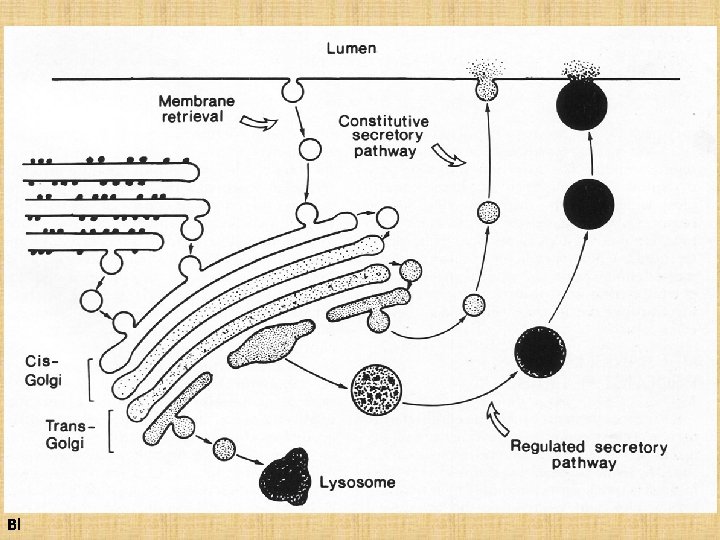
Die Transportvesikeln, die trans-Golgi Seite verlassen, enthalten nur aussortierte Proteine. Jede Vesikel „weißt”, wohin muß sie fahren mit ihrem Cargo. Die Fusion der Vesikelmembran mit dem Zielorgan-Membran ist hochspezifisch (beide Membranen enthalten spezifische Rezeptor-Ligand Paaren). Coo Bestimmte Vesikeln werden zu den Lysosomen gelingen, anderen verschmolzen mit Zellmembran. Die ständig mit Zellmembran fusionierenden Vesikeln (konstitutive Sekretion) liefern neue Membranproteine und Phospholipide zur Membran. Die auf Signale mit der Zellmembran fusionierende Vesikeln (gesteuerte Sekretion) bringen Sekret zum extrazellulären

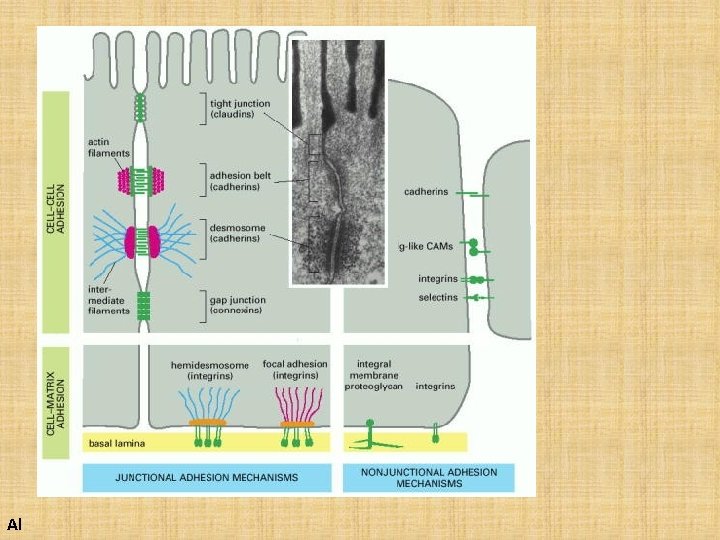
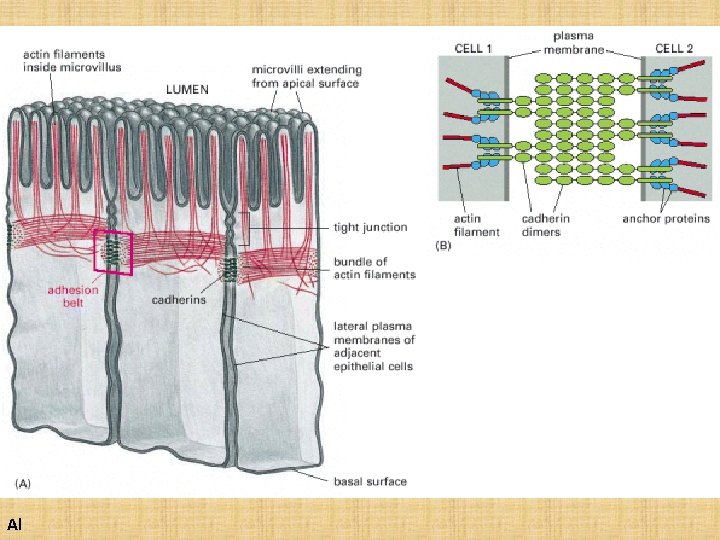
Zelladhäsionsmolekülen und Zell-Zell Kontakte Siehe auch: Histologie, Epithelien

Ud

Al

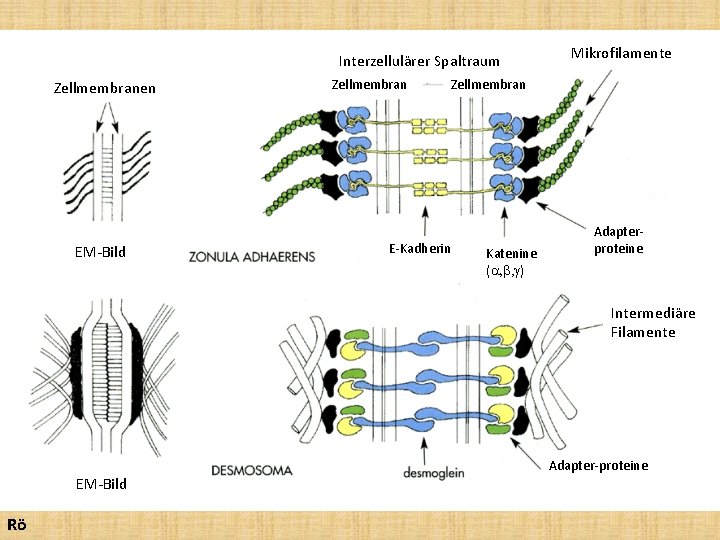
Interzellulärer Spaltraum Zellmembranen EM-Bild Zellmembran Mikrofilamente Zellmembran E-Kadherin Katenine (a, b, g) Adapterproteine Intermediäre Filamente EM-Bild Rö Adapter-proteine

Al

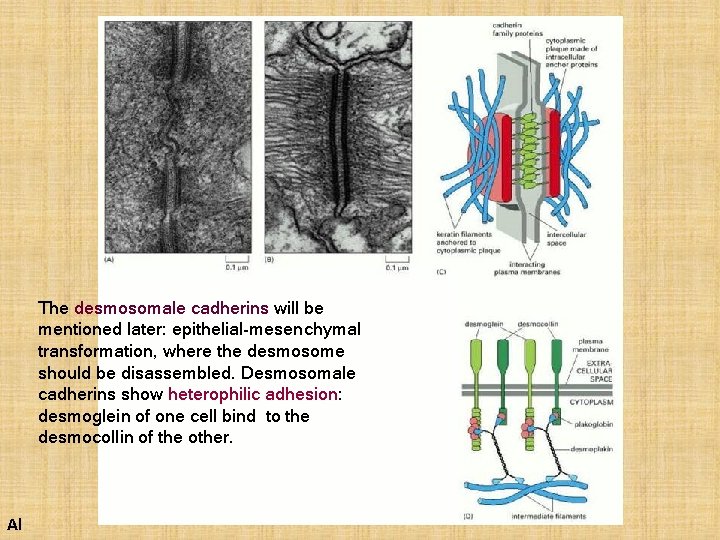
The desmosomale cadherins will be mentioned later: epithelial-mesenchymal transformation, where the desmosome should be disassembled. Desmosomale cadherins show heterophilic adhesion: desmoglein of one cell bind to the desmocollin of the other. Al

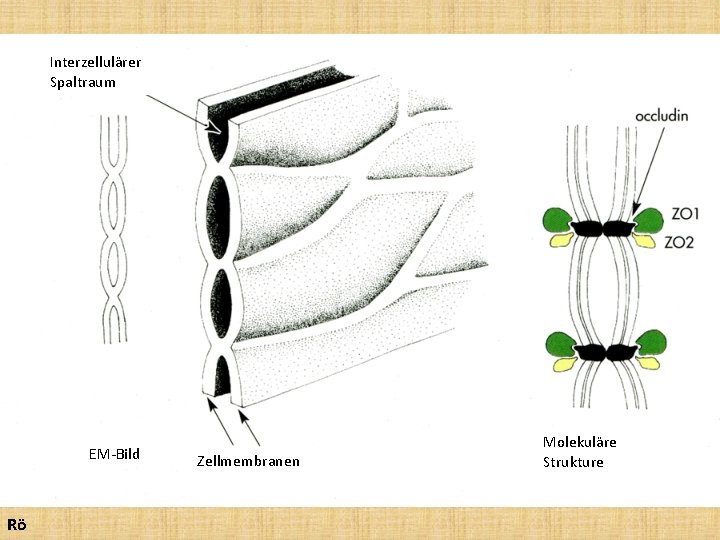
Interzellulärer Spaltraum EM-Bild Rö Zellmembranen Molekuläre Strukture

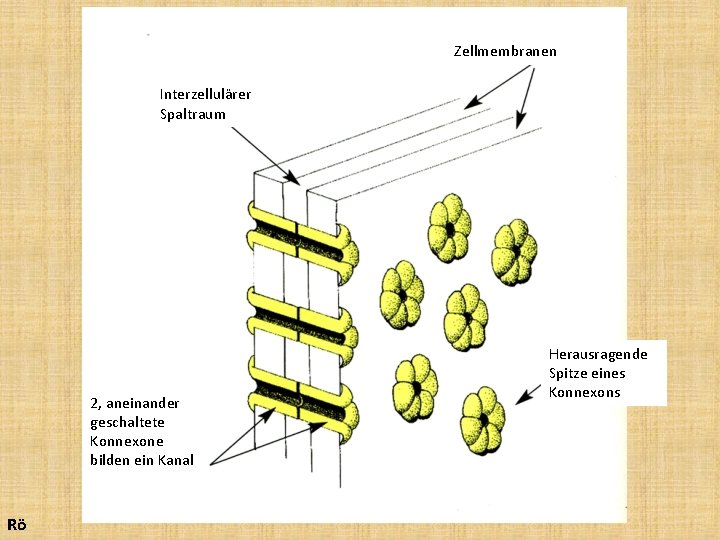
Zellmembranen Interzellulärer Spaltraum 2, aneinander geschaltete Konnexone bilden ein Kanal Rö Herausragende Spitze eines Konnexons

Quellen • Rö: Röhlich, Pál: Szövettan (Histologie), Semmelweis Kiadó, 2006 • Coo: GM Cooper, RE Hausman: The cell: a molecular approach, Sinauer Ass. , 2007 • BL: Bloom and Fawcett: A textbook of Histology, Chapman and Hall, 1994 • Rho: J. Rhodin: Histology: a text and atlas, Oxford Univ Press, 1994 • Al: B. Alberts, et al: Lehrbuch der Molekularen Zellbiologie, Wiley-VCH, 2005 • Ud: J. Ude und M. Koch: Die Zelle, Fischer, Jena 1982
 Petesejt alkotórészei
Petesejt alkotórészei Morgan stanley hungary
Morgan stanley hungary Leadership secrets of attila the hun
Leadership secrets of attila the hun Attila margos antlaşması
Attila margos antlaşması József attila éjszaka versek
József attila éjszaka versek Nav felsővezető
Nav felsővezető Dr simon éva kardiológus sopron
Dr simon éva kardiológus sopron Kocsi attila
Kocsi attila Dr kalas attila
Dr kalas attila Tompos attila
Tompos attila Szakkay attila
Szakkay attila Attila technologies
Attila technologies Papp z attila
Papp z attila Attila the hun leadership style
Attila the hun leadership style Melegh attila
Melegh attila Nke hhk
Nke hhk Somfay attila
Somfay attila Da dove provenivano i germani
Da dove provenivano i germani Piros attila bme
Piros attila bme Szűcs attila végrehajtó
Szűcs attila végrehajtó Dr somfay attila
Dr somfay attila 7m logisztika
7m logisztika Kruppa attila villámvédelem
Kruppa attila villámvédelem Sanglier attila 355 kg
Sanglier attila 355 kg Flagellum dei attila
Flagellum dei attila Dobi attila
Dobi attila Rátai attila
Rátai attila Sztojka attila
Sztojka attila Somfay attila
Somfay attila Attila ilhan poems
Attila ilhan poems Sanglier de turquie
Sanglier de turquie Trochophora
Trochophora Karsztjelenségek
Karsztjelenségek Bene attila
Bene attila Göröngy
Göröngy Cor pulmonale chronicum
Cor pulmonale chronicum