NUTRIGENMI CA Paula Armesto FOOD TRENDS 90 Alimentos














- Slides: 18

NUTRIGENÓMI CA Paula Armesto

FOOD TRENDS 90’ Alimentos SIN 00’ Alimentos CON

ALIMENTOS FUNCIONALES (NUTRACÉUTICOS, PRE/PROBIÓTICOS) Problemas de Salud Pública Ø Japón (1980) – concepto de "Alimentos para Uso Específico en la Salud” Ø EEUU (1996) – FDA obliga a fortificar con ácido fólico los productos derivados de cereales Proyecto Genoma Humano (2001) y Hap. Map (2005): información clave y herramientas NUTRIGENÓMICA Interacción entre la nutrición y el genoma de un individuo (Davis and Hord, 2005)

ESTUDIOS DE ASOCIACION DESCRIPCIÓN DEL FENOTIPO variables cuantitativas: IMC y peso (caracteres métricos) DISEÑO DEL ESTUDIO Cohorte MUESTRA 39. 342 Islandeses, 2. 998 Holandeses, 1. 890 Americanos caucásicos, 1. 160 Afro americanos. SELECCIÓN DE MARCADORES SNPs asociados a la Obesidad (GWAs población danesa y estudios de GIANT consortium) GENOTIPADO Y ANÁLISIS DE DATOS 10 SNPs con GWA significativa para IMC y peso (p<0. 05) OR obtenidos para cada SNP entre 1. 07 y 1. 27 Su efecto sólo explica una pequeña porción de la variación entre individuos en el desarrollo de Obesidad

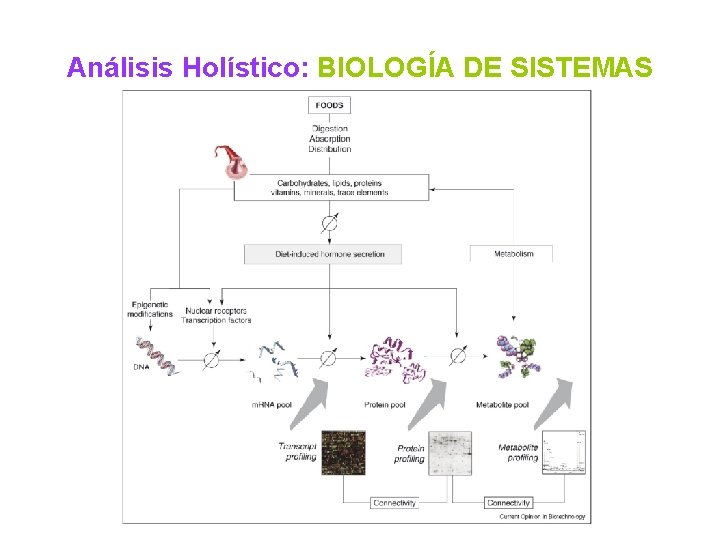
Caracterizar el resto de la respuesta a la dieta ENFOQUES Targeted Approaches/Analysis medida cuantitativa de un metabolito, proteína especifica o transcrito partiendo de una hipótesis concreta Holistic Approaches/Analysis no se selecciona ningún componente a piori, sino que se plantea una intervención nutricional y se estudian los cambios a nivel global = BIOLOGÍA DE SISTEMAS

Análisis Holístico: BIOLOGÍA DE SISTEMAS

1. NUTRITRANSCRIPTÓMICA MUESTRAS biopsias de tejidos, líneas celulares, células sanguíneas (menos eritrocitos) TÉCNICAS MICROARRAYS: perfil m. RNA vs perfil basal RETROTRANSCRIPCIÓN PCR A TIEMPO REAL: quantificación de la sub/sobre expresión

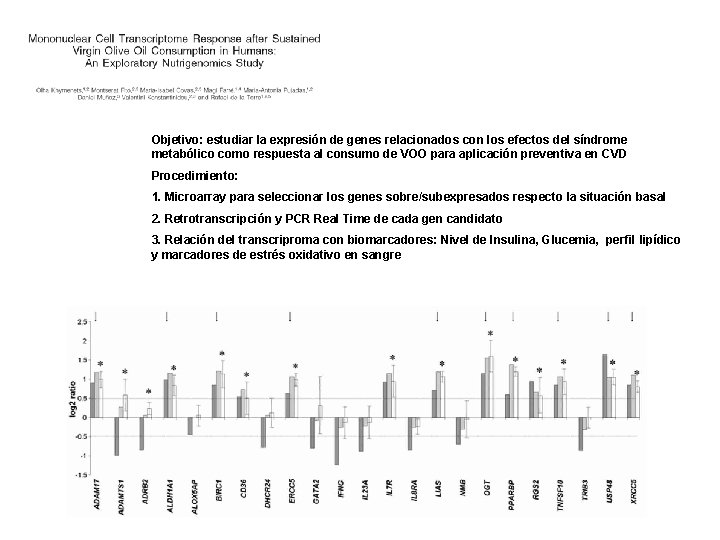
Objetivo: estudiar la expresión de genes relacionados con los efectos del síndrome metabólico como respuesta al consumo de VOO para aplicación preventiva en CVD Procedimiento: 1. Microarray para seleccionar los genes sobre/subexpresados respecto la situación basal 2. Retrotranscripción y PCR Real Time de cada gen candidato 3. Relación del transcriproma con biomarcadores: Nivel de Insulina, Glucemia, perfil lipídico y marcadores de estrés oxidativo en sangre

2. NUTRIPROTEÓMICA PAPEL CENTRAL DE LAS PROTEINAS EN LA NUTRICIÓN Transportadores (absorción intestinal, distribución y transporte en sangre) Enzimas (metabolismo en tejidos) Proteinas de intercambio (excrecion en el túbulo renal) MUESTRAS: biopsias de tejidos, líneas celulares, fluidos (sangre, orina) TÉCNICAS PROTEIN MAPPING/PROFILES : detección de cambios en variedad y abundancia relativa Separación mediante técnicas de Electroforesis (2 DE) o Cromatografía Identificación por técnicas de Espectrometría de Masas PROTEOMICA FUNCIONAL: Interacciones entre proteínas, con metabolitos o ingredientes bioactivos Métodos de interactómica Cambios postraduccionales Arrays proteicos RECAMBIO PROTEICO Seguimiento de isótopos estables para la determinación de la tasa de recambio proteico (SILAC)

APLICACIONES • Estudios de patofisiología en estados de defícit nutricional Proteoma renal de ratón sometido a deprivación prolongada de K+ (Go t al. 2005) Identificación por MS de 33 proteínas diferentes al proteoma basal, parte enzimas involucrados en la alcalosis metabólica • Efecto de suplementos dietéticos en individuos enfermos Caracterización del perfil proteico del plasma de pacientes en hemodiálisis. Normalización de los niveles de algunos polipéptidos por suplementación con vitamina C (Weissinger, 2006) • Efecto de suplementos dietéticos en individuos sanos Caracterización del perfil proteico del plasma de individuos con suplementación de atocoferol. Obtención de niveles incrementados de Apo A 1 (Aldred et al, 2006) • Efecto del exceso dietético en individuos sanos


3. NUTRIMETABOLÓMICA PUNTO FINAL DE LA INTERACCIÓN NUTRICIONAL Genoma del individuo x Genoma de su Microbiota intestinal + factores ambientales (alimentos, hábitos, estilo de vida…) MUESTRA Orina, sangre, CO 2 exhalado TÉCNICAS CONCENTRACIÓN FINAL DE UN METABOLITO Resonancia Magnética Nuclear (NMR) Para generar perfiles metabólicos de matrices complejas CAMBIO EN SU CINÉTICA Espectrometría de Masas para medir incrementos mediante Isótopos estables

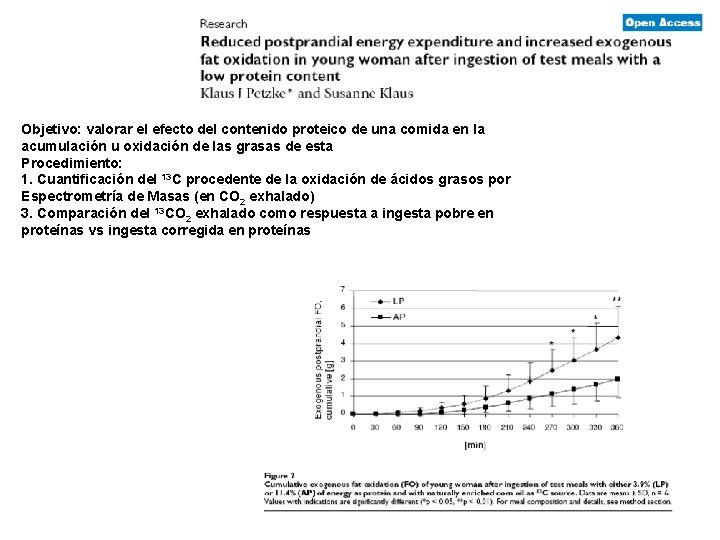
Objetivo: valorar el efecto del contenido proteico de una comida en la acumulación u oxidación de las grasas de esta Procedimiento: 1. Cuantificación del 13 C procedente de la oxidación de ácidos grasos por Espectrometría de Masas (en CO 2 exhalado) 3. Comparación del 13 CO 2 exhalado como respuesta a ingesta pobre en proteínas vs ingesta corregida en proteínas

IMPORTANCIA DEL MICROBIOMA DIETA > selección de microflora intestinal > -biodisponibilidad de nutrientes -Interacción de rutas metabólicas no relacionadas APLICACIONES Caracterización de interacciones microbiomametabolismo huésped y mejora de alimentos pre/probióticos para grupos de población concretos

LIMITACIONES ACTUALES - Obtención de muestra “viva” limitada por restricciones éticas y cuantitativas. - No se pueden analizar tejidos vitales humanos - ¿Cuándo tomar las muestras de transcritos, proteinas y metabolitos? -Baja reproducibilidad entre ensayos intra e Inter laboratorio. Fuentes de variabilidad : muestra biológica + error humano/técnico -Intervención dietética atenuada por homeostasis -Interpretación de datos Difícil integración de la información a todos los niveles

NECESIDADES Mejoras tecnológicas para los ANALISIS TEMPORALES-ESPACIALES: - Sistemas miniaturizados para aprovechar las muestras vivas escasas - Dianas mejoradas que aporten información lineal de la expresión - Mejoras software y procesadores de datos ESTANDARIZACIÓN METODOLÓGICA Y DE DATOS: MIAME, MIAPE, MIMIx, SMRS PLATAFORMAS ACTIVAS EN NUTRIGENÓMICA • • • NCMHD Center of excellence in nutritional genomics (Univ. California, Davis) Nu. GO The European Nutrigenomics Organisation GOLDN project (Genetics of Lipid Lowering Drugs and Diet Network)

ü Caracterizar enfermedades relacionadas con la nutrición DIAGNÓSTICO PREVENCIÓN (dieta personalizada) ü Justificar el uso de Alimentos funcionales

-Xuewu Zhang, Yeeleng Yap, Dong Wei, Gu Chen, Feng Chen. Novel Omics Technologies In Nutrition Research. Biotechnology advances, 2007 - Florian J. Schweigert. Nutritional Proteomics: Methods and Concepts in Nutritional Science. Annals of Nutrition and Metabolism, 2007 - Serge Rezzi, Ziad Ramadan, Laurent B. Fay, and Sunil Kochhar. Nutritional Metabonomics: Applications and Perspectives. Journal Of Proteome Research, 2006
 Unit 2 food food food
Unit 2 food food food Grazing food chain diagram
Grazing food chain diagram Food in the 1960s
Food in the 1960s Monosacaridos comida
Monosacaridos comida Zona de peligro de los alimentos
Zona de peligro de los alimentos Tecnologa en alimentos
Tecnologa en alimentos Alimento construtores
Alimento construtores Alimentos desecados
Alimentos desecados Eduardo kohler
Eduardo kohler Trabalho sobre alimentos transgênicos
Trabalho sobre alimentos transgênicos Ejemplos de mermas en alimentos
Ejemplos de mermas en alimentos Alimentos energeticos extras
Alimentos energeticos extras Componentes principales de los alimentos
Componentes principales de los alimentos 10 alimentos que contienen biomoléculas
10 alimentos que contienen biomoléculas Trabalenguas paca la vaca
Trabalenguas paca la vaca Sesión demostrativa de alimentos minsa
Sesión demostrativa de alimentos minsa Alimentos que contengan probióticos
Alimentos que contengan probióticos Comida com 9 letras
Comida com 9 letras Bpm en alimentos
Bpm en alimentos