La struttura chimica del DNA Componenti chimici degli


















- Slides: 43

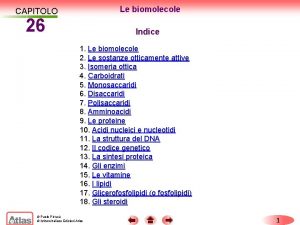
La struttura chimica del DNA

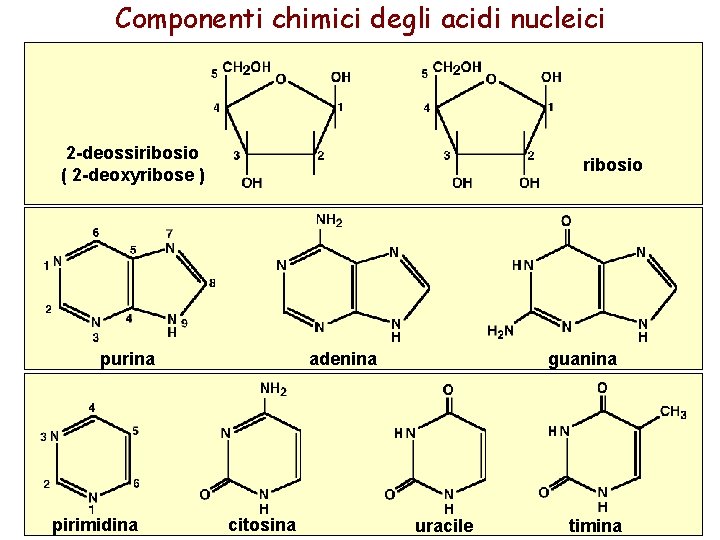
Componenti chimici degli acidi nucleici 2 -deossiribosio ( 2 -deoxyribose ) ribosio purina pirimidina adenina citosina guanina uracile timina

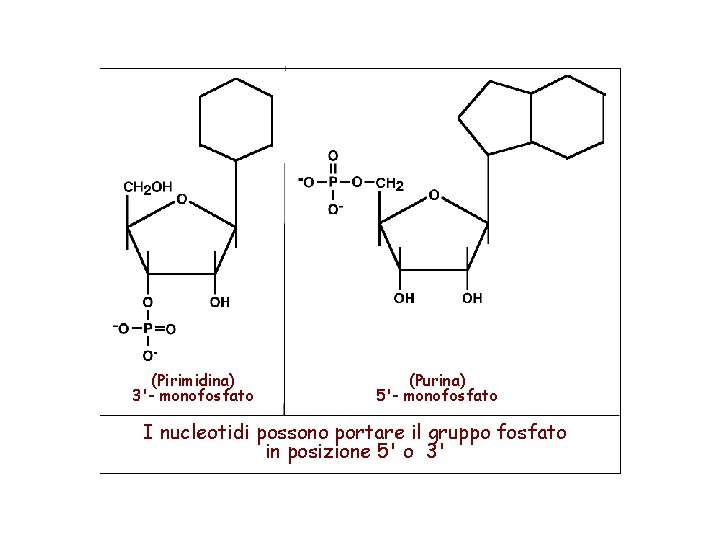
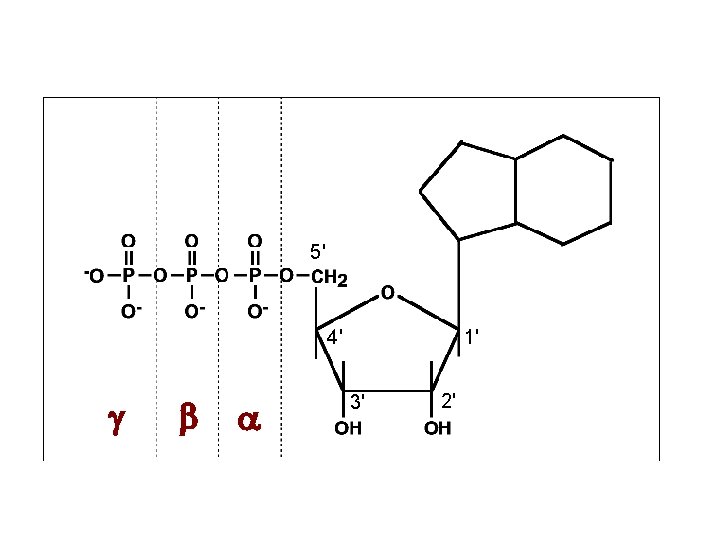
(Pirimidina) 3'- monofosfato (Purina) 5'- monofosfato I nucleotidi possono portare il gruppo fosfato in posizione 5' o 3'


5' 4' g b a 1' 3' 2'

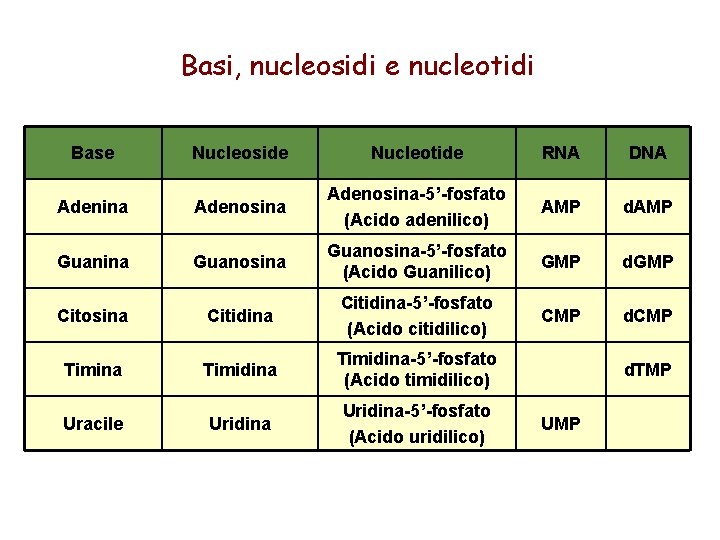
Basi, nucleosidi e nucleotidi Base Nucleoside Nucleotide RNA DNA Adenina Adenosina-5’-fosfato (Acido adenilico) AMP d. AMP Guanina Guanosina-5’-fosfato (Acido Guanilico) GMP d. GMP Citosina Citidina-5’-fosfato (Acido citidilico) CMP d. CMP Timina Timidina-5’-fosfato (Acido timidilico) Uracile Uridina-5’-fosfato (Acido uridilico) d. TMP UMP


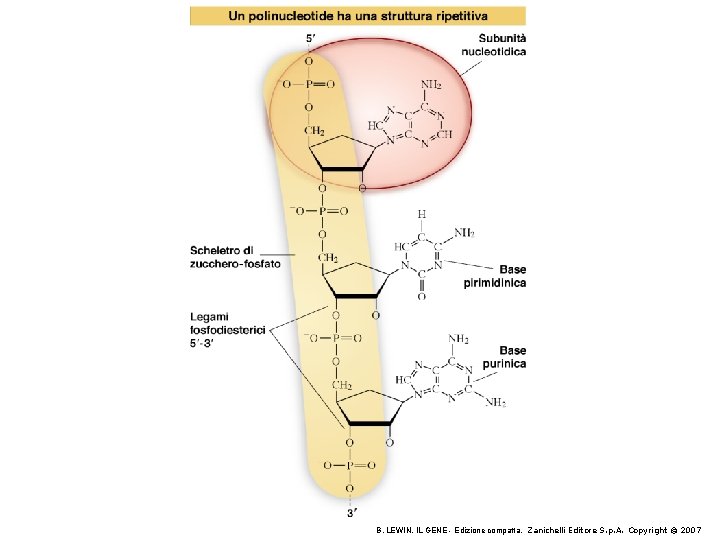
B. LEWIN, IL GENE - Edizione compatta, Zanichelli Editore S. p. A. Copyright © 2007

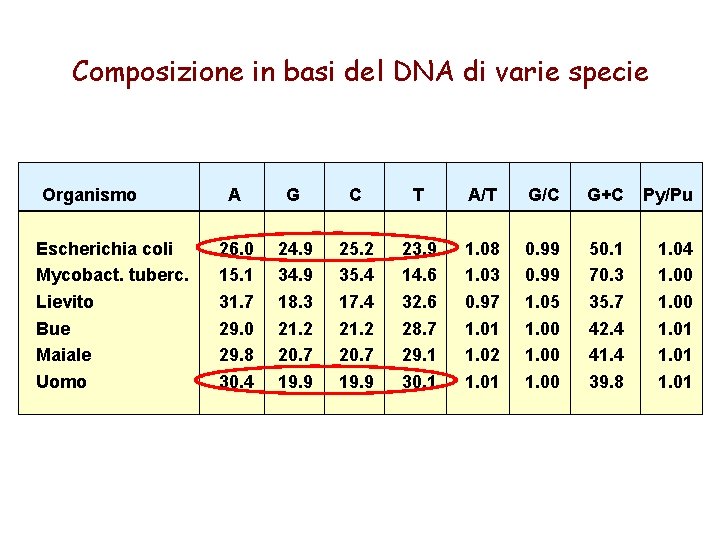
Erwin Chargaff Per primo misurò accuratamente la percentuale dei quattro nucleotidi nel DNA

Composizione in basi del DNA di varie specie Organismo Escherichia coli Mycobact. tuberc. Lievito Bue Maiale Uomo A G C T A/T G/C G+C Py/Pu 26. 0 15. 1 31. 7 29. 0 29. 8 30. 4 24. 9 34. 9 18. 3 21. 2 20. 7 19. 9 25. 2 35. 4 17. 4 21. 2 20. 7 19. 9 23. 9 14. 6 32. 6 28. 7 29. 1 30. 1 1. 08 1. 03 0. 97 1. 01 1. 02 1. 01 0. 99 1. 05 1. 00 50. 1 70. 3 35. 7 42. 4 41. 4 39. 8 1. 04 1. 00 1. 01

La struttura fisica del DNA


Protagonisti nella scoperta della struttura del DNA Linus Pauling (1901 - 1994) Maurice Wilkins (1916 - 2004) Francis Crick (1916 - 2004) Rosalind Franklin (1920 - 1958) James Watson (1928 )

Dati sulla struttura del DNA • Le percentuali G=C e A=T • Legami fosfodiesterici tra nucleotidi

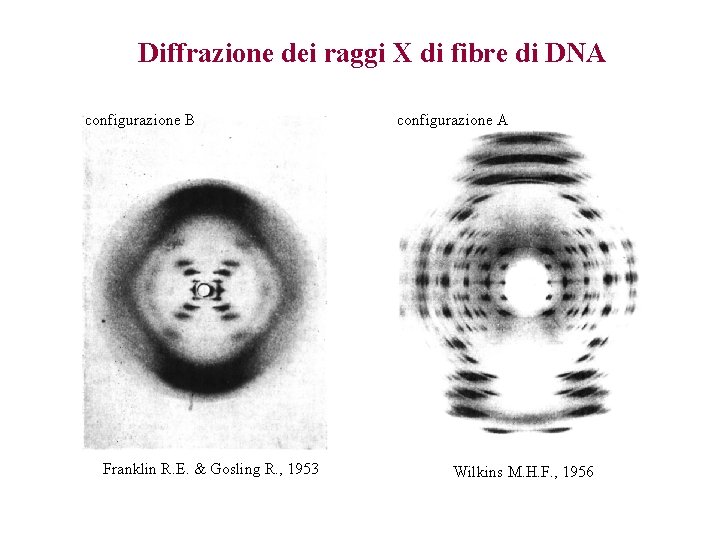
Diffrazione dei raggi X di fibre di DNA configurazione B Franklin R. E. & Gosling R. , 1953 configurazione A Wilkins M. H. F. , 1956

Figura riportata nella pubblicazione originale di Watson e Crick Nature, Aprile 1953


Lewin, IL GENE VIII, Zanichelli editore S. p. A. Copyright © 2006

Lewin, IL GENE VIII, Zanichelli editore S. p. A. Copyright © 2006

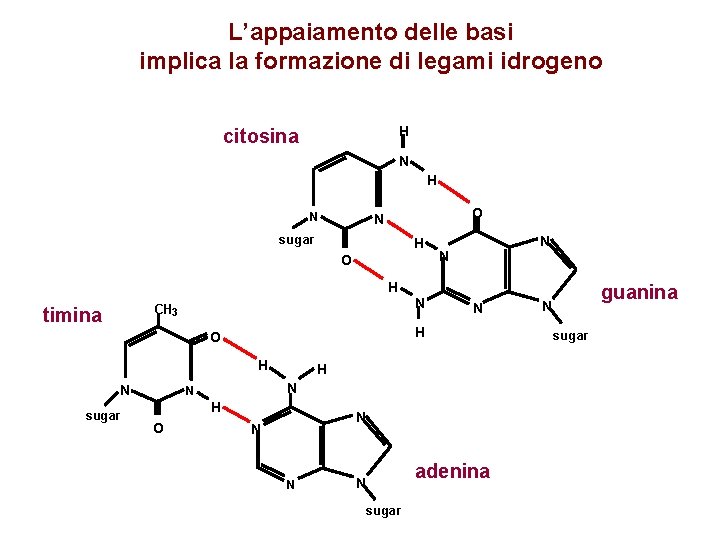
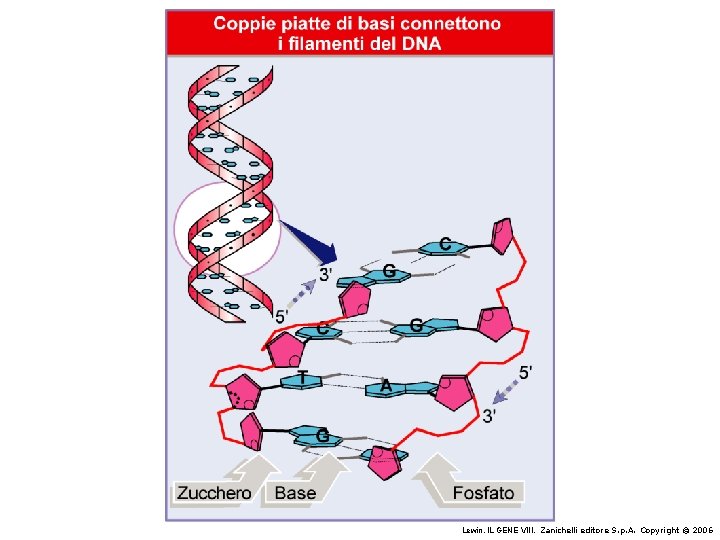
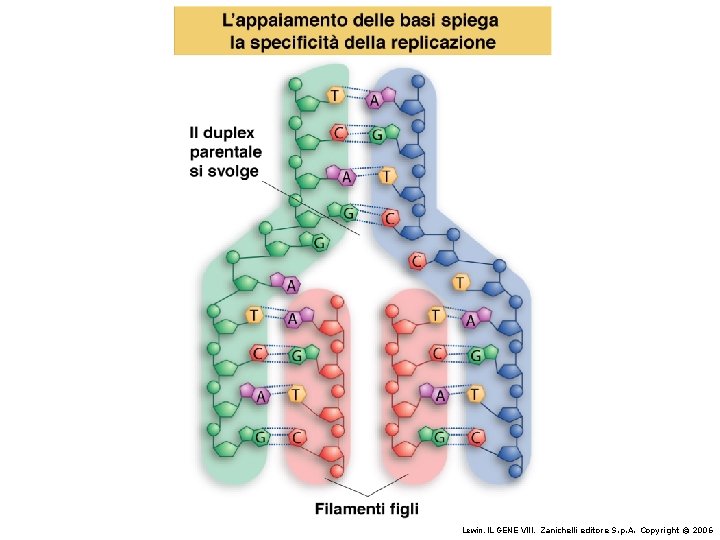
L’appaiamento delle basi implica la formazione di legami idrogeno H citosina N H N O N sugar H O H CH 3 timina H sugar N H N N H O N N N adenina N sugar guanina N sugar

Lewin, IL GENE VIII, Zanichelli editore S. p. A. Copyright © 2006

20 Lewin, IL GENE VIII, Zanichelli editore S. p. A. Copyright © 2006

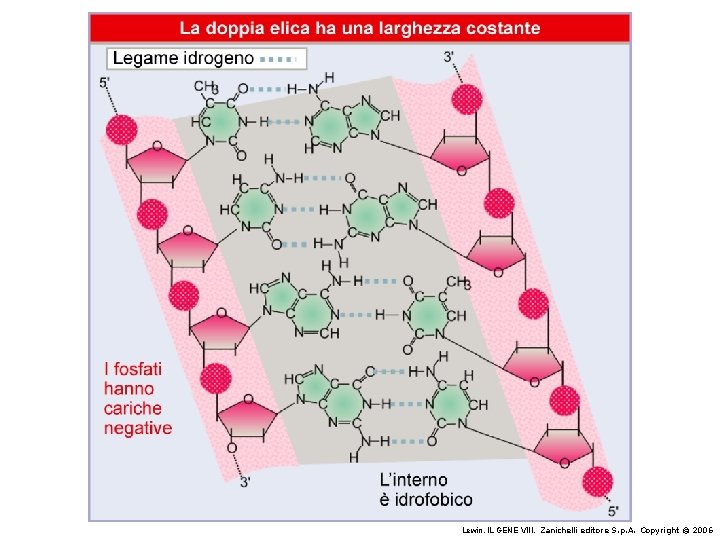
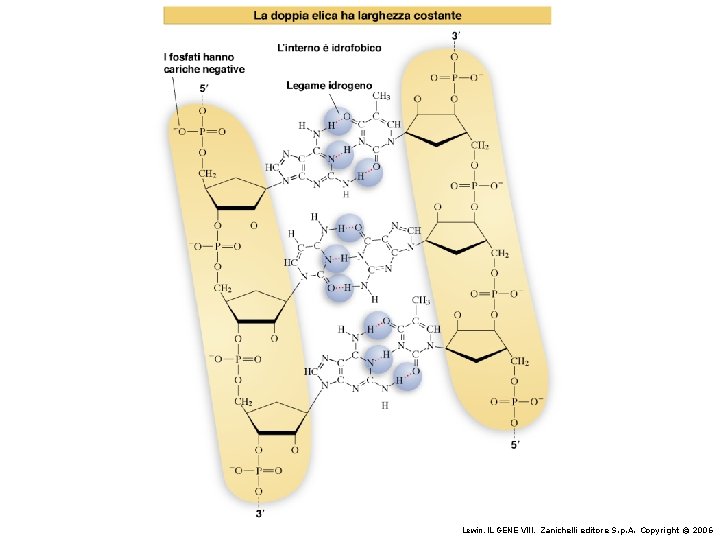
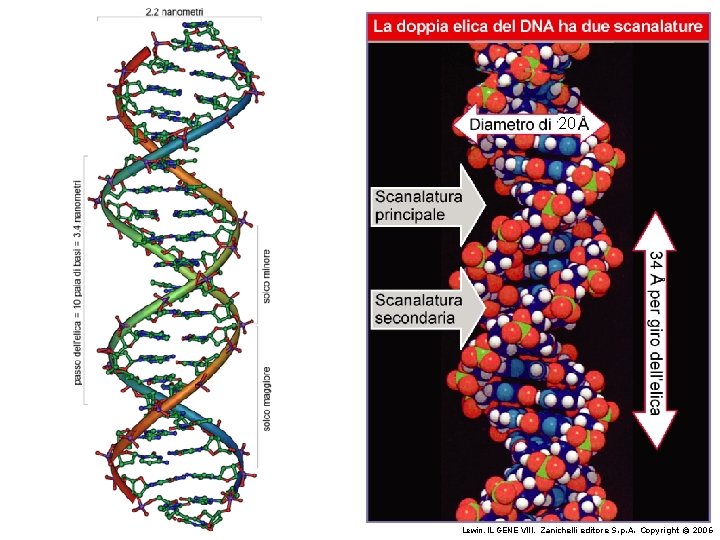
La struttura del DNA (B) • Il DNA ha una forma ad elica regolare, diametro 20Å, passo 34Å • Legami idrogeno tra le basi • L'impilamento delle basi è determinato da interazioni idrofobiche • Ogni coppia è ruotata di 36° • Solchi maggiore (22Å) e minore (12Å) • Avvolgimento in senso orario (elica destrorsa)


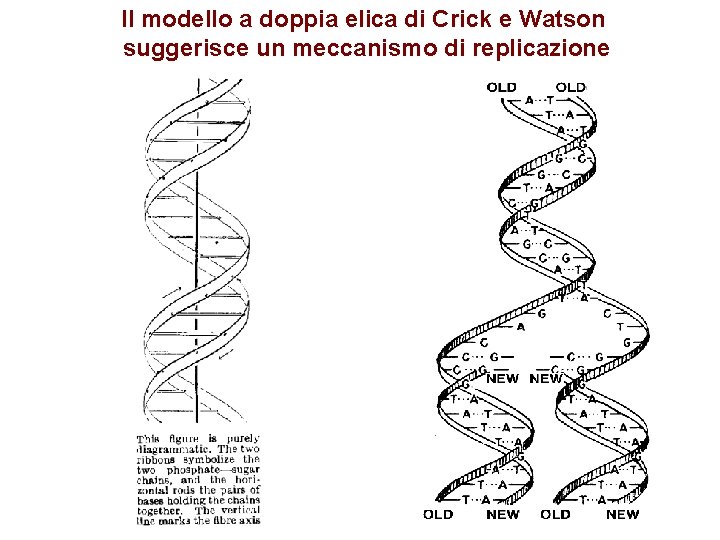
It has not escaped our notice that the specific pairing we have postulated immediately suggests a possible copying mechanism for the genetic material.

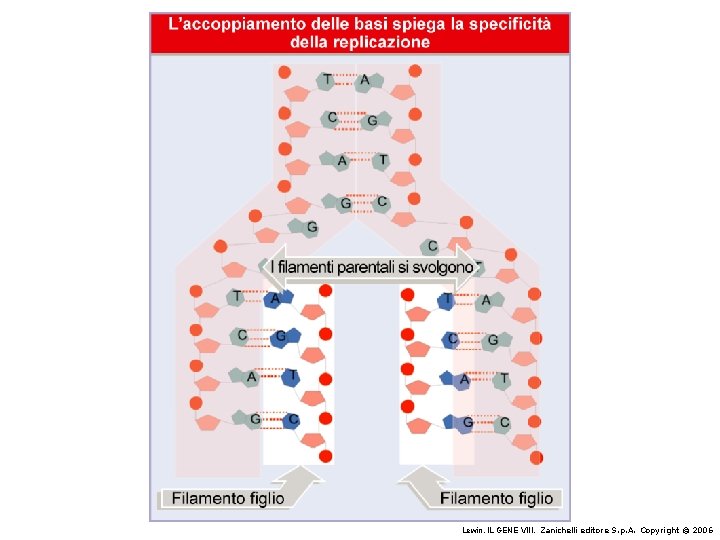
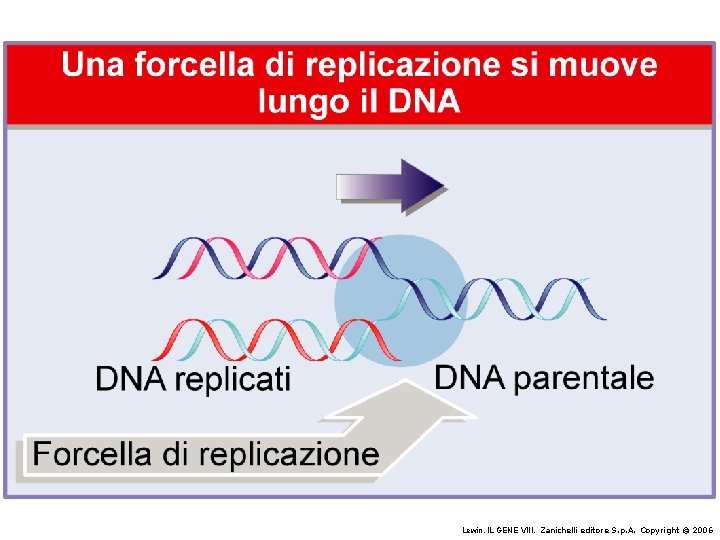
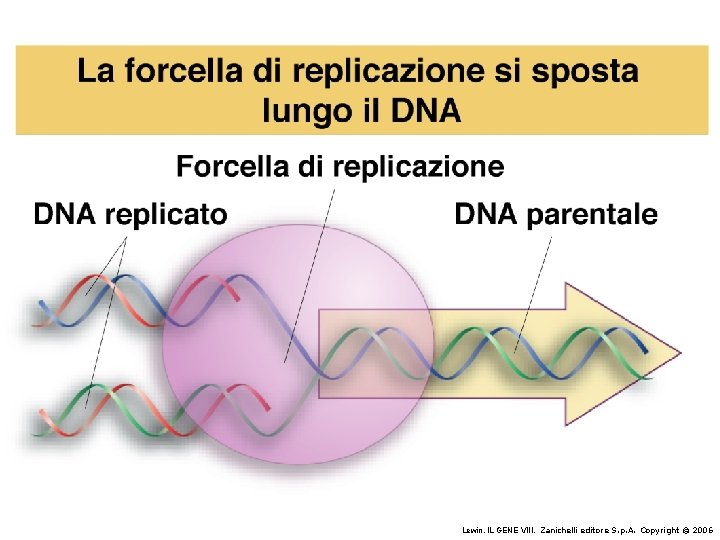
Il modello a doppia elica di Crick e Watson suggerisce un meccanismo di replicazione

Lewin, IL GENE VIII, Zanichelli editore S. p. A. Copyright © 2006

Lewin, IL GENE VIII, Zanichelli editore S. p. A. Copyright © 2006

Lewin, IL GENE VIII, Zanichelli editore S. p. A. Copyright © 2006

Lewin, IL GENE VIII, Zanichelli editore S. p. A. Copyright © 2006

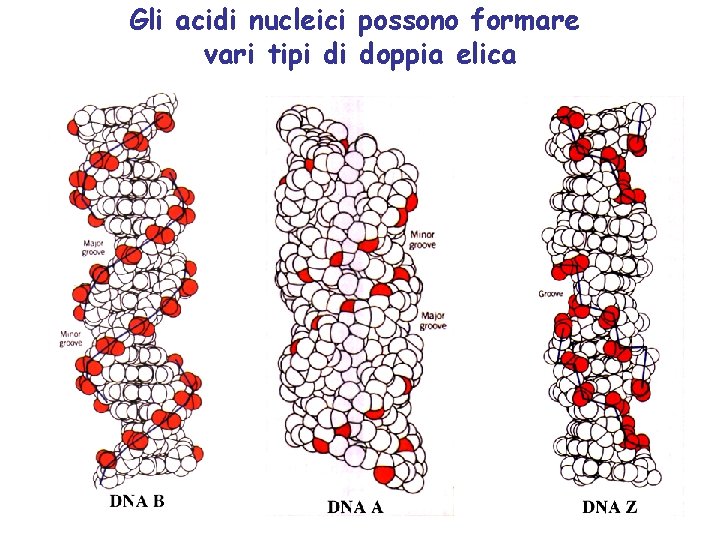
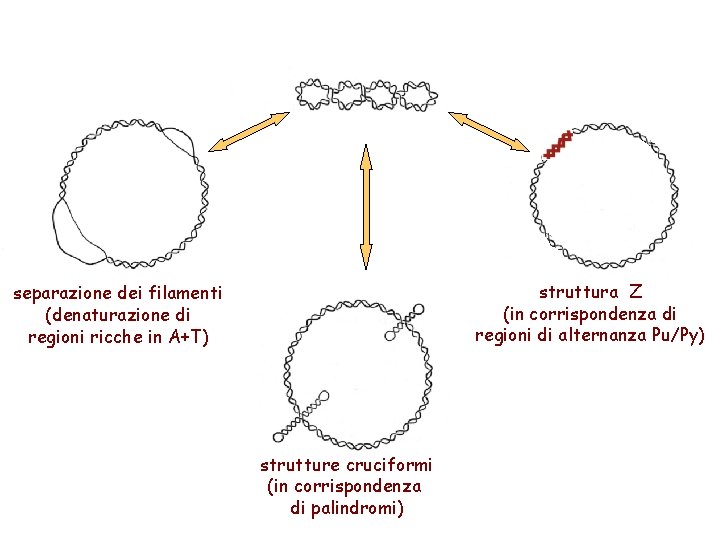
Strutture alternative del DNA

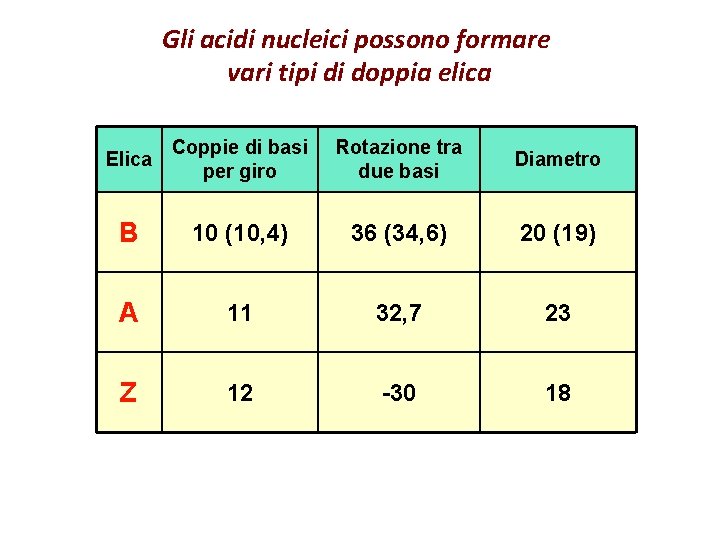
Gli acidi nucleici possono formare vari tipi di doppia elica


Gli acidi nucleici possono formare vari tipi di doppia elica Elica Coppie di basi per giro Rotazione tra due basi Diametro B 10 (10, 4) 36 (34, 6) 20 (19) A 11 32, 7 23 Z 12 -30 18

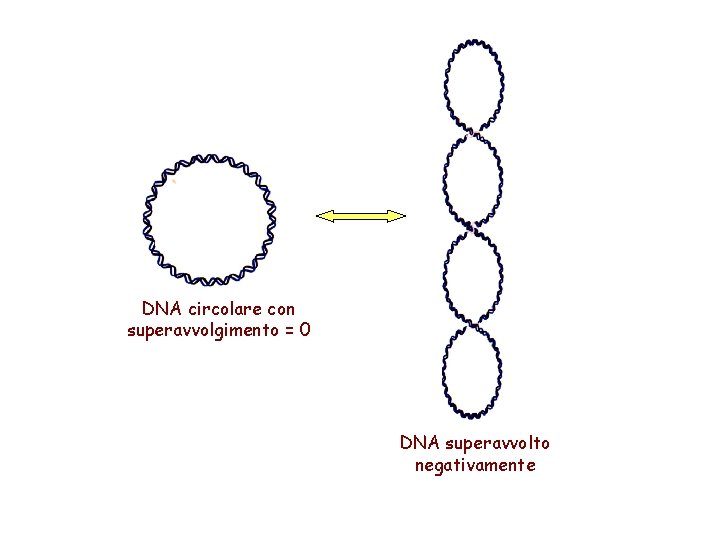
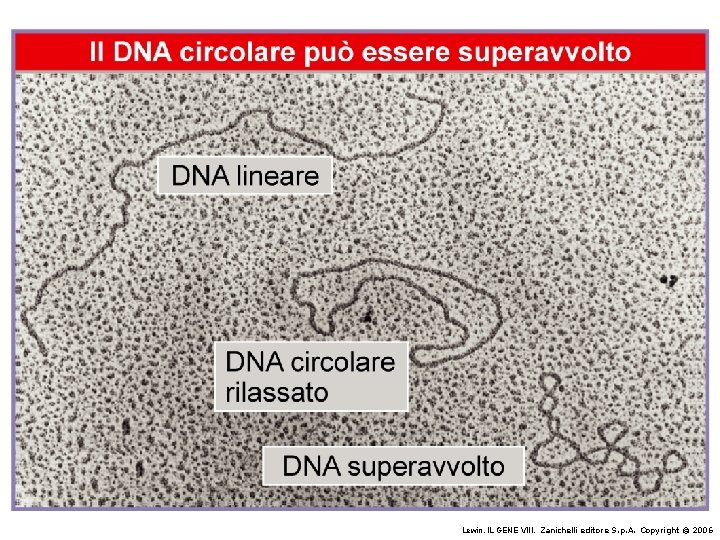
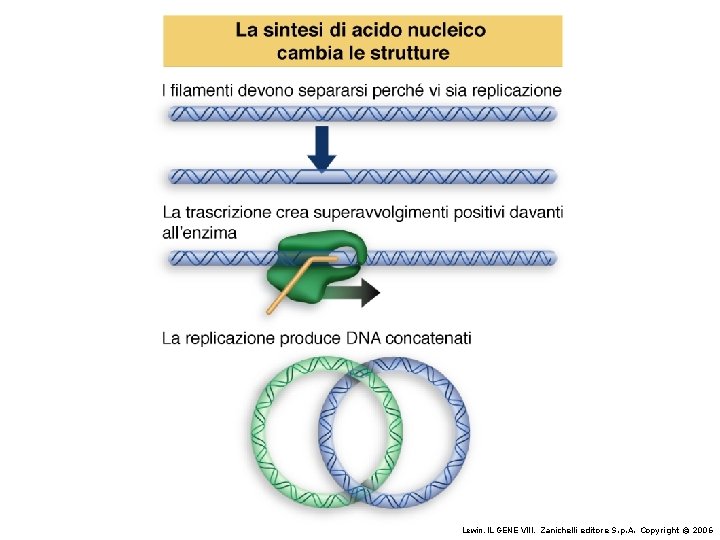
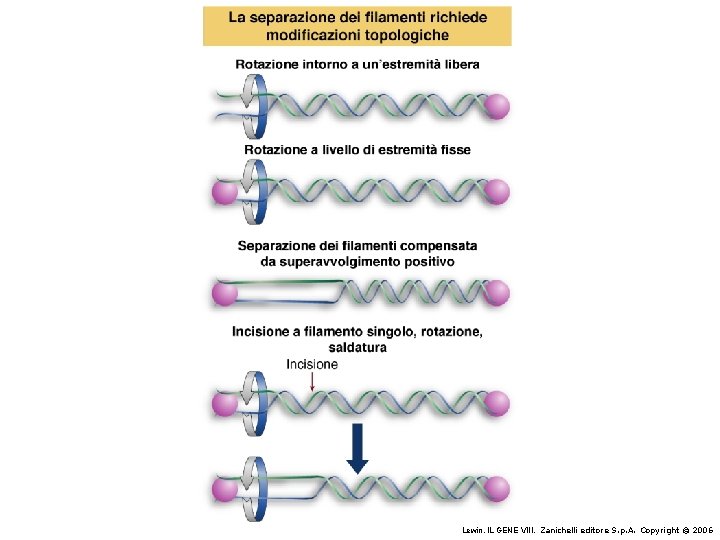
Strutture superiori del DNA

DNA circolare con superavvolgimento = 0 DNA superavvolto negativamente

Lewin, IL GENE VIII, Zanichelli editore S. p. A. Copyright © 2006

Lewin, IL GENE VIII, Zanichelli editore S. p. A. Copyright © 2006

Lewin, IL GENE VIII, Zanichelli editore S. p. A. Copyright © 2006

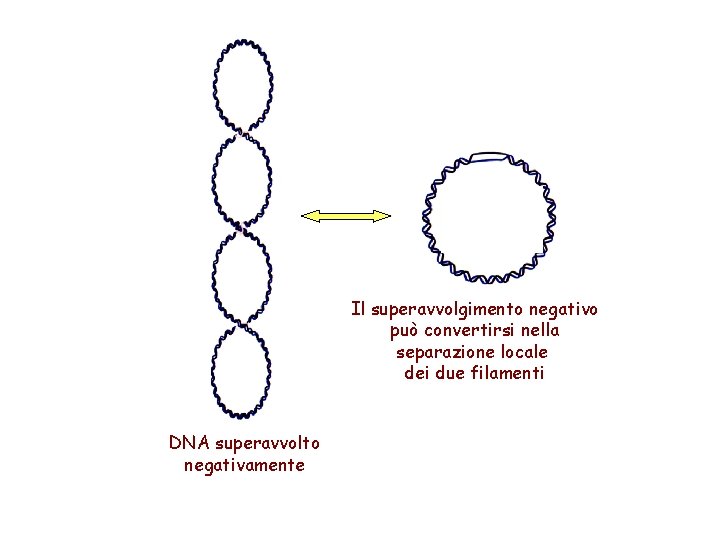
Il superavvolgimento negativo può convertirsi nella separazione locale dei due filamenti DNA superavvolto negativamente

struttura Z (in corrispondenza di regioni di alternanza Pu/Py) separazione dei filamenti (denaturazione di regioni ricche in A+T) strutture cruciformi (in corrispondenza di palindromi)

Superavvolgimento del DNA Positivo = DNA superspiralizzato Negativo = DNA sottospiralizzato

 Componenti del sangue
Componenti del sangue Struttura dna
Struttura dna Struttura del dna
Struttura del dna Fisica che studia il moto dei corpi
Fisica che studia il moto dei corpi I componenti principali di un computer
I componenti principali di un computer Analisi delle componenti principali
Analisi delle componenti principali Componenti fortemente connesse
Componenti fortemente connesse Prima legge di kirchhoff
Prima legge di kirchhoff Green village cittadella
Green village cittadella Definizione agenti chimici
Definizione agenti chimici Mappa francesco petrarca
Mappa francesco petrarca Prostaglandine vasodilatazione
Prostaglandine vasodilatazione Nomenclatura mappa concettuale
Nomenclatura mappa concettuale Nomenclatura mappa concettuale
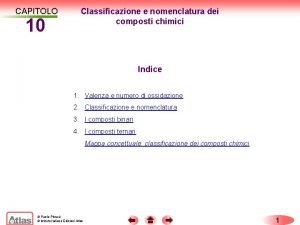
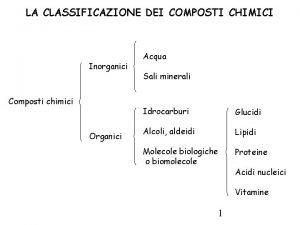
Nomenclatura mappa concettuale Classificazione dei composti chimici mappa concettuale
Classificazione dei composti chimici mappa concettuale Mappa concettuale composti binari e ternari
Mappa concettuale composti binari e ternari Messaggeri chimici
Messaggeri chimici Composti binari schema
Composti binari schema Soluzioni chimica capitolo 13
Soluzioni chimica capitolo 13 Filamenti dna complementari
Filamenti dna complementari Legami chimici
Legami chimici I pericoli fisici sono provocati da
I pericoli fisici sono provocati da Legame covalente puro
Legame covalente puro Forze di london esempi
Forze di london esempi Legami chimici schema riassuntivo
Legami chimici schema riassuntivo Composti chimici inorganici
Composti chimici inorganici Classificazione composti inorganici
Classificazione composti inorganici Legami primari
Legami primari Replication fork
Replication fork Bioflix activity dna replication dna replication diagram
Bioflix activity dna replication dna replication diagram Coding dna and non coding dna
Coding dna and non coding dna Replication process
Replication process Dna and genes chapter 11
Dna and genes chapter 11 Negano i postulati del matematico degli elementi
Negano i postulati del matematico degli elementi Termini primitivi e assiomi
Termini primitivi e assiomi La regolazione degli affetti e la riparazione del sé
La regolazione degli affetti e la riparazione del sé Murice porpora
Murice porpora Tema argomentativo struttura
Tema argomentativo struttura Scopo del testo narrativo
Scopo del testo narrativo Cos'è il testo teatrale
Cos'è il testo teatrale Prolessi
Prolessi La struttura del sole
La struttura del sole Intelligenze motrici
Intelligenze motrici Xilema e floema
Xilema e floema