Identificacin y anlisis funcional de micro RNAs desregulados
















- Slides: 36

Identificación y análisis funcional de micro. RNAs desregulados en cáncer mama Dr. César López Camarillo Laboratorio de Oncogenómica y Proteómica del Cáncer genomicas@yahoo. com. mx POSGRADO EN CIENCIAS GENÓMICAS

secuencia del genoma humano se conoce desde Lafebrero de 2001 James Watson 3, 200 millones de bases (3200 Mb) Predice unos 20, 000 - 25, 000 genes (1. 5% genoma !) Craig Venter 70% está compuesto por ADN extra-génico no codificante (DNA basura - micro. RNAs)


Examples of People Who Have had Their Genomes Sequenced Jim Watson James Watson Craig Ventor Craig Venter From: www. gencia. com sciencewithmoxie. blogspot. com. au/2010_11_01_archive. html Ozzy Osbourne

El Dogma Central de la Biología Molecular y los “omas” Gen-oma (DNA) ACGTTATGAGACGGATTAGGACAAACTATAA AAATATTAGCCGATGATCACCCAGGATTTTT Transcript-oma (m. RNA) Micro. RN-oma (mi. RNAs) Prote-oma (Proteínas)

micro. RNAs: nuevos reguladores de la expresión genética Transcritos RNAs no codificantes RNAm que codifican proteínas RNAs reguladores de la expresión génica si. RNAs, lnc. RNA, micro. RNAs de mantenimiento t. RNA, r. RNA, sn. RNA sno. RNAs Micro. RNAs: RNA pequeños (21 -25 nt) que regulan negativamente la expresión genética. Descritos en 1993 por Victor Ambros en C. elegans. Un micro. RNA puede modular hasta mas de 100 transcritos y proteínas diferentes

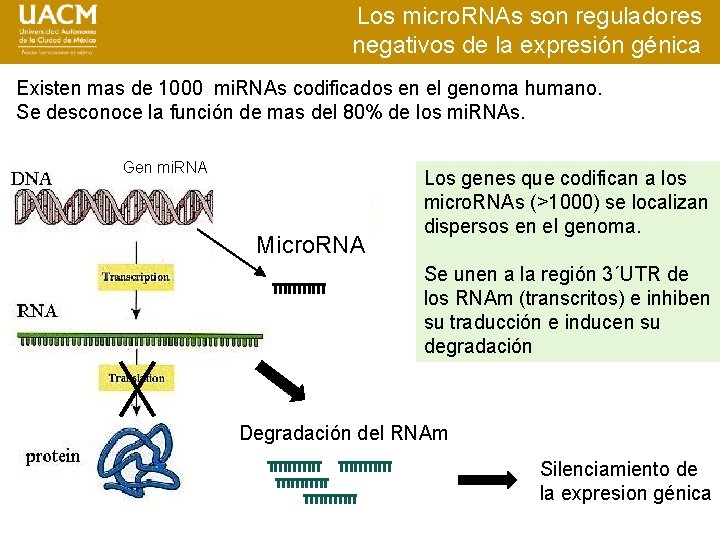
Los micro. RNAs son reguladores negativos de la expresión génica Existen mas de 1000 mi. RNAs codificados en el genoma humano. Se desconoce la función de mas del 80% de los mi. RNAs. Gen mi. RNA mi Micro. RNA Los genes que codifican a los micro. RNAs (>1000) se localizan dispersos en el genoma. Se unen a la región 3´UTR de los RNAm (transcritos) e inhiben su traducción e inducen su degradación Degradación del RNAm Silenciamiento de la expresion génica

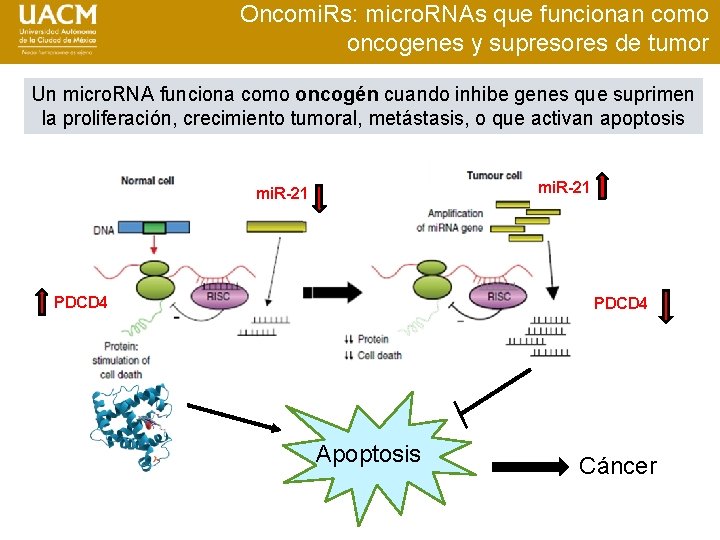
Oncomi. Rs: micro. RNAs que funcionan como oncogenes y supresores de tumor Un micro. RNA funciona como oncogén cuando inhibe genes que suprimen la proliferación, crecimiento tumoral, metástasis, o que activan apoptosis mi. R-21 PDCD 4 Apoptosis Cáncer

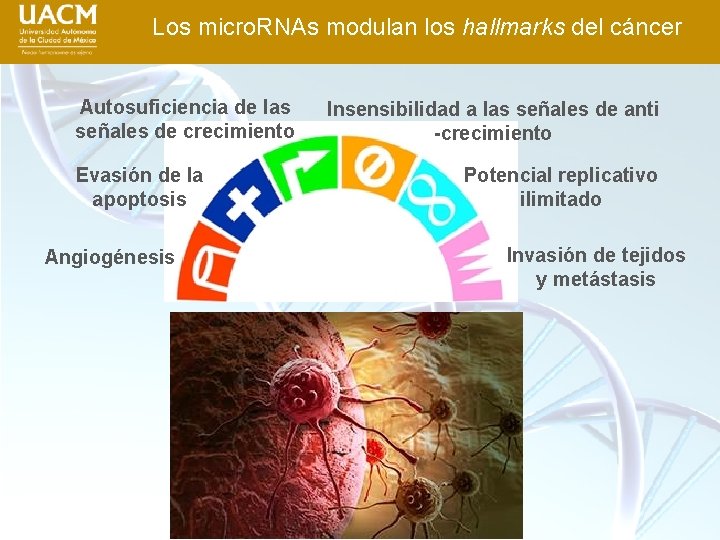
Los micro. RNAs modulan los hallmarks del cáncer Autosuficiencia de las señales de crecimiento Evasión de la apoptosis Angiogénesis Insensibilidad a las señales de anti -crecimiento Potencial replicativo ilimitado Invasión de tejidos y metástasis

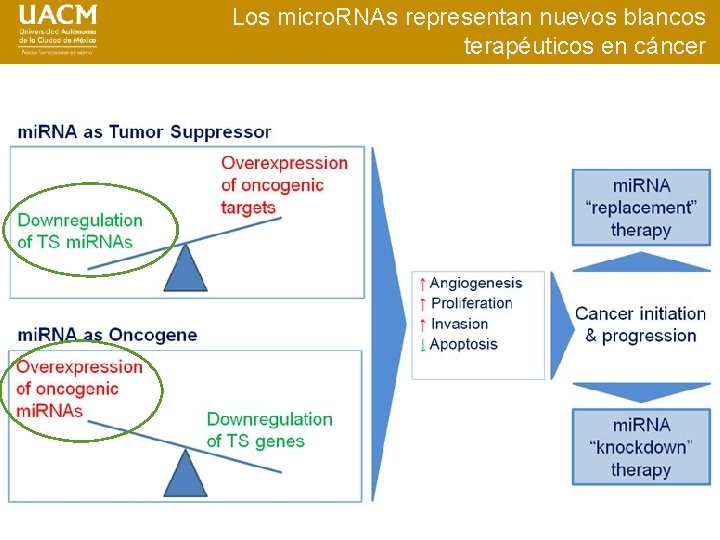
Los micro. RNAs representan nuevos blancos terapéuticos en cáncer

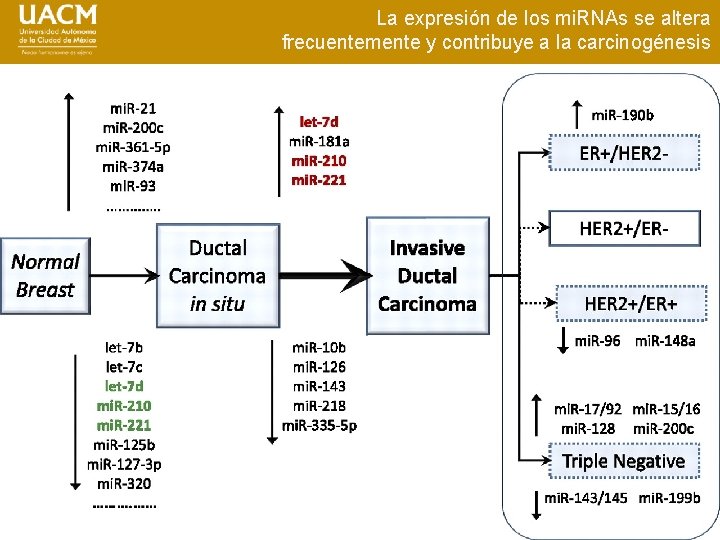
La expresión de los mi. RNAs se altera frecuentemente y contribuye a la carcinogénesis

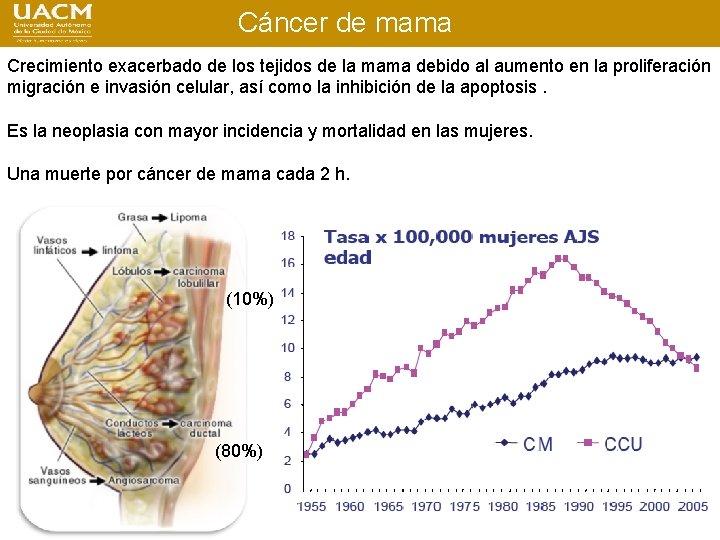
Cáncer de mama Crecimiento exacerbado de los tejidos de la mama debido al aumento en la proliferación migración e invasión celular, así como la inhibición de la apoptosis. Es la neoplasia con mayor incidencia y mortalidad en las mujeres. Una muerte por cáncer de mama cada 2 h. (10%) (80%)

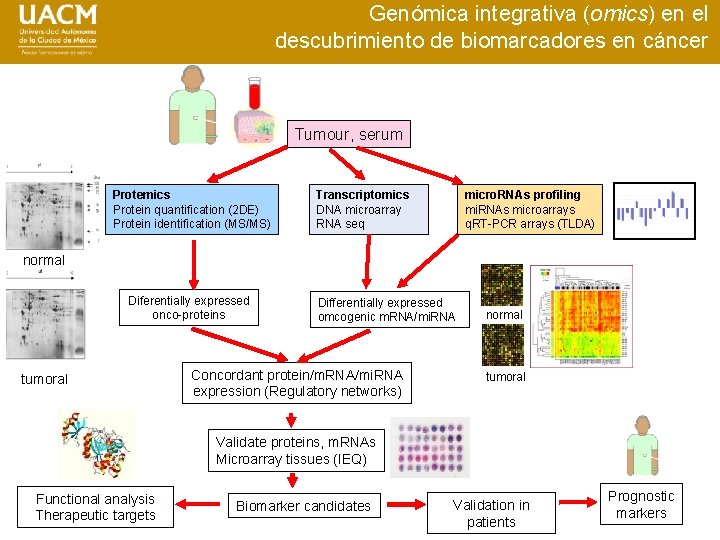
Genómica integrativa (omics) en el descubrimiento de biomarcadores en cáncer Tumour, serum Protemics Protein quantification (2 DE) Protein identification (MS/MS) micro. RNAs profiling mi. RNAs microarrays q. RT-PCR arrays (TLDA) Transcriptomics DNA microarray RNA seq normal Diferentially expressed onco-proteins tumoral Differentially expressed omcogenic m. RNA/mi. RNA Concordant protein/m. RNA/mi. RNA expression (Regulatory networks) normal tumoral Validate proteins, m. RNAs Microarray tissues (IEQ) Functional analysis Therapeutic targets Biomarker candidates Validation in patients Prognostic markers

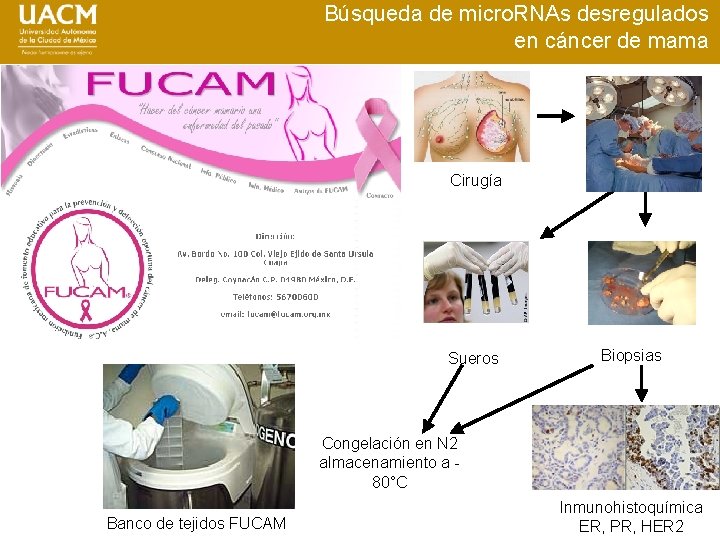
Búsqueda de micro. RNAs desregulados en cáncer de mama Cirugía Sueros Biopsias Congelación en N 2 almacenamiento a 80°C Banco de tejidos FUCAM Inmunohistoquímica ER, PR, HER 2

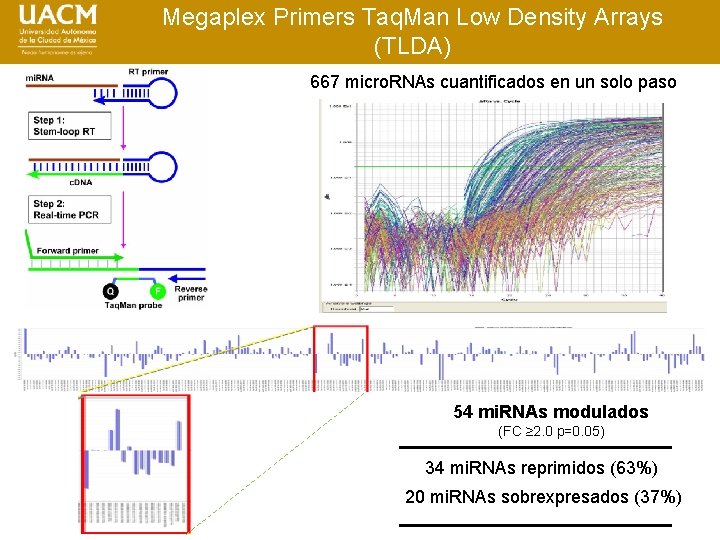
Megaplex Primers Taq. Man Low Density Arrays (TLDA) 667 micro. RNAs cuantificados en un solo paso 54 mi. RNAs modulados (FC ≥ 2. 0 p=0. 05) 34 mi. RNAs reprimidos (63%) 20 mi. RNAs sobrexpresados (37%)

Huella de expresión de 54 mi. RNAS y validación de TLDAs

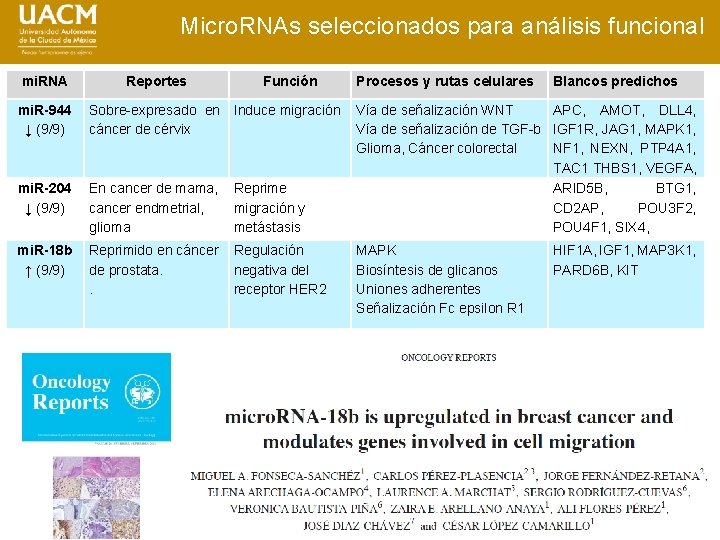
Micro. RNAs seleccionados para análisis funcional mi. RNA Reportes Función mi. R-944 ↓ (9/9) Sobre-expresado en Induce migración cáncer de cérvix mi. R-204 ↓ (9/9) En cancer de mama, cancer endmetrial, glioma Reprime migración y metástasis mi. R-18 b ↑ (9/9) Reprimido en cáncer de prostata. . Regulación negativa del receptor HER 2 Procesos y rutas celulares Blancos predichos Vía de señalización WNT APC, AMOT, DLL 4, Vía de señalización de TGF-b IGF 1 R, JAG 1, MAPK 1, Glioma, Cáncer colorectal NF 1, NEXN, PTP 4 A 1, TAC 1 THBS 1, VEGFA, ARID 5 B, BTG 1, CD 2 AP, POU 3 F 2, POU 4 F 1, SIX 4, MAPK Biosíntesis de glicanos Uniones adherentes Señalización Fc epsilon R 1 HIF 1 A, IGF 1, MAP 3 K 1, PARD 6 B, KIT

Análisis funcional del mi. R-204 en cáncer de mama M. en C. Ali Flores Pérez, UACM

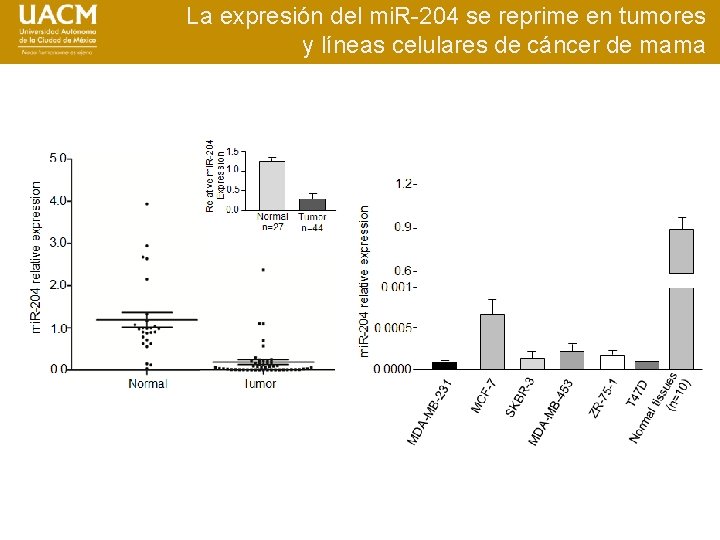
La expresión del mi. R-204 se reprime en tumores y líneas celulares de cáncer de mama

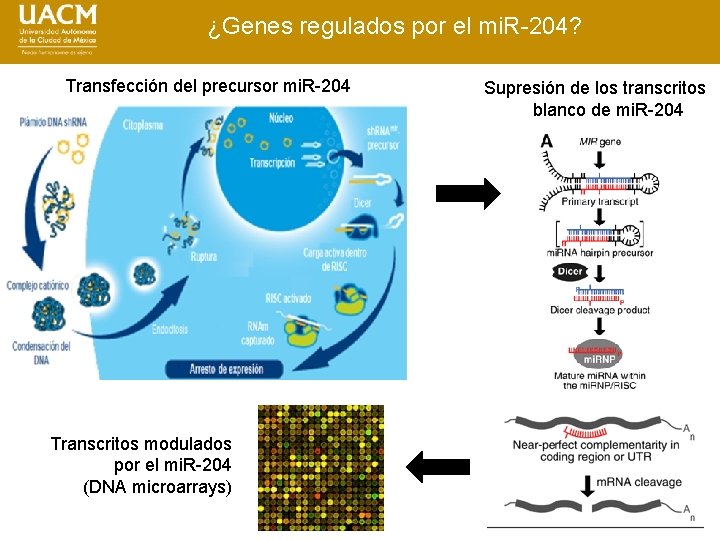
¿Genes regulados por el mi. R-204? Transfección del precursor mi. R-204 Transcritos modulados por el mi. R-204 (DNA microarrays) Supresión de los transcritos blanco de mi. R-204

DNA microarrays Nimbelgen 12 x 135 (Roche) mi. R-944 transfected No transfected control 135, 000 probes 3 probes per gene 45, 032 genes Oligos 60 -mer 549 modulated genes by mi. R-204(FC≥ 2. 0) 311 suppressed genes 238 upregulated genes

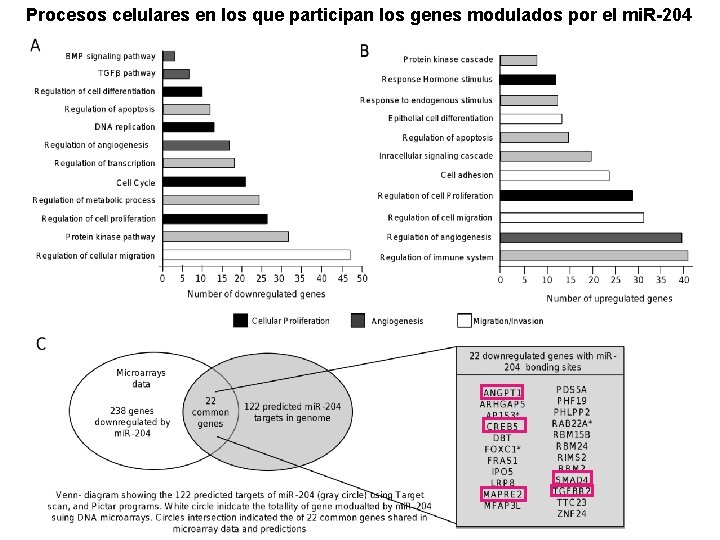
Procesos celulares en los que participan los genes modulados por el mi. R-204

¿ La restauración de la expresión del mi. R-204 modula los hallmarks del cáncer ? Transfección del mi. R-204 Análisis de fenotipos: migración, invasión, proliferación, angiogénesis Supresión de los transcritos blanco de mi. R-204

El mi. R-204 inhibe migración de las células tumorales Scratch/wound healing assays Transwell assays Inserto Membrana

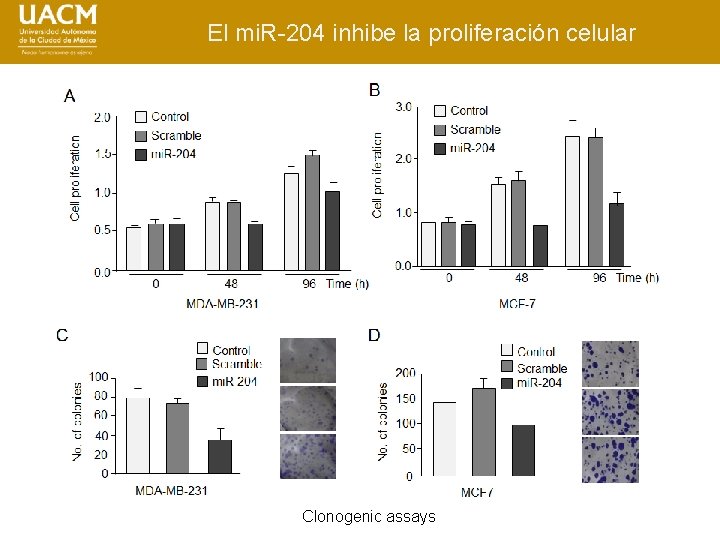
El mi. R-204 inhibe la proliferación celular Clonogenic assays

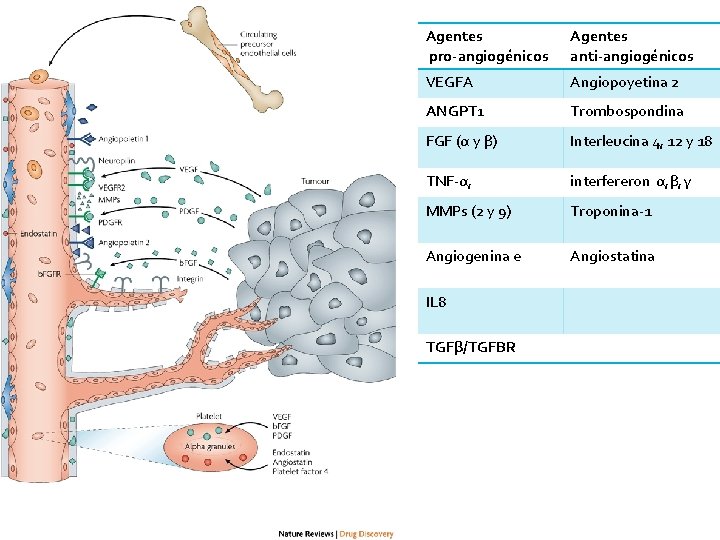
El mi. R-204 reprime genes involucrados en angiogénesis

Agentes pro-angiogénicos Agentes anti-angiogénicos VEGFA Angiopoyetina 2 ANGPT 1 Trombospondina FGF (α y β) Interleucina 4, 12 y 18 TNF-α, interfereron α, β, γ MMPs (2 y 9) Troponina-1 Angiogenina e Angiostatina IL 8 TGFβ/TGFBR

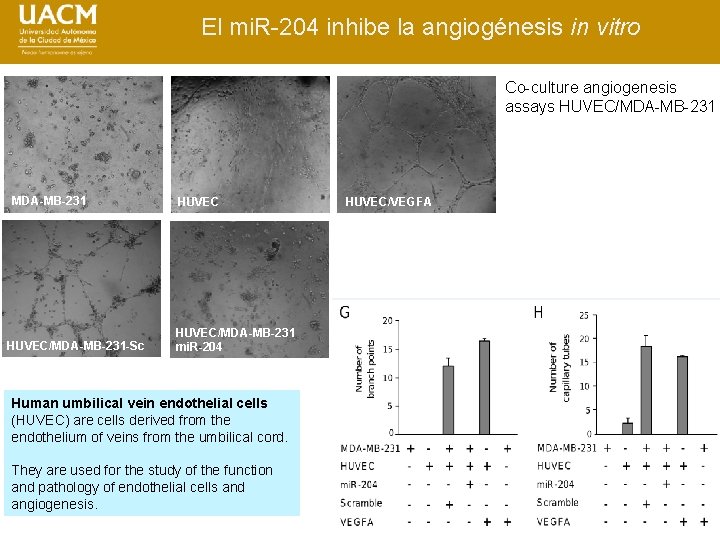
El mi. R-204 inhibe la angiogénesis in vitro Co-culture angiogenesis assays HUVEC/MDA-MB-231 -Sc HUVEC/MDA-MB-231 mi. R-204 Human umbilical vein endothelial cells (HUVEC) are cells derived from the endothelium of veins from the umbilical cord. They are used for the study of the function and pathology of endothelial cells and angiogenesis. HUVEC/VEGFA

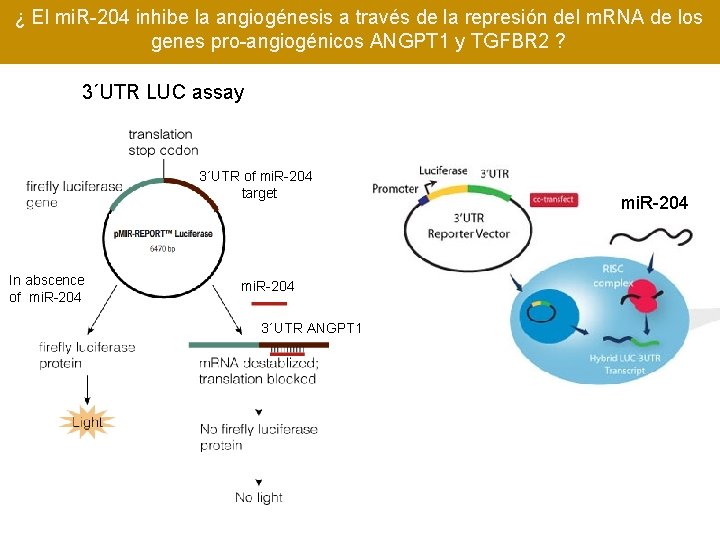
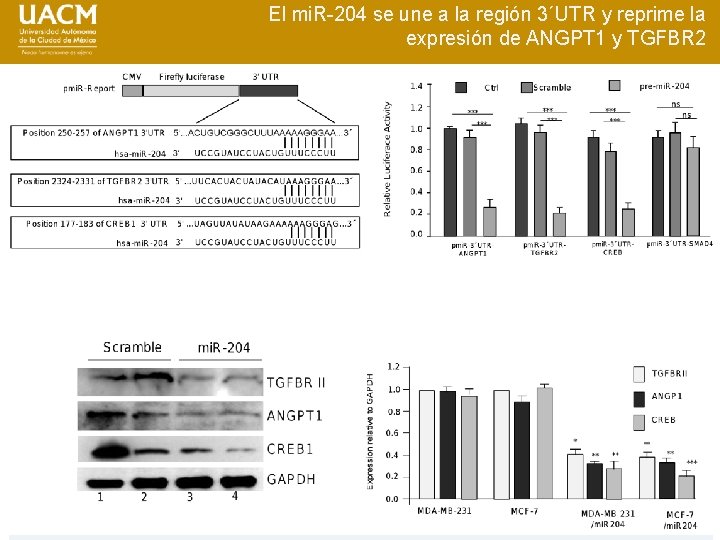
¿ El mi. R-204 inhibe la angiogénesis a través de la represión del m. RNA de los genes pro-angiogénicos ANGPT 1 y TGFBR 2 ? 3´UTR LUC assay 3´UTR of mi. R-204 target In abscence of mi. R-204 3´UTR ANGPT 1 mi. R-204

El mi. R-204 se une a la región 3´UTR y reprime la expresión de ANGPT 1 y TGFBR 2

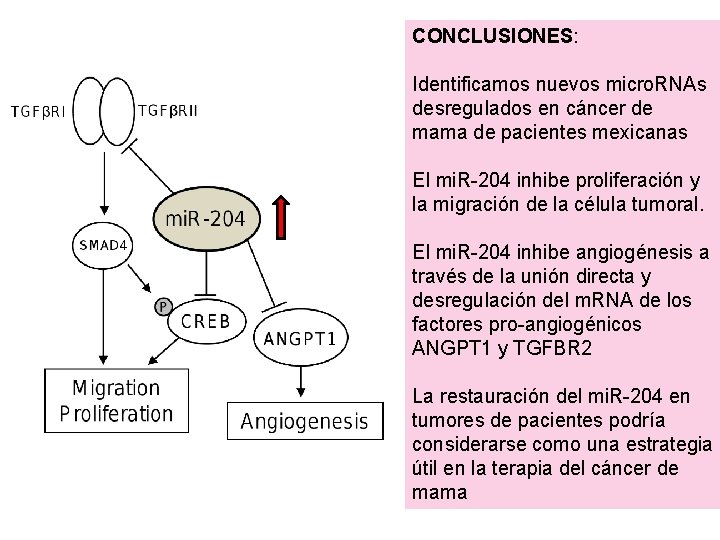
CONCLUSIONES: Identificamos nuevos micro. RNAs desregulados en cáncer de mama de pacientes mexicanas El mi. R-204 inhibe proliferación y la migración de la célula tumoral. El mi. R-204 inhibe angiogénesis a través de la unión directa y desregulación del m. RNA de los factores pro-angiogénicos ANGPT 1 y TGFBR 2 La restauración del mi. R-204 en tumores de pacientes podría considerarse como una estrategia útil en la terapia del cáncer de mama

Colaboradores Dr. Sergio Rodríguez Cuevas Dra. Verónica Bautista Instituto de Enfermedades de la Mama, FUCAM Dr. Alfredo Hidalgo Miranda Instituto Nacional de Medicina Genómica, INMEGEN Dra. Elena Arechaga Ocampo Dr. José Díaz Chávez Dra. Erika Ruiz Garcia Instituto Nacional de Cancerología, In. Can Dra. Laurence Annie Marchat Programa en Biomedicina Molecular, ENMy. H-IPN Dra. Ángeles Carlos-Reyes Instituto Nacional de Enfermedades Respiratorias Dr. Patricio Gariglio CINVESTAV-IPN Financiamiento Fonsec Salud 2009 -2012 Fonsec Salud 2014 -2017 Ciencia Básica 2014 -2017

Se aceptan estudiantes de Maestría y Doctorado en Ciencias Genómicas !! genomicas@yahoo. com. mx Gracias por su atención

Biomarcadores tumorales en la clasificación y tratamiento del cáncer de mama HER 2 ER PR Inmunohistoquímica Variables clínicas pronósticas Tamaño tumor Nódulos Metástasis ER+, PR+, HER 2+/Luminal A (50%) Terapia hormonal Tamoxifeno (anti-ER) Ablación ovárica Anastrozol, Letrozol Quimioterapia Marcadores tumorales (variables biológicas) Receptor de estrógenos (ER) progesterona (PR) y HER 2 ER+, PR+, HER 2+++ ER-, PR-, HER 2 - HER 2 Triple negativo (30%) (15 -20%) Terapia personalizada Trastuzumab (anti-HER 2) Tamoxifeno (anti-ER) Quimioterapia

La expresión de 54 mi. RNAs seen moduló 54 mi. RNAs se desregularon los significativamente en 9 tumores mamarios ductales tumores mamarios micro. RNAs reprimidos mi. R-139 -5 p mi. R-508 -3 p mi. R-10 b* mi. R-944 mi. R-486 -5 p mi. R-100 mi. R-424* mi. R-125 b-2* mi. R-218 mi. R-541 mi. R-221* mi. R-337 -3 p mi. R-369 -3 p mi. R-1 mi. R-26 b* mi. R-95 mi. R-204 mi. R-216 b mi. R-656 mi. R-488 mi. R-139 -3 p Let-7 b mi. R-543 mi. R-335 mi. R-433 mi. R-489 mi. R-132* mi. R-195 mi. R-376 c Let-7 c mi. R-432 mi. R-132 mi. R-376 a mi. R-10 b micro. RNAs sobreexpresados mi. R-155 mi. R-331 -3 p Let-7 g* mi. R-188 -5 p mi. R-708 mi. R-301 a mi. R-454* mi. R-7 mi. R-130 b mi. R-183* mi. R-181 a* mi. R-21 mi. R-142 -3 p mi. R-592 mi. R-425* mi. R-431 mi. R-142 -5 p mi. R-148 b* mi. R-638

Los micro. RNAs regulan de manera negativa la expresión génica López-Camarillo C. , et al 2013
 Símbolos de auditoría
Símbolos de auditoría Conclusiones de un proyecto ejemplos
Conclusiones de un proyecto ejemplos Identificacin
Identificacin Requisitos funcionais e não funcionais
Requisitos funcionais e não funcionais Requisitos não funcionais
Requisitos não funcionais Requisito funcional e não funcional
Requisito funcional e não funcional Foda de una persona con mentalidad positiva
Foda de una persona con mentalidad positiva Amenazas de una empresa ejemplos
Amenazas de una empresa ejemplos Anlisis financiero
Anlisis financiero Master memfi
Master memfi Estrategias fo (maxi-maxi ejemplos)
Estrategias fo (maxi-maxi ejemplos) Anlisis de riesgos
Anlisis de riesgos Anamnese do idoso
Anamnese do idoso Cetonas terminacion
Cetonas terminacion Hipoglucemia sintomas neuroglucopenicos
Hipoglucemia sintomas neuroglucopenicos O que é antropometria dinâmica
O que é antropometria dinâmica Patron funcional nutricional metabolico
Patron funcional nutricional metabolico Triple gradiente descendente
Triple gradiente descendente Teoria triédrica da empresa
Teoria triédrica da empresa La democracia en mi casa
La democracia en mi casa Hidrocarburos
Hidrocarburos Documento administrativo
Documento administrativo Grupos funcionais quimica
Grupos funcionais quimica Es un texto literario
Es un texto literario Que son los bienes patrimoniales
Que son los bienes patrimoniales Dimensión funcional en educación inicial
Dimensión funcional en educación inicial Musculo cardiaco
Musculo cardiaco O rcnei apresenta as seguintes concepções
O rcnei apresenta as seguintes concepções Layout loja de roupas
Layout loja de roupas Dietanamida
Dietanamida Caras del segundo molar inferior
Caras del segundo molar inferior Unidad funcional del musculo
Unidad funcional del musculo Modelos de actitudes con atributos múltiples
Modelos de actitudes con atributos múltiples Tipo de sinapsis
Tipo de sinapsis Constipação funcional
Constipação funcional Pseudorequerimientos
Pseudorequerimientos Grupo funcional fosfato
Grupo funcional fosfato