Curso Espectrometra de masas Electrospray Trampa Inica y





















- Slides: 32

Curso Espectrometría de masas, Electrospray Trampa Iónica y Maldi-Tof UNIVERSIDAD FRANCISCO DE VITORIA Alberto Jorge García Laboratorio de Proteómica Centro de Biología Molecular Severo Ochoa Universidad Autónoma de Madrid-CSIC

Oferta del Servicio de Proteómica • www 2. cbm. uam. es/proteomica • Análisis mediante Espectrometría de Masas • Identificación sistemática de proteínas presentes en las bases de datos a partir de geles • La Síntesis de Péptidos es un servicio independiente

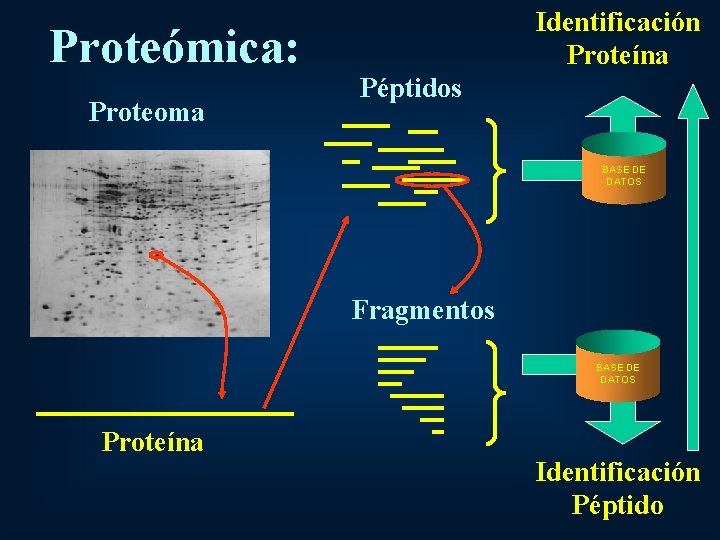
Proteómica: Proteoma Identificación Proteína Péptidos BASE DE DATOS Fragmentos BASE DE DATOS Proteína Identificación Péptido

Servicio de “Proteómica” • Digestión manual en gel • Análisis mediante MALDI-TOF: • Obtención del mapa peptídico e identificación mediante huella peptídica (PMF) • Análisis mediante LC-ESI-IT • Buen mapa con mezcla de proteínas: Exclusión dinámica (DE), búsqueda en DB mediante Turbo. Sequest • Mapa pobre ó interés en uno ó varios péptidos determinados: Monitorización de iones sencillos (SIM), búsqueda en DB por Tsequest ó confirmación mediante predicción teórica de fragmentación

Requisitos para la preparación de geles • Reactivos • Manipulación – Frescos, alto grado de pureza. – Muy cuidadosa, queratinas, recipientes de vidrio – Precauciones en la preparación de la muestra • Confección del gel – Lo más cercano posible a la entrega de la muestra – STD no preteñidos, no coloreados, no recombinantes. En cantidades conocidas y MW conocidos – No admitimos bandas ó spot recortados. Gel completo

Requisitos para la preparación de geles • Diseño del gel (monodimensional). Usuario – Minimizar el tamaño del carril para mejorar la razón proteína: gel – Optimizar la resolución y enriquecer para muestras muy complejas • Geles 2 D (bidimensionales) – Incluido en la oferta de Servicio – Es crítica la preparación de la muestra por parte del usuario (Precipitación con acetona)

Tinción de geles • Tinción Coomassie – – – Coomassie 0. 2%/metanol 50%/10%acético. Fresco Coomassie Coloidal, mayor contraste No fijar con etanol, se forma acetato de etilo Sensibilidad: 0. 1 mg, Cuantitativo Prácticamente nunca interfiere con la digestión • Tinción con plata (no recomendada) – – Sensibilidad: 1 -10 ng, No es cuantitativa Alta variedad en los protocolos de tinción Interfiere con la digestión Aumenta la necesidad de concentración de proteína, bajo rendimiento y alto nivel de contaminantes

Tinción de geles • Tinción fluorescente (mono ó 2 D) – Sensibilidad: Igual ó mayor a la plata – Variedad de tinciones (SYPRO Ruby, SYPRO Orange, SYPRO Red, Deep Purple…) – No interfiere con la digestión – La fluorescencia se puede medir en el Typhoon y recortar las bandas en un transiluminador UV (300 nm) – Disponible en el Servicio

Digestión “in situ” de geles • Procedimiento adaptado de Mann y cols. Con variaciones entre coomassie y plata. • Control interno de cada gel digiriendo std de pm (descarta problemas atribuibles a la preparación/tinción del gel) • Control externo con bandas/spots de geles ya procesados (descarta problemas atribuibles al proceso de digestión y preparación de muestra para MS)

Servicio de “Proteómica” • Digestión manual en gel • Análisis mediante MALDI-TOF: • Obtención del mapa peptídico e identificación mediante huella peptídica (PMF) • Análisis mediante LC-ESI-IT • Buen mapa con mezcla de proteínas: Exclusión dinámica (DE), búsqueda en DB mediante Turbo. Sequest • Mapa pobre ó interés en uno ó varios péptidos determinados: Monitorización de iones sencillos (SIM), búsqueda en DB por Tsequest ó confirmación mediante predicción teórica de fragmentación

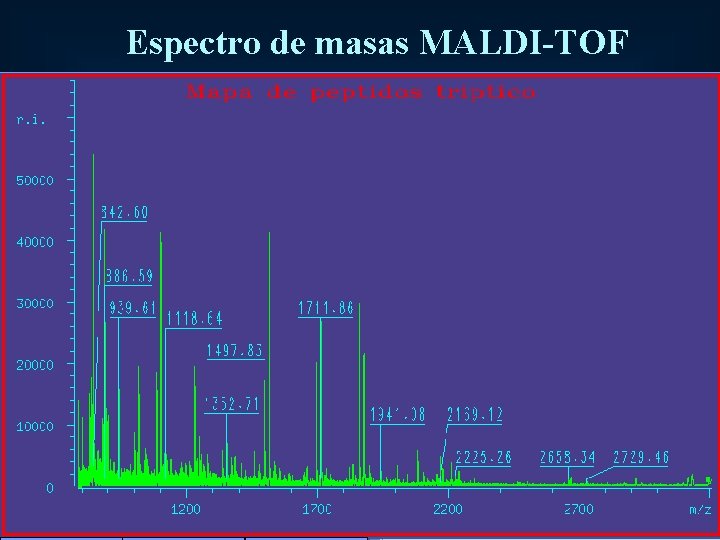
Espectro de masas MALDI-TOF

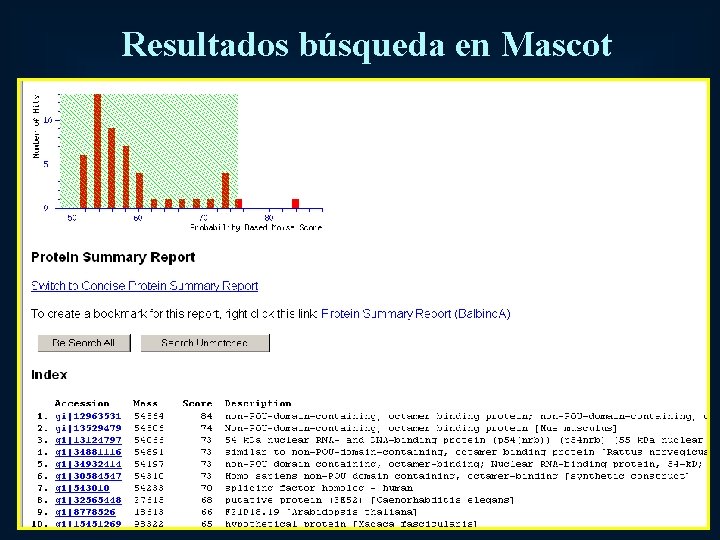
Resultados búsqueda en Mascot

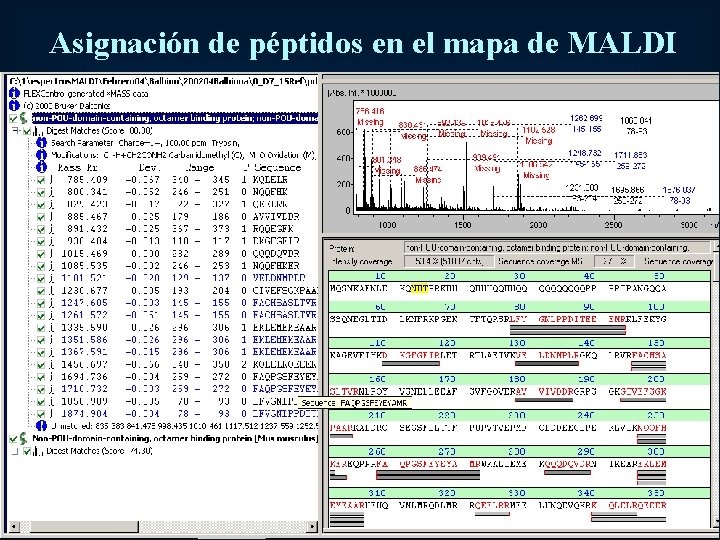
Asignación de péptidos en el mapa de MALDI

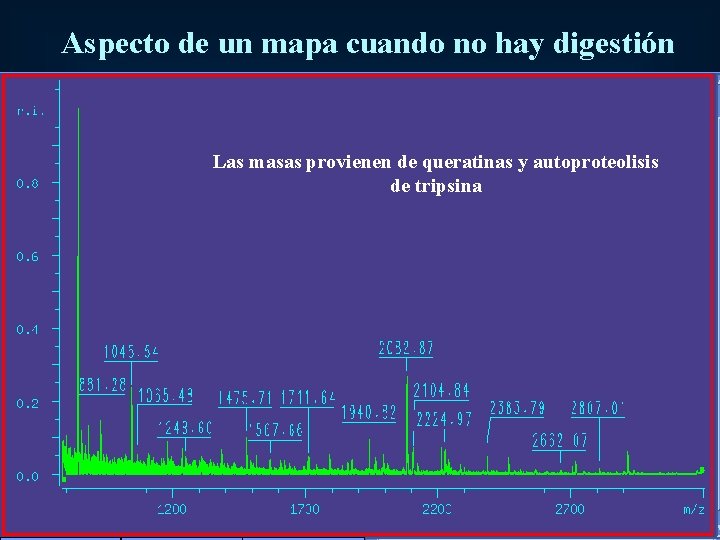
Aspecto de un mapa cuando no hay digestión Las masas provienen de queratinas y autoproteolisis de tripsina

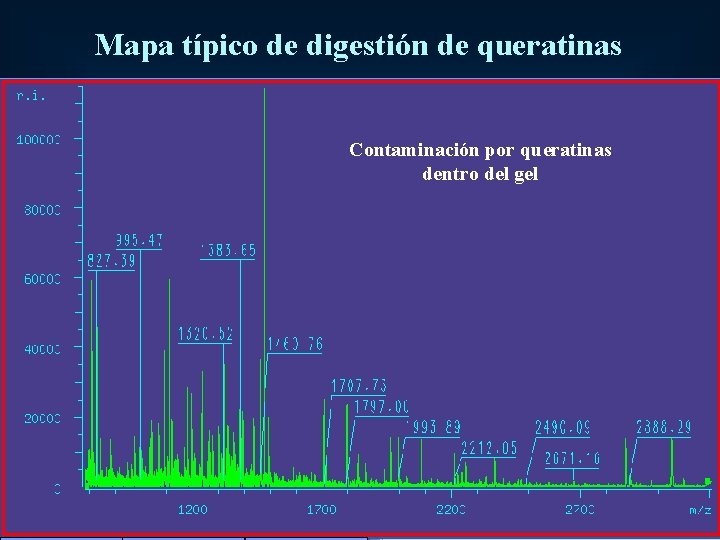
Mapa típico de digestión de queratinas Contaminación por queratinas dentro del gel

Sypro Rubi vs Plata STD PM Picomoles 2 1 0. 5 0. 25 0. 1 Gel plata 2 1 0. 5 0. 25 0. 1 Gel Sypro después de cortar

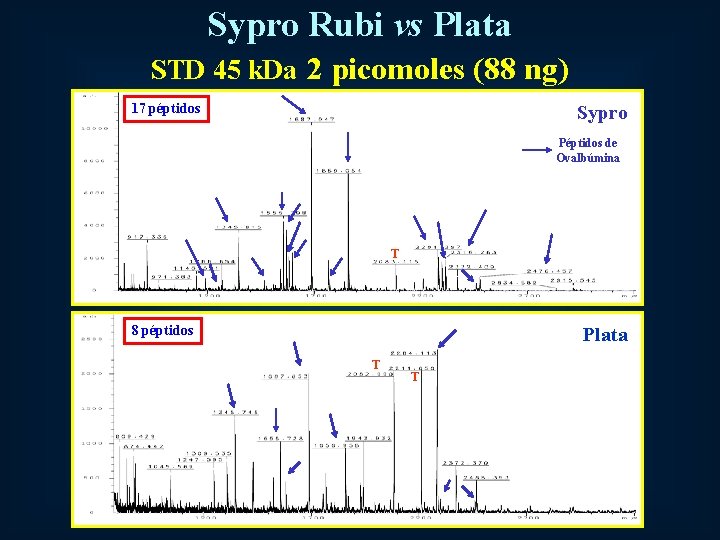
Sypro Rubi vs Plata STD 45 k. Da 2 picomoles (88 ng) 17 péptidos Sypro Péptidos de Ovalbúmina T 8 péptidos Plata T T

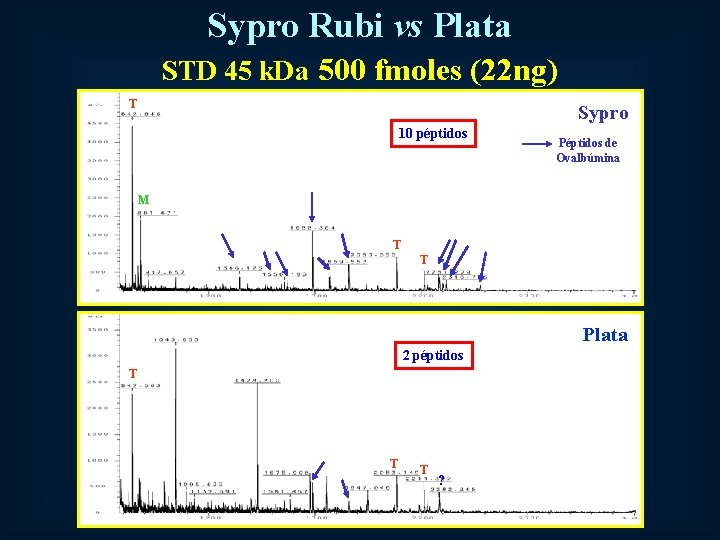
Sypro Rubi vs Plata STD 45 k. Da 500 fmoles (22 ng) T Sypro 10 péptidos Péptidos de Ovalbúmina M T T Plata 2 péptidos T T T ?

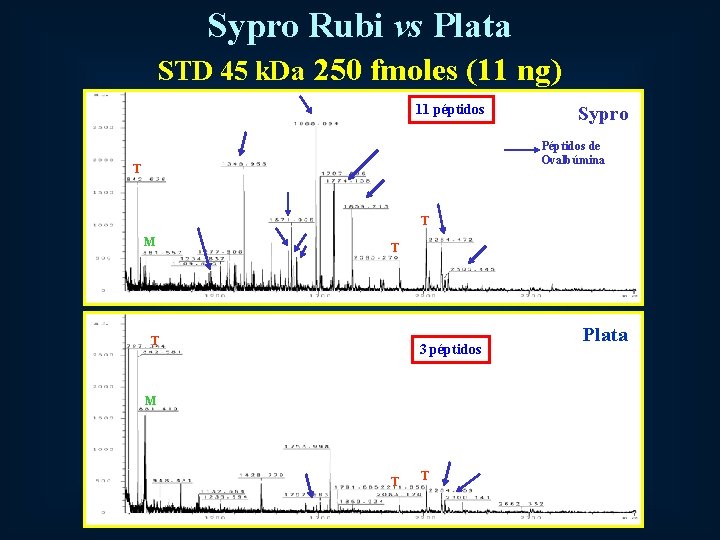
Sypro Rubi vs Plata STD 45 k. Da 250 fmoles (11 ng) 11 péptidos Sypro Péptidos de Ovalbúmina T T M T T 3 péptidos M T T Plata

Identificación mediante PMF • Tres niveles de preparación de muestras: – Sensibilidad normal: método estándar en placa de acero inoxidable (automatizable) – Alta sensibilidad: método “anchorchip” con matriz HCCA y cristalización homogénea de Bruker (automatizable) – Muy alta sensibilidad: método “anchorchip” con matriz DHB y cristalización heterogénea (no automatizable) Siempre trabajamos con este método

Identificación mediante PMF • Métodos de calibración: – Externa próxima, para búsquedas en DB con 100150 ppm. Los espectros se adquieren con esta calibración – Mediante “zonas prohibidas” (*) para búsquedas con 50 -100 ppm. Se aplica a la lista de masas obtenida por calibración externa – Interna de forma manual, mediante macro ó software (*) para búsquedas con 30 -50 ppm • Motores de búsqueda: – Internet (Mascot, Profound) * Campillo, Martín and Vázquez

Identificación mediante PMF Causas por las que no se identifica una proteína • • • Mapa con un nº insuficiente de péptidos Mezcla de proteínas Proteína no presente en DB Alto nivel de contaminación Modo en el que se genera la lista de masas

Servicio de “Proteómica” • Digestión manual en gel • Análisis mediante MALDI-TOF: • Obtención del mapa peptídico e identificación mediante huella peptídica (PMF) • Análisis mediante LC-ESI-IT • Buen mapa con mezcla de proteínas: Exclusión dinámica (DE), búsqueda en DB mediante Turbo. Sequest • Mapa pobre ó interés en uno ó varios péptidos determinados: Monitorización de iones sencillos (SIM), búsqueda en DB por Tsequest ó confirmación mediante predicción teórica de fragmentación

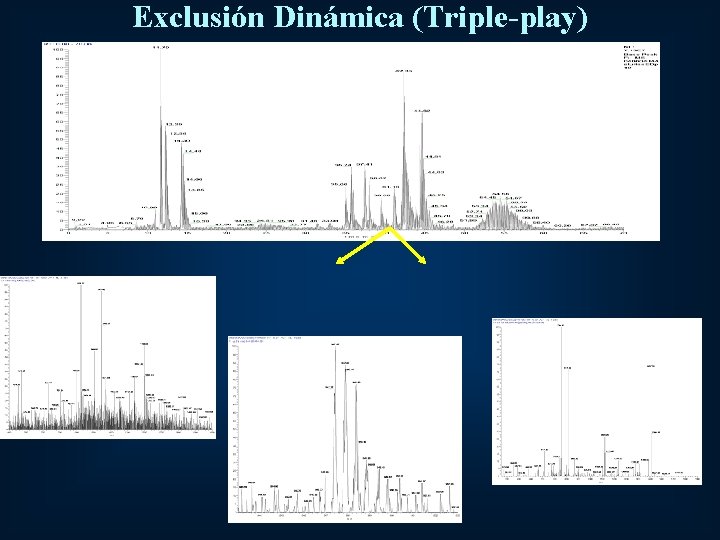
Identificación de péptidos presentes en DB mediante LCMS/MS ESI-IT • Alta sensibilidad en modo “full scan”: Exclusión dinámica (DE), 100 fmoles para un digerido en solución de estándar de BSA • Muy alta sensibilidad en modo SIM: “Monitorización de iones sencillos”, 1 -10 fmoles para estándar de BSA

Exclusión Dinámica (Triple-play)


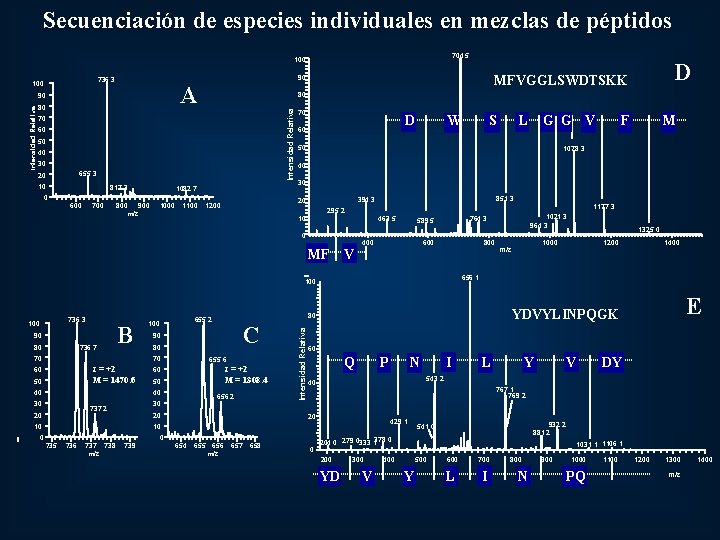
Secuenciación de especies individuales en mezclas de péptidos 704. 5 100 A 80 80 Intensidad Relativa 90 70 60 50 40 30 655. 3 20 10 0 817. 3 600 700 1082. 7 800 900 m/z 1000 1100 70 D 60 851. 3 394. 3 295. 2 463. 5 400 600 800 V 80 736. 7 B 70 80 70 z = +2 M = 1470. 6 60 50 30 30 656. 2 20 10 0 z = +2 M = 1308. 4 50 40 737. 2 655. 6 60 40 20 C 90 736 737 738 m/z 739 0 655 656 m/z 657 658 1325. 0 1000 m/z 1200 1400 E 60 Q P N 0 I L Y V DY 543. 2 40 767. 1 769. 2 20 654 1177. 3 YDVYLINPQGK 429. 1 10 735 M 656. 1 80 Intensidad Relativa 90 F 1021. 3 964. 3 764. 3 589. 5 100 655. 2 G G V 30 10 100 L 40 MF 736. 3 S 1078. 3 20 1200 W 50 0 100 D MFVGGLSWDTSKK 90 736. 3 100 932. 2 881. 2 541. 0 204. 0 279. 0333. 1378. 0 200 YD 300 V 1031. 1 1106. 1 400 500 Y 600 700 L I 800 N 900 1000 PQ 1100 1200 1300 m/z 1400

Análisis mediante Espectrometría de Masas • Análisis empleando MALDI-TOF – Muestras preparadas por usuario (incluye portas y matriz) – Muestras preparadas por el Servicio • Análisis empleando LC-ESI-IT de fracciones de péptidos provenientes de HPLC preparadas por usuario

Equipos disponibles • Un espectrómetro de masas tipo MALDI-TOF, modelo Autoflex de Bruker, equipado con reflector • Un espectrómetro de masas de trampa iónica modelo LCQ Classic de Thermo Finnigan, equipado con sistemas de ionización de nanospray y microspray de fabricación propia, acoplado a una HPLC Agilent 1100 con restrictor de flujo inteligente (cedido en mayo 04 por un año) • Interface “metal needle kit” para el acoplamiento LCESI-IT y columna RP de 180 mm, flujos de 1, 5 a 2 ml/m

Procedimiento de trabajo • Recepción y control de muestras – – Aviso previo a la entrega de geles Escaneo del gel, Confección de ficha de usuario, Instrucciones del trabajo a realizar • Análisis – Digestión manual en gel – Obtención del mapa peptídico mediante MALDITOF – Análisis mediante LC-ESI-IT


Personal del Servicio de Proteómica • Rosana Rogado: – • Laboral contratado temporal (CSIC), Ayudante de Investigación y Laboratorio Alberto Jorge: – • Laboral fijo (CSIC), Técnico de Investigación y Laboratorio Yolanda Núñez: – • Funcionaria. Técnico Especialista de Grado Medio Anabel Marina: – Laboral interino (CSIC), Titulado Técnico nivel 2
 Jaula lingual
Jaula lingual Trampa ionica farmacologia
Trampa ionica farmacologia Trampa grasas
Trampa grasas No lo hagas es una trampa
No lo hagas es una trampa Trampa estratigrafica
Trampa estratigrafica Mīnoja meita
Mīnoja meita Dinámica de las masas fluidas
Dinámica de las masas fluidas Escuela de masas
Escuela de masas Tipos de comportamiento colectivo
Tipos de comportamiento colectivo Masas
Masas Estructura argumentativa secuencial
Estructura argumentativa secuencial Definicion de mass media
Definicion de mass media Argumento de fama
Argumento de fama Constante de equilibrio quimico
Constante de equilibrio quimico Dos masas
Dos masas Fetichismo de masas texto argumentativo
Fetichismo de masas texto argumentativo Fuentes de ionizacion duras y blandas
Fuentes de ionizacion duras y blandas Keq < 1
Keq < 1 Cultura de masas imagenes
Cultura de masas imagenes El desprecio de las masas
El desprecio de las masas Tantala meita
Tantala meita Ejemplo de argumentos afectivos
Ejemplo de argumentos afectivos Masas polares alternador
Masas polares alternador Cisterna selar
Cisterna selar Dos masas
Dos masas Aplicaciones de la espectrometría de masas
Aplicaciones de la espectrometría de masas Que tica
Que tica Revenue management curso
Revenue management curso Fatfa curso
Fatfa curso Salud ocupacional en mexico
Salud ocupacional en mexico Network analizer
Network analizer Curso de profesor de yoga intensivo
Curso de profesor de yoga intensivo Curso nacional de atualização em pneumologia
Curso nacional de atualização em pneumologia