Espectrometra de masa MSMS Espectrometra en Tndem Informacin














![a. PLPC TOCL [H 2 O 2] 37°C, 1 Hr a. PLPC TOCL [H 2 O 2] 37°C, 1 Hr](https://slidetodoc.com/presentation_image_h/2af83f18147f9afa00fc05323af09837/image-22.jpg)











- Slides: 44

Espectrometría de masa MS/MS Espectrometría en Tándem: Información estructural de pequeñas moléculas Secuenciación de péptidos 1

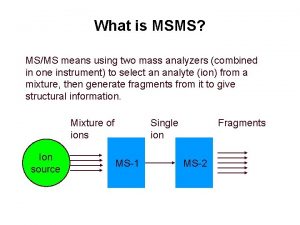
Espectrometría de masa MS/MS: Espectrometría en Tándem Seleccionar uno de los iones de la muestra, fragmentarlo y estudiar la formación de iones “hijos” mp+ m d+ + m n 0 Aplicaciones: Identificación de moléculas Secuenciación de péptidos “al vuelo” Modificaciones posttraduccionales 2

Espectrometría de masa MS/MS: Secuenciación peptídica Mezcla de péptidos 1 péptido seleccionado para MS/MS + + + + Espectro MS Carlos Cerveñansky Espectro MS/MS: masa de los fragmentos 3

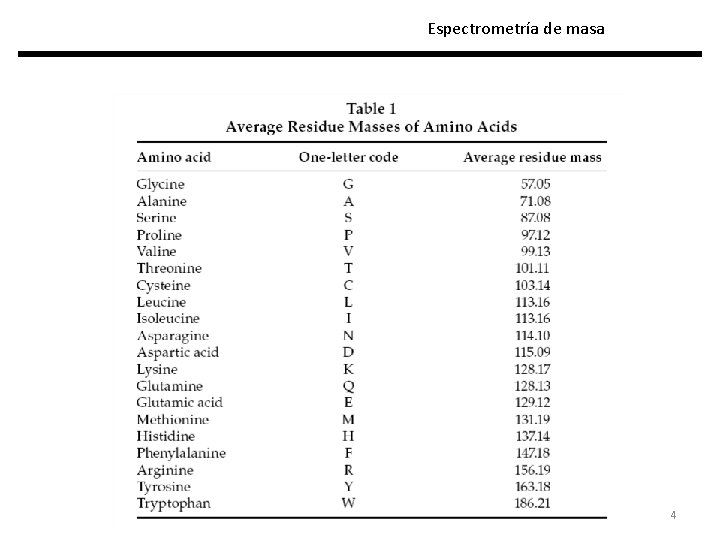
Espectrometría de masa 4

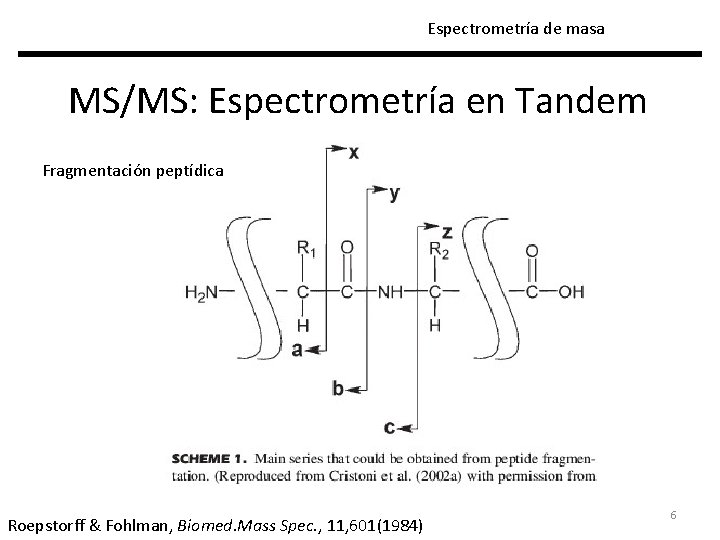
Espectrometría de masa MS/MS: Espectrometría en Tandem Fragmentación peptídica C-terminal N-terminal La fragmentación más común se da en el enlace peptídico, generando fragmentos b e y Roepstorff & Fohlman, Biomed. Mass Spec. , 11, 601(1984)

Espectrometría de masa MS/MS: Espectrometría en Tandem Fragmentación peptídica Roepstorff & Fohlman, Biomed. Mass Spec. , 11, 601(1984) 6


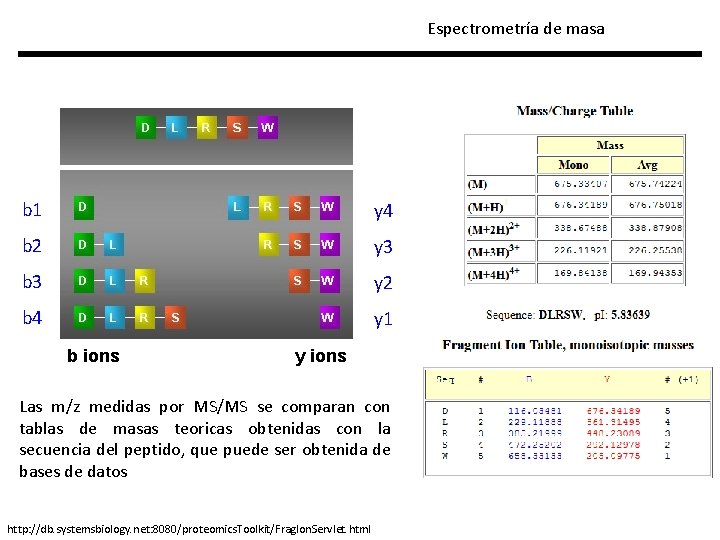
Espectrometría de masa b 1 y 4 b 2 y 3 b 3 y 2 b 4 y 1 b ions y ions Las m/z medidas por MS/MS se comparan con tablas de masas teoricas obtenidas con la secuencia del peptido, que puede ser obtenida de bases de datos http: //db. systemsbiology. net: 8080/proteomics. Toolkit/Frag. Ion. Servlet. html

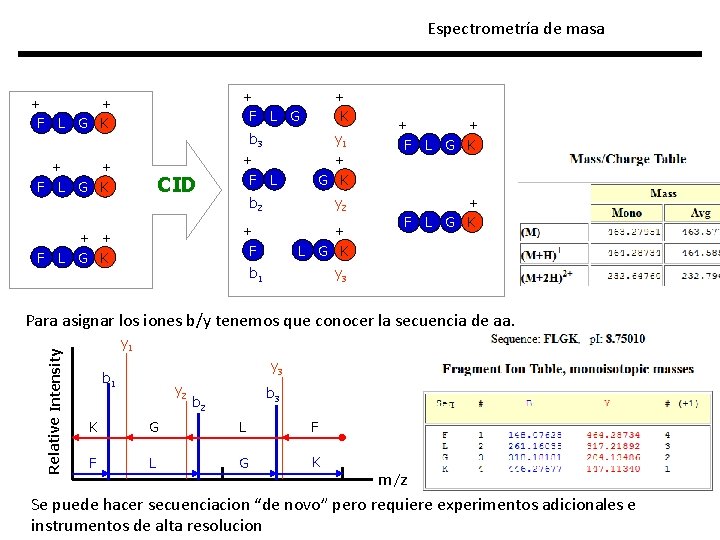
Espectrometría de masa + F + L G K L G b 3 + + F L G K F + K CID + F y 1 L b 2 + L G K F + L G K y 2 + F + + L G K + F + L G K b 1 y 3 Relative Intensity Para asignar los iones b/y tenemos que conocer la secuencia de aa. y 1 y 3 b 1 y 2 b 3 b 2 K G L F F L G K m/z Se puede hacer secuenciacion “de novo” pero requiere experimentos adicionales e instrumentos de alta resolucion

Fragmentacion de Peptidos por MS/MS y 13 y 12 y 11 y 10 y 9 y 8 y 7 y 6 y 5 y 4 y 3 y 2 y 1 His-Gly-Thr-Val-Leu-Thr-Ala-Leu-Gly-Ile -Leu-Lys b 1 b 2 b 3 b 4 b 5 b 6 b 7 b 9 b 10 b 11 b 12 b 13 y 9 887. 6 100 Relative Abundance b 8 y 10 986. 6 90 80 y 8 774. 5 70 b 5 494. 3 60 607. 4 40 b 4 30 395. 2 10 y 11 1168. 7 1085. 7 b 6 50 20 y 12* 1186. 7 b 5* 476. 3 a 5 466. 3 400 500 b 6* 589. 3 y 7 673. 5 b 11 1006. 6 b 8 779. 5 b 7 772. 5 885. 6 708. 4 984. 6 b 12 1119. 6 a 12 y 13 1243. 7 b 13 1184. 7 b 10 600 700 800 900 1000 1100 1200 1300 1400

Espectrometría de masa MS/MS: Espectrometría en Tandem Cómo se hace?

Espectrometría de masa MS/MS: Espectrometría en Tandem ESI-Q Q 1 Q 2 Q 3: Triple Cuadrupolo. Q 1 Select Parent Q 2 Fragment Q 3 Analyse Fragment Masses Q 1 Selecciona el ion molecular “padre” Q 2 sirve de cámara de colisión: Se introduce un gas para que los iones acelerados choquen y se fragmenten (CID) Q 3 Analiza los iones “hijo” 12

Espectrometría de masa MS/MS: Espectrometría en Tandem ESI-Q Q 1 Q 2 Q 3: Triple Cuadrupolo. Q 2 sirve de cámara de colisión: Se introduce un gas para que los iones acelerados choquen y se fragmenten CID: Collision Induced Dissociation Se usa mucho para realizar cuantificaciones por LC-MS-MS 13

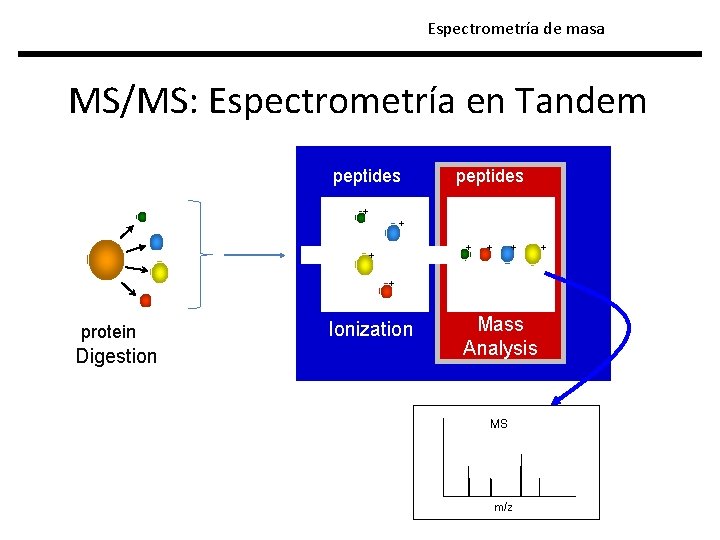
Espectrometría de masa MS/MS: Espectrometría en Tandem peptides + + + + protein Digestion Ionization Mass Analysis MS m/z +

Espectrometría de masa MS/MS: Espectrometría en Tandem protein peptide fragments peptides ++ + + ++ + Ionization MS m/z + + + ++ Digestion ++ + Isolation m/z Fragmentation Mass Analysis MS/MS m/z +

Espectrometría de masa MS/MS: Espectrometría en Tandem ESI-IT IT: Trampa de Iones La trampa de iones puede analizar los iones de una muestra MS También puede seleccionar uno, y concentrarlo en la trampa Luego se aceleran los iones para que choquen con Helio y se fragmenten MS 2 Luego se analizan los iones hijo CID: Collision Induced Dissociation Las trampas de iones tienen capacidad de hacer MSn se seleccionan iones hijo se fragmentan, analizan, seleccionan uno hasta que les de la sensibilidad 16

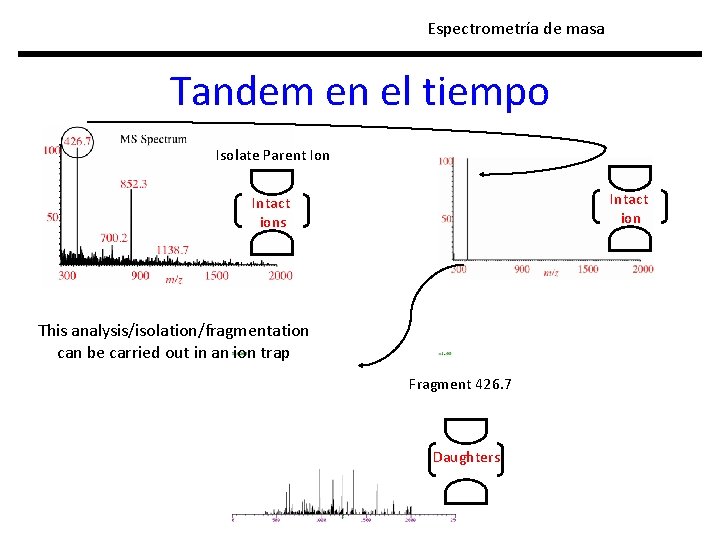
Espectrometría de masa Tandem en el tiempo Isolate Parent Ion Intact ions This analysis/isolation/fragmentation can be carried out in an ion trap Fragment 426. 7 Daughters

Espectrometría de masa MS/MS: Espectrometría en Tandem MALDI-TOF Iones moleculares que se fragmentan durante el vuelo llegan juntos al “reflector” y pueden ser analizados por el mismo. PSD: Post Source Decay 18

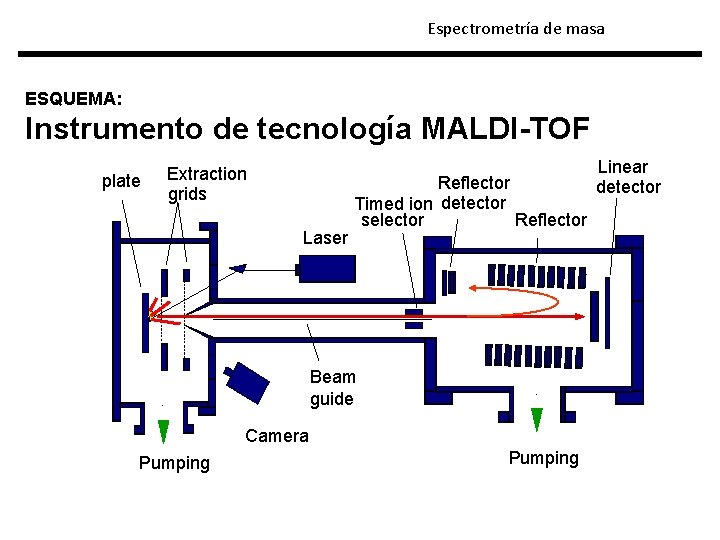
Espectrometría de masa ESQUEMA: Instrumento de tecnología MALDI-TOF plate Extraction grids Laser Reflector Timed ion detector selector Reflector Beam guide Camera Pumping Linear detector

Espectrometría de masa 20

Identificación de sitios de modificación Oxidación de citocromo c Located in mitochondria, plays an important role in apoptosis intrinsic pathway and has been linked to oxidative stress responses Y 74 Y 67 Y 97 Y 48
![a PLPC TOCL H 2 O 2 37C 1 Hr a. PLPC TOCL [H 2 O 2] 37°C, 1 Hr](https://slidetodoc.com/presentation_image_h/2af83f18147f9afa00fc05323af09837/image-22.jpg)
a. PLPC TOCL [H 2 O 2] 37°C, 1 Hr

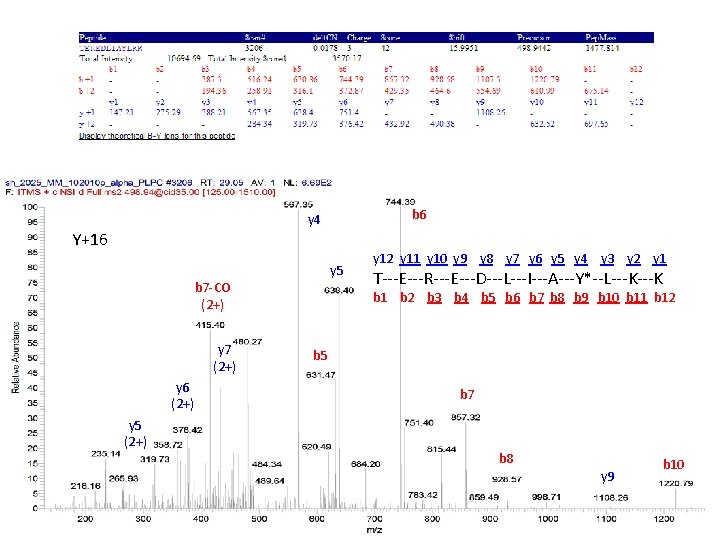
b 6 y 4 Y+16 y 5 b 7 -CO (2+) y 7 (2+) y 6 (2+) y 5 (2+) y 12 y 11 y 10 y 9 y 8 y 7 y 6 y 5 y 4 y 3 y 2 y 1 T---E---R---E---D---L---I---A---Y*--L---K b 1 b 2 b 3 b 4 b 5 b 6 b 7 b 8 b 9 b 10 b 11 b 12 b 5 b 7 b 8 y 9 b 10

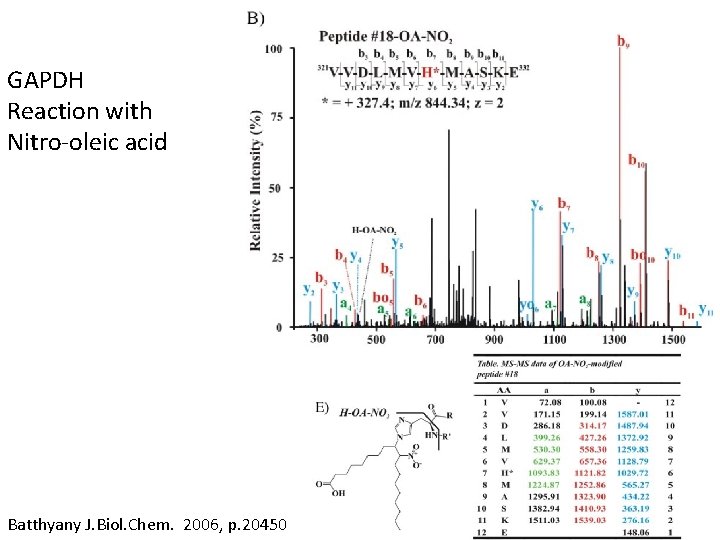
GAPDH Reaction with Nitro-oleic acid Batthyany J. Biol. Chem. 2006, p. 20450

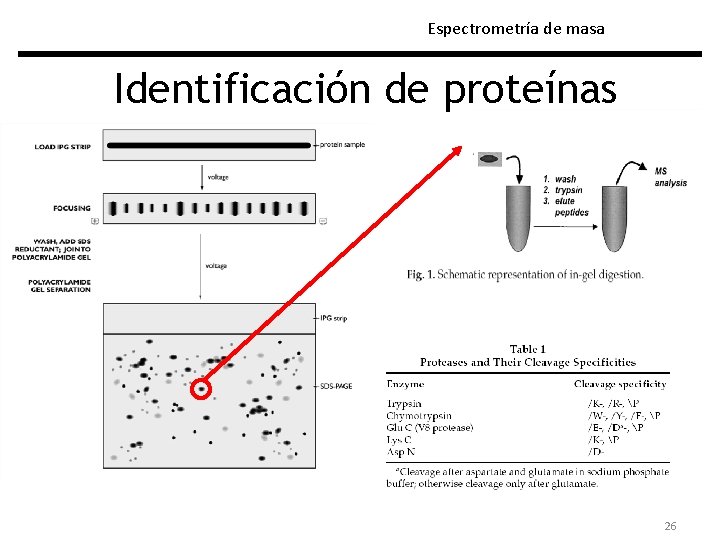
Espectrometría de masa Identificación de proteínas Peptide mass fingerprinting y proteomica 25

Espectrometría de masa Identificación de proteínas 26

Huella peptídica Cada proteína, cortada con una proteasa de especificidad conocida, da una serie de péptidos de masa determinada que puede ser usada para identificar la proteína (huella peptídica) En el 2003 se terminó de secuenciar el primer genoma humano. De la secuencia del ADN se puede determinar la secuencia de aminoácidos de todas las proteínas potencialmente expresables. Hoy contamos con cerca de 20. 000 Genomas secuenciados, y mas por salir.

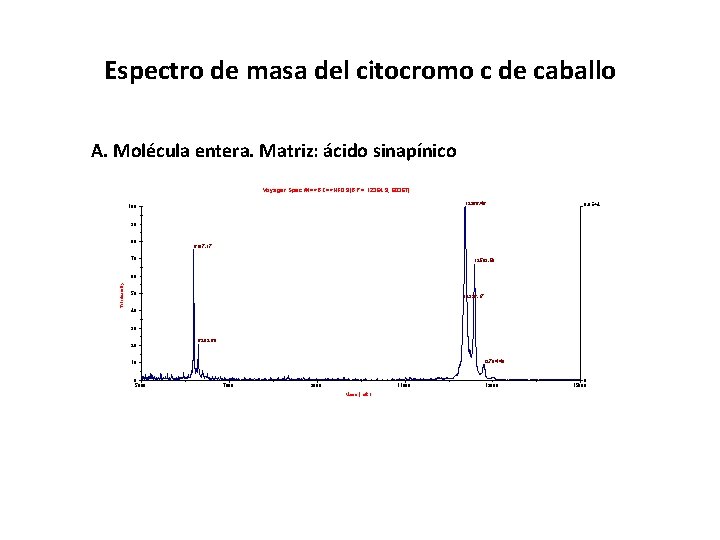
Espectro de masa del citocromo c de caballo A. Molécula entera. Matriz: ácido sinapínico Voyager Spec #1=>BC=>NF 0. 9[BP = 12364. 9, 60367] 12366. 46 100 6. 0 E+4 90 80 6187. 17 70 12583. 58 % Intensity 60 50 12326. 17 40 30 20 6292. 68 12784. 48 10 0 5000 7000 9000 11000 Mass (m/z) 13000 0 15000

Secuencia AA citocromo c de caballo GDVEKGKKIFVQKCAQCHTVEKGGKHKTGPNL HGLFGRKTGQAPGFTYTDANKNKGITWK EETLMEYLENPKKYIPGTKMIFAGIKKKTEREDLI AYLKKATNE Digestión in silico por tripsina (corta despues de K o R) Lista de péptidos teóricos GDVEKGK GK GKK IFVQKCAQCHTVEKGGK GGKHK TGPNLHGLFGRK TGQAPGFTYTDANK Se calculan las masas teóricas para todos los péptidos posibles y se comparan con los resultados de MS de cytc tratado con tripsina

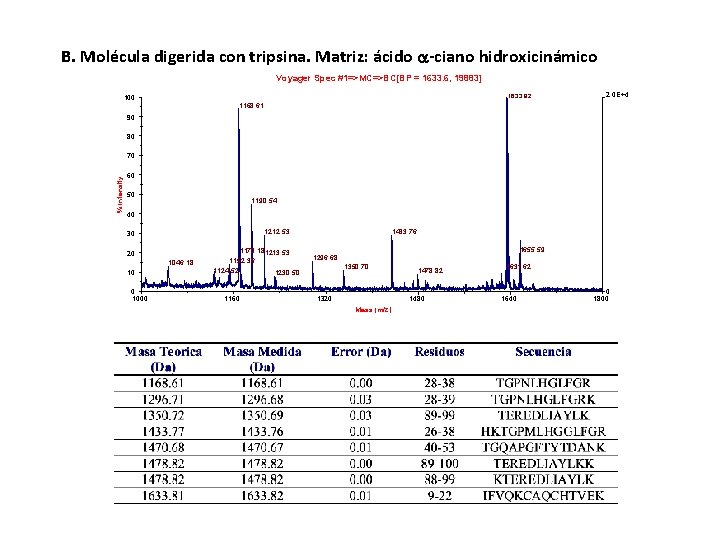
B. Molécula digerida con tripsina. Matriz: ácido -ciano hidroxicinámico Voyager Spec #1=>MC=>BC[BP = 1633. 6, 19883] 163 3. 82 100 2. 0 E+4 1168. 61 90 80 % Intensity 70 60 50 1190. 54 40 1212. 53 30 20 1046. 18 10 0 1000 1170. 181213. 53 1152. 36 1124. 52 1230. 50 1160 1433. 76 1655. 59 1296. 68 1350. 70 1320 1478. 82 1480 Mass (m/z) 1631. 62 1640 0 1800

Niveles de información……… en la caracterización de proteínas por MS. • medida de masa de una molécula entera experimental • medidas de masas en mapeo peptídico • secuenciado de péptidos por MS/MS 1 valor 5 -20 valores experimentales más de 100 valores experimentales …mejora en cantidad y calidad de la información

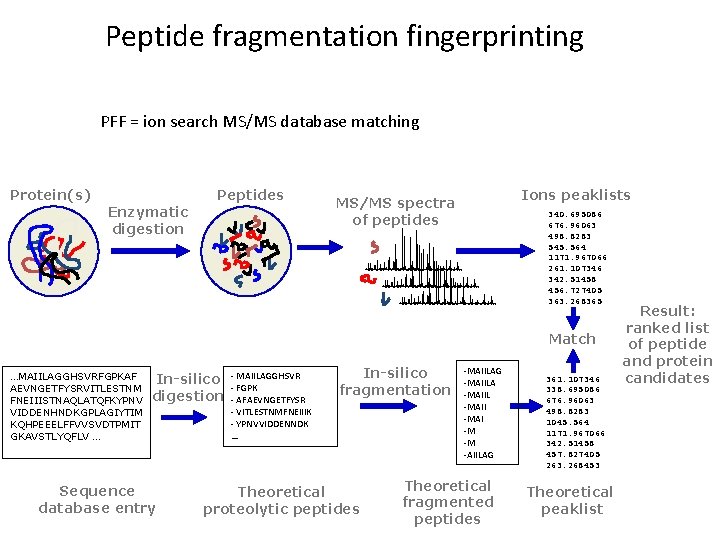
Peptide fragmentation fingerprinting PFF = ion search MS/MS database matching Protein(s) Peptides Enzymatic digestion Ions peaklists MS/MS spectra of peptides 340. 695086 676. 96063 498. 8283 545. 564 1171. 967066 261. 107346 342. 51458 456. 727405 363. 268365 Match …MAIILAGGHSVRFGPKAF AEVNGETFYSRVITLESTNM FNEIIISTNAQLATQFKYPNV VIDDENHNDKGPLAGIYTIM KQHPEEELFFVVSVDTPMIT GKAVSTLYQFLV … In-silico digestion Sequence database entry - MAIILAGGHSVR - FGPK - AFAEVNGETFYSR - VITLESTNMFNEIIIK - YPNVVIDDENNDK … In-silico fragmentation Theoretical proteolytic peptides -MAIILAG -MAIILA -MAIIL -MAII -MAI -M -M -AIILAG Theoretical fragmented peptides 361. 107346 338. 695086 676. 96063 498. 8283 1045. 564 1171. 967066 342. 51458 457. 827405 263. 268453 Theoretical peaklist Result: ranked list of peptide and protein candidates

Proteoma • El término “proteoma” fue acuñado por Marc Wilkins en 1995, como el complemento de PROTEínas al gen. OMA • • Es el producto final del genoma más variedad Es una entidad dinámica Fenotipo Modificaciones posttraduccionales No todos los genes se expresan como proteínas en todos los estadios/tipos celulares

Proteoma • Del gen a la proteína final, pueden ocurrir muchas modificaciones que no son predecibles por la secuencia de ADN original • 1 gen ya no equivale a 1 proteína

Sujetos de estudio de la Proteómica • • • Identificación de Proteínas (pept mass fingerprint) Comparación de niveles de expresión de proteinas Función de Proteinas Modificaciones posttraduccionales de Proteinas Localización y Compartimentalización de Proteinas Interacciones Proteina-Proteina


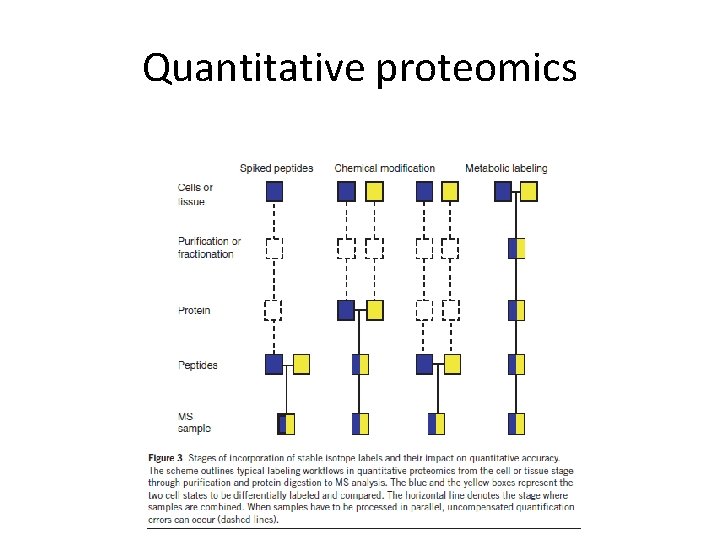
Quantitative proteomics

Quantitative proteomics


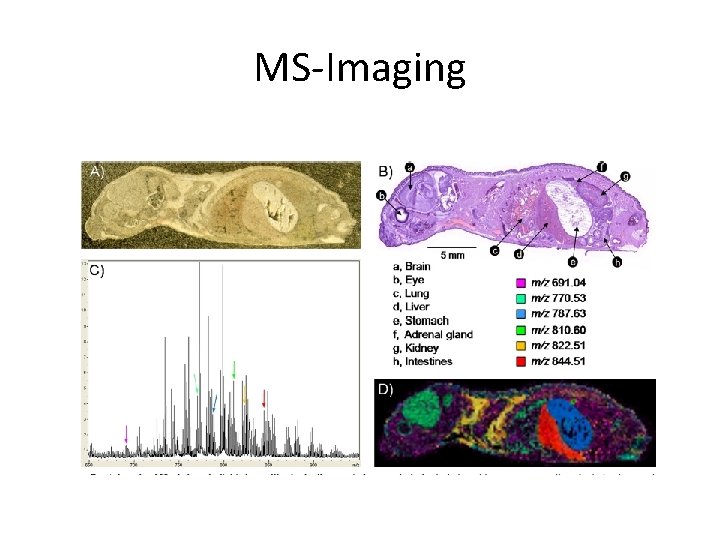
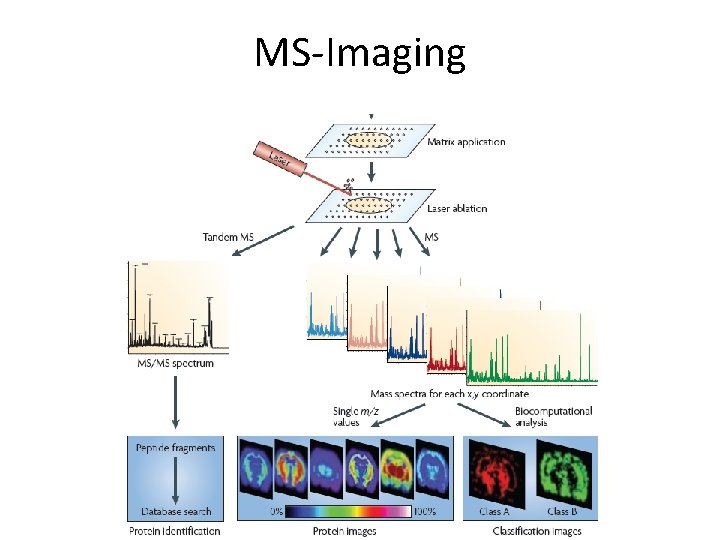
MS-Imaging

MS-Imaging


Conclusiones La espectrometría de masa es una técnica con muchísimas aplicaciones y aún tiene mucho potencial por desarrollar

 Fuentes de informacin
Fuentes de informacin Informacin
Informacin Matriz de masa
Matriz de masa Tndem
Tndem Lc-msms
Lc-msms Msms icys
Msms icys Lc-msms
Lc-msms Concentración de una disolución fórmula
Concentración de una disolución fórmula Masa molar
Masa molar Perkembangan mobil dari masa ke masa
Perkembangan mobil dari masa ke masa Perkembangan desain dari masa ke masa
Perkembangan desain dari masa ke masa Ejercicios masa/masa
Ejercicios masa/masa Perkembangan hardware
Perkembangan hardware Pendidikan masa kini dan masa depan
Pendidikan masa kini dan masa depan Relativna molekulska masa
Relativna molekulska masa Rancangan pentadbiran perniagaan
Rancangan pentadbiran perniagaan Predistinasi
Predistinasi Masa molecular uma
Masa molecular uma Ecuacion balanceada
Ecuacion balanceada Molska masa
Molska masa Srpsko petorečje
Srpsko petorečje Masa awal kemerdekaan
Masa awal kemerdekaan Schizogoni
Schizogoni Las leyes de newton
Las leyes de newton Fuerza peso fórmula
Fuerza peso fórmula Vady masa
Vady masa Que es el transporte en masa
Que es el transporte en masa Bentuk demokrasi pada masa orde lama
Bentuk demokrasi pada masa orde lama Sueu unidades
Sueu unidades Dari arah tempat masa
Dari arah tempat masa Un automovil de masa m esta sobre un camino cubierto
Un automovil de masa m esta sobre un camino cubierto Masa tribulasi
Masa tribulasi Jakostní třídy hovězího masa
Jakostní třídy hovězího masa Masa vegetatif bawang merah
Masa vegetatif bawang merah Kreacja par
Kreacja par Prustov zakon
Prustov zakon Glandula verde crustaceos
Glandula verde crustaceos Contoh surat bayaran balik emolumen
Contoh surat bayaran balik emolumen Formula de distribucion de poisson
Formula de distribucion de poisson Perkembangan pengaruh barat pada masa kolonial
Perkembangan pengaruh barat pada masa kolonial Masa volumen y peso
Masa volumen y peso Masa disfagia
Masa disfagia 13 atom
13 atom Tantangan pendidikan pancasila di perguruan tinggi
Tantangan pendidikan pancasila di perguruan tinggi Alat ukur masa
Alat ukur masa