Introduccin a la Espectrometra de Masa Algunas aplicaciones








![Voyager Spec #1=>MC[BP = 2469. 5, 12005] 2 x. C 13 100 90 2093 Voyager Spec #1=>MC[BP = 2469. 5, 12005] 2 x. C 13 100 90 2093](https://slidetodoc.com/presentation_image_h/ff775a94677c40f54b63397eaa324596/image-14.jpg)





























- Slides: 62

Introducción a la Espectrometría de Masa Algunas aplicaciones de la Espectrometría de Masa en biología y biomedicina

Espectrometría de Masa • La Espectrometría de Masa es una técnica que permite determinar la masa de moléculas por medio de la medida de la relación masa/carga (m/z). • La medida de masas moleculares involucra la producción, separación y detección de iones moleculares en fase gaseosa. • Cada molécula puede originar iones moleculares con distinto número de cargas ( y distintas m/z). • La masa molecular es una propiedad física fundamental e inalterable de la materia……

Bioespectrometría • Aplicación de la Espectrometría de Masa al estudio de bio-moléculas: identificación, niveles, modificaciones. • Incluye la combinación de herramientas de la Bioquímica (cromatografía, electroforesis, bioafinidad y digestiones enzimáticas, entre otras) con la Espectrometría de Masa.

Espectrometría de masa y Biología Las medidas de masa de péptidos y proteínas (. . . y de otras macromoléculas) eran extremadamente difíciles de realizar hasta fines de los años 80’, cuando aparecieron dos nuevos métodos para producir iones en fase gaseosa: • Matrix Assisted Laser Desorption/Ionization (MALDI), para muestras sólidas • Ionización por “Electrospray” (ESI) para muestras líquidas

Espectrometría de masa: equipos de tipo MALDI-TOF Espectrómetro de masa MALDI TOF/TOF (Applied Biosystems Voyager DE-PRO) (Applied Biosystems 4800 Analizer)

ESQUEMA: Instrumento de tecnología MALDI-TOF plate Extraction grids Laser Reflector Timed ion detector selector Reflector Beam guide Camera Pumping Linear detector

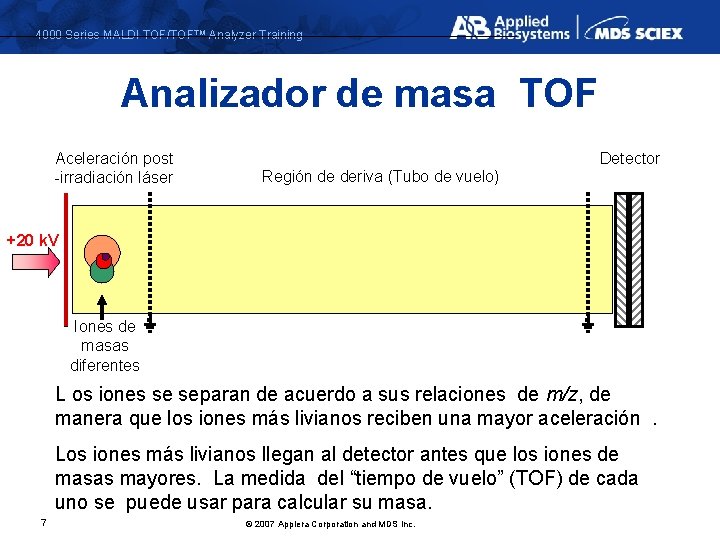
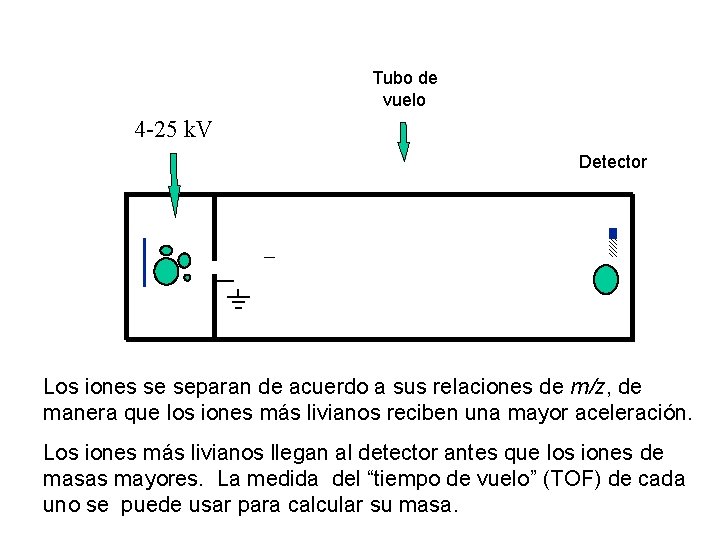
4000 Series MALDI TOF/TOF™ Analyzer Training Analizador de masa TOF Aceleración post -irradiación láser Detector Región de deriva (Tubo de vuelo) +20 k. V Iones de masas diferentes L os iones se separan de acuerdo a sus relaciones de m/z, de manera que los iones más livianos reciben una mayor aceleración. Los iones más livianos llegan al detector antes que los iones de masas mayores. La medida del “tiempo de vuelo” (TOF) de cada uno se puede usar para calcular su masa. 7 © 2007 Applera Corporation and MDS Inc.


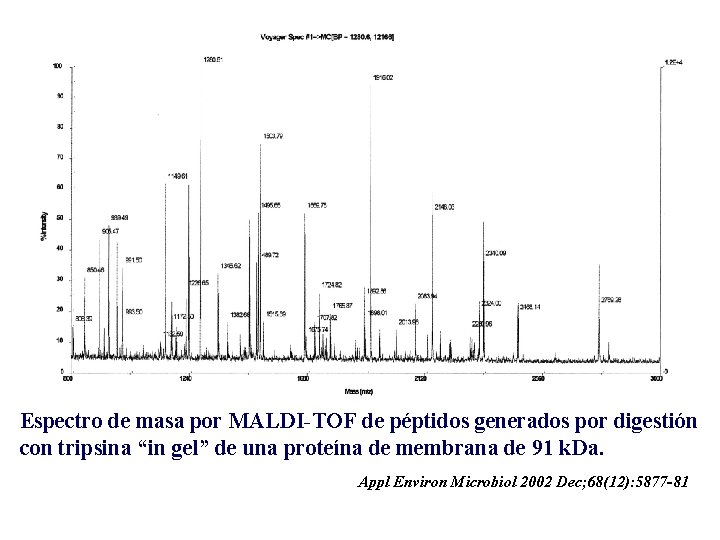
Espectro de masa por MALDI-TOF de péptidos generados por digestión con tripsina “in gel” de una proteína de membrana de 91 k. Da. Appl Environ Microbiol 2002 Dec; 68(12): 5877 -81

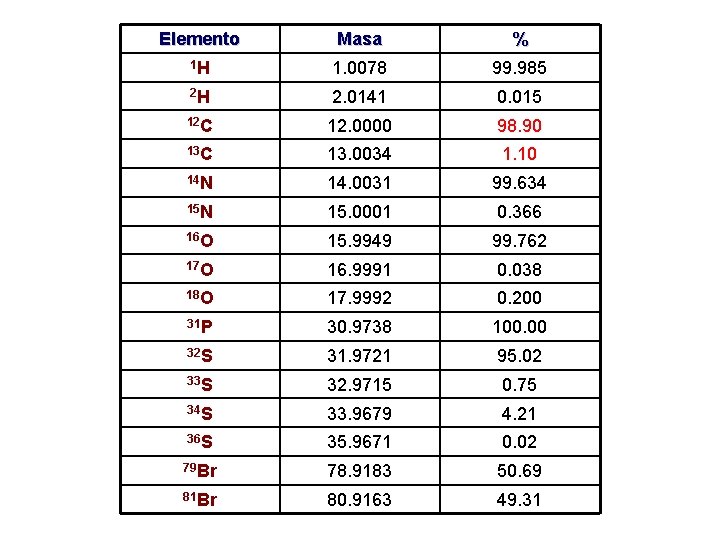
Elemento Masa % 1 H 1. 0078 99. 985 2 H 2. 0141 0. 015 12 C 12. 0000 98. 90 13 C 13. 0034 1. 10 14 N 14. 0031 99. 634 15 N 15. 0001 0. 366 16 O 15. 9949 99. 762 17 O 16. 9991 0. 038 18 O 17. 9992 0. 200 31 P 30. 9738 100. 00 32 S 31. 9721 95. 02 33 S 32. 9715 0. 75 34 S 33. 9679 4. 21 36 S 35. 9671 0. 02 79 Br 78. 9183 50. 69 81 Br 80. 9163 49. 31

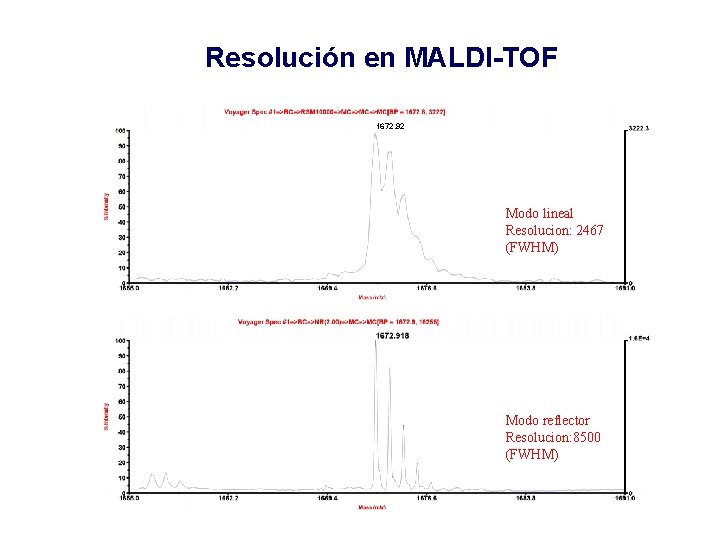
Resolución en MALDI-TOF 1672. 92 Modo lineal Resolucion: 2467 (FWHM) Modo reflector Resolucion: 8500 (FWHM)


![Voyager Spec 1MCBP 2469 5 12005 2 x C 13 100 90 2093 Voyager Spec #1=>MC[BP = 2469. 5, 12005] 2 x. C 13 100 90 2093](https://slidetodoc.com/presentation_image_h/ff775a94677c40f54b63397eaa324596/image-14.jpg)
Voyager Spec #1=>MC[BP = 2469. 5, 12005] 2 x. C 13 100 90 2093 C 13 80 % Intensity 70 60 50 40 C 12 30 20 10 0 5725 5729 5733 Mass (m/z) 5737 5741 5745 Espectro de masa con resolución isotópica de insulina bovina. (aprox. 0. 15 pmoles, 0. 8 ng, en 1 ml de muestra; matriz: CHCA; R= 13800 FWHM) 0

Algunas aplicaciones. . .

CARACTERIZACIÓN DE PROTEÍNAS POR ESPECTROMETRÍA DE MASA • Identificación de proteínas – comparación correlativa en una base de datos de secuencias • Control de calidad – proteínas recombinantes, reacciones de modificación • Modificaciones postraduccionales – fosforilación, glicosilación, metilación, etc. • Identificación de dominios – dominios estructurales funcionales, pro-enzima vs. enzima • Niveles de expresión – métodos cuantitativos

Espectro de masa del citocromo c de caballo A. Molécula entera. Matriz: ácido sinapínico Voyager Spec #1=>BC=>NF 0. 9[BP = 12364. 9, 60367] 12366. 46 100 6. 0 E+4 90 80 6187. 17 70 12583. 58 % Intensity 60 50 12326. 17 40 30 20 6292. 68 12784. 48 10 0 5000 7000 9000 11000 Mass (m/z) 13000 0 15000

Mapeo peptídico por EM (Peptide Mass Fingerprinting/MS) proteína intacta péptidos enzima proteolítica MEMEKEFEQIDKSGSWAAIYQDIRHEASDFPCRVAKLPKNKNRNRYRDVS PFDHSRIKLHQEDNDYINASLIKMEEAQRSYILTQGPLPNTCGHFWEMVW EQKSRGVVMLNRVMEKGSLKCAQYWPQKEEKEMIFEDTNLKLTLISEDIK SYYTVRQLELENLTTQETREILHFHYTTWPDFGVPESPASFLNFLFKVRE SGSLSPEHGPVVVHCSAGIGRSGTFCLADTCLLLMDKRKDPSSVDIKKVL LEMRKFRMGLIQTADQLRFSYLAVIEGAKFIMGDSSVQDQWKELSHEDLE PPPEHIPPPPRPPKRILEPHNGKCREFFPNHQWVKEETQEDKDCPIKEEK GSPLNAAPYGIESMSQDTEVRSRVVGGSLRGAQAASPAKGEPSLPEKDED HALSYWKPFLVNMCVATVLTAGAYLCYRFLFNSNT

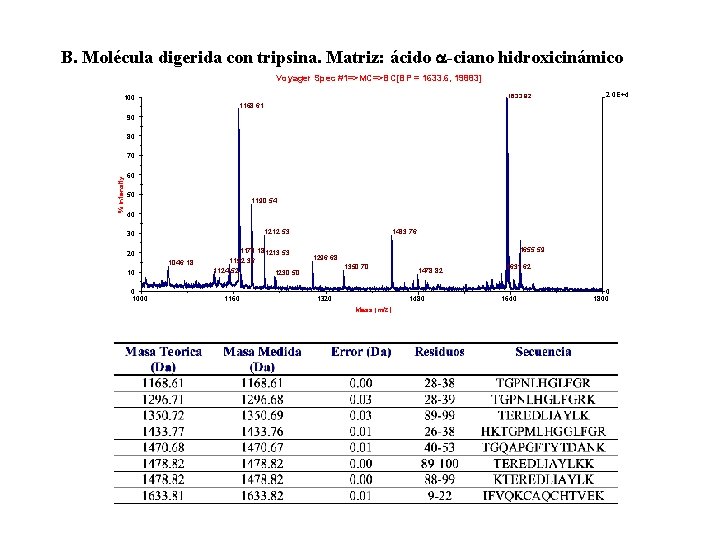
B. Molécula digerida con tripsina. Matriz: ácido -ciano hidroxicinámico Voyager Spec #1=>MC=>BC[BP = 1633. 6, 19883] 163 3. 82 100 2. 0 E+4 1168. 61 90 80 % Intensity 70 60 50 1190. 54 40 1212. 53 30 20 1046. 18 10 0 1000 1170. 181213. 53 1152. 36 1124. 52 1230. 50 1160 1433. 76 1655. 59 1296. 68 1350. 70 1320 1478. 82 1480 Mass (m/z) 1631. 62 1640 0 1800

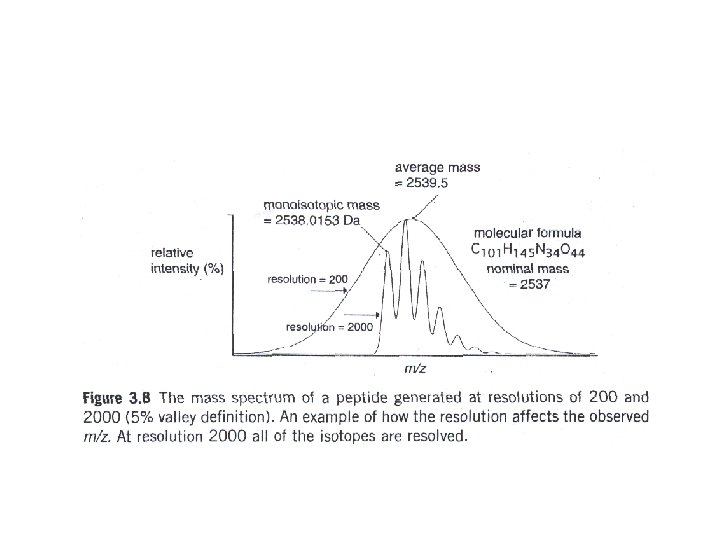
RESUMEN: Niveles de información……… en la caracterización de proteínas por MS. • medida de masa de una molécula entera • medidas de masas en mapeo peptídico • secuenciado de péptidos por MS/MS 1 valor experimental 5 -20 valores experimentales más de 100 valores experimentales …mejora en cantidad y calidad de la información

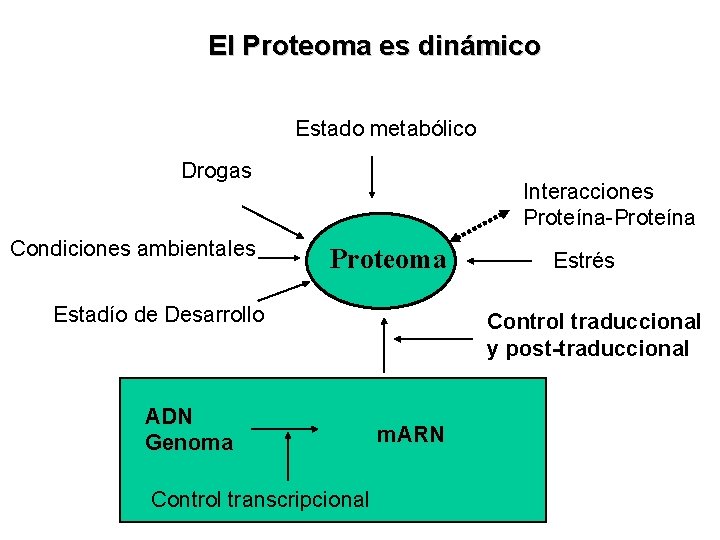
El Proteoma es dinámico Estado metabólico Drogas Condiciones ambientales Interacciones Proteína-Proteína Proteoma Estadío de Desarrollo ADN Genoma Control transcripcional Estrés Control traduccional y post-traduccional m. ARN

Análisis por electroforesis 2 D

Identificación de proteínas 1000 m/z Mass (m/z) 1500 2000 Extracción de péptidos y análisis de masas Separación en gel 2 D Selección de la muestra Tratamiento con una enzima proteolítica Búsqueda en banco de datos

Protein Prospector Homepage MS-Fit Search

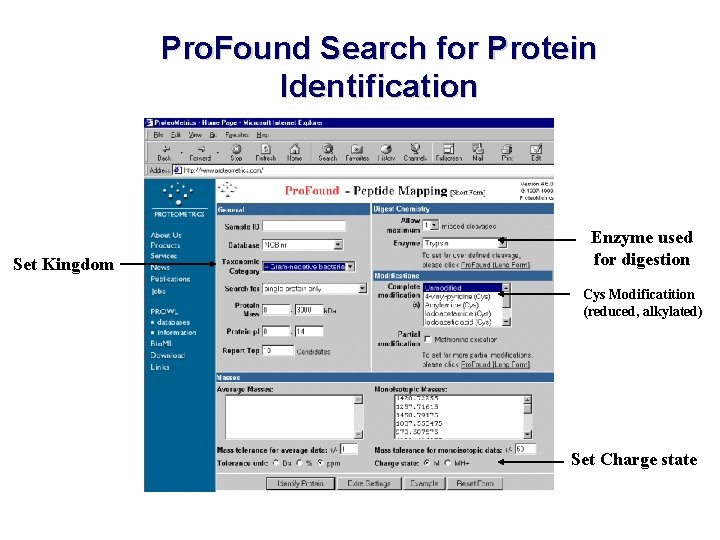
Pro. Found Search for Protein Identification Set Kingdom Enzyme used for digestion Cys Modificatition (reduced, alkylated) Set Charge state

Search Result after Peptide Mass Fingerprinting The result showed that in fact the spot contained 2 proteins. Both were identified, Vimentin and Tubulin. Up to 5 proteins in the same spot could potentially be identified. Note that the genome of CHO-cells (hamster) is not sequenced. Therefore the most homologues proteins of related species are scored.

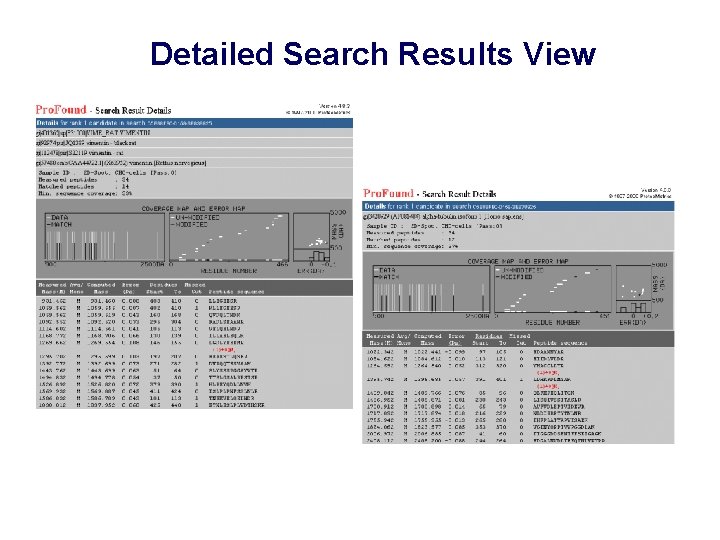
Detailed Search Results View

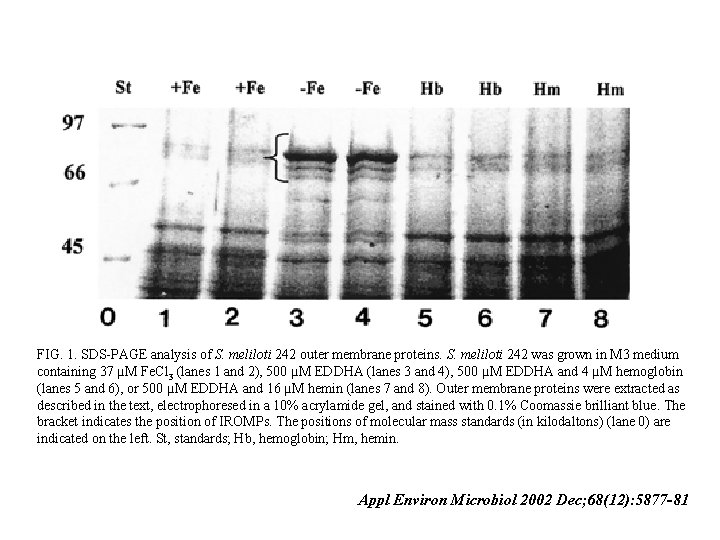
Identificación de una proteína regulada por hierro en membrana externa de Sinorhizobium meliloti Appl Environ Microbiol 2002 Dec; 68(12): 5877 -81

FIG. 1. SDS-PAGE analysis of S. meliloti 242 outer membrane proteins. S. meliloti 242 was grown in M 3 medium containing 37 µM Fe. Cl 3 (lanes 1 and 2), 500 µM EDDHA (lanes 3 and 4), 500 µM EDDHA and 4 µM hemoglobin (lanes 5 and 6), or 500 µM EDDHA and 16 µM hemin (lanes 7 and 8). Outer membrane proteins were extracted as described in the text, electrophoresed in a 10% acrylamide gel, and stained with 0. 1% Coomassie brilliant blue. The bracket indicates the position of IROMPs. The positions of molecular mass standards (in kilodaltons) (lane 0) are indicated on the left. St, standards; Hb, hemoglobin; Hm, hemin. Appl Environ Microbiol 2002 Dec; 68(12): 5877 -81

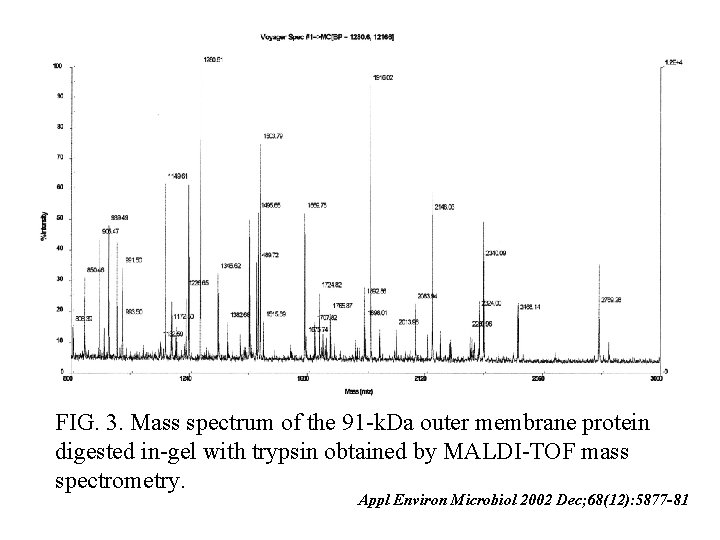
FIG. 3. Mass spectrum of the 91 -k. Da outer membrane protein digested in-gel with trypsin obtained by MALDI-TOF mass spectrometry. Appl Environ Microbiol 2002 Dec; 68(12): 5877 -81


Appl Environ Microbiol 2002 Dec; 68(12): 5877 -81

Identificación de proteínas por MALDI-TOF • Proteína aislada • Proteína identificada por la masa de los péptidos (PMF), generados por un método específico: – Enzimático o químico • Para su identificación, la secuencia de la proteína debe estar en una base de datos • La identificación es probabilística: según criterios estadísticos

Caracterización de proteínas por espectrometría de masa • Identificación de proteínas – comparación correlativa en una base de datos de secuencias • Control de calidad – proteínas recombinantes, reacciones de modificación • Modificaciones postraduccionales – fosforilación, glicosilación, metilación, etc. • Identificación de dominios – dominios estructurales funcionales, pro-enzima vs. enzima • Niveles de expresión – métodos cuantitativos

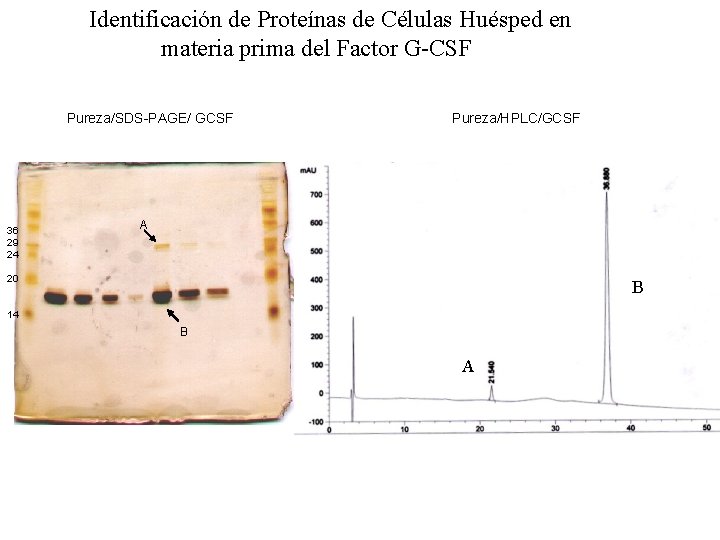
Identificación de Proteínas de Células Huésped en materia prima del Factor G-CSF Pureza/SDS-PAGE/ GCSF 36 29 24 Pureza/HPLC/GCSF A 20 B 14 B A

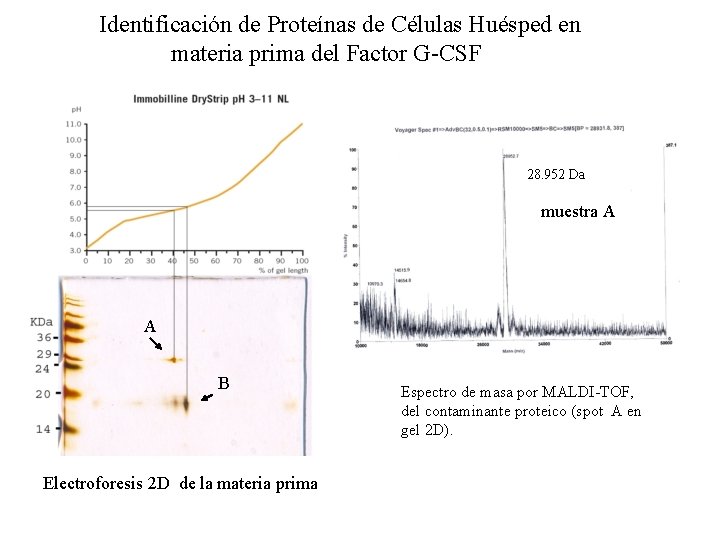
Identificación de Proteínas de Células Huésped en materia prima del Factor G-CSF 28. 952 Da muestra A A B Electroforesis 2 D de la materia prima Espectro de masa por MALDI-TOF, del contaminante proteico (spot A en gel 2 D).

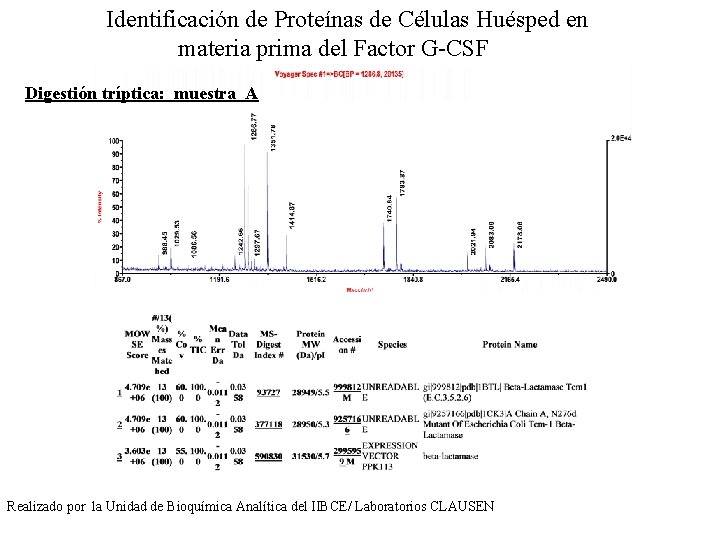
Identificación de Proteínas de Células Huésped en materia prima del Factor G-CSF Digestión tríptica: muestra A Realizado por la Unidad de Bioquímica Analítica del IIBCE/ Laboratorios CLAUSEN

Caracterización de proteínas por espectrometría de masa • Identificación de proteínas – comparación correlativa en una base de datos de secuencias • Control de calidad – proteínas recombinantes, reacciones de modificación • Modificaciones postraduccionales – fosforilación, glicosilación, metilación, etc. • Identificación de dominios – dominios estructurales funcionales, pro-enzima vs. enzima • Niveles de expresión – métodos cuantitativos

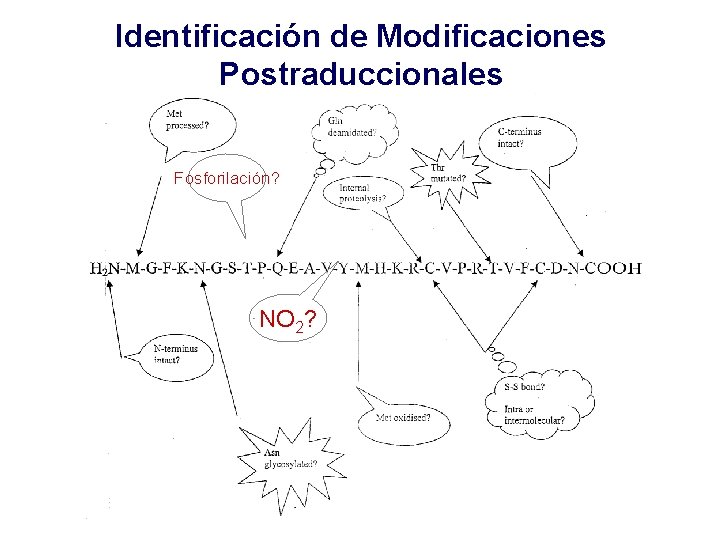
Identificación de Modificaciones Postraduccionales Fosforilación? NO 2?

Common Protein Post-Translational Modifications ∆ (monoisotopic) -17 -2 -0. 98 2 14 16 28 42 43 44 71 79. 9568 79. 9663 modification pyrrolidone carboxylic acid disulfide formation (thiol oxidation) amidation Some modifications are natural. deamidation Many are the result of handling. disulfide reduction (thiol formation) methylation oxidation (e. g. sulfoxide formation) formylation acetylation disulfide vs. two thiols carbamylation phosphate group vs. sulfate group g-carboxyglutamic acid good mass spectrometer needed Cys-propionamide especially with large molecules sulfation phosphorylation M. R. Wilkins et al. , J. Mol. Biol. 289: 645 -657 (1999). Delta Mass at http: //www. abrf. org

Identificación de sitios de fosforilación Pkn. B de Mycobaterium tuberculosis 1 51 101 151 201 251 301 GSSHHHHHHSSGLVPRGSHMTTPSHLSDRYELGEILGFGGMSEVHLARDLRLH RDVAVKVLRADLARDPSFYLRFRREAQNAAALNHPAIVAVYDTGEAE TPAGPLPYIVMEYVDGVTLRDIVHTEGPMTPKRAIEVIADACQALNFSHQ NGIIHRDVKPANIMISATNAVKVMDFGIARAIADSGNSVTQTAAVIGTAQ YLSPEQARGDSVDARSDVYSLGCVLYEVLTGEPPFTGDSPVSVAYQHVR EDPIPPSARHEGLSADLDAVVLKALAKNPENRYQTAAEMRADLVRVHNGEP PEAPKVLTDAERTSLLSSAAGNLSGPRTDPLPRQDLDDTDRDRSIGSVGR


1 51 101 151 201 251 301 GSSHHHHHHSSGLVPRGSHMTTPSHLSDRYELGEILGFGGMSEVHLARDLRLH RDVAVKVLRADLARDPSFYLRFRREAQNAAALNHPAIVAVYDTGEAE TPAGPLPYIVMEYVDGVTLRDIVHTEGPMTPKRAIEVIADACQALNFSHQ NGIIHRDVKPANIMISATNAVKVMDFGIARAIADSGNSVp. TQp. TAAVIGTAQ YLSPEQARGDSVDARSDVYSLGCVLYEVLTGEPPFTGDSPVSVAYQHVR EDPIPPSARHEGLSADLDAVVLKALAKNPENRYQTAAEMRADLVRVHNGEP PEAPKVLTDAERTSLLSSAAGNLSGPRTDPLPRQDLDDTDRDRSIGSVGR

Caracterización de proteínas por espectrometría de masa • Identificación de proteínas – comparación correlativa en una base de datos de secuencias • Control de calidad – proteínas recombinantes, reacciones de modificación • Modificaciones postraduccionales – fosforilación, glicosilación, metilación, etc. • Identificación de dominios – dominios estructurales funcionales, pro-enzima vs. enzima • Niveles de expresión – métodos cuantitativos

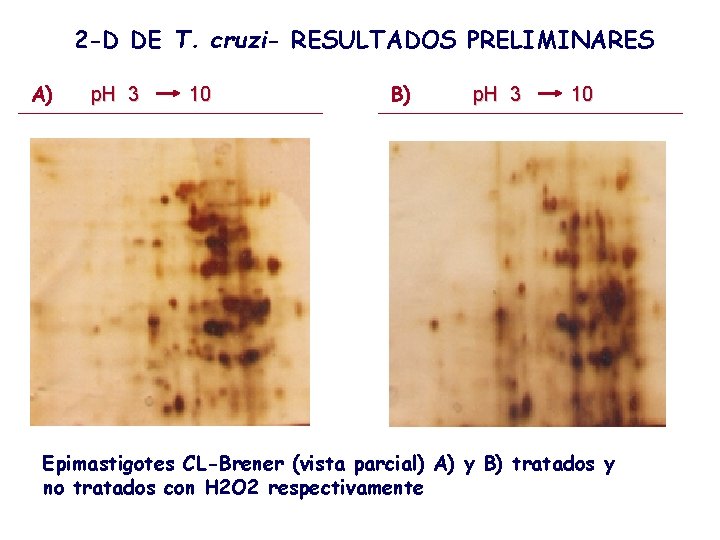
2 -D DE T. cruzi- RESULTADOS PRELIMINARES A) p. H 3 10 B) p. H 3 10 Epimastigotes CL-Brener (vista parcial) A) y B) tratados y no tratados con H 2 O 2 respectivamente

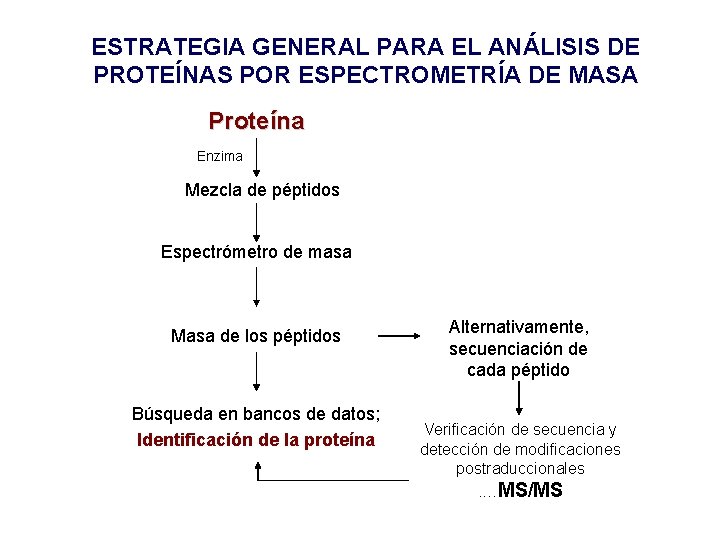
ESTRATEGIA GENERAL PARA EL ANÁLISIS DE PROTEÍNAS POR ESPECTROMETRÍA DE MASA Proteína Enzima Mezcla de péptidos Espectrómetro de masa Masa de los péptidos Búsqueda en bancos de datos; Identificación de la proteína Alternativamente, secuenciación de cada péptido Verificación de secuencia y detección de modificaciones postraduccionales. …MS/MS

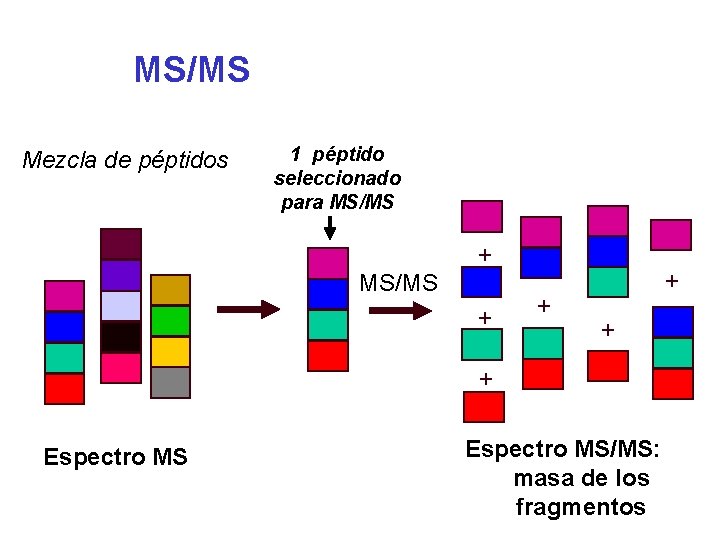
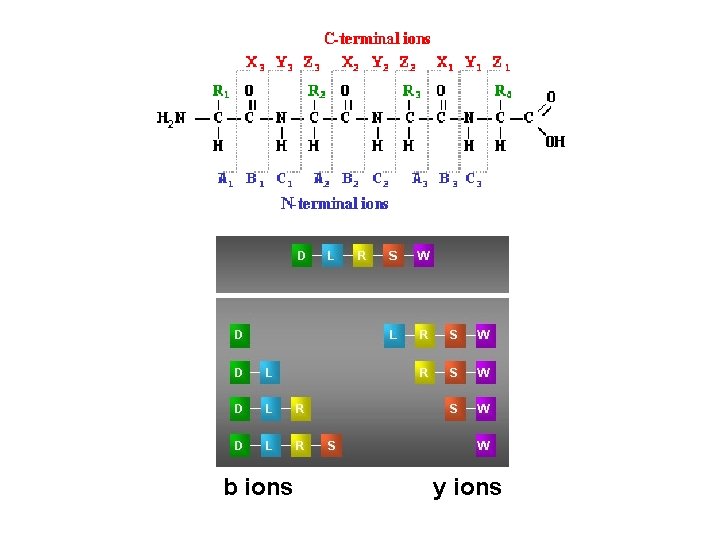
MS/MS Mezcla de péptidos 1 péptido seleccionado para MS/MS + + + Espectro MS/MS: masa de los fragmentos

b ions y ions


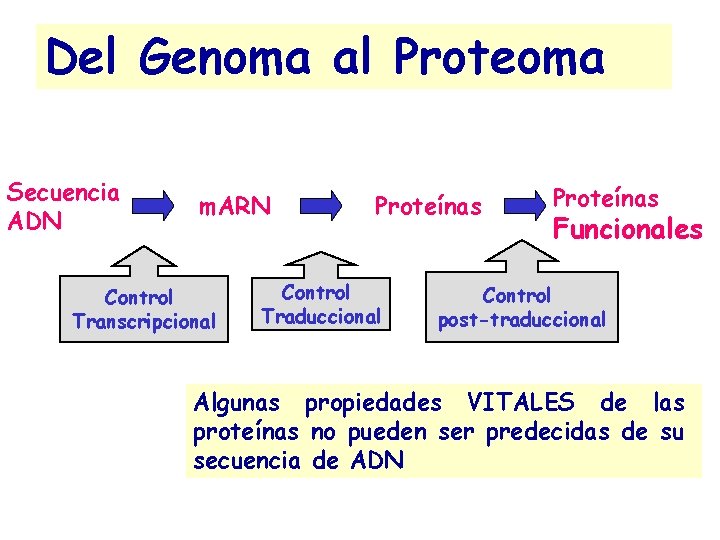
Del Genoma al Proteoma Secuencia ADN m. ARN Control Transcripcional Proteínas Control Traduccional Proteínas Funcionales Control post-traduccional Algunas propiedades VITALES de las proteínas no pueden ser predecidas de su secuencia de ADN

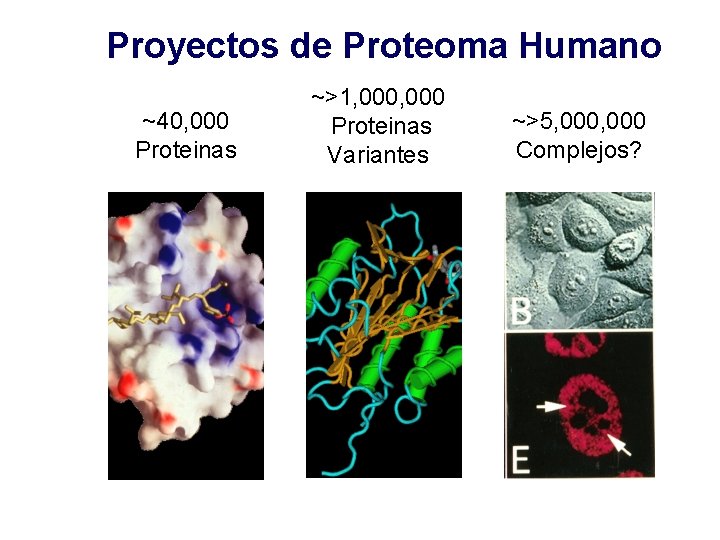
Proyectos de Proteoma Humano ~40, 000 Proteinas ~>1, 000 Proteinas Variantes ~>5, 000 Complejos?

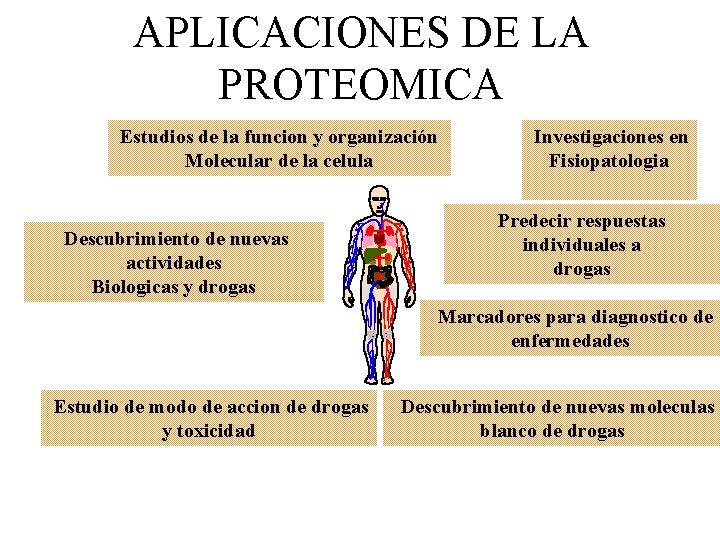
APLICACIONES DE LA PROTEOMICA Estudios de la funcion y organización Molecular de la celula Descubrimiento de nuevas actividades Biologicas y drogas Investigaciones en Fisiopatologia Predecir respuestas individuales a drogas Marcadores para diagnostico de enfermedades Estudio de modo de accion de drogas y toxicidad Descubrimiento de nuevas moleculas blanco de drogas






Linear Detector 4800 MALDI TOF/TOF™ Analyzer Schematic 2 -Stage Mirror Lens 3 Mirror Deflectors Metastable Suppressor Collision Cell Source 2 CID Lens (Lens 2) Decel Stack Lens 1 Camera Laser Decel Deflectors Lamp TIS X 2, Y 2 Deflectors Attenuator Reflector Detector X 1, Y 1 Deflectors Sample Loading Chamber Mirrors Source 1 Lens Sample Plate and Stage

Tubo de vuelo 4 -25 k. V Detector Los iones se separan de acuerdo a sus relaciones de m/z, de manera que los iones más livianos reciben una mayor aceleración. Los iones más livianos llegan al detector antes que los iones de masas mayores. La medida del “tiempo de vuelo” (TOF) de cada uno se puede usar para calcular su masa.

Espectrómetro de masa MALDI TOF/TOF (Applied Biosystems 4800 Analizer)

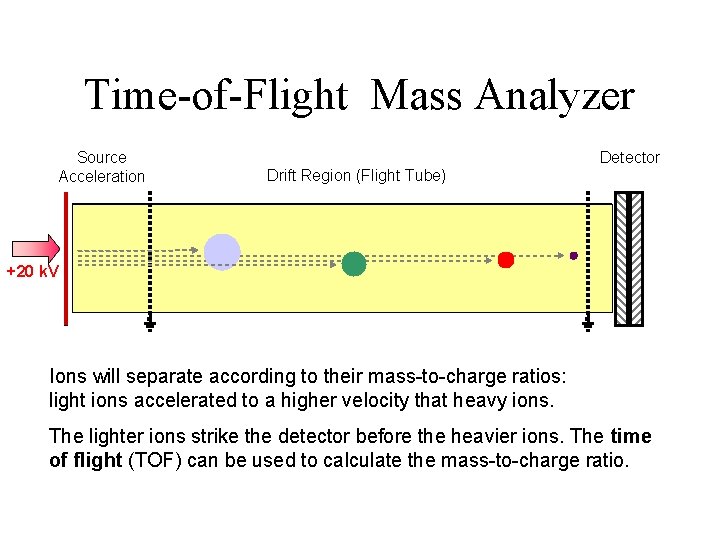
Time-of-Flight Mass Analyzer Source Acceleration Detector Drift Region (Flight Tube) +20 k. V Ions will separate according to their mass-to-charge ratios: light ions accelerated to a higher velocity that heavy ions. The lighter ions strike the detector before the heavier ions. The time of flight (TOF) can be used to calculate the mass-to-charge ratio.

• Modelo: Voyager DE-PRO (Applied Biosystems, USA). • Datos técnicos: • Posibilidades de operación: - Modo lineal - Laser de nitrógeno ( 337 nm) - Modo reflector - Voltaje de aceleración: hasta 25 k. V - "Delay extraction" (DE) - Tubo de vuelo: 1. 3 m (modo lineal); 2. 0 m (modo reflector) - Sistema alto vacío: 2 bombas turbo-moleculares (10 -8 Torr) - "Post Source Decay“ (PSD) - "Collision-Induced Dissociation“ (CID) - Rango de masa: 500 Da hasta > 400 k. Da - Exactitud de masa : hasta 0. 002% (20 ppm) - Sensibilidad (péptidos): subpicomolar
 Introduccin
Introduccin Induccin
Induccin Introduccin
Introduccin Enzimas introducción
Enzimas introducción Introduccin
Introduccin Introduccin
Introduccin Tantangan pendidikan di masa depan
Tantangan pendidikan di masa depan Relativna atomska masa i relativna molekulska masa
Relativna atomska masa i relativna molekulska masa Factores que afectan la solubilidad
Factores que afectan la solubilidad Peso molecular
Peso molecular Perkembangan mobil dari masa ke masa
Perkembangan mobil dari masa ke masa Perkembangan desain dari masa ke masa
Perkembangan desain dari masa ke masa Masa molar ba(oh)2
Masa molar ba(oh)2 Perkembangan hardware dari masa ke masa
Perkembangan hardware dari masa ke masa Antiguamente se realizaban algunas transfusiones de sangre
Antiguamente se realizaban algunas transfusiones de sangre Empieza aumentar la temperatura y algunas plantas florecen
Empieza aumentar la temperatura y algunas plantas florecen Causas de las enfermedades en la biblia
Causas de las enfermedades en la biblia Que es el calor especifico
Que es el calor especifico Demostrativos indefinidos
Demostrativos indefinidos A veces llegan personas a tu vida
A veces llegan personas a tu vida Verbo que cambia su raiz en algunas personas
Verbo que cambia su raiz en algunas personas Objeto estético
Objeto estético Pila redox
Pila redox Aplicaciones de la integral longitud de arco
Aplicaciones de la integral longitud de arco El efecto fotoeléctrico
El efecto fotoeléctrico Evaporador de tubos verticales con circulación natural
Evaporador de tubos verticales con circulación natural Aplicaciones del eter
Aplicaciones del eter Aparatos que utilizan imanes
Aparatos que utilizan imanes Aplicaciones de la espectrometría de masas
Aplicaciones de la espectrometría de masas Función cuadrática en la vida cotidiana
Función cuadrática en la vida cotidiana Grafo rueda
Grafo rueda Aplicaciones de la termodinámica
Aplicaciones de la termodinámica Aplicaciones logica difusa
Aplicaciones logica difusa Aplicaciones del bloque d
Aplicaciones del bloque d Aplicaciones de ecuaciones diferenciales de primer orden
Aplicaciones de ecuaciones diferenciales de primer orden Sensor fotoelétrico
Sensor fotoelétrico Lenguaje ensamblador aplicaciones
Lenguaje ensamblador aplicaciones Desarrollo de aplicaciones web con asp.net
Desarrollo de aplicaciones web con asp.net Características del ácido clorhídrico
Características del ácido clorhídrico Aplicaciones de la electrólisis
Aplicaciones de la electrólisis Aplicaciones de la polarimetria
Aplicaciones de la polarimetria Vlvulas
Vlvulas Taller sobre aplicaciones en la administración
Taller sobre aplicaciones en la administración Aplicaciones de regresion lineal simple
Aplicaciones de regresion lineal simple Aplicaciones de la dinámica
Aplicaciones de la dinámica Induccion electromagnetica
Induccion electromagnetica Aplicaciones de la energía eólica
Aplicaciones de la energía eólica Tabla benzodiacepinas
Tabla benzodiacepinas Las aplicaciones mas usadas
Las aplicaciones mas usadas Materiales compuestos aplicaciones
Materiales compuestos aplicaciones Diodo schottky definicion
Diodo schottky definicion Boroeste
Boroeste Ley de ohm aplicaciones
Ley de ohm aplicaciones Aplicaciones del principio de pascal
Aplicaciones del principio de pascal Integrales impropias aplicaciones
Integrales impropias aplicaciones Ejemplos de reacciones redox
Ejemplos de reacciones redox Aplicaciones del principio del trabajo virtual
Aplicaciones del principio del trabajo virtual Aplicaciones de la derivada concepto
Aplicaciones de la derivada concepto Aplicaciones de los resortes
Aplicaciones de los resortes Aplicaciones de la pnl
Aplicaciones de la pnl Valor absoluto esquema
Valor absoluto esquema Aplicaciones tecnologicas
Aplicaciones tecnologicas Derivadas en la vida
Derivadas en la vida