Chr Exon Intron Gene 5UTR 3UTR CDS GFFgeneral



































- Slides: 40


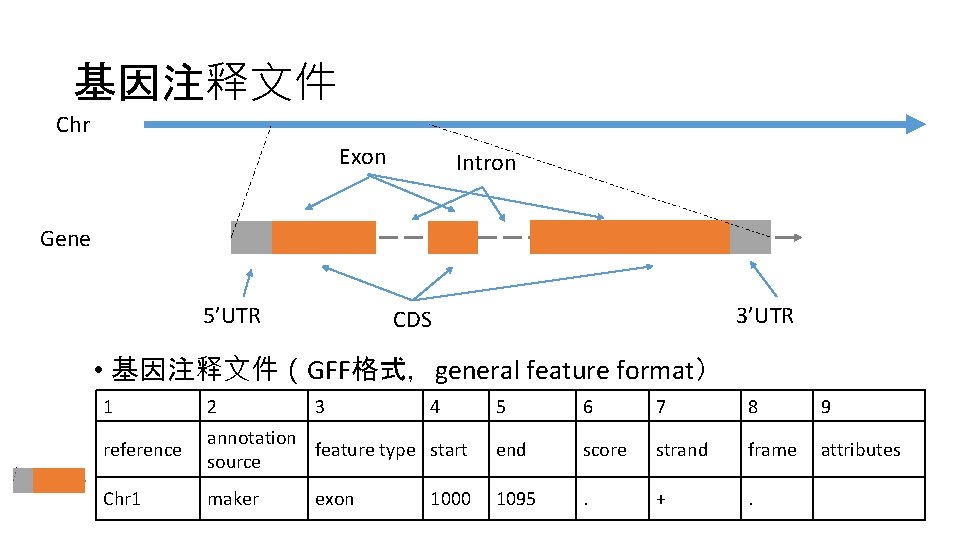
基因注释文件 Chr Exon Intron Gene 5’UTR 3’UTR CDS • 基因注释文件(GFF格式,general feature format) 1 2 reference Chr 1 3 5 6 7 8 9 annotation feature type start source end score strand frame attributes maker 1095 . + . exon 4 1000

作业 – 下载基因组 • 文献阅读 (https: //plabipd. de/timeline_view. ep) • 在线数据库 • http: //www. tripal. info/sites_using_tripal • https: //phytozome. jgi. doe. gov/pz/portal. html • http: //plants. ensembl. org/index. html • 其它数据库,例如NCBI,Giga. DB等







作业 – 下载基因组 • FASTA文件,GFF文件格式说明 • 基因组,基因注释,m. RNA,蛋白序列 • 数据库演示 • http: //www. tripal. info/sites_using_tripal • https: //phytozome. jgi. doe. gov/pz/portal. html • http: //plants. ensembl. org/index. html

作业 – 统计Feature数量 什么是Feature -- a piece of a sequence http: //gmod. org/wiki/Chado_Sequence_Module#Features 基因注释文件中Feature信息 1 2 reference Chr 1 3 5 6 7 8 9 annotation feature type start source end score strand frame attributes maker 1095 . + . exon 4 1000 统计Feature数量 cut -f 3 input. gff 3 | grep –v -P “^#” | sort | uniq –c

作业 – 获取m. RNA及蛋白序列 1 GFF格式转换为GTF $gffread -T -o output. gtf input. gff $cut –f 3 output. gtf | sort | uniq -c 2 提取m. RNA序列 $gffread -w m. RNA. fa -g genome. fa input. gtf 3 提取蛋白序列 $gffread -y protein. fa -g genome. fa input. gtf

作业 – 作业报告 1 文件名:genome. yaml Genus: Malus Species: Domestica URL: ftp: //ftp. bioinfo. wsu. edu/species/Malus_x_domestica/ Malus_x_domestica-genome_GDDH 13_v 1. 1 Pub: https: //www. nature. com/articles/ng. 3886 gene: 52741 m. RNA: 45116 nc. RNA: 7625 Exon: 234986 CDS: 215867 five_prime_UTR: 38129 three_prime_UTR: 36043 SNum: 2018 NNNNNN Name: Zhang San

作业 – 数据整理 1 建立目录,将所有文件放入该目录 genus_species_cultivar_version 2 利用软连接对文件进行统一命名 $ln $ln –s –s original_genome_name. fa genome. fa original_m. RNA. fa original_protein. fa original_genes. gff gene_models. gff


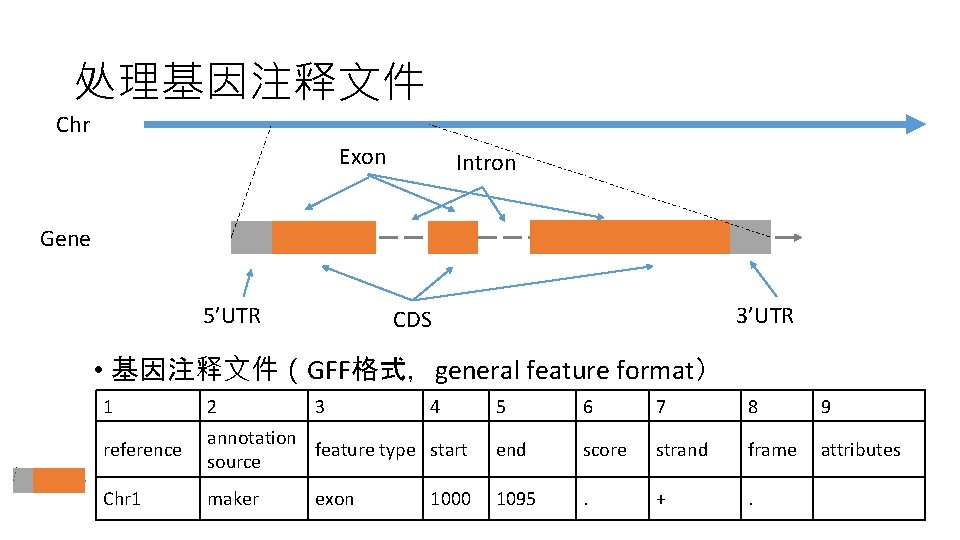
处理基因注释文件 Chr Exon Intron Gene 5’UTR 3’UTR CDS • 基因注释文件(GFF格式,general feature format) 1 2 reference Chr 1 3 5 6 7 8 9 annotation feature type start source end score strand frame attributes maker 1095 . + . exon 4 1000


处理基因注释文件 • 基因注释文件(GFF格式,general feature format) 1 2 3 reference sequence name annotation feature source type 4 5 6 start end score coordinate 7 8 9 strand frame attributes • 基因注释文件(BED格式,Browser Extensible Data) • 12列,前三列是必须的 chrom, chrom. Start, chrom. End, feature. ID, score, strand http: //grch 37. ensembl. org/info/website/upload/bed. html

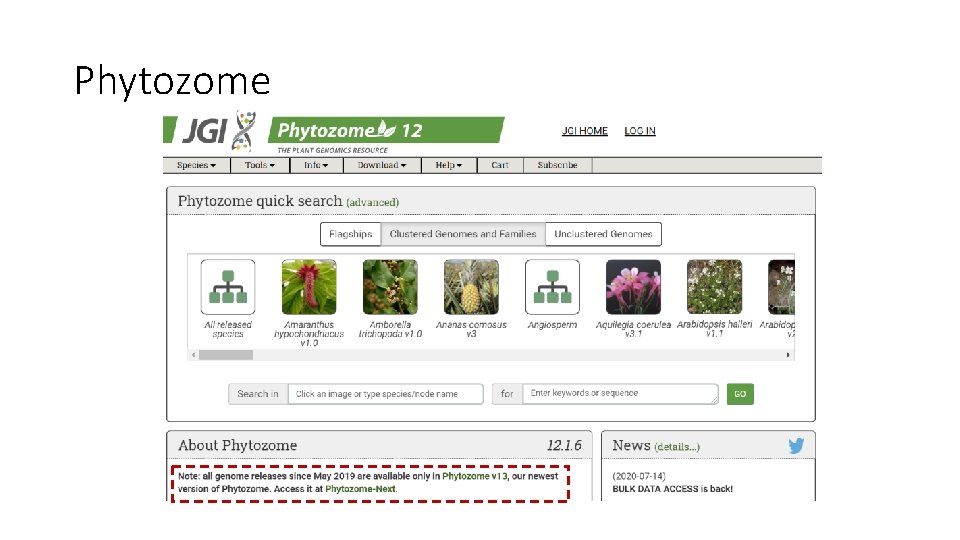
Phytozome



Beijing University of Agriculture Huilongguan, Changping District








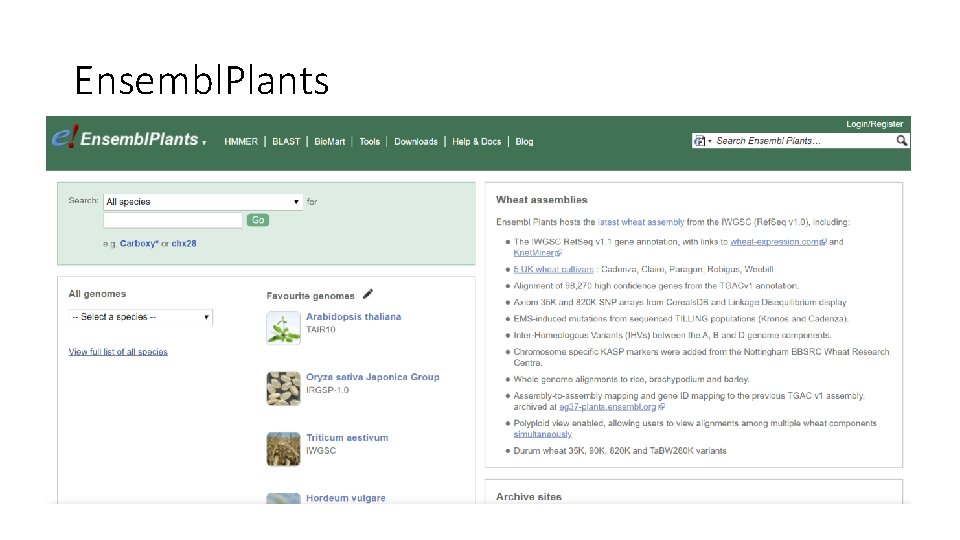
Ensembl. Plants









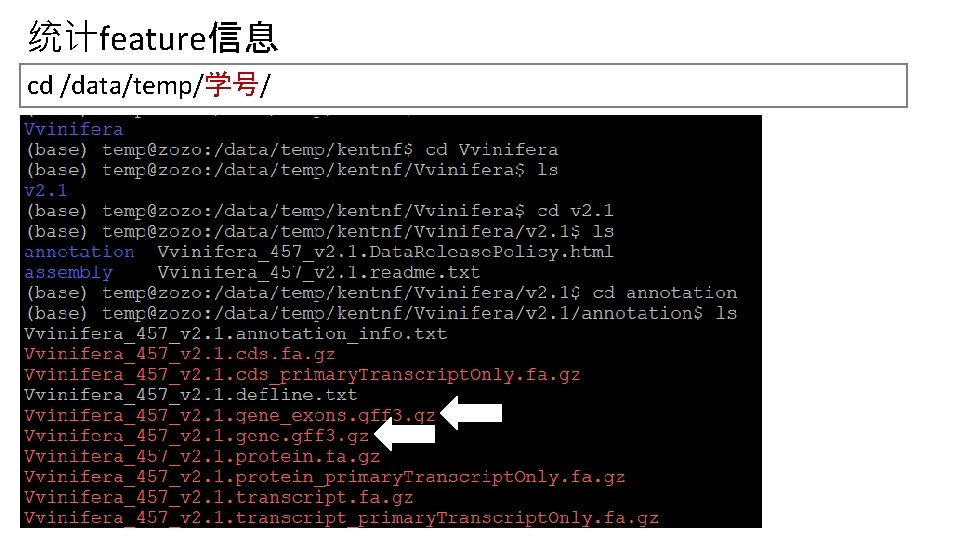
统计feature信息 cd /data/temp/学号/

统计feature信息 找到对应的基因注释文件(GFF 3格式),先解压缩,然后生成 feature_count 文件 gunzip Vvinifera_457_v 2. 1. gene_exons. gff 3. gz cut -f 3 Vvinifera_457_v 2. 1. gene_exons. gff 3 | sort | uniq -c > feature_count. txt 1 297312 31845 1 55564 ##annot-version v 2. 1 CDS exon gene ##gff-version 3 m. RNA Deadline: Nov 30
 Exon intron promoter
Exon intron promoter Taqman gene expression assay
Taqman gene expression assay Intron splicing
Intron splicing Kromatin
Kromatin Exon
Exon Met exon 14
Met exon 14 Exon shuffling spiegazione
Exon shuffling spiegazione Intrones
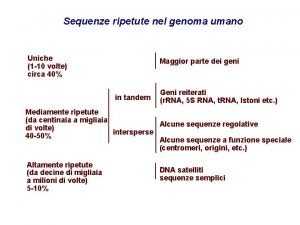
Intrones Sequenze ripetute
Sequenze ripetute Exon and lifo
Exon and lifo Paradosso del valore c
Paradosso del valore c Exon 18 g719x
Exon 18 g719x Gene by gene test results
Gene by gene test results Chapter 17 from gene to protein
Chapter 17 from gene to protein 1500 n chr
1500 n chr Krhz
Krhz Chr 13
Chr 13 John chr. aasted
John chr. aasted Cholecystitis chr calculosa
Cholecystitis chr calculosa 2 chr 7:14
2 chr 7:14 Chr(10) in sql
Chr(10) in sql Soins infirmiers spécifiques metz
Soins infirmiers spécifiques metz 10000 v chr
10000 v chr Chr pascal
Chr pascal Glr chr
Glr chr 753 v chr
753 v chr Kidney with ureter
Kidney with ureter Mbshc
Mbshc Educause cds
Educause cds Cds onap
Cds onap Gianni sansonne schemi operativi 2020
Gianni sansonne schemi operativi 2020 A library has 6 422 music cds stored on 26 shelves
A library has 6 422 music cds stored on 26 shelves Tipuri de curriculum
Tipuri de curriculum Cds care
Cds care Obtional sau optional
Obtional sau optional Audio learning resources
Audio learning resources The committee debates or debate these questions carefully
The committee debates or debate these questions carefully Cln cds
Cln cds Art 186 cds
Art 186 cds Risk meter flood
Risk meter flood Haltbarkeit blue ray
Haltbarkeit blue ray