TRANSCRIPCIN DEL ADN EN ARN Transcripcin l Es













- Slides: 46

TRANSCRIPCIÓN DEL ADN EN ARN

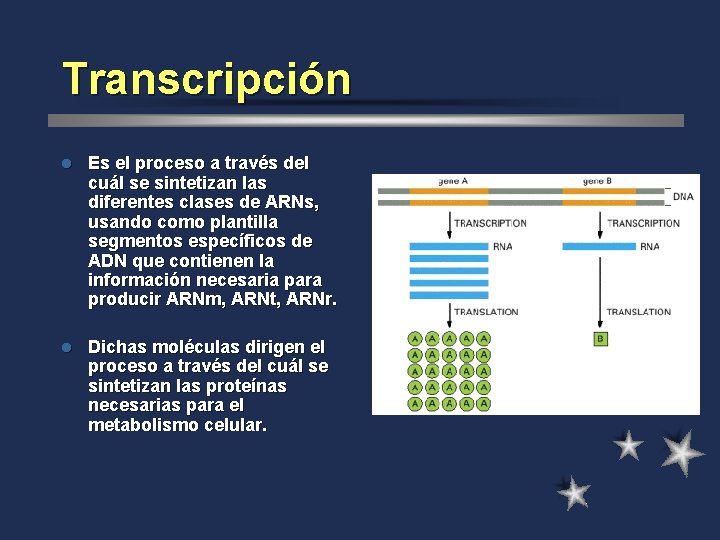
Transcripción l Es el proceso a través del cuál se sintetizan las diferentes clases de ARNs, usando como plantilla segmentos específicos de ADN que contienen la información necesaria para producir ARNm, ARNt, ARNr. l Dichas moléculas dirigen el proceso a través del cuál se sintetizan las proteínas necesarias para el metabolismo celular.

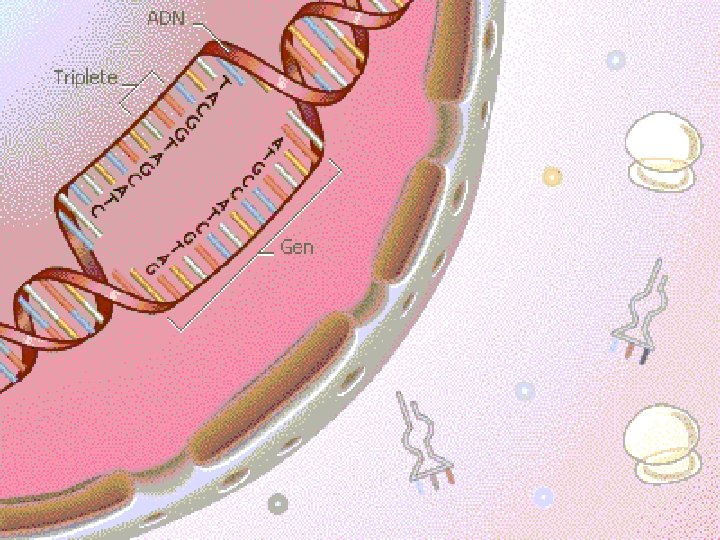
Tipos de ARN r : l constituye a los ribosomas l ARN m : contiene el mensaje genético, la estructura primaria de la l proteínas l ARN t : transfiere los amino-acidos en la cadena de síntesis proteica l Todos los ARNs están contenidos en moléculas mas grandes llamadas pre ARN Además existen otros tipos de ARNs con actividad catalítica, que son ARNsn, ARNsc, ARNsno.

Transcripción en Procariotas Los procariotas poseen una sola enzima para transcribir sus ARNs, la ARN polimerasa. l Cataliza la polimerización de ribonucleótidos a partir de un molde ADN únicamente en dirección 5´ -- 3´. l La ARN polimerasa es una enzima compleja compuesta de 4 cadenas diferentes α, β, β´y s. l

Transcripción en Procariotas La subunidad s sirve para unirse específicamente al sitio de inicio de la transcripción, denominado sitio promotor l El sitio promotor en procariontes se encuentra ubicado a -10 o -35 hacia la izquierda a partir del nucleótido +1 que es el primero de la secuencia génica. l

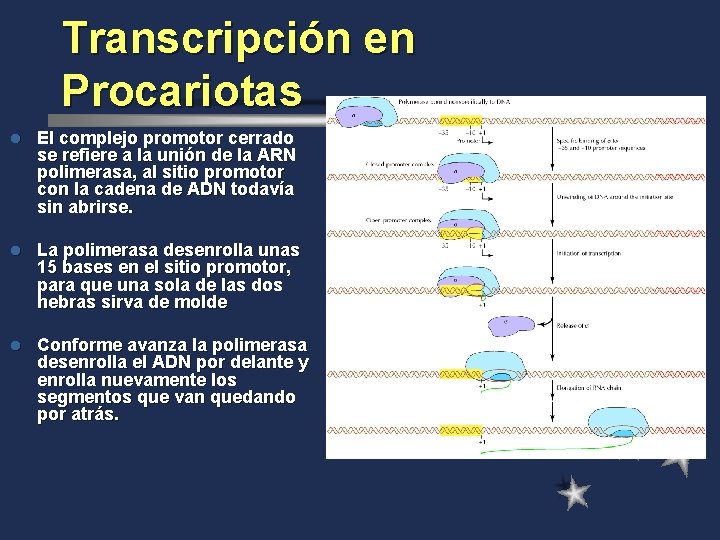
Transcripción en Procariotas l El complejo promotor cerrado se refiere a la unión de la ARN polimerasa, al sitio promotor con la cadena de ADN todavía sin abrirse. l La polimerasa desenrolla unas 15 bases en el sitio promotor, para que una sola de las dos hebras sirva de molde l Conforme avanza la polimerasa desenrolla el ADN por delante y enrolla nuevamente los segmentos que van quedando por atrás.

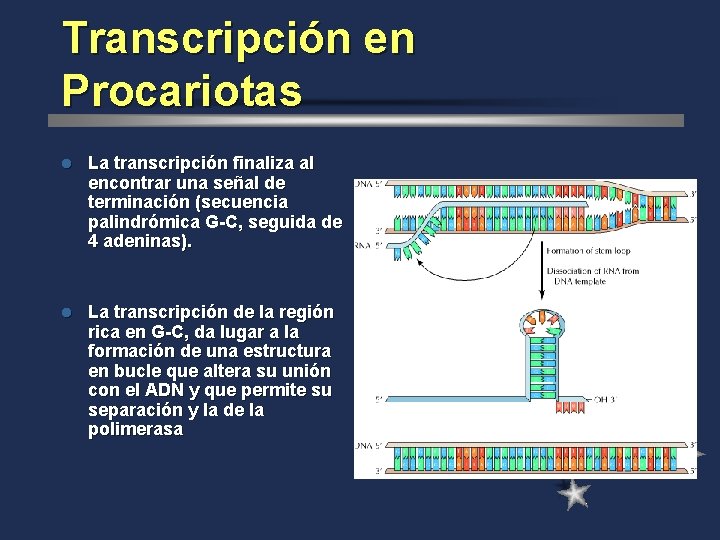
Transcripción en Procariotas l La transcripción finaliza al encontrar una señal de terminación (secuencia palindrómica G-C, seguida de 4 adeninas). l La transcripción de la región rica en G-C, da lugar a la formación de una estructura en bucle que altera su unión con el ADN y que permite su separación y la de la polimerasa

Transcripción en eucariotas

Clases de genes transcritos por ARN polimerasas Tipo de ARN sintetizado l l l l RNAm RNAt RNAr 5. 8 S, 18 S, 28 S RNAr 5 S RNAsn y RNAsc genes mitocondriales genes de cloroplastos RNA polimerasa II I III II y III Mitocondrial Cloroplasto

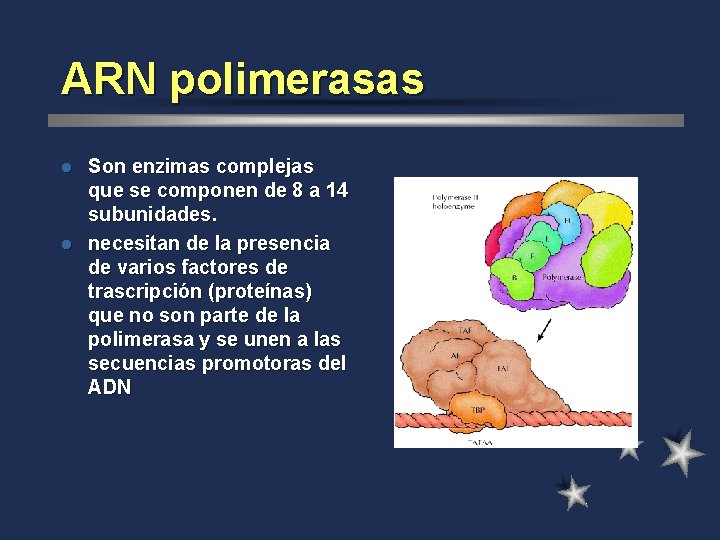
ARN polimerasas Son enzimas complejas que se componen de 8 a 14 subunidades. l necesitan de la presencia de varios factores de trascripción (proteínas) que no son parte de la polimerasa y se unen a las secuencias promotoras del ADN l

ARN polimerasas l Los promotores eucariontes poseen a -25 o -30, la secuencia de consenso llamada TATA. l Para la formación del complejo de transcripción, un factor general de transcripción llamado TFIID se une a la secuencia TATA. l El TFIID, se compone de múltiples subunidades: 10 o 12 polipéptidos llamados factores asociados a TBP (TAF)

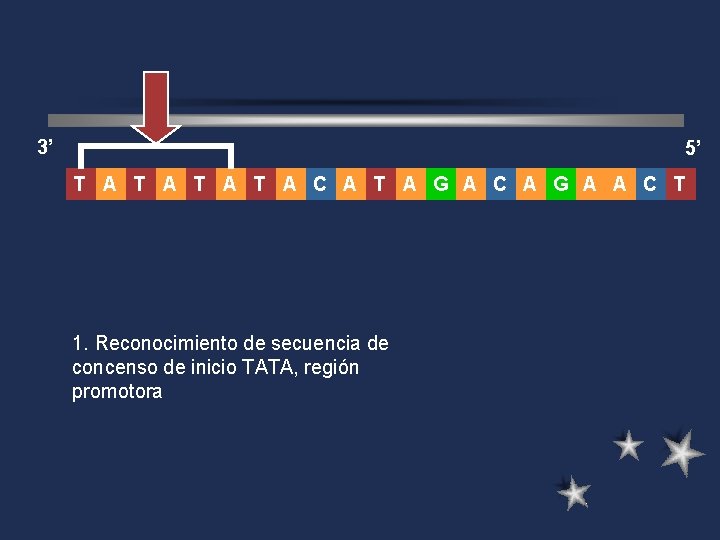
3’ 5’ T A T A C A T A G A C A G A A C T 1. Reconocimiento de secuencia de concenso de inicio TATA, región promotora

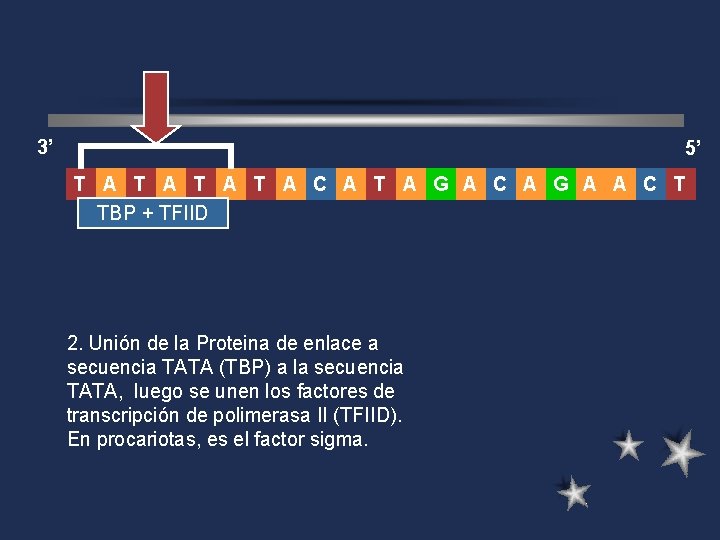
3’ 5’ T A T A C A T A G A C A G A A C T TBP + TFIID 2. Unión de la Proteina de enlace a secuencia TATA (TBP) a la secuencia TATA, luego se unen los factores de transcripción de polimerasa II (TFIID). En procariotas, es el factor sigma.

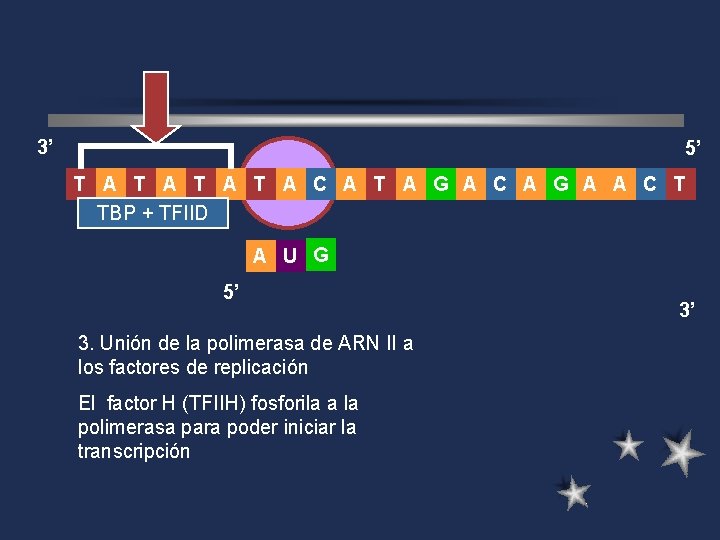
3’ 5’ T A T A C A T A G A C A G A A C T TBP + TFIID A U G 5’ 3. Unión de la polimerasa de ARN II a los factores de replicación El factor H (TFIIH) fosforila a la polimerasa para poder iniciar la transcripción 3’

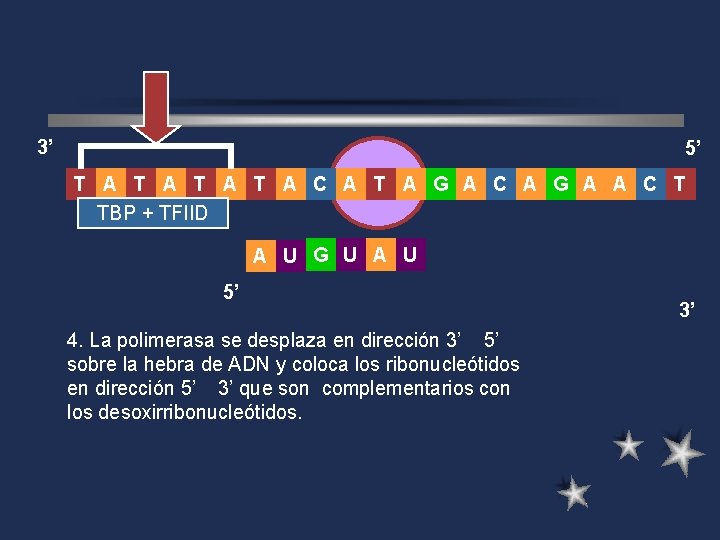
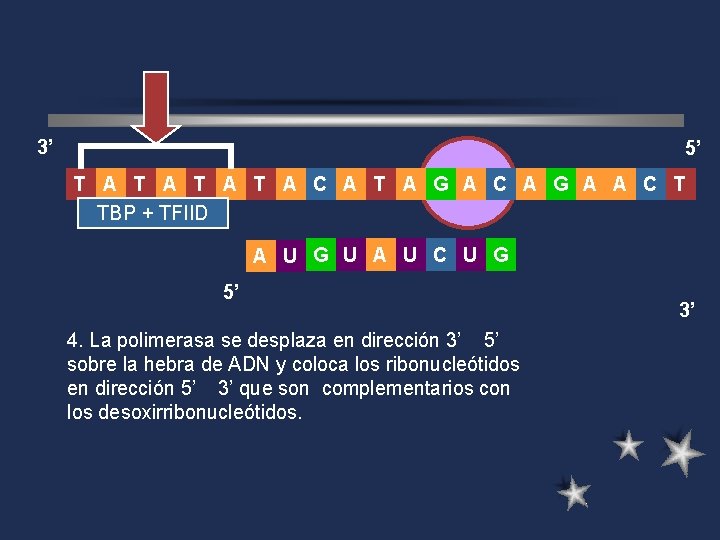
3’ 5’ T A T A C A T A G A C A G A A C T TBP + TFIID A U G U A U 5’ 1. Reconocimiento de secuencia de depolimerasa inicio TATA, 3. concenso Unión de la deregión ARN II a 4. La polimerasa se desplaza en dirección 3’ 5’ lospromotora factores de de replicación sobre la hebra ADN y coloca los ribonucleótidos Unión de 5’la Proteina de complementarios enlace a en 2. dirección 3’ que son con El factor H (TFIIH) fosforila a la TATA (PET) a la secuencia lossecuencia desoxirribonucleótidos. polimerasa para poder iniciar la TATA, luego se unen los factores de transcripción de polimerasa II (TFIID). En procariotas, es el factor sigma. 3’

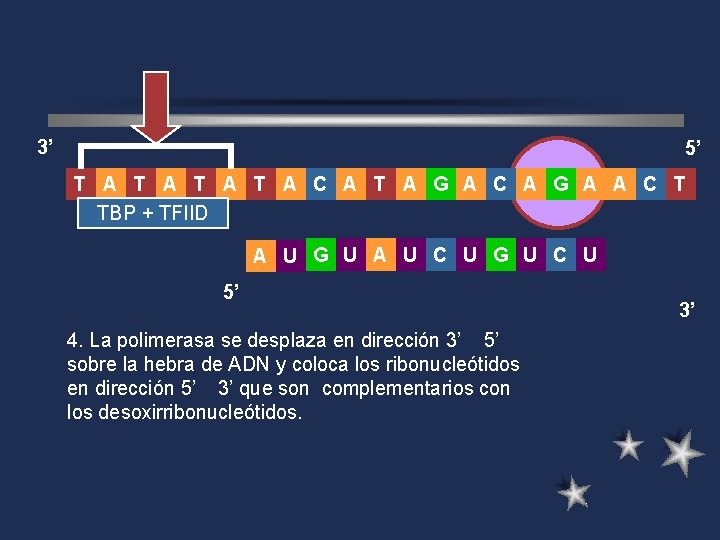
3’ 5’ T A T A C A T A G A C A G A A C T TBP + TFIID A U G U A U C U G 5’ 1. Reconocimiento de secuencia de depolimerasa inicio TATA, 3. concenso Unión de la deregión ARN II a 4. La polimerasa se desplaza en dirección 3’ 5’ lospromotora factores de de replicación sobre la hebra ADN y coloca los ribonucleótidos Unión de 5’la Proteina de complementarios enlace a en 2. dirección 3’ que son con El factor H (TFIIH) fosforila a la TATA (PET) a la secuencia lossecuencia desoxirribonucleótidos. polimerasa para poder iniciar la TATA, luego se unen los factores de transcripción de polimerasa II (TFIID). En procariotas, es el factor sigma. 3’

3’ 5’ T A T A C A T A G A C A G A A C T TBP + TFIID A U G U A U C U G U C U 5’ 1. Reconocimiento de secuencia de depolimerasa inicio TATA, 3. concenso Unión de la deregión ARN II a 4. La polimerasa se desplaza en dirección 3’ 5’ lospromotora factores de de replicación sobre la hebra ADN y coloca los ribonucleótidos Unión de 5’la Proteina de complementarios enlace a en 2. dirección 3’ que son con El factor H (TFIIH) fosforila a la TATA (PET) a la secuencia lossecuencia desoxirribonucleótidos. polimerasa para poder iniciar la TATA, luego se unen los factores de transcripción de polimerasa II (TFIID). En procariotas, es el factor sigma. 3’

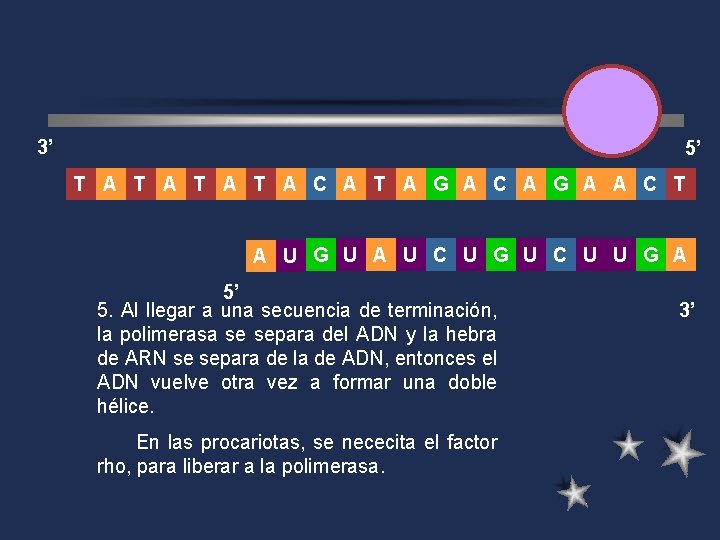
3’ 5’ T A T A C A T A G A C A G A A C T A U G U A U C U G U C U U G A 5’ 3’

3’ 5’ T A T A C A T A G A C A G A A C T A U G U A U C U G U C U U G A 5’ 5. Al llegar a una secuencia de terminación, la polimerasa se separa del ADN y la hebra de ARN se separa de la de ADN, entonces el ADN vuelve otra vez a formar una doble hélice. En las procariotas, se nececita el factor rho, para liberar a la polimerasa. 3’

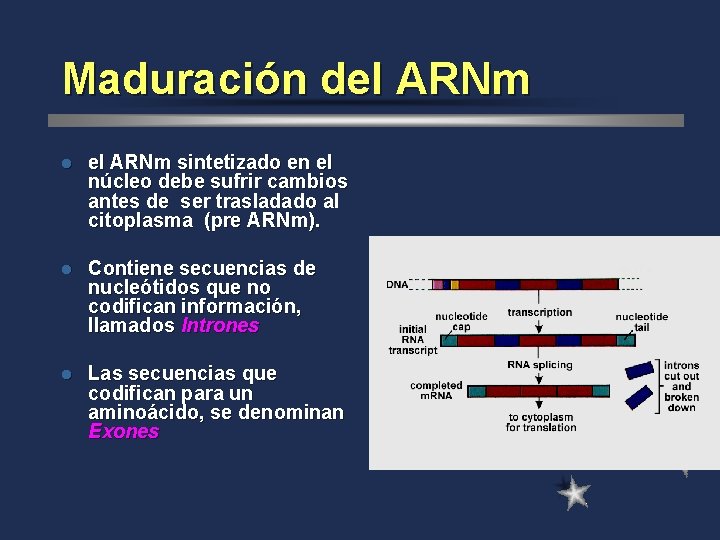
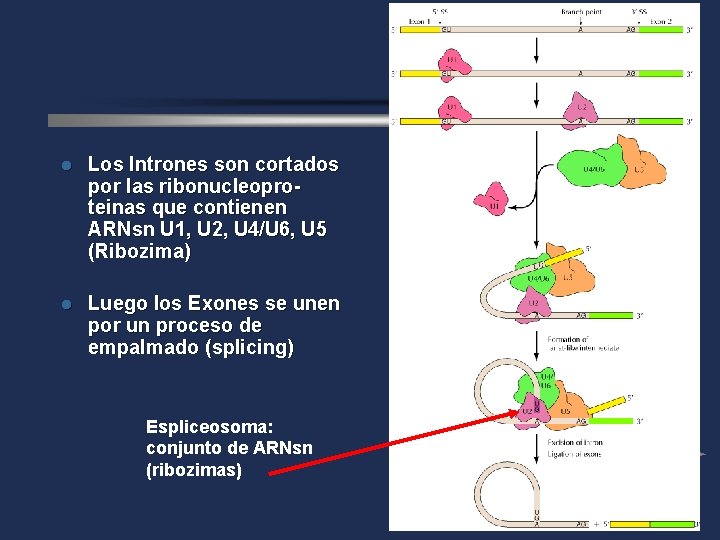
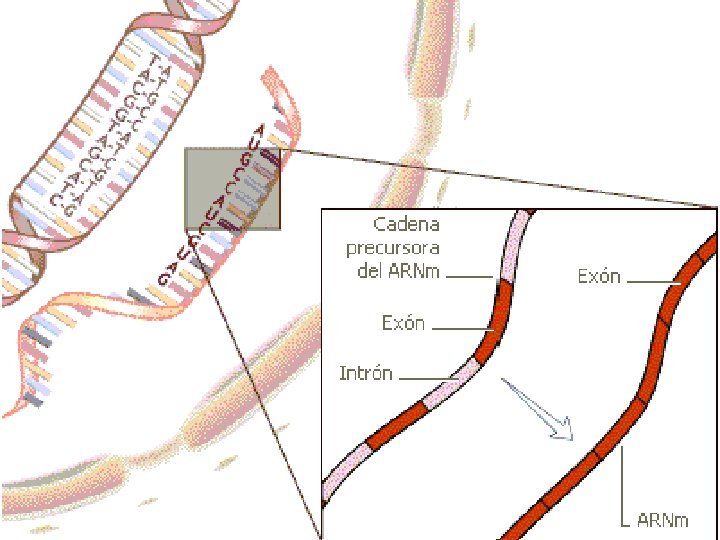
Maduración del ARNm l el ARNm sintetizado en el núcleo debe sufrir cambios antes de ser trasladado al citoplasma (pre ARNm). l Contiene secuencias de nucleótidos que no codifican información, llamados Intrones l Las secuencias que codifican para un aminoácido, se denominan Exones

l Los lntrones son cortados por las ribonucleoproteinas que contienen ARNsn U 1, U 2, U 4/U 6, U 5 (Ribozima) l Luego los Exones se unen por un proceso de empalmado (splicing) Espliceosoma: conjunto de ARNsn (ribozimas)

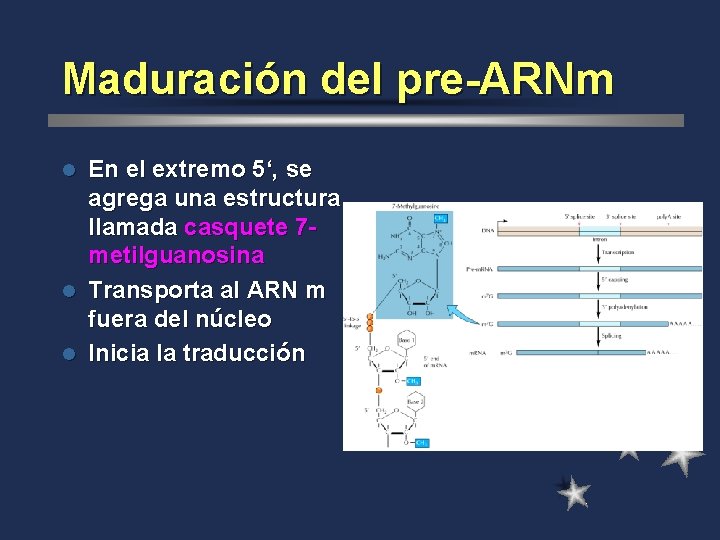
Maduración del pre-ARNm En el extremo 5‘, se agrega una estructura llamada casquete 7 metilguanosina l Transporta al ARN m fuera del núcleo l Inicia la traducción l

Maduración del pre-ARNm l l Poliadenilación, agrega una cola poli A al extremo 3‘, Son Residuos de adenosina Estabiliza al ARN m en el citosol Protección contra el desdobamiento en el citosol

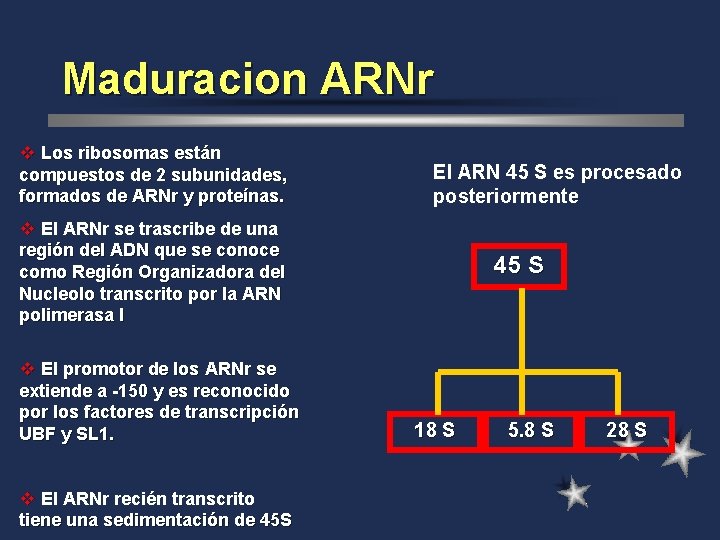
Maduracion ARNr v Los ribosomas están compuestos de 2 subunidades, formados de ARNr y proteínas. El ARN 45 S es procesado posteriormente v El ARNr se trascribe de una región del ADN que se conoce como Región Organizadora del Nucleolo transcrito por la ARN polimerasa I v El promotor de los ARNr se extiende a -150 y es reconocido por los factores de transcripción UBF y SL 1. v El ARNr recién transcrito tiene una sedimentación de 45 S 45 S 18 S 5. 8 S 28 S

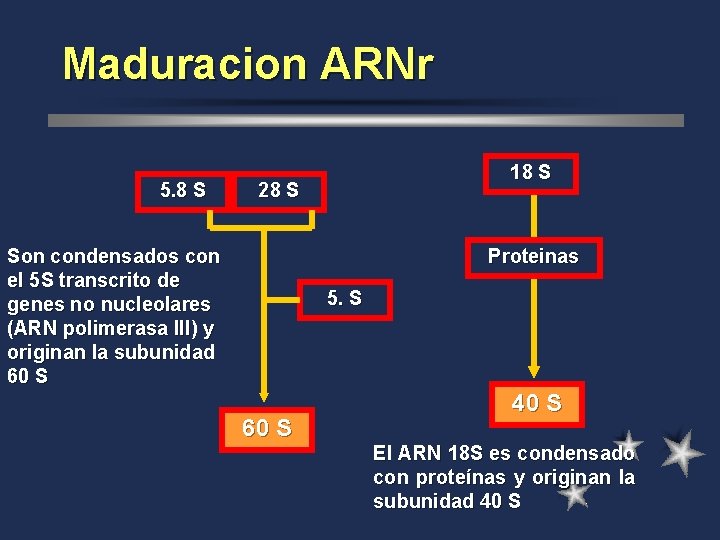
Maduracion ARNr 5. 8 S 18 S 28 S Son condensados con el 5 S transcrito de genes no nucleolares (ARN polimerasa III) y originan la subunidad 60 S Proteinas 5. S 60 S 40 S El ARN 18 S es condensado con proteínas y originan la subunidad 40 S

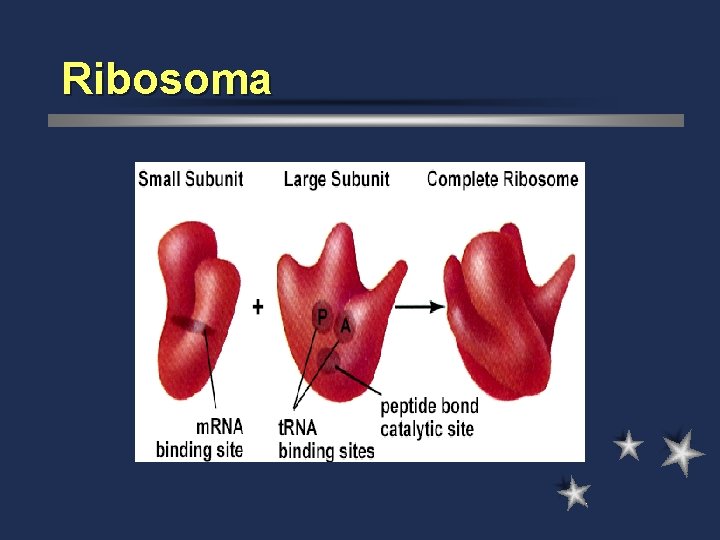
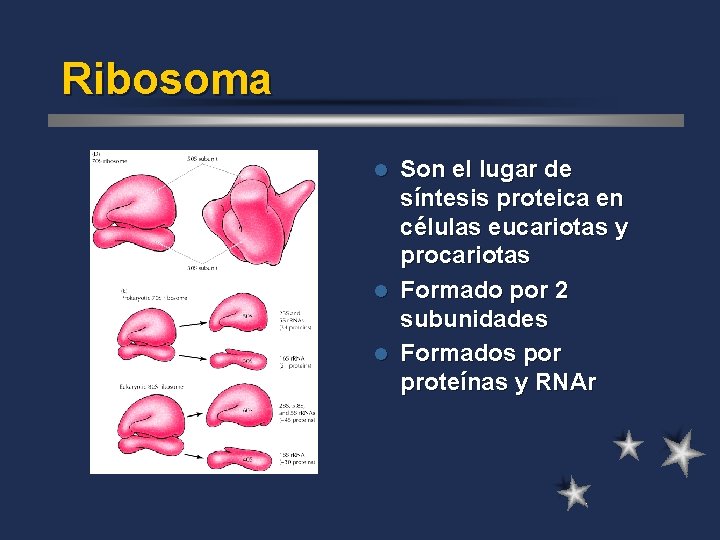
Ribosoma

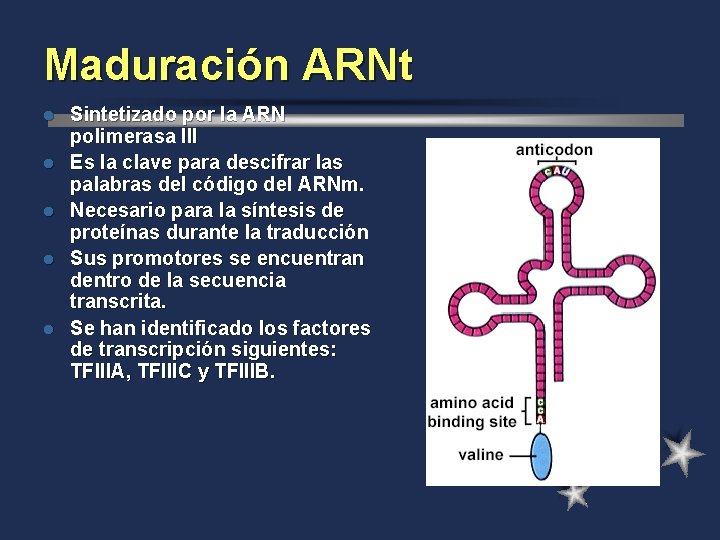
Maduración ARNt l l l Sintetizado por la ARN polimerasa III Es la clave para descifrar las palabras del código del ARNm. Necesario para la síntesis de proteínas durante la traducción Sus promotores se encuentran dentro de la secuencia transcrita. Se han identificado los factores de transcripción siguientes: TFIIIA, TFIIIC y TFIIIB.

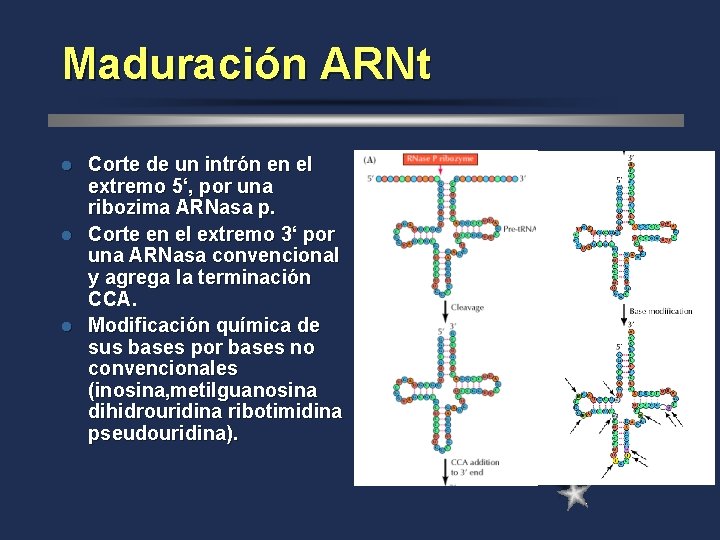
Maduración ARNt Corte de un intrón en el extremo 5‘, por una ribozima ARNasa p. l Corte en el extremo 3‘ por una ARNasa convencional y agrega la terminación CCA. l Modificación química de sus bases por bases no convencionales (inosina, metilguanosina dihidrouridina ribotimidina pseudouridina). l

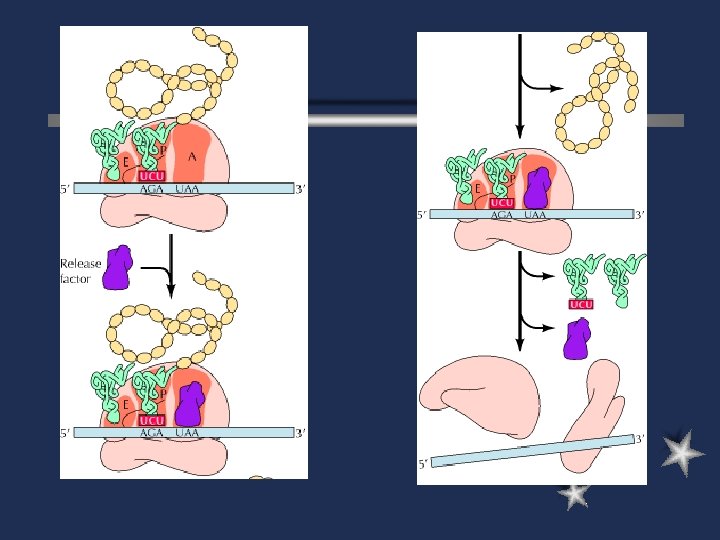
TRADUCCION

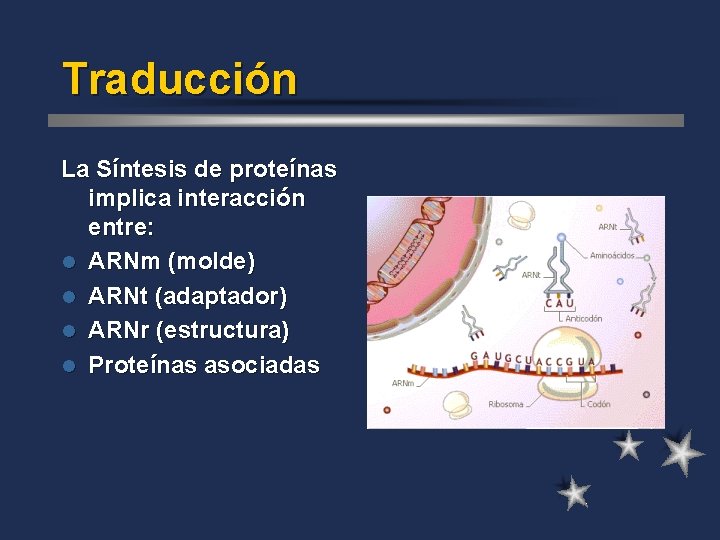
Traducción l l l Síntesis de proteínas mediada por un molde de ARNm. Dirección de lectura del ARNm: 5´-3´ Dirección de Síntesis del péptido: de Amino terminal a carboxilo terminal Localización: citosol Estructura involucrada: Ribosomas Adaptadores entre molde de ARNm y aa: ARNt.





Traducción La Síntesis de proteínas implica interacción entre: l ARNm (molde) l ARNt (adaptador) l ARNr (estructura) l Proteínas asociadas

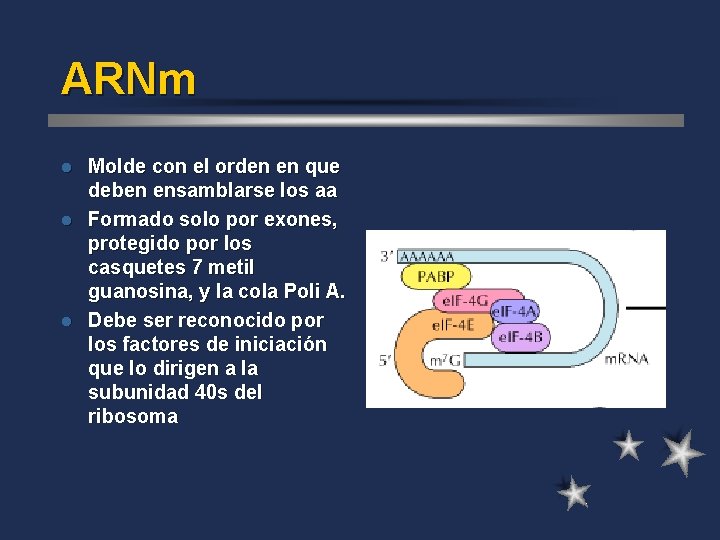
ARNm Molde con el orden en que deben ensamblarse los aa l Formado solo por exones, protegido por los casquetes 7 metil guanosina, y la cola Poli A. l Debe ser reconocido por los factores de iniciación que lo dirigen a la subunidad 40 s del ribosoma l

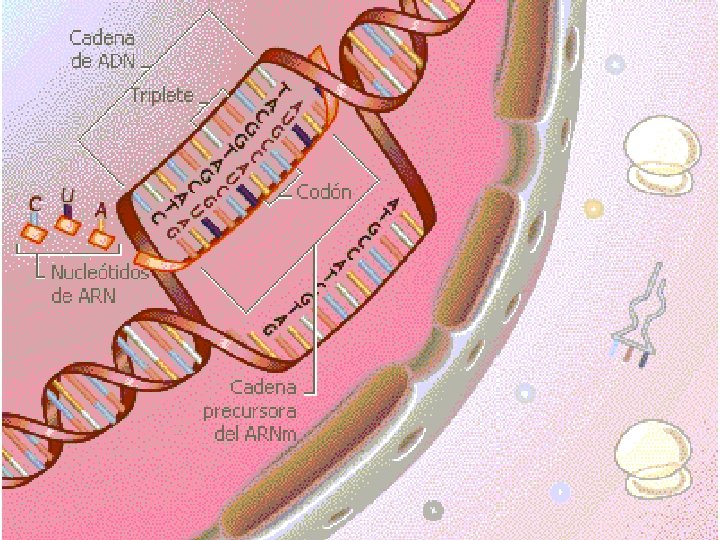
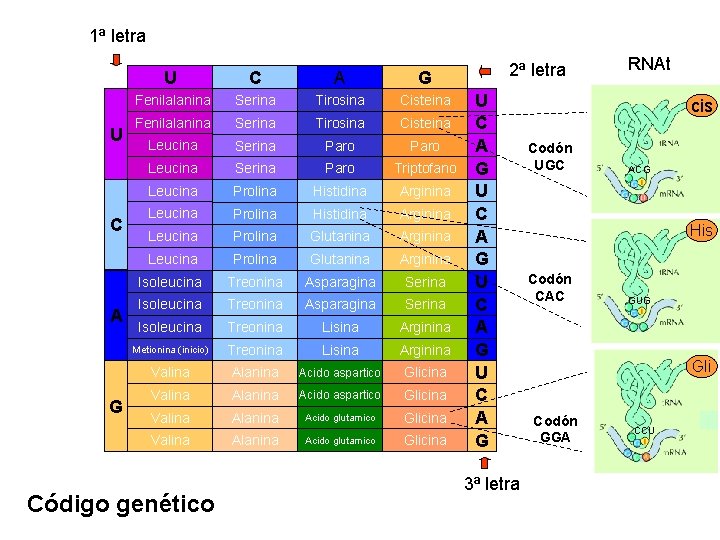
Codigo Genético Está constituido por una secuencia de 3 nucleótidos en el ARNm llamados codones l Existen 64 combinaciones posibles con cada uno de los 4 nucléotidos l Cada codón es específico para un aminoácido l Cada aminoácido puede poseer varios codones que lo codifican l Las combinaciones pueden ser: AUA AGA AAA ACA AUU AGU AAU ACU AUG AGG AAG ACG AUC AGC AAC ACC l

Codigo Genético Note que hay 16 posibilidades de combinación siendo adenina el primer nucléotido l Siendo 4 nucléotidos es fácil ver por qué son 64 combinaciones 16 x 4 = 64 l Solo hay que cambiar el primer nucleótido y se formarán los demás codones l AUA AUU AUG AUC A GA A GU A GG A GC AAA ACA AAU ACU AAG ACG AAC ACC UUA UUU UUG UUC UGA UG U UG G UG C UAA UCA UAU UCU UAG UCG UAC UCC

1ª letra U C A G Fenilalanina Serina Tirosina Cisteina Leucina Serina Paro Triptofano Leucina Prolina Histidina Arginina Leucina Prolina Glutanina Arginina Isoleucina Treonina Asparagina Serina Isoleucina Treonina Lisina Arginina Metionina (inicio) Treonina Lisina Arginina Valina Alanina Acido aspartico Glicina Valina Alanina Acido glutamico Glicina Código genético 2ª letra U C A G 3ª letra RNAt cis Codón UGC ACG His Codón CAC GUG Gli Codón GGA CCU

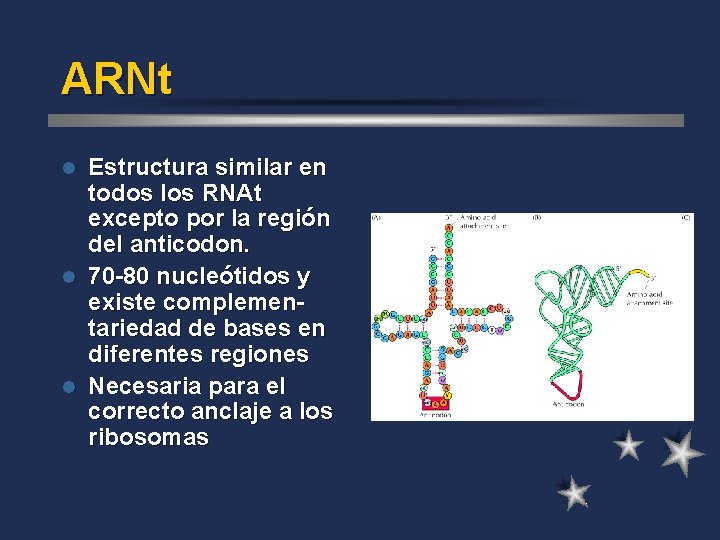
ARNt Estructura similar en todos los RNAt excepto por la región del anticodon. l 70 -80 nucleótidos y existe complementariedad de bases en diferentes regiones l Necesaria para el correcto anclaje a los ribosomas l

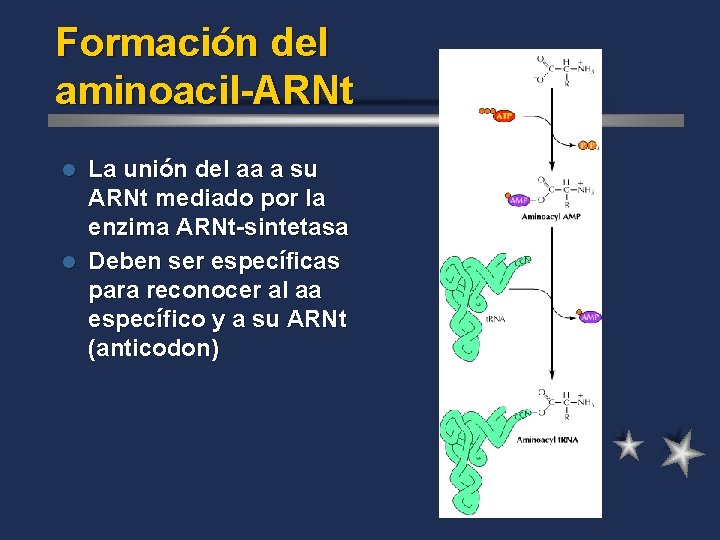
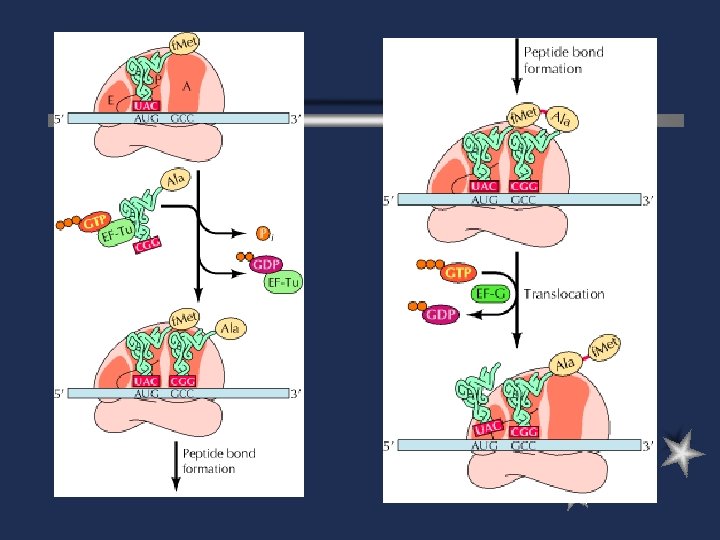
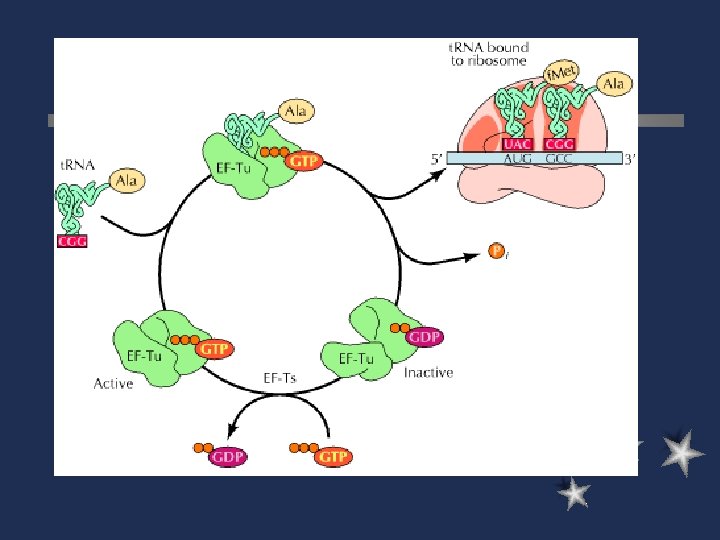
Formación del aminoacil-ARNt La unión del aa a su ARNt mediado por la enzima ARNt-sintetasa l Deben ser específicas para reconocer al aa específico y a su ARNt (anticodon) l

Ribosoma Son el lugar de síntesis proteica en células eucariotas y procariotas l Formado por 2 subunidades l Formados por proteínas y RNAr l

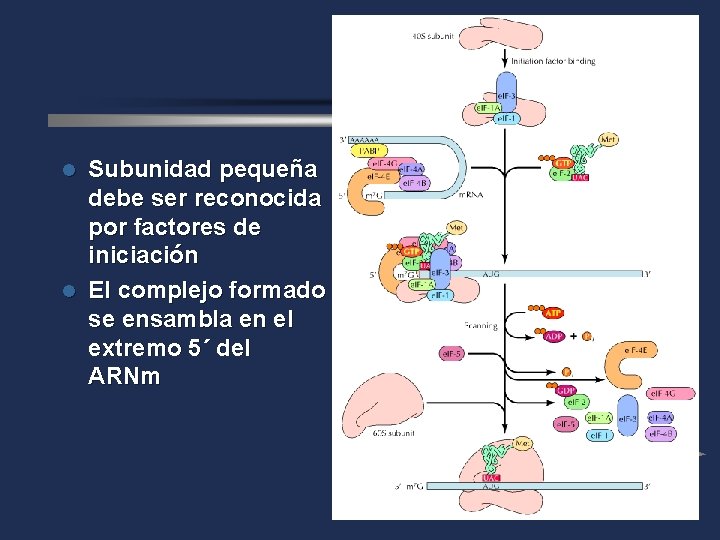
Subunidad pequeña debe ser reconocida por factores de iniciación l El complejo formado se ensambla en el extremo 5´ del ARNm l


