Secuenciacin de ARN Secuenciacin de ARN Directamente a













- Slides: 33

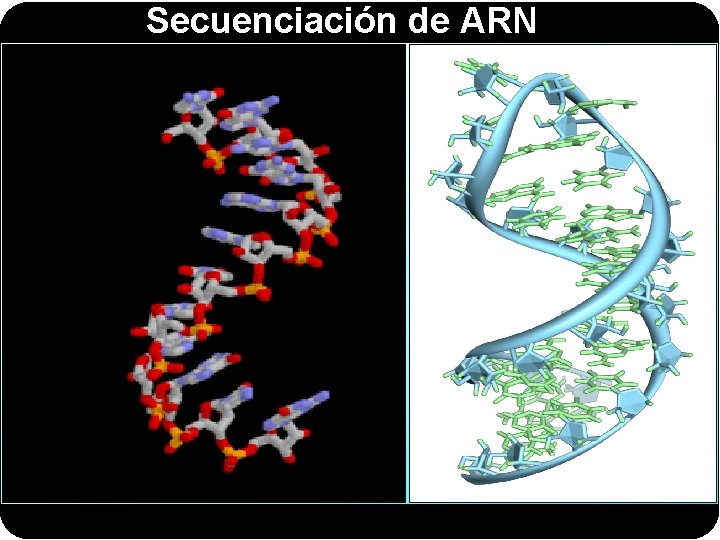
Secuenciación de ARN

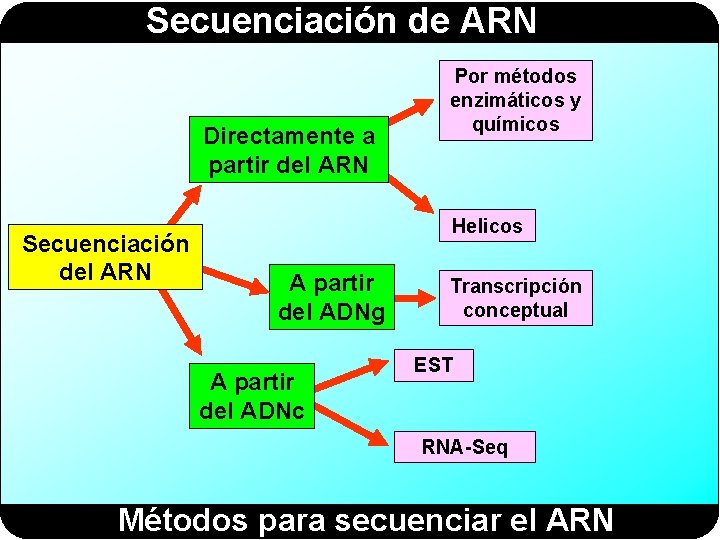
Secuenciación de ARN Directamente a partir del ARN Secuenciación del ARN Por métodos enzimáticos y químicos Helicos A partir del ADNg A partir del ADNc Transcripción conceptual EST RNA-Seq Métodos para secuenciar el ARN

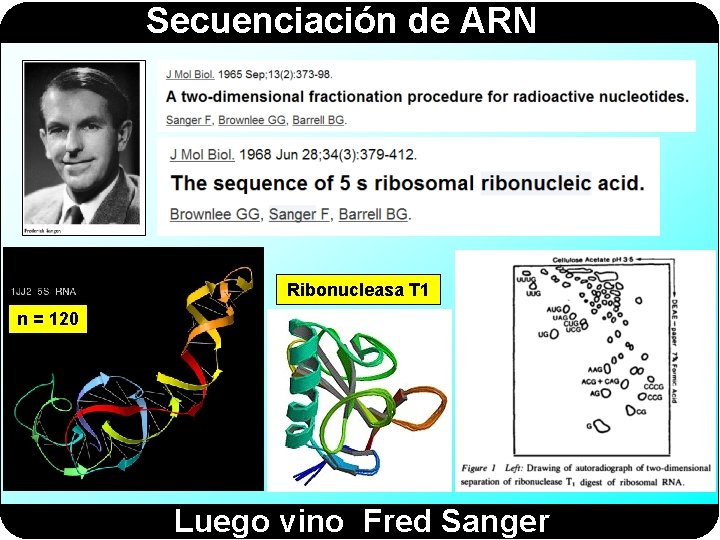
Secuenciación de ARN Science, 147 (1965): 1462 -1465 Los primeros n = 77

Secuenciación de ARN Ribonucleasa T 1 n = 120 Luego vino Fred Sanger

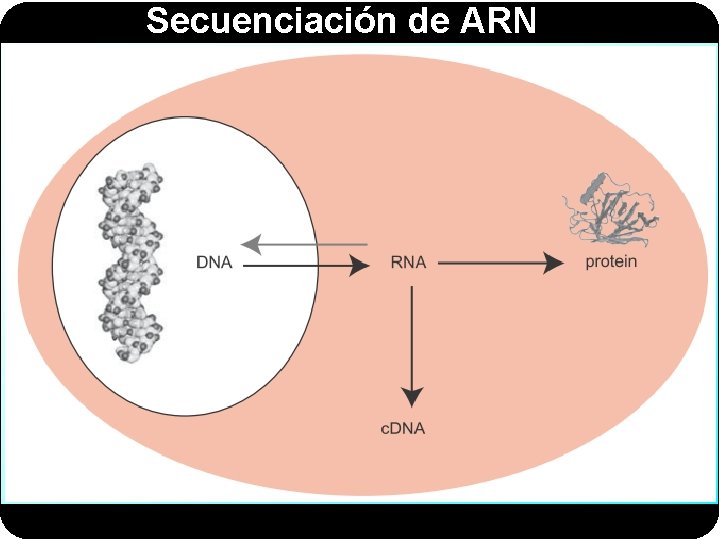
Secuenciación de ARN

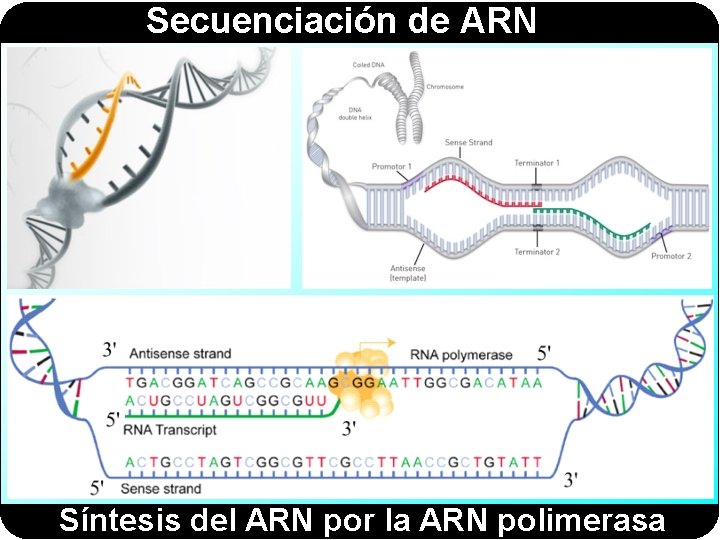
Secuenciación de ARN Síntesis del ARN por la ARN polimerasa

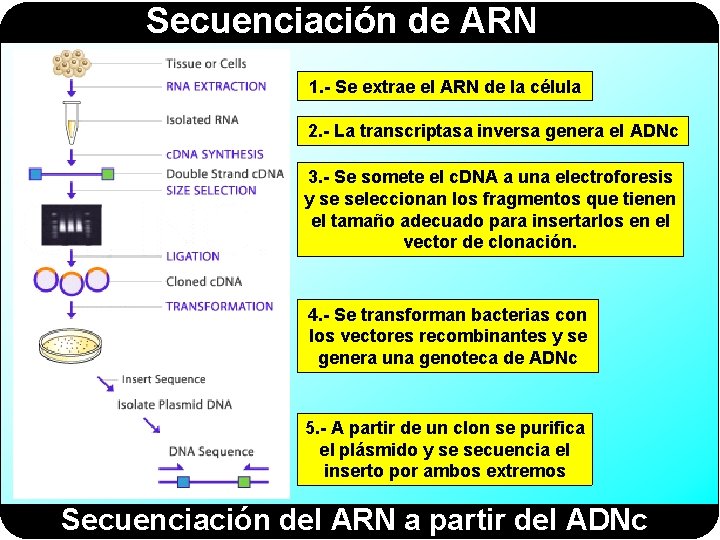
Secuenciación de ARN 1. - Se extrae el ARN de la célula 2. - La transcriptasa inversa genera el ADNc 3. - Se somete el c. DNA a una electroforesis y se seleccionan los fragmentos que tienen el tamaño adecuado para insertarlos en el vector de clonación. 4. - Se transforman bacterias con los vectores recombinantes y se genera una genoteca de ADNc 5. - A partir de un clon se purifica el plásmido y se secuencia el inserto por ambos extremos Secuenciación del ARN a partir del ADNc

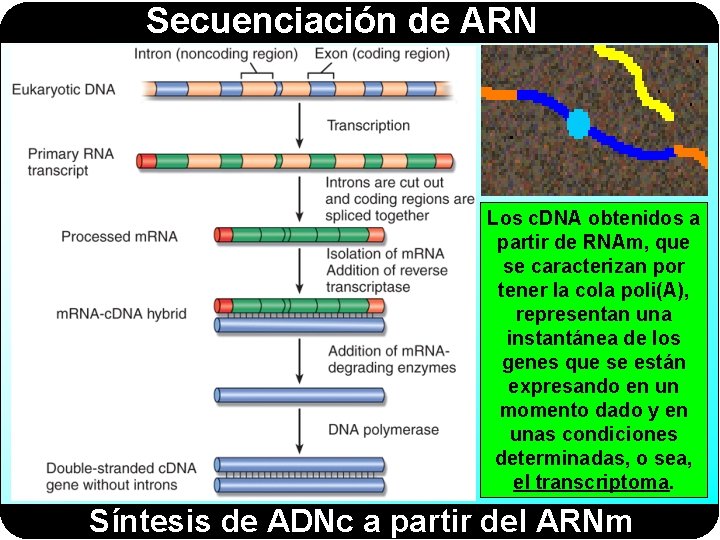
Secuenciación de ARN Los c. DNA obtenidos a partir de RNAm, que se caracterizan por tener la cola poli(A), representan una instantánea de los genes que se están expresando en un momento dado y en unas condiciones determinadas, o sea, el transcriptoma. Síntesis de ADNc a partir del ARNm

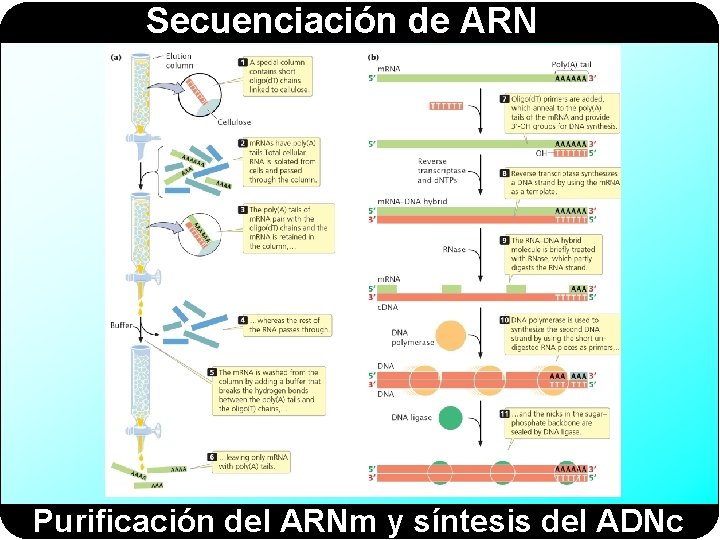
Secuenciación de ARN Purificación del ARNm y síntesis del ADNc

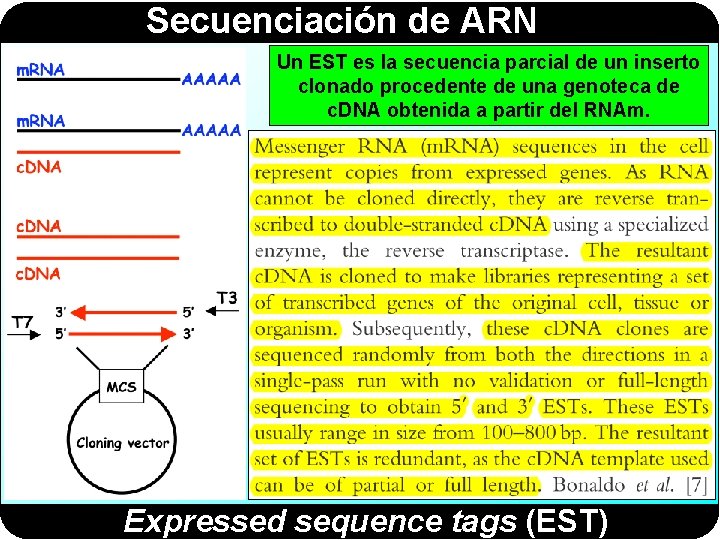
Secuenciación de ARN Un EST es la secuencia parcial de un inserto clonado procedente de una genoteca de c. DNA obtenida a partir del RNAm. Expressed sequence tags (EST)

Secuenciación de ARN Expressed sequence tags (EST)

Secuenciación de ARN 1. - Se preparan a partir de varios organismos, tejidos, tipos celulares, condiciones patológicas o condiciones experimentales. 2. - La complejidad y la calidad de las genotecas varía mucho de una a otra. 3. - La mayoría son “nativas”, ya que la frecuencia de los clones refleja la abundancia de los distintos ARNm. 4. - Las genotecas “normalizadas” se han modificado para reducir la presencia de los m. RNA más abundantes y aumentar la probabilidad de detectar los m. RNA menos frecuentes. 5. - La mayoría se han generado con un cebador poli(T), de modo que los extremos 3’ están sobrerrepresentados. Características de las genotecas de ADNc

Secuenciación de ARN Características de los EST

Secuenciación de ARN Limitaciones de los EST

Secuenciación de ARN

Secuenciación de ARN RNA-Seq

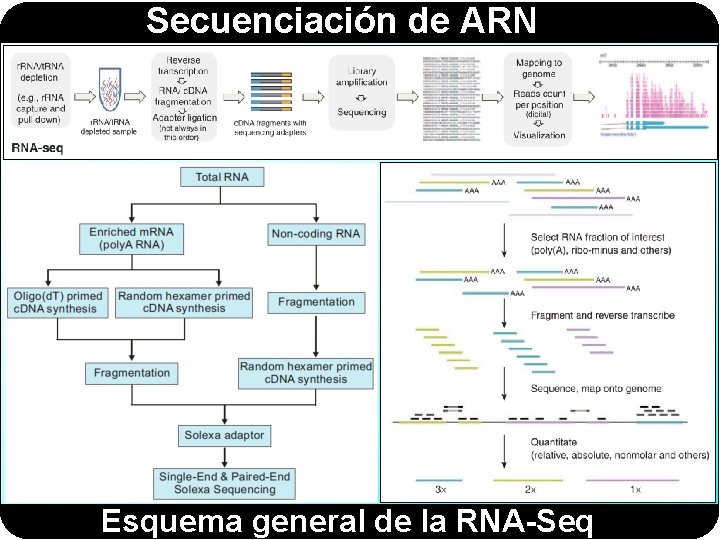
Secuenciación de ARN Esquema general de la RNA-Seq

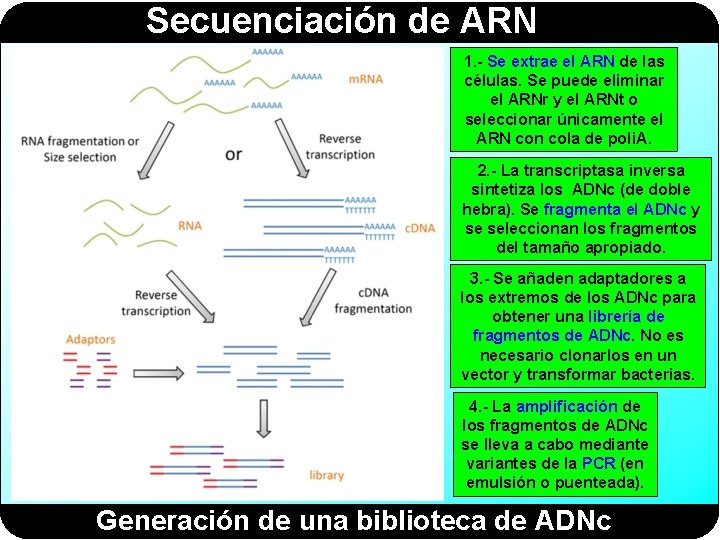
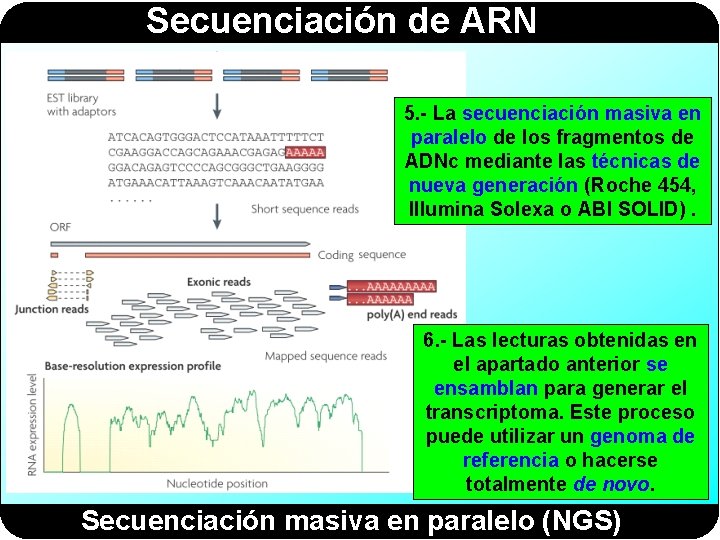
Secuenciación de ARN 1. - Se extrae el ARN de las células. Se puede eliminar el ARNr y el ARNt o seleccionar únicamente el ARN con cola de poli. A. 2. - La transcriptasa inversa sintetiza los ADNc (de doble hebra). Se fragmenta el ADNc y se seleccionan los fragmentos del tamaño apropiado. 3. - Se añaden adaptadores a los extremos de los ADNc para obtener una librería de fragmentos de ADNc. No es necesario clonarlos en un vector y transformar bacterias. 4. - La amplificación de los fragmentos de ADNc se lleva a cabo mediante variantes de la PCR (en emulsión o puenteada). Generación de una biblioteca de ADNc

Secuenciación de ARN 5. - La secuenciación masiva en paralelo de los fragmentos de ADNc mediante las técnicas de nueva generación (Roche 454, Illumina Solexa o ABI SOLID). 6. - Las lecturas obtenidas en el apartado anterior se ensamblan para generar el transcriptoma. Este proceso puede utilizar un genoma de referencia o hacerse totalmente de novo. Secuenciación masiva en paralelo (NGS)

Secuenciación de ARN Single-Molecule Sequencing (Helicos)

Secuenciación de ARN Evita los problemas asociados a la síntesis del ADNc

Secuenciación de ARN El artículo que describe el método

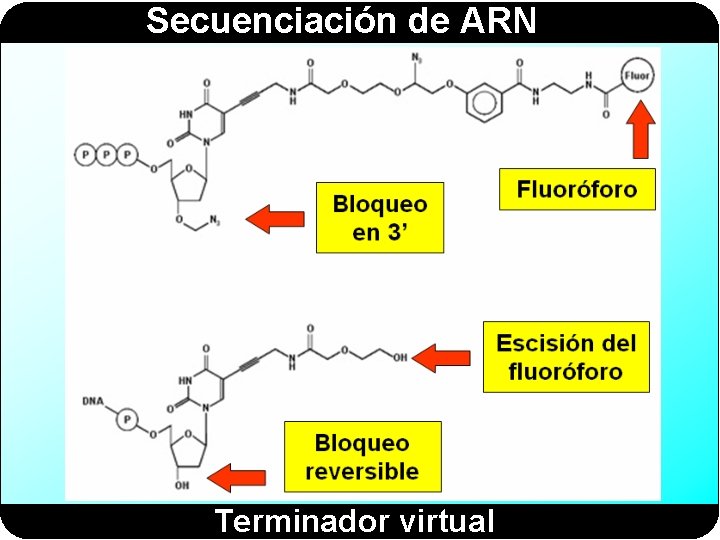
Secuenciación de ARN Terminador virtual

Secuenciación de ARN 108 moléculas por cm 2 El secuenciador Heli. Scope y la celda de flujo

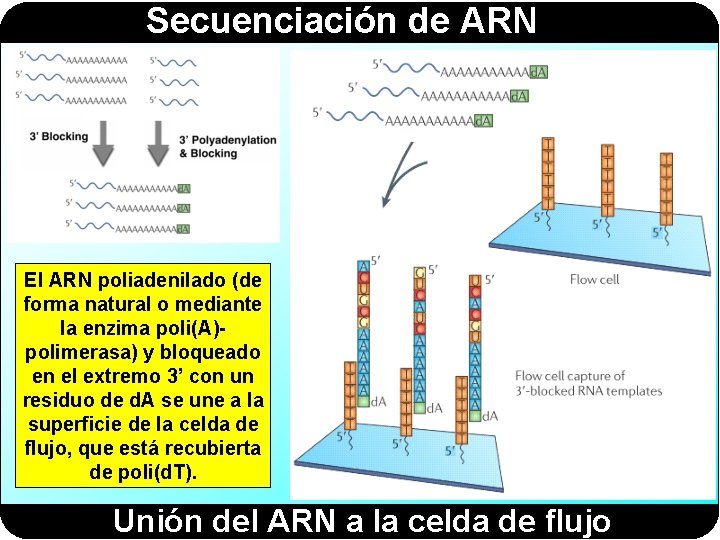
Secuenciación de ARN El ARN poliadenilado (de forma natural o mediante la enzima poli(A)polimerasa) y bloqueado en el extremo 3’ con un residuo de d. A se une a la superficie de la celda de flujo, que está recubierta de poli(d. T). Unión del ARN a la celda de flujo

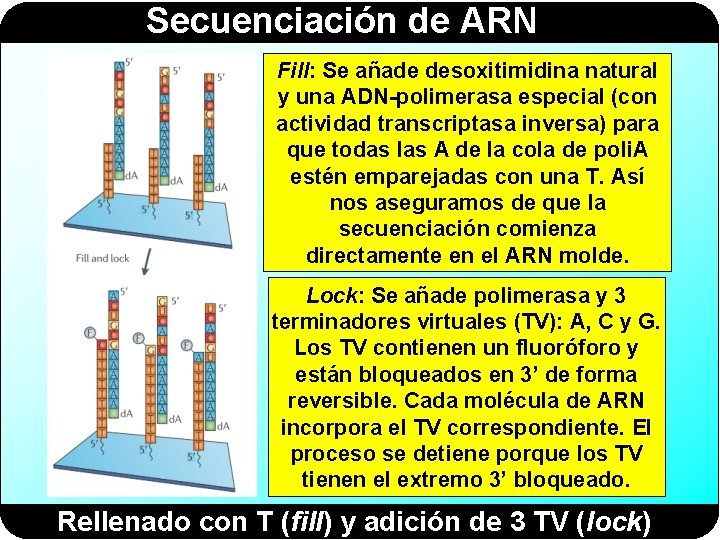
Secuenciación de ARN Fill: Se añade desoxitimidina natural y una ADN-polimerasa especial (con actividad transcriptasa inversa) para que todas las A de la cola de poli. A estén emparejadas con una T. Así nos aseguramos de que la secuenciación comienza directamente en el ARN molde. Lock: Se añade polimerasa y 3 terminadores virtuales (TV): A, C y G. Los TV contienen un fluoróforo y están bloqueados en 3’ de forma reversible. Cada molécula de ARN incorpora el TV correspondiente. El proceso se detiene porque los TV tienen el extremo 3’ bloqueado. Rellenado con T (fill) y adición de 3 TV (lock)

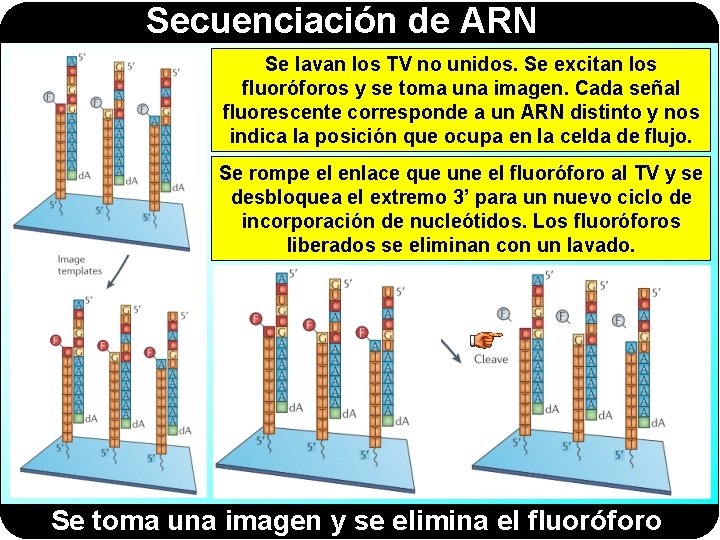
Secuenciación de ARN Se lavan los TV no unidos. Se excitan los fluoróforos y se toma una imagen. Cada señal fluorescente corresponde a un ARN distinto y nos indica la posición que ocupa en la celda de flujo. Se rompe el enlace que une el fluoróforo al TV y se desbloquea el extremo 3’ para un nuevo ciclo de incorporación de nucleótidos. Los fluoróforos liberados se eliminan con un lavado. Se toma una imagen y se elimina el fluoróforo

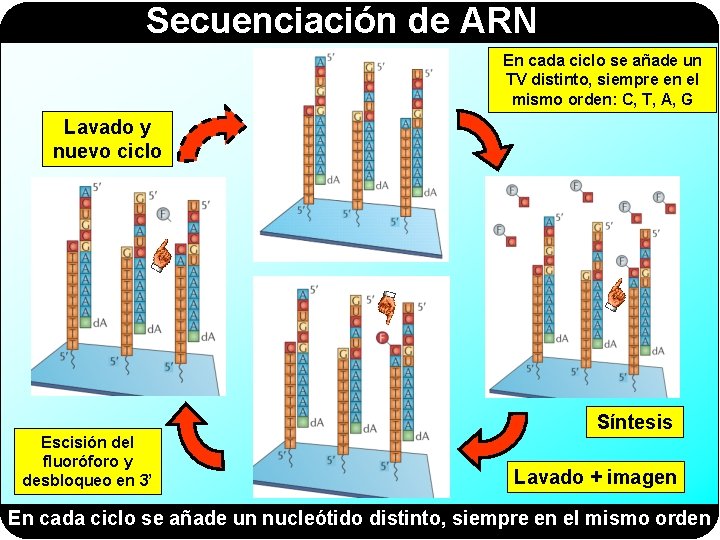
Secuenciación de ARN En cada ciclo se añade un TV distinto, siempre en el mismo orden: C, T, A, G Lavado y nuevo ciclo Síntesis Escisión del fluoróforo y desbloqueo en 3’ Lavado + imagen En cada ciclo se añade un nucleótido distinto, siempre en el mismo orden

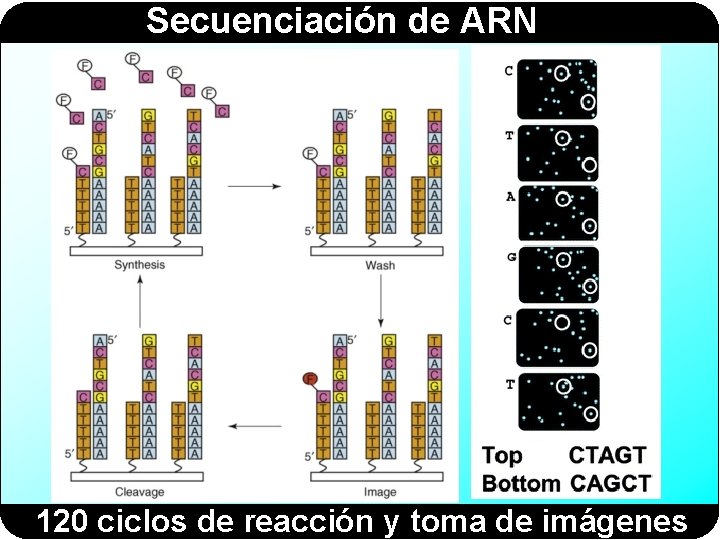
Secuenciación de ARN 120 ciclos de reacción y toma de imágenes

Secuenciación de ARN La longitud media de las lecturas es de 33 nucleótidos

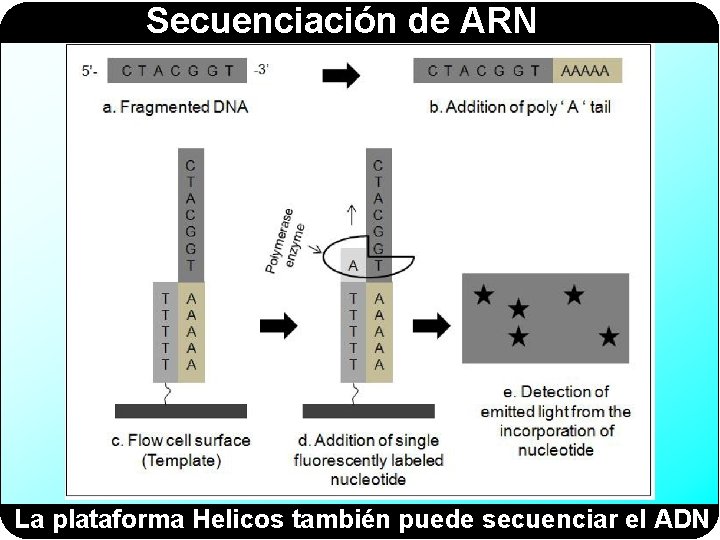
Secuenciación de ARN La plataforma Helicos también puede secuenciar el ADN

Secuenciación de ARN Helicos cierra por bancarrota

Secuenciación de ARN Seq. LL utiliza su tecnología