Processos de Sequenciamento de DNA Camila Dantas Malossi















- Slides: 17

Processos de Sequenciamento de DNA Camila Dantas Malossi BMP 5777 - Biologia Molecular e Bioquímica de proteínas alvo a drogas do parasita da malária humana Plasmodium falciparum


Tecnologias de sequenciamento Método de degradação química: procedimento Maxam-Gilbert - Método de degradação enzimática: procedimento de Sanger - - Sequenciador Mega. BACE (1 Mpb/24 horas) - Pirosequenciamento - Sequenciador 454 (150 Mpb/24 horas)

Procedimento Maxam-Gilbert Marcar radioativamente o DNA alvo com isótopos radioativos, tais como 32 P e 35 S; � Separam-se as duas cadeias de DNA; � Distribui-se por 4 tubos, onde as moléculas serão clivadas quimicamente para cada base (A, C, G ou T). Por exemplo, no tubo em que o tratamento foi direcionado para a adenina, após a clivagem, a extremidade do fragmento de DNA marcada com o radioisótopo, apresentará uma base que corresponde à que precede a adenina. � Gel de eletroforese para observar a sequência. �



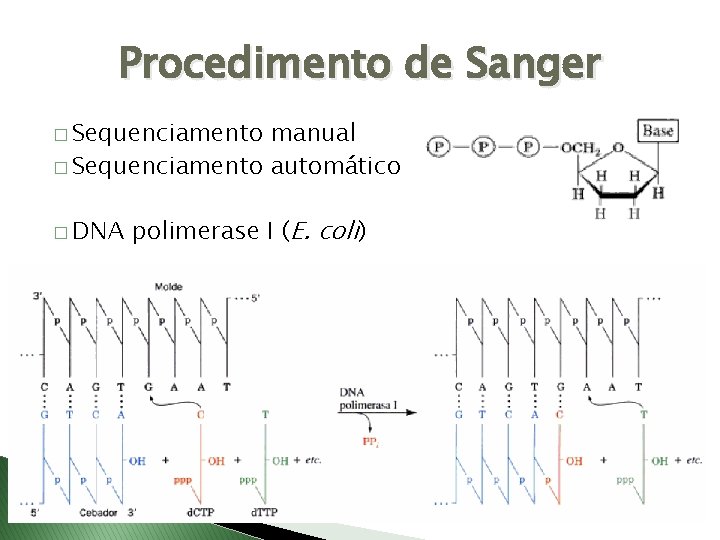
Procedimento de Sanger � Sequenciamento manual � Sequenciamento automático � DNA polimerase I (E. coli)

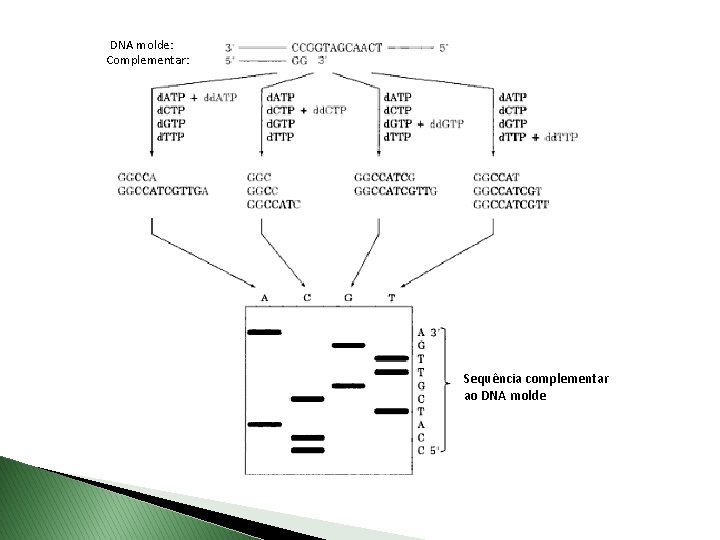
DNA molde: Complementar: Sequência complementar ao DNA molde

Sequenciamento automático: � Cada um dos quatros dideoxy terminators – dd. G, dd. A, dd. C, dd. T – estão marcados com dois tipos de dye – fluoresceína e rodomina. �A fluoresceína alto coeficiente de extinção (488 nm) no laser de argon. Atua como um dye doador absorve a energia da luz do laser incidente e a transfere para o dye aceptor (rodomina) �A rodomina emite a luz em um comprimento de onda característico para a identificação dos 4 nucleotídeos que terminam a fita de DNA.




Pirosequenciamento - O adaptador permite que o DNA se ligue em grânulos minúsculos (diâmetro de 28 mm). Apenas um DNA é ligado em cada grânulo - Os grânulos são envolvidos em gotas de óleo que contêm todos os reagentes necessários para amplificar o DNA - Cada gota contendo o grânulo é mantida isolada para evitar contaminação e consegue produzir 10 milhões de cópias numa reação de pirosequenciamento - Um pmol de DNA numa reação de pirosequenciamento produz 1011 moléculas de ATP gerando mais de 109 fótons, num comprimento de onda de 560 nm, e num período de 3 -4 segundos, facilmente detectado por uma câmera de CCD


Sanger vs Pirosequenciamento Sanger • Depende de clonagem em bactéria (2 semanas de trabalho) • 1 milhão de pb em 24 horas Pirosequenciamento • Não há clonagem • 25 milhões de bp em 4 horas (100 x mais rápido) • Clones de fita dupla permitem seqüenciamento em ambas direções (facilita orientação e montagem) • Fragmentos fita simples não permitem seqüenciamento em ambas direções • 6 meses de sequenciamento, 24 horas por dia, para sequenciar o genoma de um fungo • 24 horas para sequenciar o genoma de um fungo


Referências bibliográficas Goldberg, S. M. D. ; Johnson, J. ; Busam, D. ; Feldblyum, T. ; Ferriera, S. ; Friedman, R. ; Halpern, A. ; Khouri, H. ; Kravitz, S. A. ; Lauro, F. M. ; Li, K. ; Rogers, Y. ; Strausberg, R. ; Sutton, G. ; Tallon, L. ; Thomas, T. ; Venter, E. ; Frazier, M. & Venter, J. C. 2006. A Sanger/pyrosequencing hybrid approach for the generation of high-quality draft assemblies of marine microbial genomes. PNAS. 103 (30): 11240 -11245. Messing, J. & Llaca, V. 1998. Importance of anchor genomes for any plant genome project. Proc. Natl. Acad. Sci. USA. 95: 2017– 2020. Metzker, M. L. 2010. Sequencing technologies — the next generation. Nature. 11: 31 -46. Rogers, Y. & Venter, J. C. 2005. Massively parallel sequencing. Nature. 437: 326 -327. Voet, D. ; Voet, J. G. & Pratt, C. W. 2002. Fundamentos de Bioquímica. Ed. Artmed. 1ª ed. p. 59 -64.