OUEST Genopole J Nicolas IRISA Inria Rennes Assist




















- Slides: 36

OUEST Genopole® J. Nicolas IRISA / Inria Rennes Assisté de O. Colin, H. Leroy, E. Kabore, E. Morin, C. Bioinformatique Delamarche, des C. génopoles Hitte et D. Lavenier Journées – Lyon -Octobre 2003

OUEST-Génopole® : un réseau de 54 unités de recherche 10 CNRS 2 IFREMER 16 INRA 13 INSERM 1 INRIA 1 AFSSA 11 unités de recherche des Univ. d'Angers, Brest, Nantes et Rennes) 2000 personnes dont 800 chercheurs Journées Bioinformatique des génopoles – Lyon -Octobre 2003

Une histoire récente Juillet 2000 : Dépôt dossier Génopole Ouest au ministère Mars 2001 : Expertise sur site de la génopole Janvier 2002 : Labellisation OUEST-Genopole® CDDs plate-forme bioinformatique génopole Septembre 2002 : recrutement 1 an de E. Morin +recrutement 2 ans de E. Kabore (CDD région) Juillet 2003 : recrutement 1 an de A. -S. Valin Journées Bioinformatique des génopoles – Lyon -Octobre 2003

OUEST-Génopole® : organisation Composantes Mer – Agronomie – Santé – Bio. Informatique Cinq plates-formes technologiques - Séquençage/Génotypage - Transcriptome - Protéome - Exploration fonctionnelle - Bio-informatique Groupement d'intérêt scientifique (GIS) en 2002 Journées Bioinformatique des génopoles – Lyon -Octobre 2003

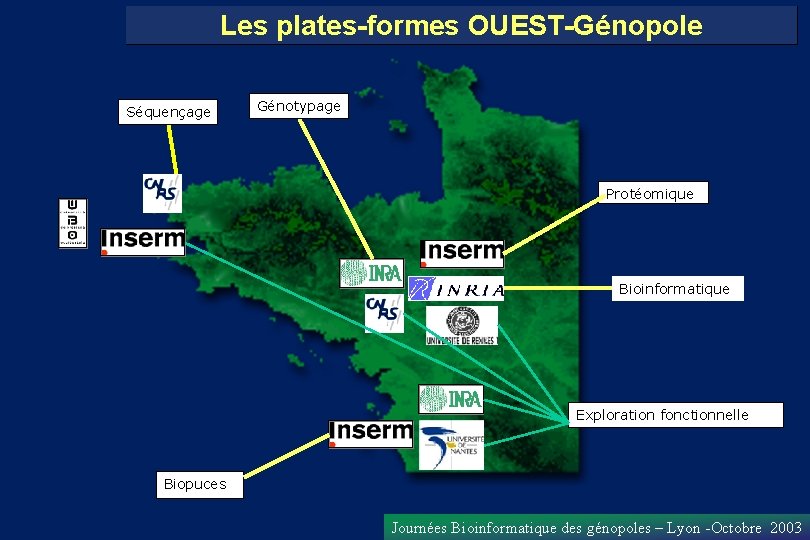
Les plates-formes OUEST-Génopole Séquençage Génotypage Protéomique Bioinformatique Exploration fonctionnelle Biopuces Journées Bioinformatique des génopoles – Lyon -Octobre 2003

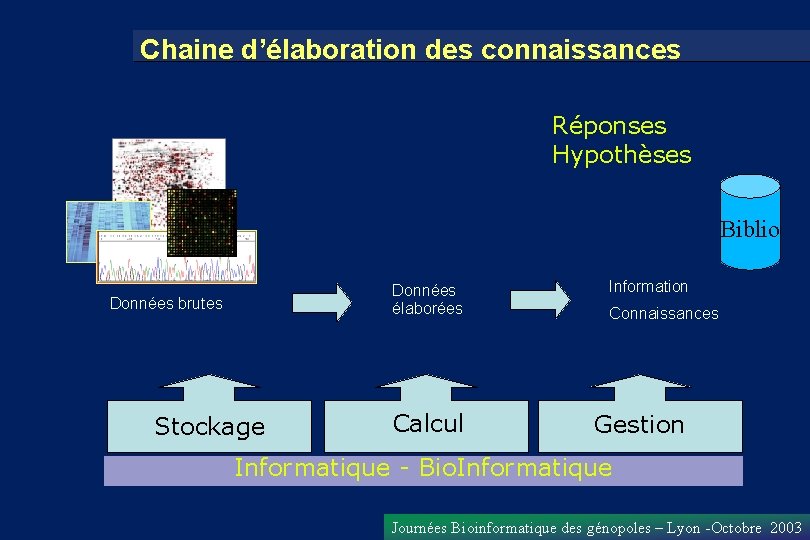
Chaine d’élaboration des connaissances Réponses Hypothèses Biblio Données brutes Stockage Données élaborées Information Calcul Gestion Connaissances Informatique - Bio. Informatique Journées Bioinformatique des génopoles – Lyon -Octobre 2003

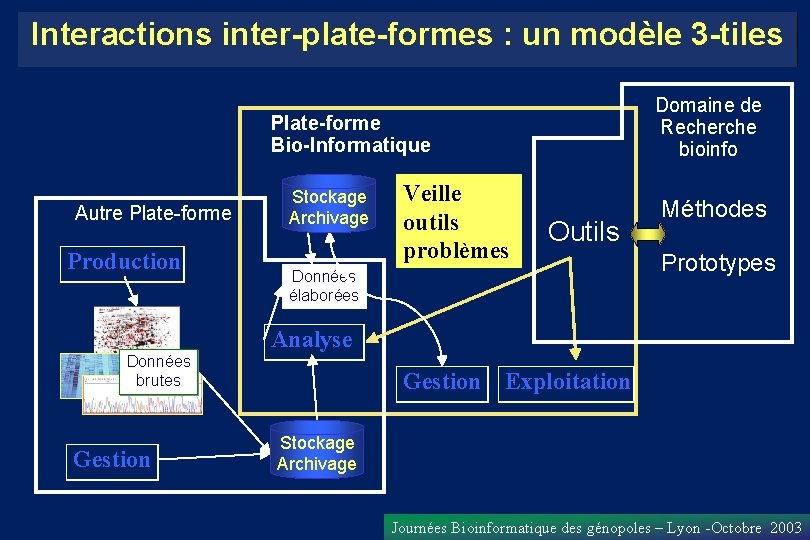
Interactions inter-plate-formes : un modèle 3 -tiles Domaine de Recherche bioinfo Plate-forme Bio-Informatique Autre Plate-forme Production Stockage Archivage Veille outils problèmes Outils Données élaborées Méthodes Prototypes Analyse Données brutes Gestion Exploitation Stockage Archivage Journées Bioinformatique des génopoles – Lyon -Octobre 2003

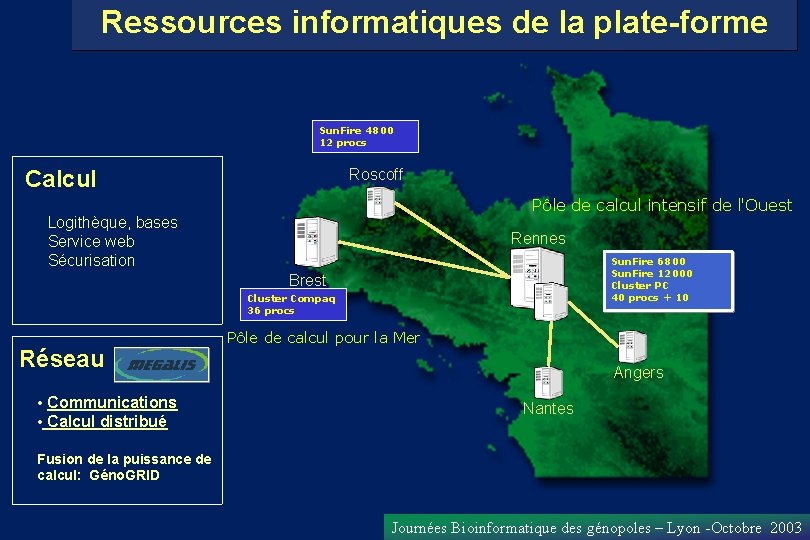
Ressources informatiques de la plate-forme Sun. Fire 4800 12 procs Calcul Roscoff Pôle de calcul intensif de l'Ouest Logithèque, bases Service web Sécurisation Rennes Sun. Fire 6800 Sun. Fire 12000 Cluster PC 40 procs + 10 Brest Cluster Compaq 36 procs Réseau • Communications • Calcul distribué Pôle de calcul pour la Mer Angers Nantes Fusion de la puissance de calcul: Géno. GRID Journées Bioinformatique des génopoles – Lyon -Octobre 2003

Parallélisme et architectures pour la génomique • Motivation – le volume des données génomique double approximativement tous les ans (plusieurs centaines de T bytes en 2010) – la puissance des ordinateurs double tous les 18 mois (loi de Moore) solutions Nouvelles méthodes algorithmiques Usage des machines parallèles Développement de machines spécialisées Journées Bioinformatique des génopoles – Lyon -Octobre 2003

Parallélisme • Projet Géno. GRID (resp. D. Lavenier) – une grille expérimentale pour la génomique – objectif : mutualiser les ressources (banque de données, machines) sur des calculs intensifs – deux niveaux de parallélisation • grille = plusieurs nœuds • nœuds = machines parallèles » cluster de PC » supercalculateurs Roscoff Lille Rouen – applications : • repliement des protéines • comparaison génomes • détection de séquences répétées Rennes Brest Angers Journées Bioinformatique des génopoles – Lyon -Octobre 2003

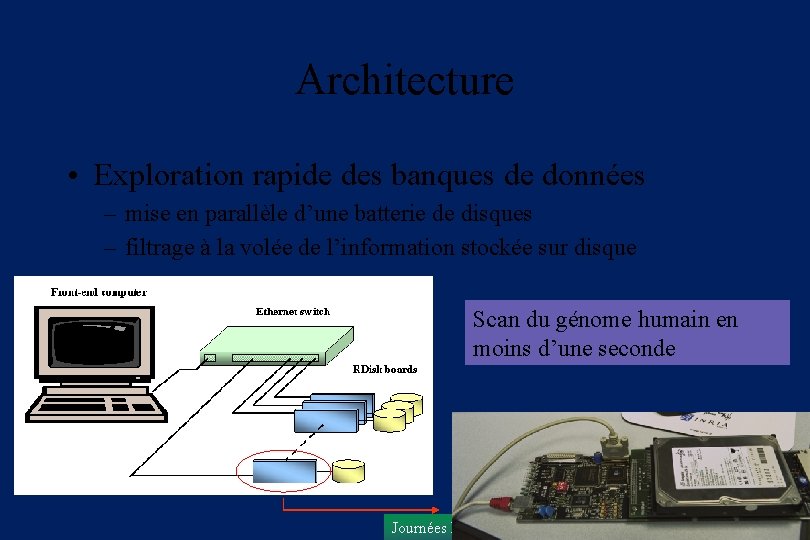
Architecture • Exploration rapide des banques de données – mise en parallèle d’une batterie de disques – filtrage à la volée de l’information stockée sur disque Scan du génome humain en moins d’une seconde Journées Bioinformatique des génopoles – Lyon -Octobre 2003

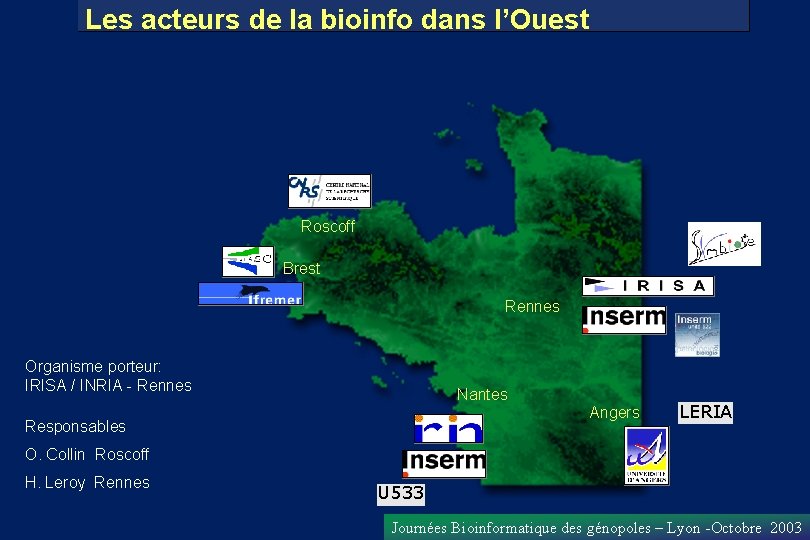
Les acteurs de la bioinfo dans l’Ouest Roscoff Brest Rennes Organisme porteur: IRISA / INRIA - Rennes Nantes Angers Responsables LERIA O. Collin Roscoff H. Leroy Rennes U 533 Journées Bioinformatique des génopoles – Lyon -Octobre 2003

Le réseau : animation Responsables plate-forme: O. Collin (SBR) + pôle Mer, CS Genopole et H. Leroy (Irisa) + système, Genogrid Comité d’animation • Relations inter plate-formes • Stratégie domaine bio-informatique Comité correspondants • Relations utilisateurs • Mise en place des actions Laure Berti-Equille Audrey Bihouée François Brücker Olivier Collin François Coste Christian Delamarche Didier Flament Marc Ferré Guillaume Fertin Christiane Guillouzo Nathalie Guitton Jin-Kao Hao Yannick Jacques Esther Kaboré Gilles Lassalle Dominique Lavenier Jean Léger Sandrine Laguarrigue Hugues Leroy Jérôme Mikolajczak Emmanuelle Morin Fouzia Moussouni Jacques Nicolas Philippe Picouet Charles Pineau Stéphanie Prioul Jean-Michel Richer Irèna Rusu Michel Samson Anne Siegel Dominique Tessier Tranh Vin Journées Bioinformatique des génopoles – Lyon -Octobre 2003

Postes CDD sur Rennes Esther Kaboré (sept 2002) Ingénieur bases de données : gérer les comptes et les moyens de stockage sur le serveur du PCIO. ; accès et mise à jour d’un miroir local des principales banques publiques ; Coordination des choix sur chaque site de développement des bases de données proposition d’outils génériques pour le développement de bases de données spécialisées dans les laboratoires. Emmanuelle Morin (sept 2002) Ingénieur en bioinformatique : choix, gestion et maintenance des logiciels applicatifs nécessaires en particulier pour l’étude de génomes complets; développement d’interfaces adaptées à un usage direct par les laboratoires de biologie des chaînes de traitement logiciel; Proposition de formations sur les outils de la plate-forme; intégration des outils de bio-info produits dans le cadre de la Génopole. � Anne-Sophie Valin (juil 2003) Ingénieur en informatique : développement de la plate-forme de recherche et d'extraction de motifs (thème bioinfo génopole) veille logicielle dans ce domaine Formation aux outils, aide à l’utilisation. Journées Bioinformatique des génopoles – Lyon -Octobre 2003

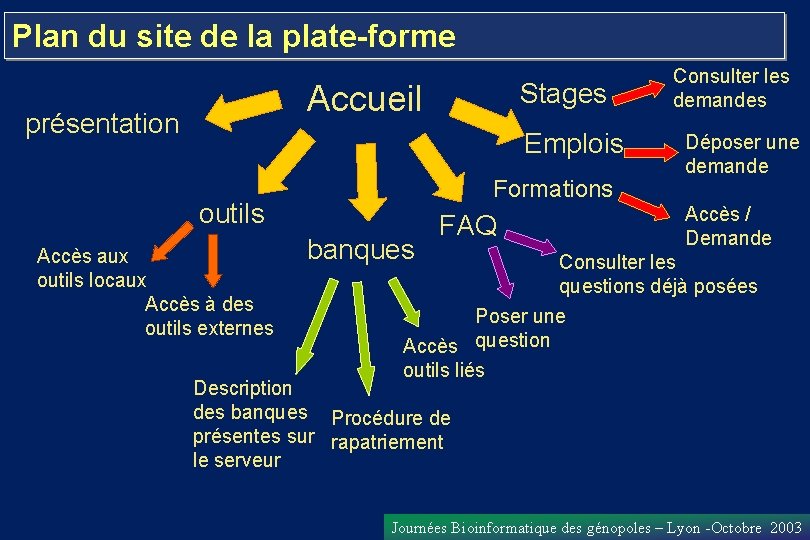
Plan du site de la plate-forme Stages Accueil présentation Emplois Formations outils Accès aux outils locaux Accès à des outils externes banques FAQ Consulter les demandes Déposer une demande Accès / Demande Consulter les questions déjà posées Poser une Accès question outils liés Description des banques Procédure de présentes sur rapatriement le serveur Journées Bioinformatique des génopoles – Lyon -Octobre 2003

Outils qui utilisent les ressources de calcul de la plate-forme Wisconsin package standard Blast Multiple rare Fast. Me rare Plate-forme de recherche exclusif et découverte de motifs (Smile, Model, Pratt…) Geno. Frag exclusif Journées Bioinformatique des génopoles – Lyon -Octobre 2003

Les banques de données publiques Genbank : version 137. 0 (août 2003) PIR : version 77 (juillet 2003) Swiss-Prot : version 41 (février 2003) Banques de génomes : - 10 génomes eucaryotes - Beaucoup de génomes bactériens Mise à jour régulière Développement de banques à façon Rsync: mise à jour des sites distants (Ifremer, Roscoff) Journées Bioinformatique des génopoles – Lyon -Octobre 2003

Quelques bases de données de la génopole Santé • INSERM Rennes : Entrepôt de données « foie » • GERM Rennes : base fédérée Expasy, base de donnée « Reproduction » , base de données défensines • INSERM Nantes/Rennes : base de données biopuces • CNRS Rennes : base de données «canaux membranaires » Agro • INRA : Agena • INRA : Stressgenes Mer • CNRS Roscoff : Génomer base de données EST Structuration initiale par domaine puis ouverture progressive Point clé: sécurisation des données Harmonisation des approches, développement d’outils communs Journées Bioinformatique des génopoles – Lyon -Octobre 2003

Exemple d’utilisateur de la plate-forme : Identification et Cartographie de 10, 000 gènes canins Journées Bioinformatique des génopoles – Lyon -Octobre 2003

Cartographie sur hybrides irradiés : ordonnancement des marqueurs par approche TSP (Hitte et al. J. Hered 2003) TSP variant maps 0 20 40 60 80 100% MLE OCB Consensus map Mk_# Mk_Nam e |======== [ 35 35 35 38 35] (mk_35) EST 7 A 10# 22 |========== [ 33 33 33] (mk_33) EST 3 C 10 -B# 53 |======== [ 13 13 13 35 13] (mk_13) BAC_375 -K 3# 39 |======== [ 12 12 12 13 12] (mk_12) BAC_375 -F 13# 22 |==== [ 6 6 51 12 70] (mk_6) BAC_372 -E 22# 22 |====== [ 70 70 70 6 51] (mk_70) VCAM 1 54 |====== [ 51 51 6] (mk_51) FH 3445# 54 |======== [ 48 48 48 70 48] (mk_48) FH 3246# 39 |======== [ 36 36 36 48 36] (mk_36) FH 2119 39 |======== [ 28 28 28 36 28] (mk_28) EST 17 G 5# 23 |======== [ 34 34 34 28 34] (mk_34) EST 4 F 4 -B# 39 |======== [ 49 49 49 34 49] (mk_49) FH 3282# 39 |======== [ 26 26 26 49 26] (mk_26) EST 14 G 8# 0 20 40 60 80 Phase d’Analyse : Ordonner 100 marqueurs 1/2 h (- 5 CPUs PCIO-IDEFIX) 100% Journées Bioinformatique des génopoles – Lyon -Octobre 2003

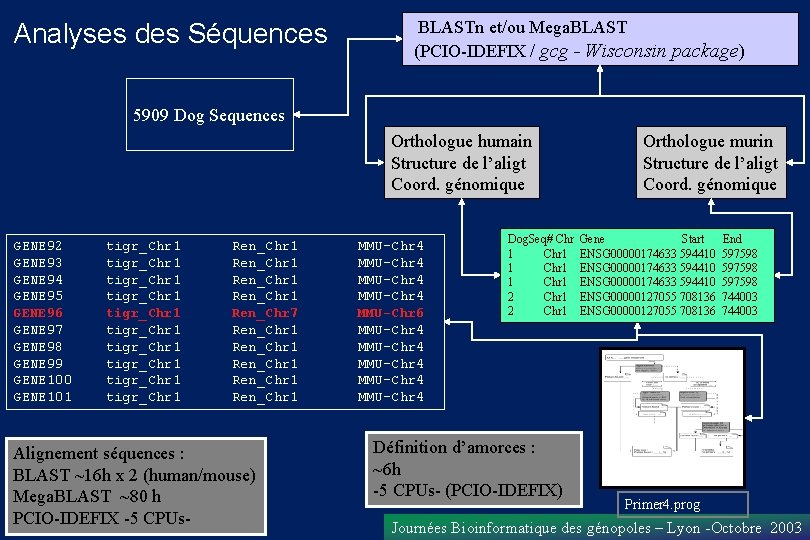
Analyses des Séquences BLASTn et/ou Mega. BLAST (PCIO-IDEFIX / gcg - Wisconsin package) 5909 Dog Sequences Orthologue humain Structure de l’aligt Coord. génomique GENE 92 GENE 93 GENE 94 GENE 95 GENE 96 GENE 97 GENE 98 GENE 99 GENE 100 GENE 101 tigr_Chr 1 tigr_Chr 1 tigr_Chr 1 Ren_Chr 1 Ren_Chr 7 Ren_Chr 1 Ren_Chr 1 Alignement séquences : BLAST ~16 h x 2 (human/mouse) Mega. BLAST ~80 h PCIO-IDEFIX -5 CPUs- MMU-Chr 4 MMU-Chr 6 MMU-Chr 4 MMU-Chr 4 Dog. Seq# Chr 1 1 Chr 1 2 Chr 1 Définition d’amorces : ~6 h -5 CPUs- (PCIO-IDEFIX) Orthologue murin Structure de l’aligt Coord. génomique Gene Start ENSG 00000174633 594410 ENSG 00000127055 708136 End 597598 744003 Primer 4. prog Journées Bioinformatique des génopoles – Lyon -Octobre 2003

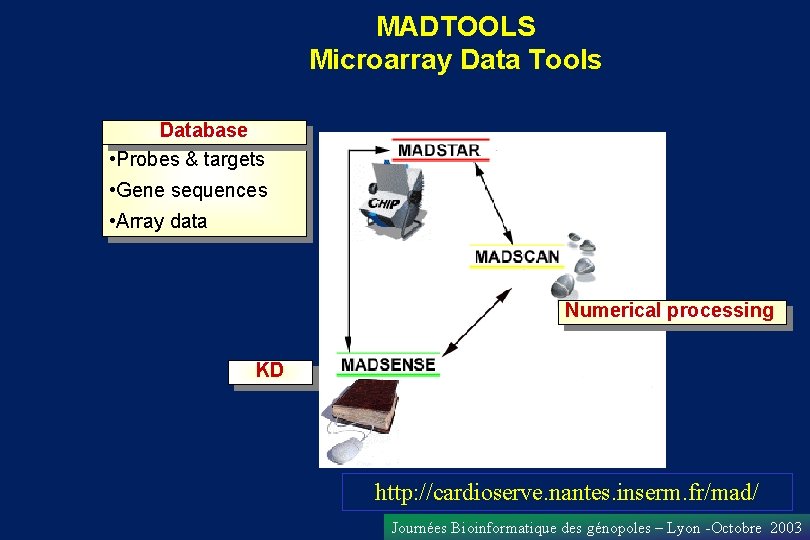
Exemple de développement au niveau d’une plate -forme bio http: //www. madtools. org Ouest Génopole. IFR – 26 INSERM U. 533 2003 Journées Bioinformatique des génopoles Lyon -Octobre

MADTOOLS Microarray Data Tools Database • Probes & targets • Gene sequences • Array data Numerical processing KD http: //cardioserve. nantes. inserm. fr/mad/ Journées Bioinformatique des génopoles – Lyon -Octobre 2003

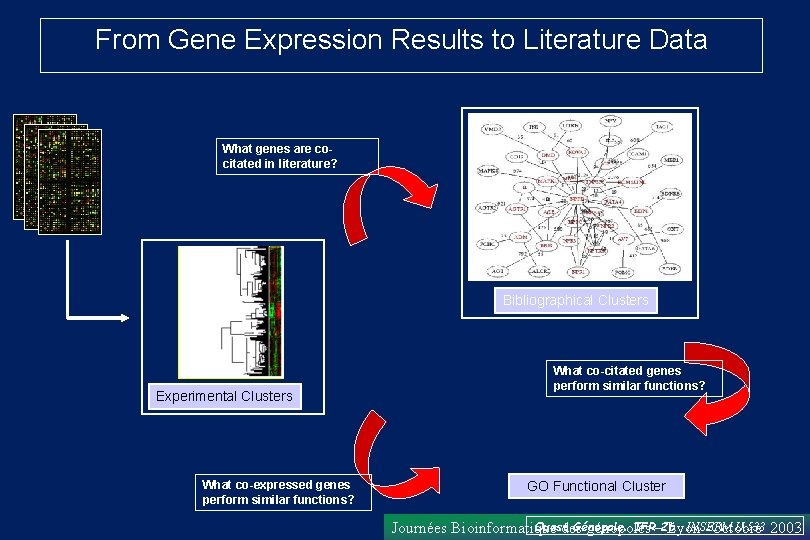
From Gene Expression Results to Literature Data What genes are cocitated in literature? Bibliographical Clusters Experimental Clusters What co-expressed genes perform similar functions? What co-citated genes perform similar functions? GO Functional Cluster Ouest Génopole. IFR– 26 INSERM U. 533 2003 Journées Bioinformatique des génopoles Lyon -Octobre

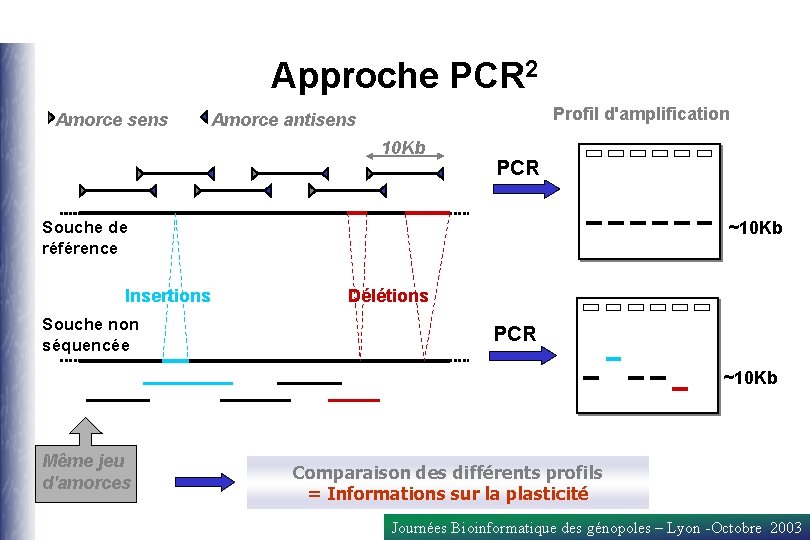
Exemple de demande de service ayant conduit à une collaboration puis au développement d’un outil Logiciel de Recherche d'Amorces Optimisées pour l’amplification de Chromosomes Bactériens par PCR Longue Portée • Nouri BEN ZAKOUR Laboratoire de Microbiologie UMR 1055 INRA ENSAR • Dominique LAVENIER IRISA / CNRS - équipe Symbiose Journées Bioinformatique des génopoles – Lyon -Octobre 2003

Approche PCR 2 Amorce sens Profil d'amplification Amorce antisens 10 Kb PCR ~10 Kb Souche de référence Insertions Souche non séquencée Délétions PCR ~10 Kb Même jeu d'amorces Comparaison des différents profils = Informations sur la plasticité Journées Bioinformatique des génopoles – Lyon -Octobre 2003

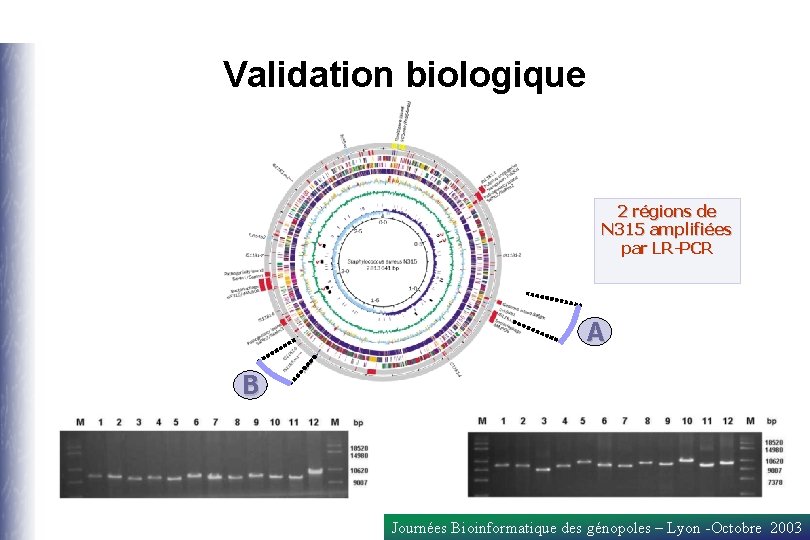
Validation biologique 2 régions de N 315 amplifiées par LR-PCR A B Journées Bioinformatique des génopoles – Lyon -Octobre 2003

Bases de données spécialisées Esther Kaboré Didier Flament Journées Bioinformatique des génopoles – Lyon -Octobre 2003

Recherche de motifs et de signatures Cynthia Alland Emmanuelle Morin Anne-Sophie Valin Journées Bioinformatique des génopoles – Lyon -Octobre 2003

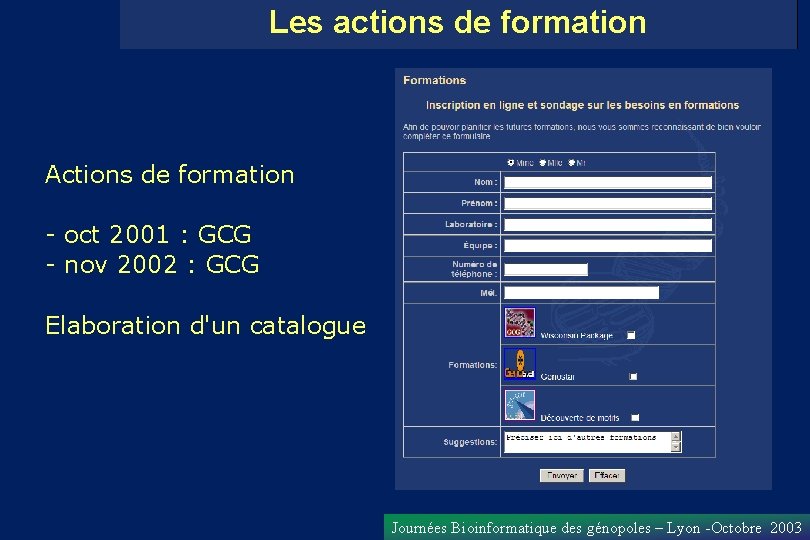
Les actions de formation Actions de formation - oct 2001 : GCG - nov 2002 : GCG Elaboration d'un catalogue Journées Bioinformatique des génopoles – Lyon -Octobre 2003

L'existant depuis 2000 • DEA Get. I • Maîtrise de Biologie • Maîtrise de d'informatique 31 étudiants formés 15 thèses en cours Journées Bioinformatique des génopoles – Lyon -Octobre 2003

Promotion 2003 • 12 étudiants • 6 Biologistes • 6 informaticiens Journées Bioinformatique des génopoles – Lyon -Octobre 2003

A partir de 2004 • Licence de Biologie et Informatique • Master de bio-informatique Journées Bioinformatique des génopoles – Lyon -Octobre 2003

Le site de OUEST-Génopole® http: //genouest. no-ip. org Journées Bioinformatique des génopoles – Lyon -Octobre 2003

Perspectives : Une richesse largement inexploitée : Banques de génomes complets Génomes Eukaryotes: Homo sapiens, Mus musculus, Ratus Norvegicus, Oryza sativa, Plasmodium falciparum, Caenorhabditis elegans, Saccharomyces serevisiae, Drosophila melagongaster, Encephalitozoon cuniculi Génomes Bactériens: Escherichia coli, Prochloroccocus marinus, Salmonella typhi, Staphylococcus aureus, vibrio cholerae, Neisseria meningitidis Yersinia pestis, … Journées Bioinformatique des génopoles – Lyon -Octobre 2003

Journées Bioinformatique des génopoles – Lyon -Octobre 2003
 Organigramme genopole
Organigramme genopole Cordelia schmid
Cordelia schmid Inria holidays dataset
Inria holidays dataset Inria email
Inria email Inria grenoble
Inria grenoble Carte de la belgique
Carte de la belgique Nord ouest bretagne
Nord ouest bretagne Ce cofely sud ouest
Ce cofely sud ouest Thomas cook voyages 9 rue du puits mauger 35000 rennes
Thomas cook voyages 9 rue du puits mauger 35000 rennes Alfadi rennes
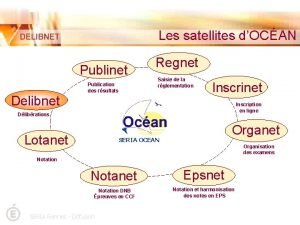
Alfadi rennes Delibnet
Delibnet Ent rennes 1
Ent rennes 1 Sablage rennes
Sablage rennes Ose rennes 1
Ose rennes 1 Kristell dicharry
Kristell dicharry Cegidd rennes
Cegidd rennes Conseil dynamiser collaborateurs strasbourg
Conseil dynamiser collaborateurs strasbourg Cursus rennes 2
Cursus rennes 2 Rennes métropole
Rennes métropole Point splatting
Point splatting Bibliothèque champs manceaux rennes
Bibliothèque champs manceaux rennes Dr pierre lebaud
Dr pierre lebaud Domoticien onisep
Domoticien onisep Espace svt ac rennes dissection souris
Espace svt ac rennes dissection souris Mission assist
Mission assist Assist user guide
Assist user guide Cisco smart assist
Cisco smart assist The whips assist the party leaders by
The whips assist the party leaders by What cognitive strategies assist our problem solving
What cognitive strategies assist our problem solving Medi assist escalation matrix
Medi assist escalation matrix Bmv ambu
Bmv ambu Intelligent assist device
Intelligent assist device Mcdp-1 states that trust
Mcdp-1 states that trust Hormonal stimulus
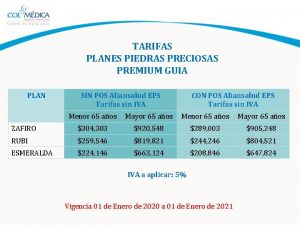
Hormonal stimulus Plan rubi colmedica
Plan rubi colmedica Aig travel assist
Aig travel assist Peer assist
Peer assist