UNIVERSIDADE DE VORA TODAS AS VIAS VO DAR







- Slides: 31

UNIVERSIDADE DE ÉVORA TODAS AS VIAS VÃO DAR AO PROTEASSOMA … OU QUASE TODAS! Docente: Prof. Paulo de Oliveira Discentes: • Ana Filipa Robalo 30703 • Ana Rita Hipólito 30237 23 de Junho de 2014 Biologia do Desenvolvimento 2013 -2014

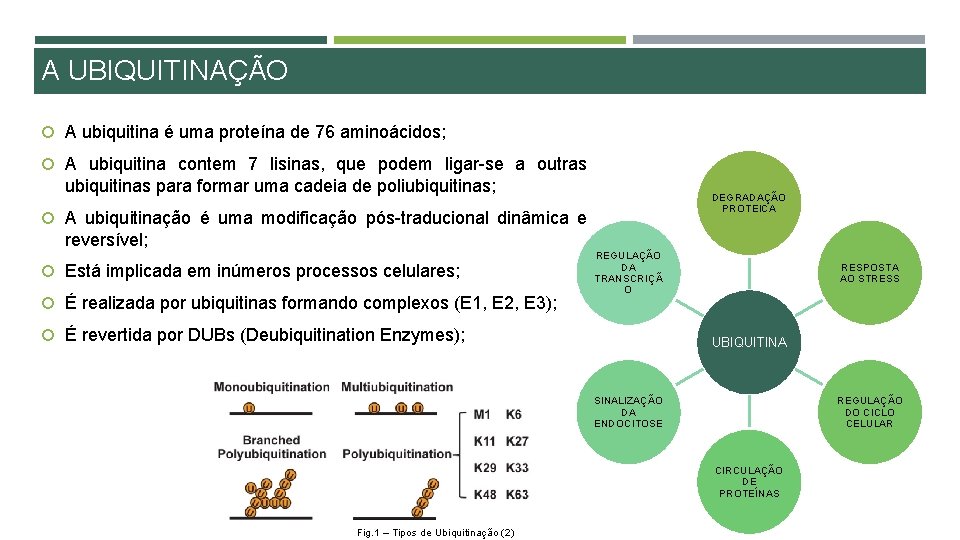
A UBIQUITINAÇÃO A ubiquitina é uma proteína de 76 aminoácidos; A ubiquitina contem 7 lisinas, que podem ligar-se a outras ubiquitinas para formar uma cadeia de poliubiquitinas; DEGRADAÇÃO PROTEICA A ubiquitinação é uma modificação pós-traducional dinâmica e reversível; Está implicada em inúmeros processos celulares; É realizada por ubiquitinas formando complexos (E 1, E 2, E 3); REGULAÇÃO DA TRANSCRIÇÃ O É revertida por DUBs (Deubiquitination Enzymes); RESPOSTA AO STRESS UBIQUITINA SINALIZAÇÃO DA ENDOCITOSE REGULAÇÃO DO CICLO CELULAR CIRCULAÇÃO DE PROTEÍNAS Fig. 1 – Tipos de Ubiquitinação (2)

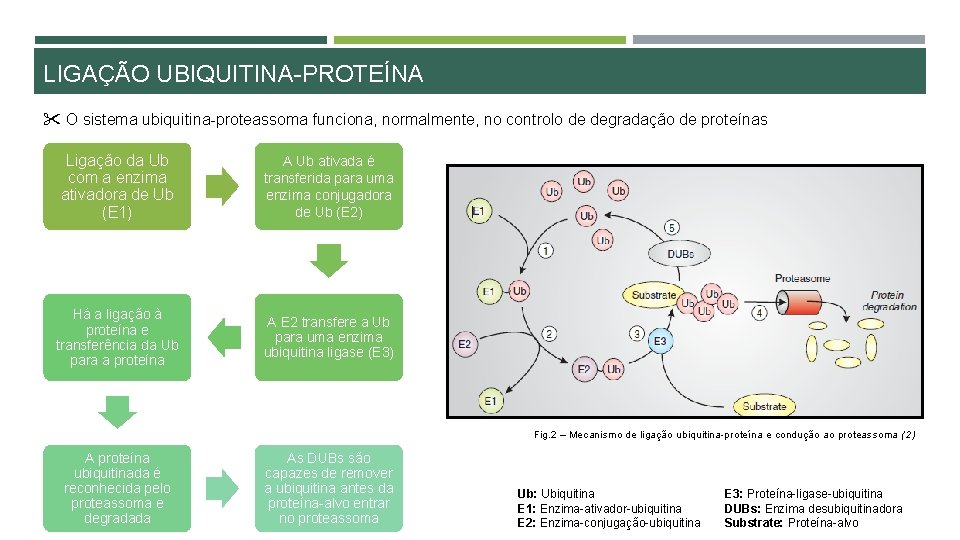
LIGAÇÃO UBIQUITINA-PROTEÍNA " O sistema ubiquitina-proteassoma funciona, normalmente, no controlo de degradação de proteínas Ligação da Ub com a enzima ativadora de Ub (E 1) A Ub ativada é transferida para uma enzima conjugadora de Ub (E 2) Há a ligação à proteína e transferência da Ub para a proteína A E 2 transfere a Ub para uma enzima ubiquitina ligase (E 3) Fig. 2 – Mecanismo de ligação ubiquitina-proteína e condução ao proteassoma (2) A proteína ubiquitinada é reconhecida pelo proteassoma e degradada As DUBs são capazes de remover a ubiquitina antes da proteína-alvo entrar no proteassoma Ub: Ubiquitina E 1: Enzima-ativador-ubiquitina E 2: Enzima-conjugação-ubiquitina E 3: Proteína-ligase-ubiquitina DUBs: Enzima desubiquitinadora Substrate: Proteína-alvo

UBIQUITINAÇÃO E A VIA NF-KB NF-k. B é um fator de transcrição Fatores de transcrição da família do NF-k. B são reguladores de muitos processos incluindo imunidade, inflamação e apoptose Da família do NF-k. B fazem parte: Rel-A (p 65), c-Rel, Rel-B, p 50 e p 52 A ubiquitina está envolvida em pelo menos 3 passos nesta via A ativação da via NF-k. B é classificada como canónica e não canónica, dependendo se a ativação envolve a degradação de Ik. B ou processamento de p 100

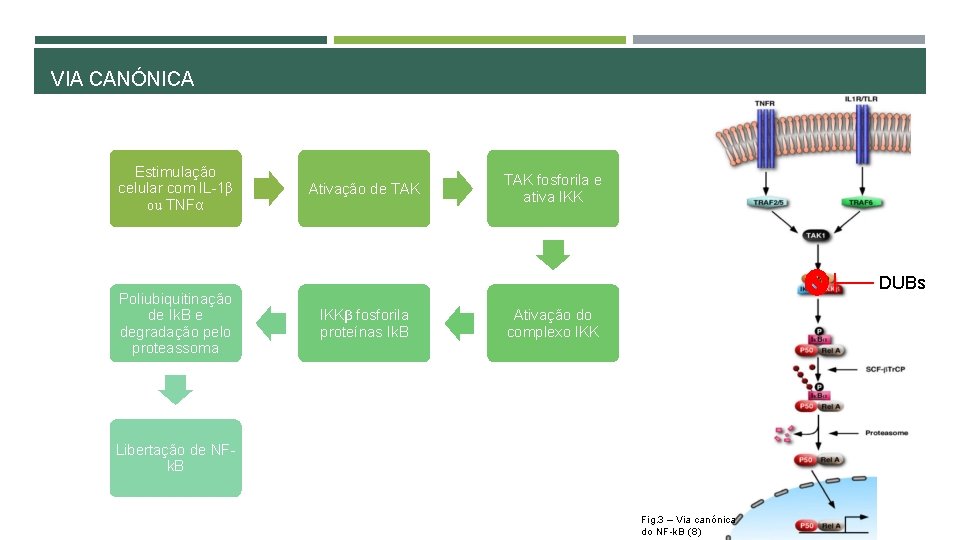
VIA CANÓNICA Estimulação celular com IL-1β ou TNFα Poliubiquitinação de Ik. B e degradação pelo proteassoma Ativação de TAK fosforila e ativa IKK DUBs IKKβ fosforila proteínas Ik. B Ativação do complexo IKK Libertação de NFk. B Fig. 3 – Via canónica do NF-k. B (8)

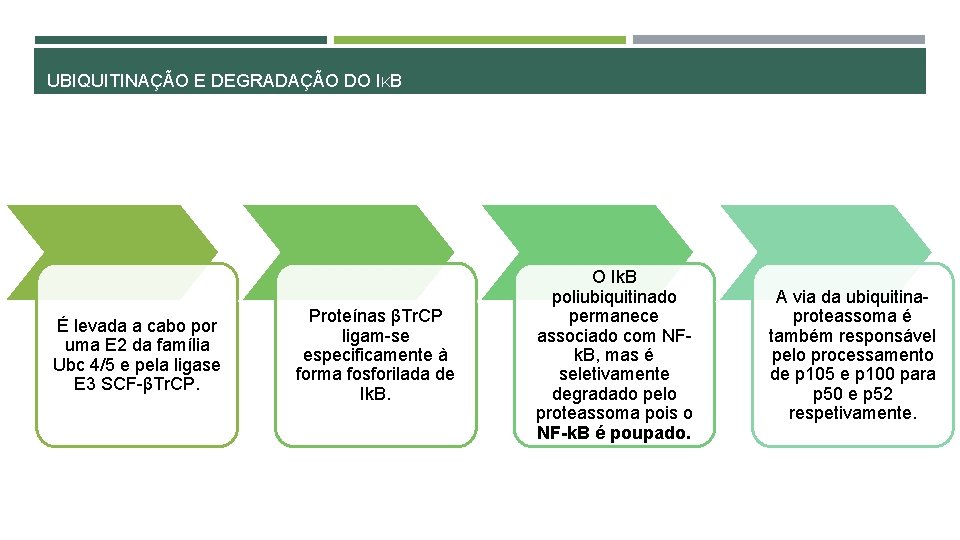
UBIQUITINAÇÃO E DEGRADAÇÃO DO IKB É levada a cabo por uma E 2 da família Ubc 4/5 e pela ligase E 3 SCF-βTr. CP. Proteínas βTr. CP ligam-se especificamente à forma fosforilada de Ik. B. O Ik. B poliubiquitinado permanece associado com NFk. B, mas é seletivamente degradado pelo proteassoma pois o NF-k. B é poupado. A via da ubiquitinaproteassoma é também responsável pelo processamento de p 105 e p 100 para p 50 e p 52 respetivamente.

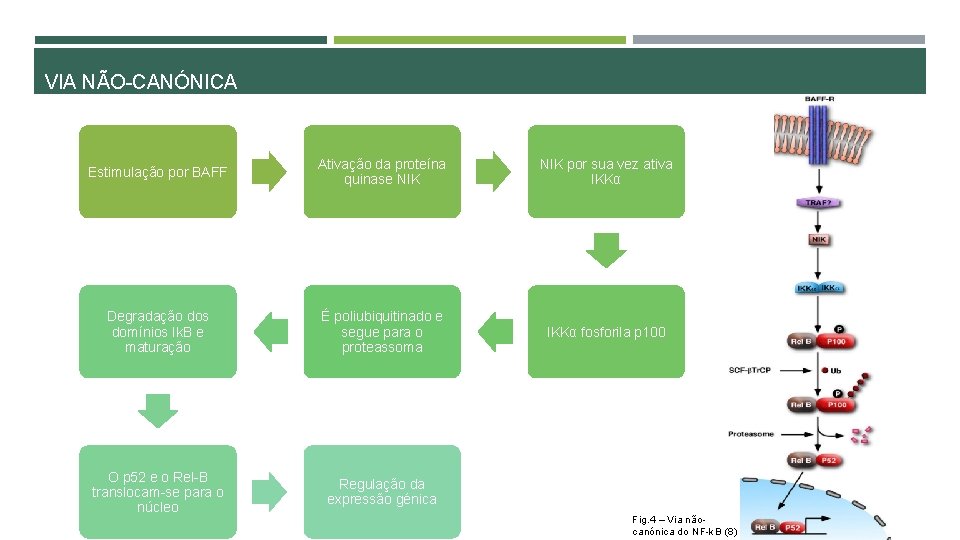
VIA NÃO-CANÓNICA Estimulação por BAFF Ativação da proteína quinase NIK por sua vez ativa IKKα Degradação dos domínios Ik. B e maturação É poliubiquitinado e segue para o proteassoma IKKα fosforila p 100 O p 52 e o Rel-B translocam-se para o núcleo Regulação da expressão génica Fig. 4 – Via nãocanónica do NF-k. B (8)

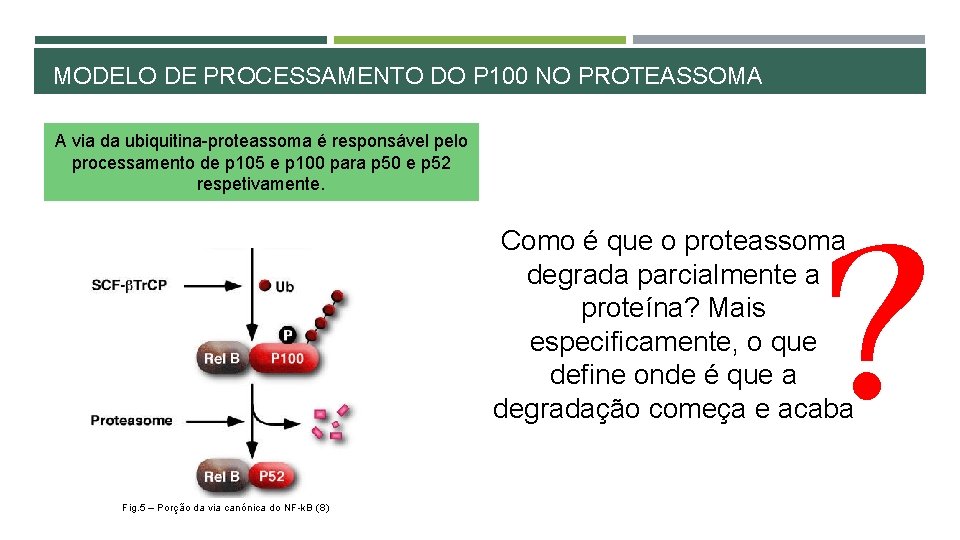
MODELO DE PROCESSAMENTO DO P 100 NO PROTEASSOMA A via da ubiquitina-proteassoma é responsável pelo processamento de p 105 e p 100 para p 50 e p 52 respetivamente. ? Como é que o proteassoma degrada parcialmente a proteína? Mais especificamente, o que define onde é que a degradação começa e acaba Fig. 5 – Porção da via canónica do NF-k. B (8)

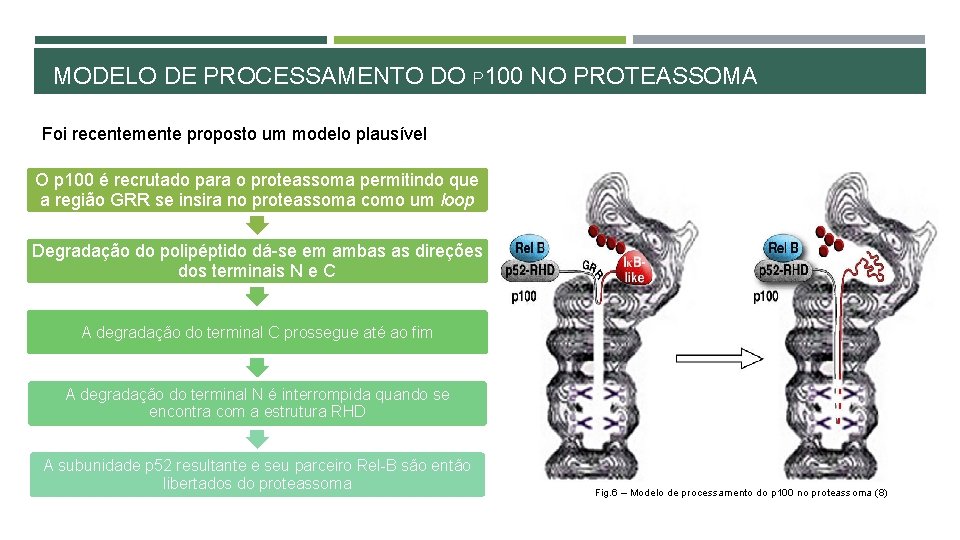
MODELO DE PROCESSAMENTO DO P 100 NO PROTEASSOMA Foi recentemente proposto um modelo plausível O p 100 é recrutado para o proteassoma permitindo que a região GRR se insira no proteassoma como um loop Degradação do polipéptido dá-se em ambas as direções dos terminais N e C A degradação do terminal C prossegue até ao fim A degradação do terminal N é interrompida quando se encontra com a estrutura RHD A subunidade p 52 resultante e seu parceiro Rel-B são então libertados do proteassoma Fig. 6 – Modelo de processamento do p 100 no proteassoma (8)

β UBIQUITINAÇÃO E A VIA TGF Um dos mais importantes reguladores da comunicação celular em todos os organismos multicelulares. Induz a expressão de vários genes É uma citocina envolvida na regulação da proliferação celular, diferenciação, apoptose e migração de diversos tipos celulares. A sinalização anormal pelo TGF-β resulta no desenvolvimento de tumores, e está relacionada com vários tipos de cancro no homem É indispensável a existência de regulação desta sinalização – Recetores, Smads e outros reguladores de sinal

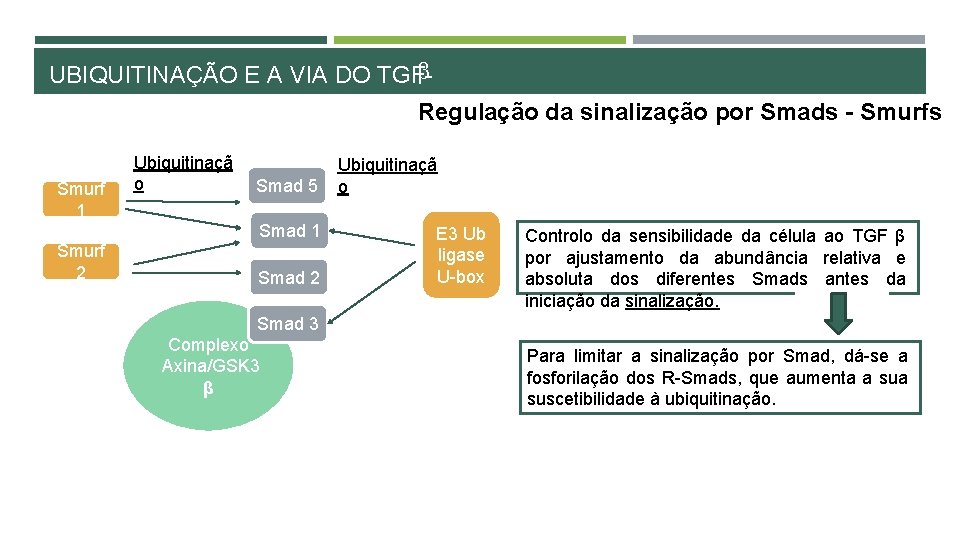
β UBIQUITINAÇÃO E A VIA DO TGFRegulação da sinalização por Smads - Smurfs Smurf 1 Ubiquitinaçã o Smad 5 Smad 1 Smurf 2 Smad 2 Ubiquitinaçã o E 3 Ub ligase U-box Controlo da sensibilidade da célula ao TGF β por ajustamento da abundância relativa e absoluta dos diferentes Smads antes da iniciação da sinalização. Smad 3 Complexo Axina/GSK 3 β Para limitar a sinalização por Smad, dá-se a fosforilação dos R-Smads, que aumenta a suscetibilidade à ubiquitinação.

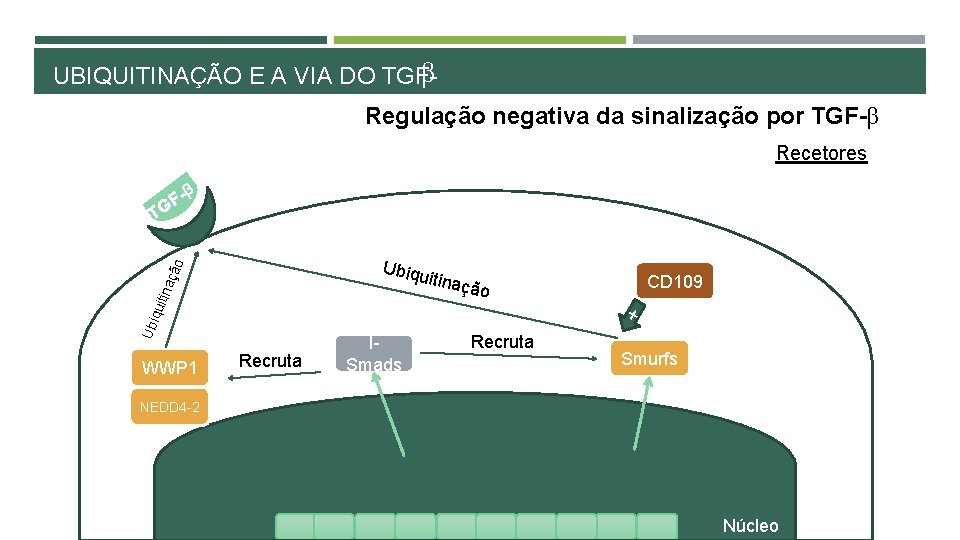
β UBIQUITINAÇÃO E A VIA DO TGFRegulação negativa da sinalização por TGF-β Recetores GF β T ção Ubiqu tina itinaç qui + Ubi WWP 1 CD 109 ão Recruta ISmads Recruta Smurfs NEDD 4 -2 Núcleo

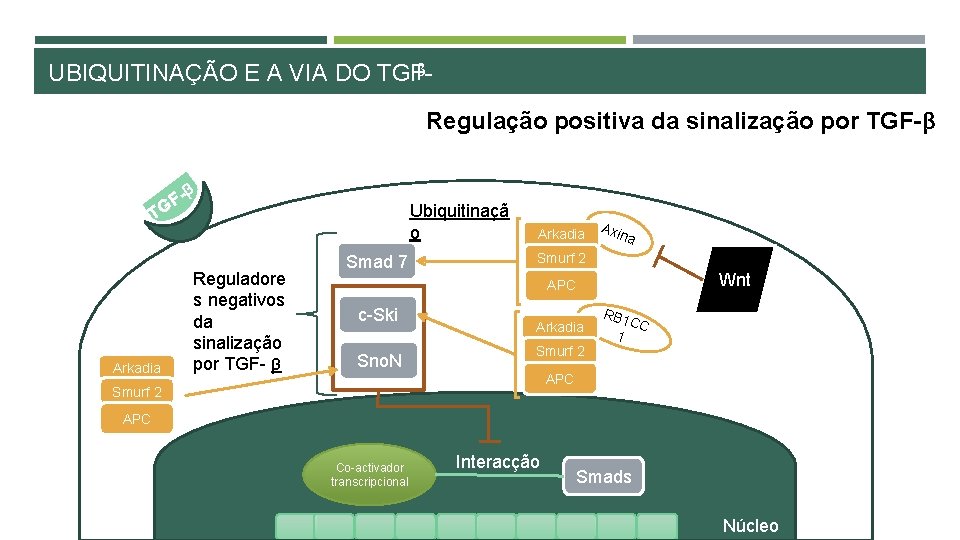
β UBIQUITINAÇÃO E A VIA DO TGF- Regulação positiva da sinalização por TGF-β GF β Ubiquitinaçã o T Arkadia Reguladore s negativos da sinalização por TGF- β Smad 7 Arkadia Axi na Smurf 2 Wnt APC c-Ski Sno. N Arkadia RB 1 1 CC Smurf 2 APC Co-activador transcripcional Interacção Smads Núcleo

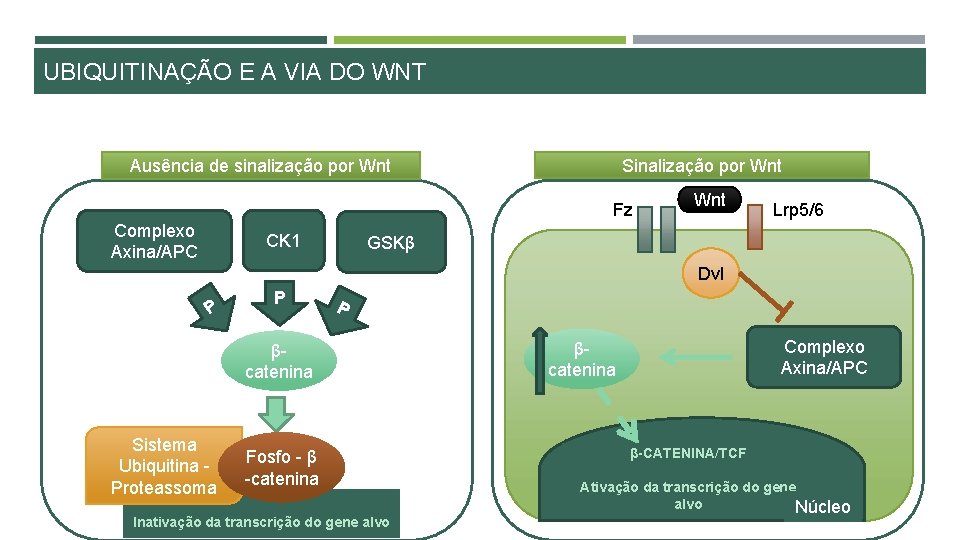
UBIQUITINAÇÃO E A VIA DO WNT A sinalização por Wnt medeia processos de desenvolvimento e homeostase incluindo manutenção das stem cells, crescimento e especificação do destino das células, polaridade celular e migração. A ativação imprópria da sinalização por Wnt está relacionada com uma vasto conjunto de doenças humanas como cancro e doenças neurodegenerativas. Na cascata Wnt/β-catenina, os eventos de sinalização convergem na regulação da degradação mediada por ubiquitinação do fator transcricional β-catenina.

UBIQUITINAÇÃO E A VIA DO WNT Sinalização por Wnt Ausência de sinalização por Wnt Fz Complexo Axina/APC CK 1 Wnt Lrp 5/6 GSKβ Dvl P P P βcatenina Sistema Ubiquitina Proteassoma Fosfo - β -catenina Inativação da transcrição do gene alvo Complexo Axina/APC βcatenina β-CATENINA/TCF Ativação da transcrição do gene alvo Núcleo

UBIQUITINAÇÃO E A VIA DO WNT Várias ubiquitinas ligases marcam a β-catenina para degradação no proteassoma: β-Tr. CP • Reconhece a β-catenina fosforilada e marca-a para degradação Siah-1 • Induzida pelo stress genotóxico – p 53 • Pode agir em conjunto com a E 2 Ubc. H 5 a 53 Jade-1 • Interage diretamente com a β-catenina fosforilada • Age em conjunto com a E 2 Ubc. H 6 ou Ubc. H 2 • Age por um mecanismo dependente de VHL

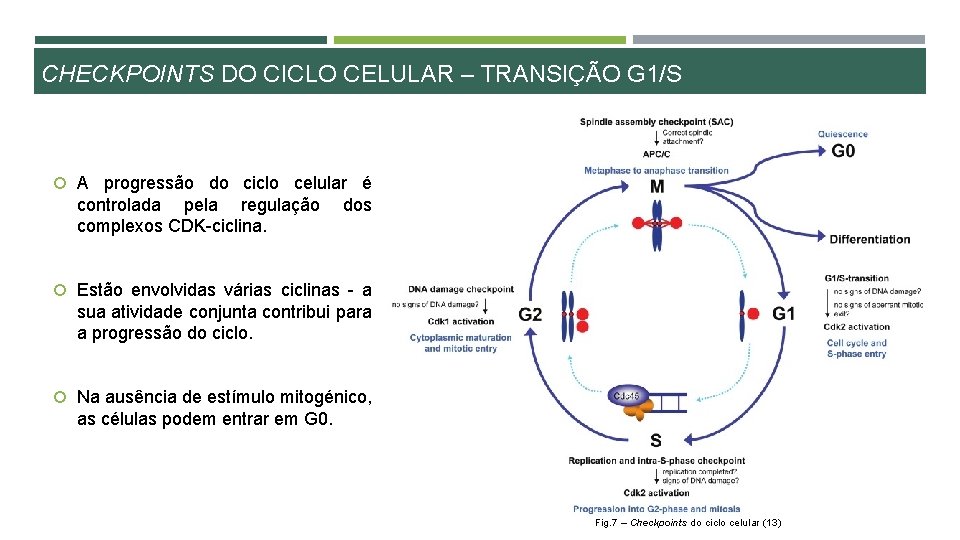
CHECKPOINTS DO CICLO CELULAR – TRANSIÇÃO G 1/S A progressão do ciclo celular é controlada pela regulação complexos CDK-ciclina. dos Estão envolvidas várias ciclinas - a sua atividade conjunta contribui para a progressão do ciclo. Na ausência de estímulo mitogénico, as células podem entrar em G 0. Fig. 7 – Checkpoints do ciclo celular (13)

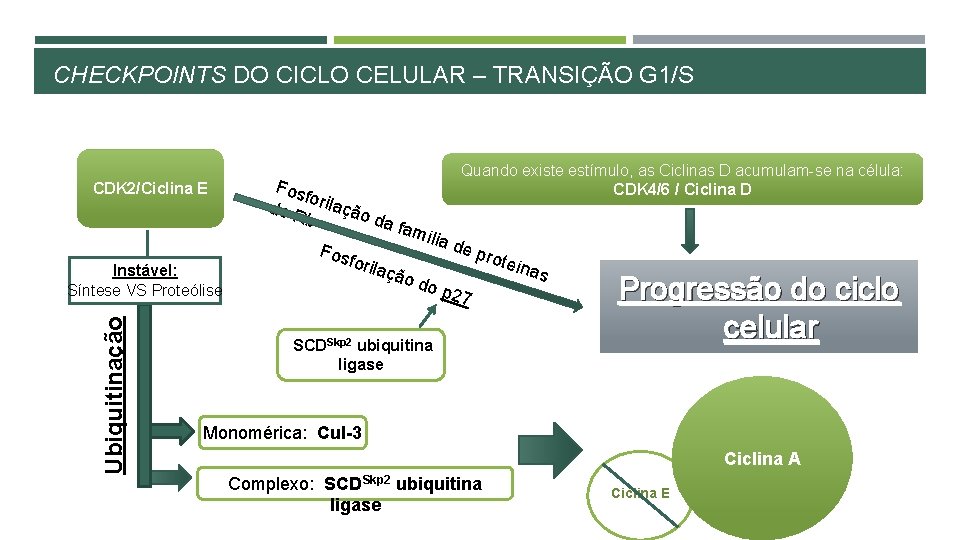
CHECKPOINTS DO CICLO CELULAR – TRANSIÇÃO G 1/S CDK 2/Ciclina E Ubiquitinação Instável: Síntese VS Proteólise Quando existe estímulo, as Ciclinas D acumulam-se na célula: CDK 4/6 / Ciclina D Fos fo do R rilação da f b a Fos foril mília açã o do de p p 27 SCDSkp 2 ubiquitina ligase rote ínas Progressão do ciclo celular Monomérica: Cul-3 Ciclina A Complexo: SCDSkp 2 ubiquitina ligase Ciclina E

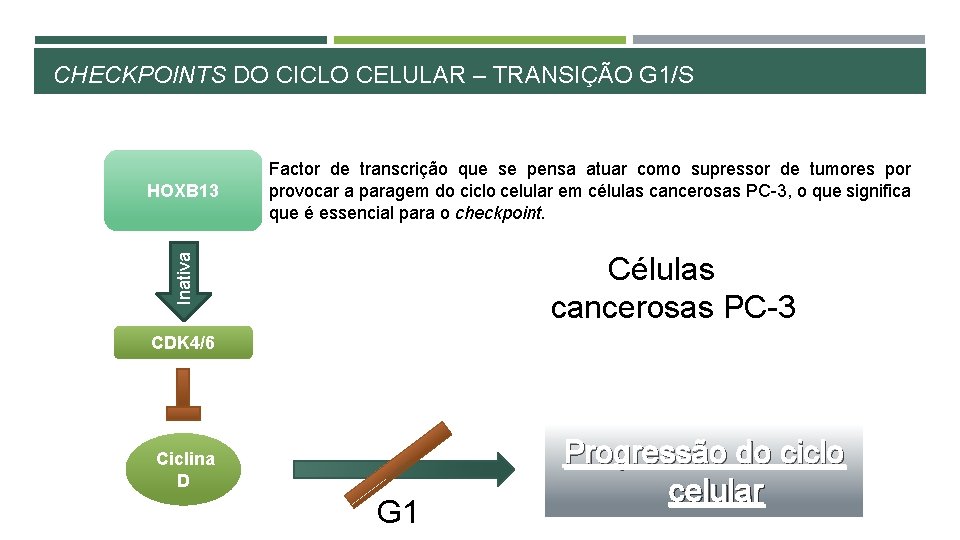
CHECKPOINTS DO CICLO CELULAR – TRANSIÇÃO G 1/S Células cancerosas PC-3 Inativa HOXB 13 Factor de transcrição que se pensa atuar como supressor de tumores por provocar a paragem do ciclo celular em células cancerosas PC-3, o que significa que é essencial para o checkpoint. CDK 4/6 Ciclina D G 1 Progressão do ciclo celular

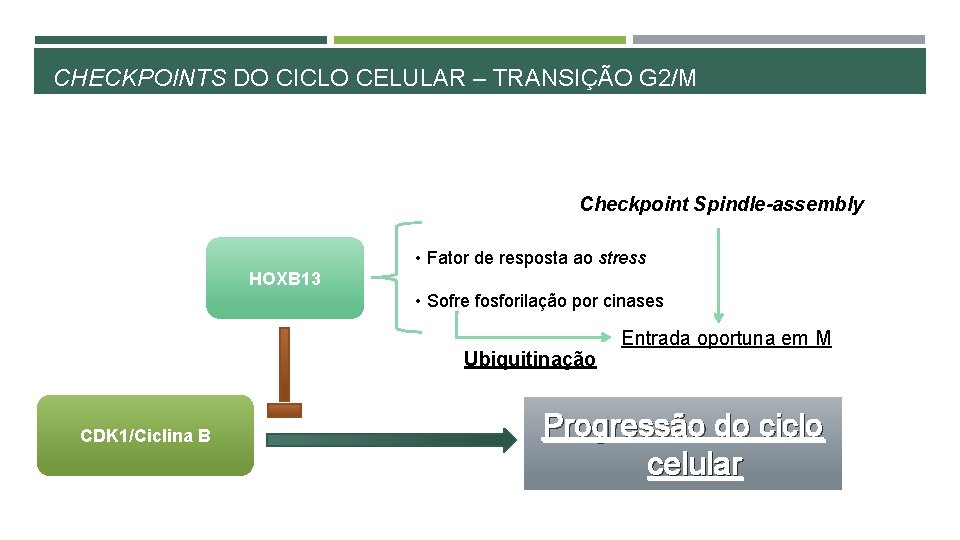
CHECKPOINTS DO CICLO CELULAR – TRANSIÇÃO G 2/M Checkpoint Spindle-assembly • Fator de resposta ao stress HOXB 13 • Sofre fosforilação por cinases Ubiquitinação CDK 1/Ciclina B Entrada oportuna em M Progressão do ciclo celular

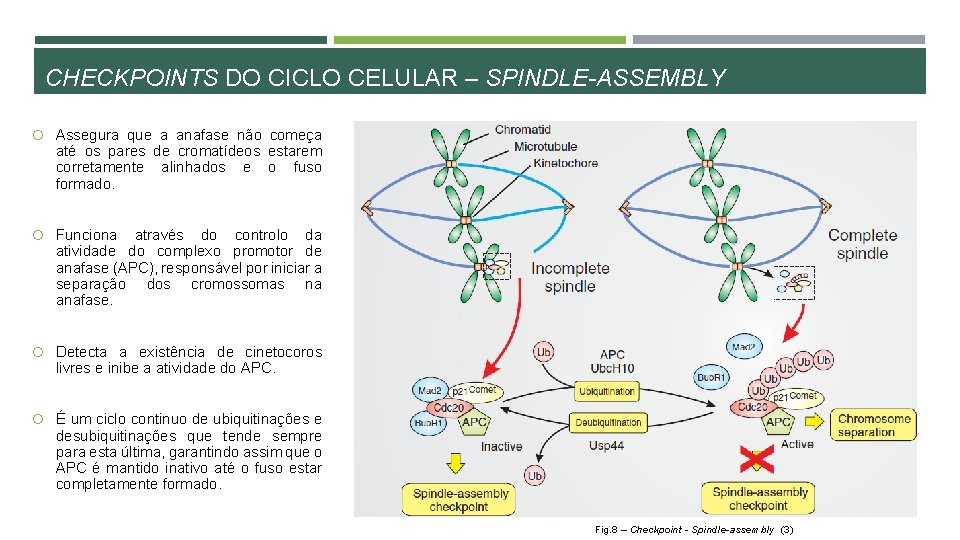
CHECKPOINTS DO CICLO CELULAR – SPINDLE-ASSEMBLY Assegura que a anafase não começa até os pares de cromatídeos estarem corretamente alinhados e o fuso formado. Funciona através do controlo da atividade do complexo promotor de anafase (APC), responsável por iniciar a separação dos cromossomas na anafase. Detecta a existência de cinetocoros livres e inibe a atividade do APC. É um ciclo continuo de ubiquitinações e desubiquitinações que tende sempre para esta última, garantindo assim que o APC é mantido inativo até o fuso estar completamente formado. Fig. 8 – Checkpoint - Spindle-assembly (3)

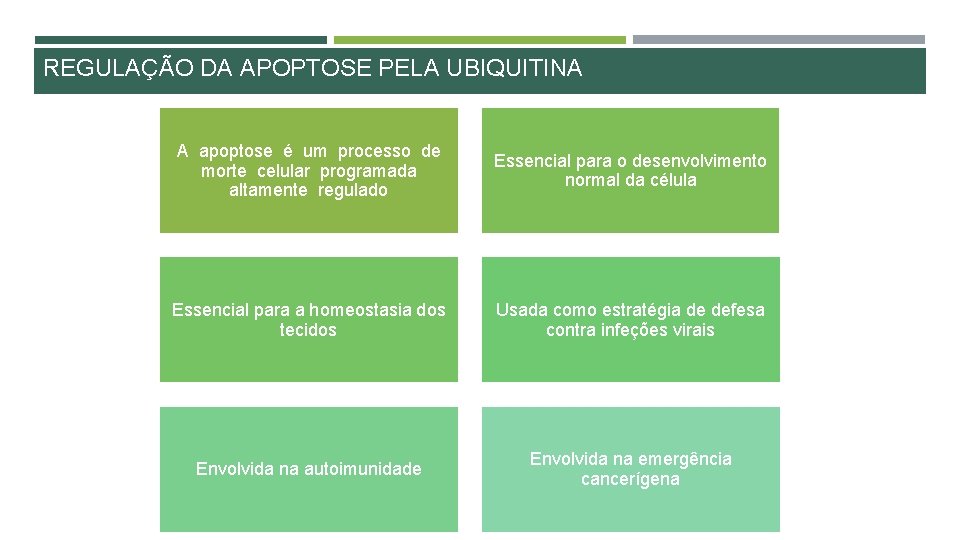
REGULAÇÃO DA APOPTOSE PELA UBIQUITINA A apoptose é um processo de morte celular programada altamente regulado Essencial para o desenvolvimento normal da célula Essencial para a homeostasia dos tecidos Usada como estratégia de defesa contra infeções virais Envolvida na autoimunidade Envolvida na emergência cancerígena

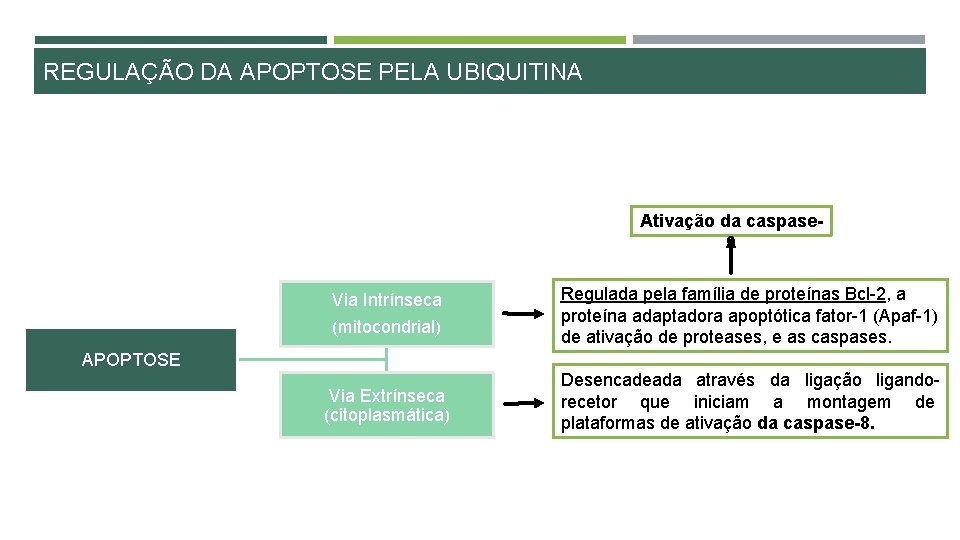
REGULAÇÃO DA APOPTOSE PELA UBIQUITINA Ativação da caspase 9 Via Intrínseca (mitocondrial) Regulada pela família de proteínas Bcl-2, a proteína adaptadora apoptótica fator-1 (Apaf-1) de ativação de proteases, e as caspases. APOPTOSE Via Extrínseca (citoplasmática) Desencadeada através da ligação ligandorecetor que iniciam a montagem de plataformas de ativação da caspase-8.

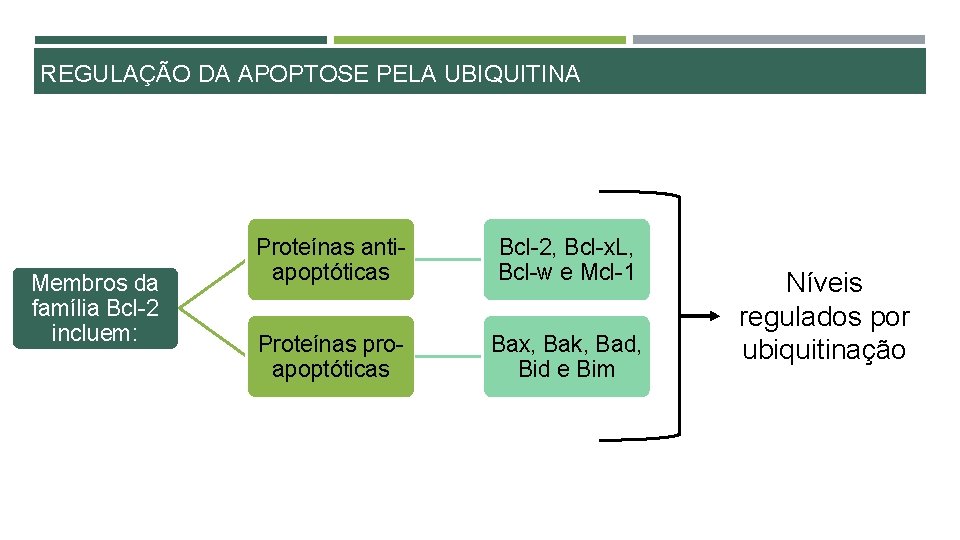
REGULAÇÃO DA APOPTOSE PELA UBIQUITINA Membros da família Bcl-2 incluem: Proteínas antiapoptóticas Bcl-2, Bcl-x. L, Bcl-w e Mcl-1 Proteínas proapoptóticas Bax, Bak, Bad, Bid e Bim Níveis regulados por ubiquitinação

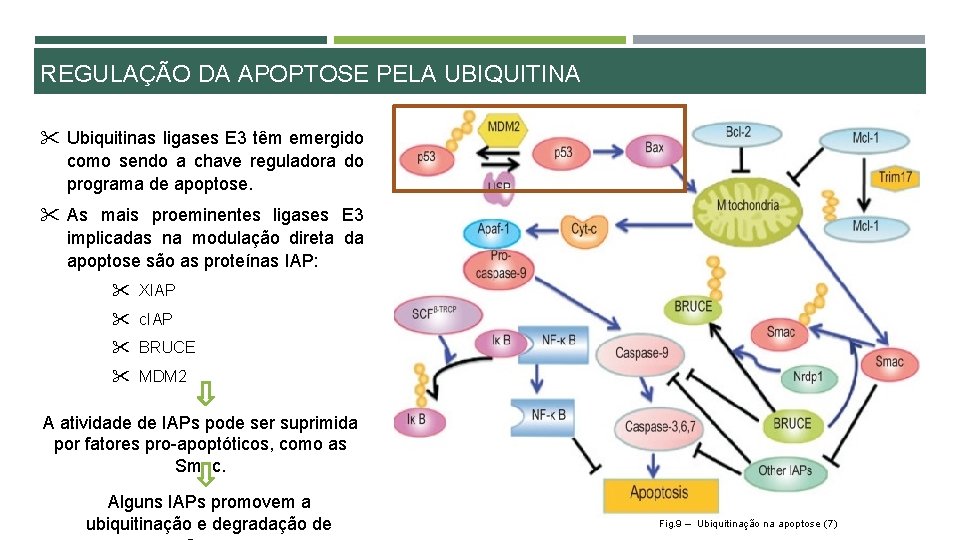
REGULAÇÃO DA APOPTOSE PELA UBIQUITINA " Ubiquitinas ligases E 3 têm emergido como sendo a chave reguladora do programa de apoptose. " As mais proeminentes ligases E 3 implicadas na modulação direta da apoptose são as proteínas IAP: " XIAP " c. IAP " BRUCE " MDM 2 A atividade de IAPs pode ser suprimida por fatores pro-apoptóticos, como as Smac. Alguns IAPs promovem a ubiquitinação e degradação de Fig. 9 – Ubiquitinação na apoptose (7)

UBIQUITINAÇÃO DE HISTONAS " As histonas podem ser modificadas através da ubiquitinação; " A histona H 2 A foi a primeira proteína a ser identificada ubiquitinada; " A maioria da u. H 2 A está na forma monoubiquitinada, a H 2 A poliubiquitinadas também tem sido detetada em muitos tecidos e tipos de células; " A histona H 2 B também é ubiquitinada; " Devido ao facto de a transcrição ocorrer no contexto de nucleossomas, o efeito da ubiquitinação de histonas na transcrição tem sido investigado. Fig. 10 – Estrutura do nucleossoma in http: //www. fomosplanejados. com. br/capitulos/assunto. asp? codcapitulo=22&codassunto=101&n

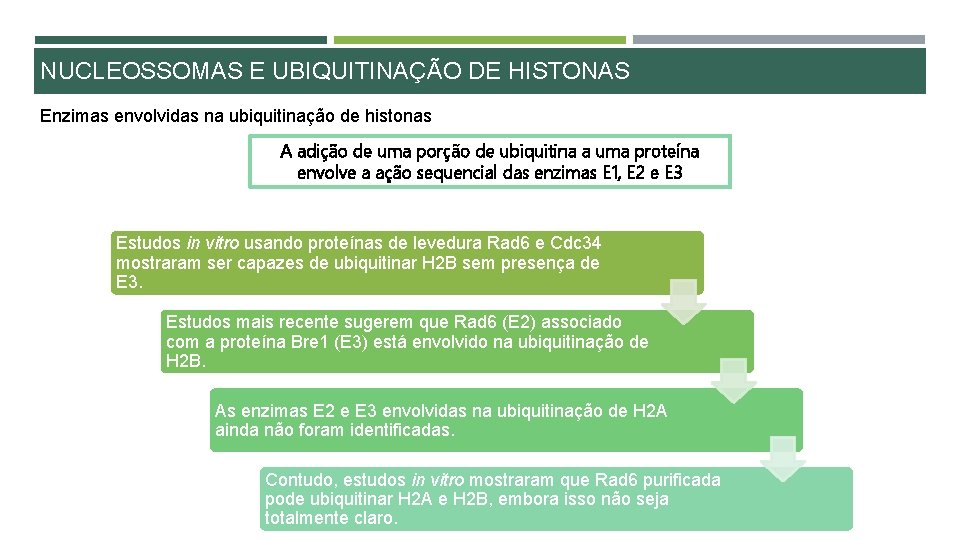
NUCLEOSSOMAS E UBIQUITINAÇÃO DE HISTONAS Enzimas envolvidas na ubiquitinação de histonas A adição de uma porção de ubiquitina a uma proteína envolve a ação sequencial das enzimas E 1, E 2 e E 3 Estudos in vitro usando proteínas de levedura Rad 6 e Cdc 34 mostraram ser capazes de ubiquitinar H 2 B sem presença de E 3. Estudos mais recente sugerem que Rad 6 (E 2) associado com a proteína Bre 1 (E 3) está envolvido na ubiquitinação de H 2 B. As enzimas E 2 e E 3 envolvidas na ubiquitinação de H 2 A ainda não foram identificadas. Contudo, estudos in vitro mostraram que Rad 6 purificada pode ubiquitinar H 2 A e H 2 B, embora isso não seja totalmente claro.

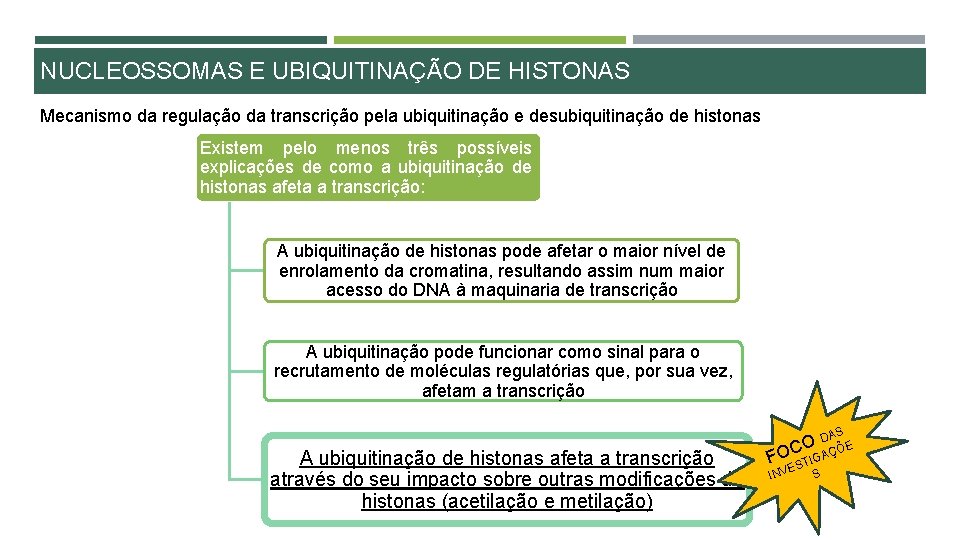
NUCLEOSSOMAS E UBIQUITINAÇÃO DE HISTONAS Mecanismo da regulação da transcrição pela ubiquitinação e desubiquitinação de histonas Existem pelo menos três possíveis explicações de como a ubiquitinação de histonas afeta a transcrição: A ubiquitinação de histonas pode afetar o maior nível de enrolamento da cromatina, resultando assim num maior acesso do DNA à maquinaria de transcrição A ubiquitinação pode funcionar como sinal para o recrutamento de moléculas regulatórias que, por sua vez, afetam a transcrição AS A ubiquitinação de histonas afeta a transcrição através do seu impacto sobre outras modificações de histonas (acetilação e metilação) F O DÇÕE C O GA I EST S V N I

CONCLUSÃO A ubiquitinação é um processo pós-traducional que desempenha importantes funções ao nível da sinalização celular, da regulação da transcrição, da regulação do ciclo celular, da reparação do DNA e do “controlo de qualidade” das proteínas, através da sua marcação para degradação no proteassoma. É um mediador ideal para o crosstalking entre diferentes vias. As E 3 ligases mostram especificidade para certos alvos, no entanto, a sua ação in vivo depende de outras proteínas que atuam como adaptadores ou ativadores da sua atividade. Deficiências neste processo podem levar ao desenvolvimento de cancro e a outras alterações fisiológicas.

REFERÊNCIAS 1. ABERLE, Hermann et al (1997) “β-catenin is a target for the ubiquitin-proteasome pathway”, The EMBO Journal 2. BERRIDJE, Michael J. (2012 a) Cell Signalling Biology: Module 1 – “Introduction”, Biochemical Journal, 1 3. BERRIDJE, Michael J. (2012 d) Cell Signalling Biology: Module 9 – “Cell Cycle and Proliferation” , Biochemical Journal, 1 4. BOECK, Miriam D. ; DIJKE, Peter T. (2012) “Key role for ubiquitin protein modification in TGF-β signal transduction”, Journal of Medical Sciences 5. BROEMER, Meike; MEIER, Pascal (2009) “Ubiquitin-mediated regulation of apoptosis”, Trends in Cell Biology, 6. CHEN, Jueqi; CHEN, Zhijian J. (2013) “Regulation of NF-k. B by Ubiquitination”, Curr Opin Immunol 7. CHEN, Yu-Shan; QIU, Xiao-Bo (2013) “Ubiquitin at the crossroad of death and survival”, Chinese Journal of Cancer 8. CHEN, Zhijian J. (2005) “Ubiquitin Signlaling in the NF-k. B Pathway”, Nat Cell Biol, 9. HAMID, Sayed M. , et al (2014) “HOXB 13 contributes to G 1/S and G 2/M checkpoint controls in prostate”, Molecular and Cellular Endocrinology 10. HATAKEYAMA, Shigetsugu (2012) “Ubiquitin-mediated regulation of JAK-STAT signaling in embryonic stem cells”, Landes Bioscience 11. LI, Vivian et al (2012) “Wnt Signalling through Inhibition of β-Catenin Degradation in na Intact Axin 1 Complex” 12. NAPETSCHNIG, Johanna; WU, Hao (2013) “Molecular Basis of NF-k. B Signaling”, Annu Ver Biophys, 13. SCHNERCH, Dominik et al (2012) “Cell cycle control in acute myeloid leukemia”, American Journal of Cancer Research 14. SOOND, Surinder M. ; CHANTRY, Andrew (2011) “How ubiquitination regulates the TGF- β signalling pathway: New insights and new players”, Prospects & Overviews 15. TAURIELLO, Daniele; MAURICE, Madelon (2010) “The various roles of ubiquitin in Wnt pathway regulation”, Landes Bioscience 16. WE, Juncheng et al (2012) “The Ubiquitin Ligase TRAF 6 Negatively Regulates the JAK-STAT signaling Pathway by Binding to STAT 3 and Mediating its Ubiquitination”, 17. WU, Zhao-Hui; SHI, Yuling (2013) “When Ubiquitin Meets NF-k. B: A trove for anti-cancer drug development”, Curr Pharm Des, 18. ZHANG, Yi (2003) “Transcriptional regulation by histone ubiquitination and deubiquitination”, Genes & Development 19. ZHANG, Ying, et al (2001) “Regulation of Smad degradation and activity by Smurf 2, an E 3 ubiquitin ligase”

OBRIGADA PELA ATENÇÃO! Proteassoma Proteína Péptidos Docente: Prof. Paulo de Oliveira Discentes: • Ana Filipa Robalo 30703 • Ana Rita Hipólito 30237
 Bhavya vora
Bhavya vora Subodh vora & co
Subodh vora & co A la vora del mar poema
A la vora del mar poema Teodor llorente vora el barranc dels algadins
Teodor llorente vora el barranc dels algadins Subodh vora & co
Subodh vora & co Poorvi vora
Poorvi vora Vora el barranc dels algadins comentari
Vora el barranc dels algadins comentari Phinix
Phinix Subodh vora & co
Subodh vora & co Vora la mar jacint verdaguer
Vora la mar jacint verdaguer Vias reflejas digestivas
Vias reflejas digestivas Factor 3 plaquetario
Factor 3 plaquetario 5 vias de santo tomas de aquino
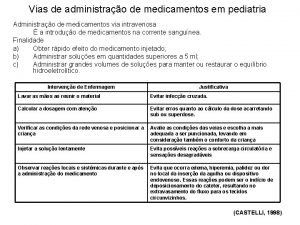
5 vias de santo tomas de aquino Vias de administração de medicamentos
Vias de administração de medicamentos Antipsicóticos atipicos
Antipsicóticos atipicos Vias metabolicas
Vias metabolicas Cranberry en polvo
Cranberry en polvo Ventilacion alveolar
Ventilacion alveolar Venas para la venoclisis
Venas para la venoclisis Reeducación vesical con sonda foley
Reeducación vesical con sonda foley Tracto espinotalamico anterior
Tracto espinotalamico anterior Las 5 vias de santo tomas
Las 5 vias de santo tomas Vias aferentes y eferentes
Vias aferentes y eferentes Vias de transmision
Vias de transmision Pombo torcaz vias extinção
Pombo torcaz vias extinção Hemotsis
Hemotsis Vias respiratorias
Vias respiratorias Vias de ativação do sistema complemento
Vias de ativação do sistema complemento Partes de la cara nariz
Partes de la cara nariz Glucogenolisis y gluconeogenesis
Glucogenolisis y gluconeogenesis Tunica muscular
Tunica muscular Vias
Vias