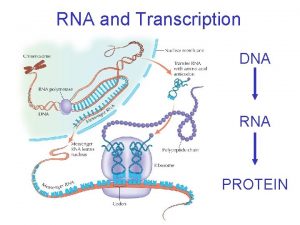
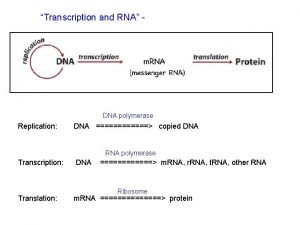
Transcription termination RNA polymerase I terminates transcription at











































- Slides: 51

Transcription termination • RNA polymerase I terminates transcription at an 18 base terminator sequence. • RNA polymerase III terminates transcription in poly(U)4 sequence embedded in a G·C-rich sequence. • In pol II transcripts the sequence AAUAAA is a signal for cleavage to generate a 3 end of m. RNA that is polyadenylated.




RNA polymerasi II termination • The reaction requires a protein complex that contains a specificity factor, an endonuclease, and poly(A) polymerase. • The specificity factor and endonuclease cleave RNA downstream of AAUAAA. • The specificity factor and poly(A) polymerase add ~200 A residues processively to the 3 end.








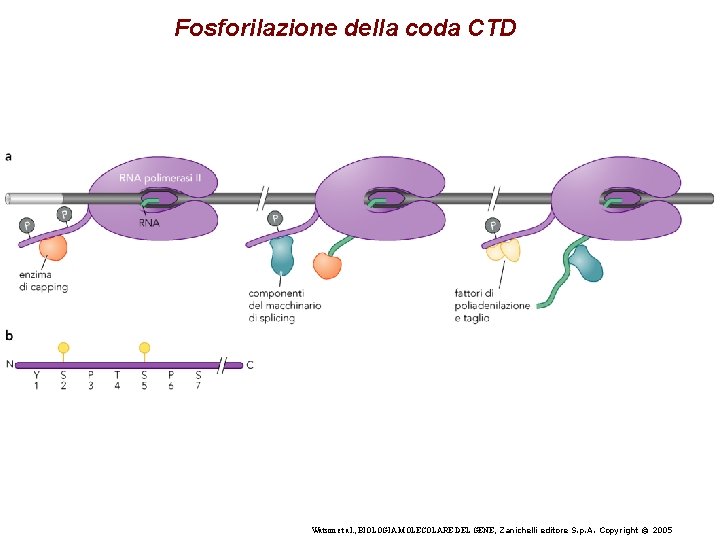
Fosforilazione della coda CTD Watson et al. , BIOLOGIA MOLECOLARE DEL GENE, Zanichelli editore S. p. A. Copyright © 2005

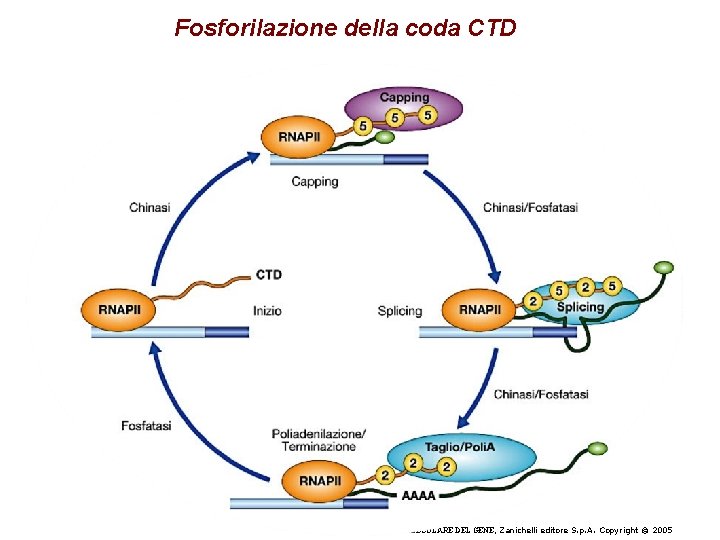
Fosforilazione della coda CTD Watson et al. , BIOLOGIA MOLECOLARE DEL GENE, Zanichelli editore S. p. A. Copyright © 2005

“Fabbrica dell’RNA” (RNA factory) • Interdipendenza trascrizione e modificazioni post-trascrizionali • Pol II senza CTD inibisce splicing, terminazione e poliadenilazione • CPSF e Cst. F legano il CTD • CPSF si trova associato a TFIID

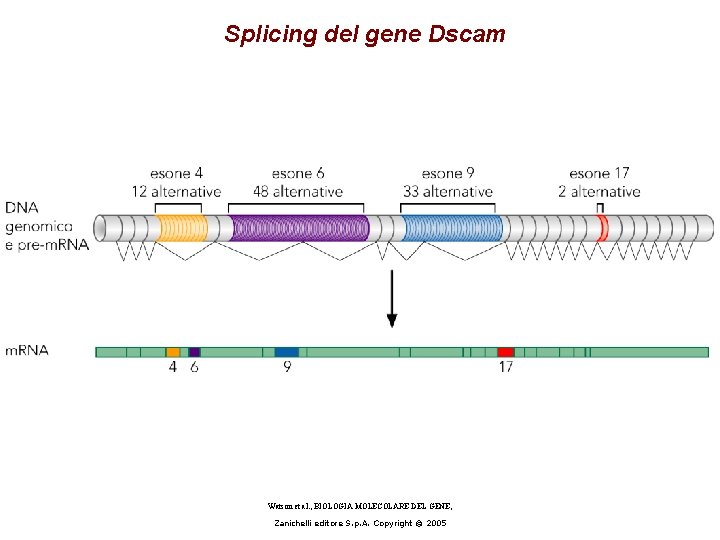
Splicing del gene Dscam Watson et al. , BIOLOGIA MOLECOLARE DEL GENE, Zanichelli editore S. p. A. Copyright © 2005

Complessità genoma-proteoma • Percentuale di geni con splicing alternativo varia tra 35 e 59 • Il gene Dscam di Drosofila (recettore per la guida degli assoni) contiene 95 possibili esoni che fanno splicing alternativo per un totale di 38000 isoforme proteiche possibili • Il 15% delle mutazioni che causano malattie genetiche provocano difetti di splicing


Watson et al. , BIOLOGIA MOLECOLARE DEL GENE, Zanichelli editore S. p. A. Copyright © 2005

Watson et al. , BIOLOGIA MOLECOLARE DEL GENE, Zanichelli editore S. p. A. Copyright © 2005

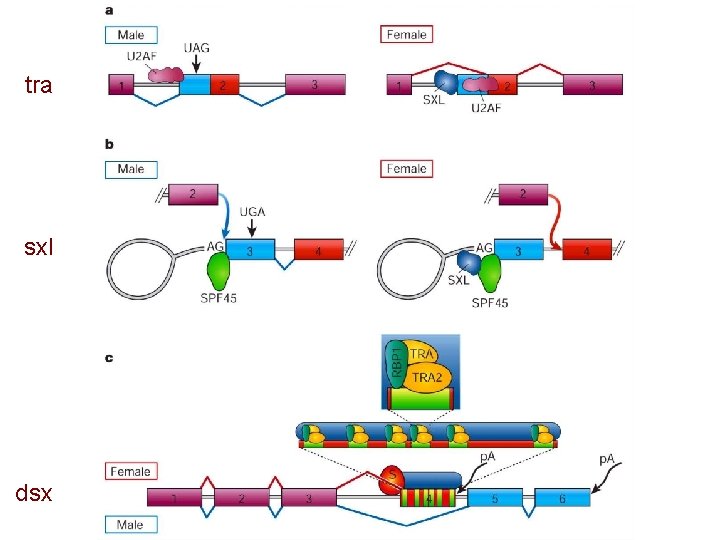
tra sxl dsx

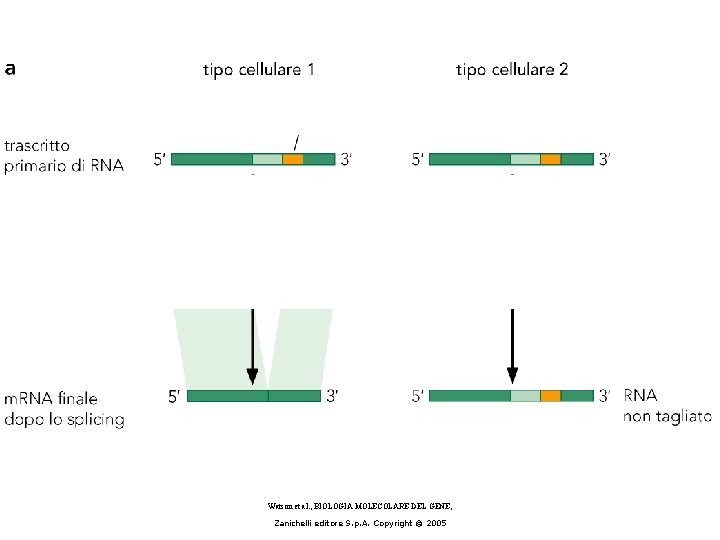
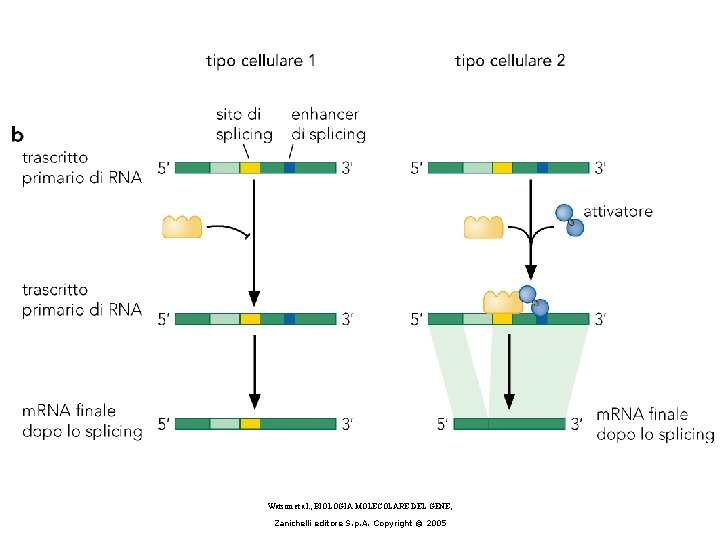
Alternative splicing • Specific exons may be excluded or included in the RNA product by using or failing to use a pair of splicing junctions. • Exons may be extended by changing one of the splice junctions to use an alternative junction. • Alternative splicing may depend on SR proteins or specific factors


Ruolo della poliadenilazione alternativa • stabilità dell'm. RNA • localizzazione dell'm. RNA • espressione di proteine diverse • regolazione della traduzione


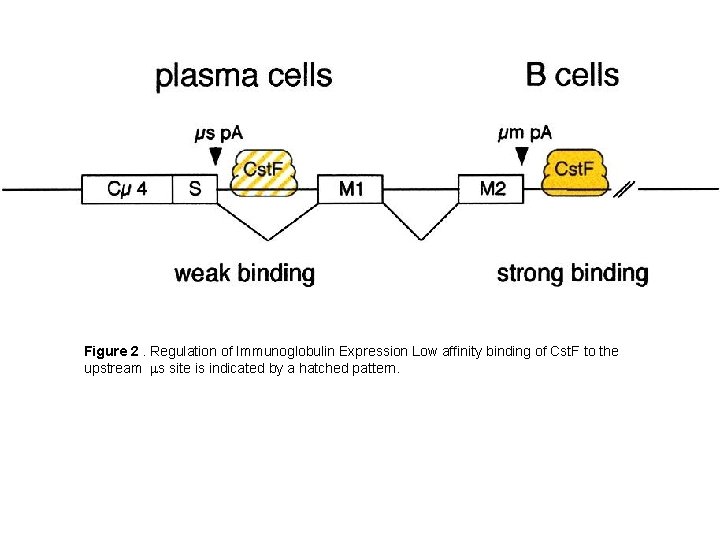
Figure 2. Regulation of Immunoglobulin Expression Low affinity binding of Cst. F to the upstream ms site is indicated by a hatched pattern.



Scelta del sito di poliadenilazione • Sequenze cis-agenti che definiscono il sito di taglio • Concentrazione o attività dei fattori costitutivi di poliadenilazione • Espressione di fattori specifici di poliadenilazione e fattori di splicing






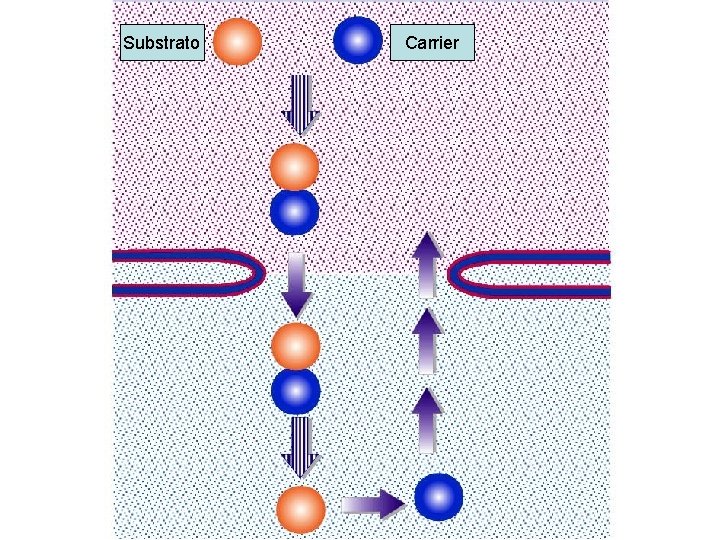
Poro nucleare • Il complesso del poro nucleare (NPC) è grande 125 MDa (66 MDa in lievito) • 2000 NPC nei vertebrati (200 in lievito) • NPC è composto da circa 1000 proteine di 30 -50 tipi diversi (di ciascuna almeno 8 copie) • Molecole fino a 9 nm (30 -40 k. Da) diffondono liberamente attraverso l’NPC • Molecole fino a 25 nm vengono attivamente trasportate attraverso l’NPC


Substrato Carrier














Transport through the nuclear pores • The NLS and NES consist of short sequences that are necessary and sufficient for proteins to be transported through the nuclear pores. • Transport receptors have the dual properties of recognizing NLS or NES sequences and binding to the nuclear pore. • The direction of transport is controlled by the state of the monomeric G protein, Ran. • The nucleus contains Ran-GTP, which stabilizes export complexes, while the cytosol contains Ran-GDP, which stabilizes import complexes. • The mechanism of movement does not involve a motor.
 Rna polymerase 1 2 3
Rna polymerase 1 2 3 Dna transcription
Dna transcription Rna polymerase
Rna polymerase Termination of transcription in prokaryotes
Termination of transcription in prokaryotes Rna transcription
Rna transcription Dna to rna transcription
Dna to rna transcription Ribosomem
Ribosomem Codon wheel
Codon wheel Adn polymérase
Adn polymérase Polymerase chain reaction
Polymerase chain reaction Transcription
Transcription The three steps of polymerase chain reaction
The three steps of polymerase chain reaction Site:slidetodoc.com
Site:slidetodoc.com Dna prokaryotic
Dna prokaryotic Taq dna polymerase
Taq dna polymerase Dna polymerase
Dna polymerase Types of dna polymerase in eukaryotes
Types of dna polymerase in eukaryotes What are the enzymes involved in dna replication
What are the enzymes involved in dna replication Pcr phases
Pcr phases Function of dna polymerase 3
Function of dna polymerase 3 Dna polymerase
Dna polymerase Real time pcr primer efficiency test
Real time pcr primer efficiency test Polymerase chain reaction (pcr) application
Polymerase chain reaction (pcr) application Template strand, new strand, base pair, and dna polymerase.
Template strand, new strand, base pair, and dna polymerase. Clonaggio
Clonaggio Dna polymerase proofreading
Dna polymerase proofreading Dna polymerase
Dna polymerase Polymerase chain reaction
Polymerase chain reaction Polymerase chain reaction
Polymerase chain reaction Dna primase
Dna primase Project termination
Project termination Emergency action termination
Emergency action termination Leg venous drainage
Leg venous drainage Unplanned termination of members from a group
Unplanned termination of members from a group Basic i/o supervisor
Basic i/o supervisor Termination device
Termination device Termination area mutcd
Termination area mutcd Termination of mudarabah
Termination of mudarabah Microdistribution termination video
Microdistribution termination video Dr seema haque
Dr seema haque Termination transtheoretical model
Termination transtheoretical model Project audit life cycle
Project audit life cycle Functional architecture diagram
Functional architecture diagram Termination of mudarabah
Termination of mudarabah Palmar arch artery
Palmar arch artery Ending stage of group work
Ending stage of group work Treaties in international law
Treaties in international law Relationship escalation model
Relationship escalation model Musharakah vs mudharabah
Musharakah vs mudharabah Features of musharakah
Features of musharakah Dorsalis pedis artery pulse location
Dorsalis pedis artery pulse location Chain termination method
Chain termination method