Traduo Sntese de Protenas Cdigo Gentico Dicionrio correspondncia


















- Slides: 37

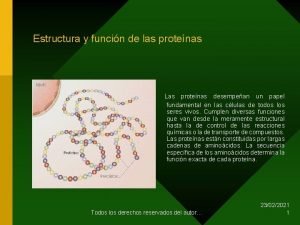
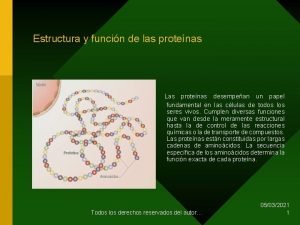
Tradução / Síntese de Proteínas


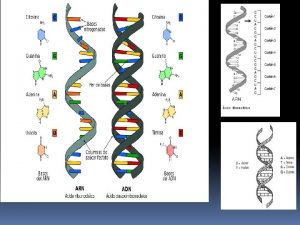
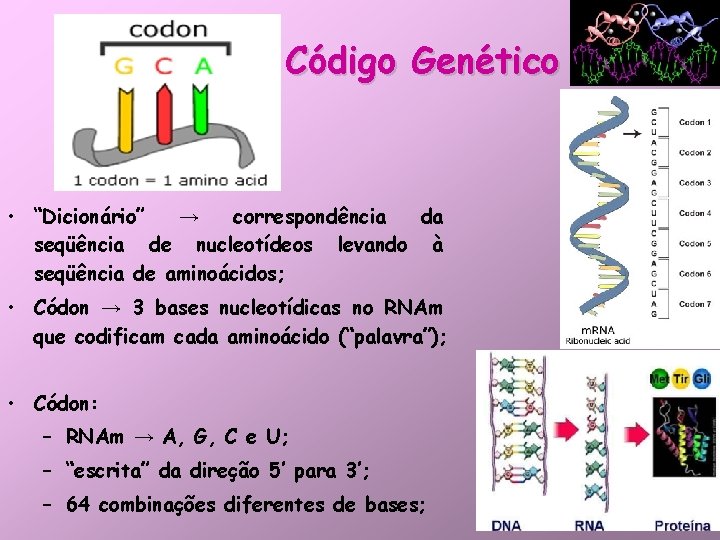
Código Genético • “Dicionário” → correspondência da seqüência de nucleotídeos levando à seqüência de aminoácidos; • Códon → 3 bases nucleotídicas no RNAm que codificam cada aminoácido (“palavra”); • Códon: – RNAm → A, G, C e U; – “escrita” da direção 5’ para 3’; – 64 combinações diferentes de bases;

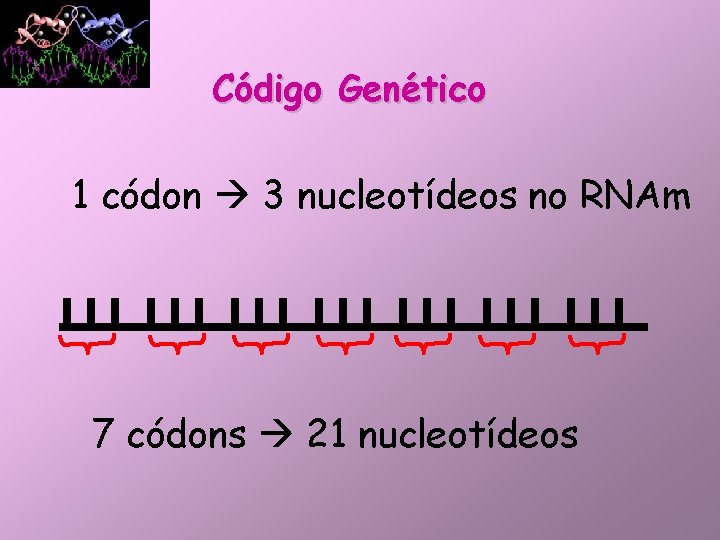
Código Genético 1 códon 3 nucleotídeos no RNAm 7 códons 21 nucleotídeos

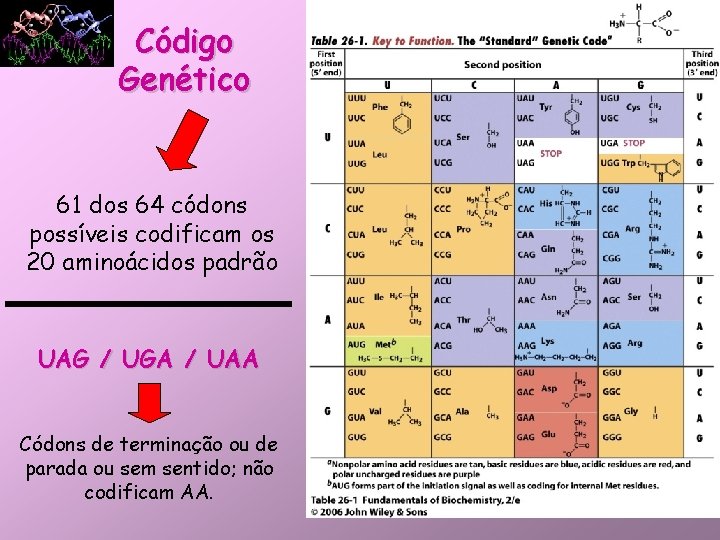
Código Genético 61 dos 64 códons possíveis codificam os 20 aminoácidos padrão UAG / UGA / UAA Códons de terminação ou de parada ou sem sentido; não codificam AA.

Código Genético • Características: – Especificidade – um determinado códon sempre codifica o mesmo AA; – Universalidade – é conservado em todas as espécies; – Redundância ou Degeneração – um AA pode ter mais de 1 trinca que o codifica; – Contínuo – sempre lido de 3 em 3 bases. Degeneração do código

Mutações no Código Genético • Mutação silenciosa: – Códon com 1 base alterada ainda codifica o mesmo AA; • Mutação com perda de sentido: – Códon com 1 base alterada codifica um AA diferente; • Mutação sem sentido: – Códon com 1 base alterada se torna um dos códons de terminação;

Outras Mutações no Código Genético • Expansão de repetições trinucleotídicas: – Inserções de várias repetições de 1 códon. Ex: doença de Huntington; • Mutações em sítios de corte-junção: – Alteração de íntrons removidos; • Mutações com alteração de módulo de leitura: – 1 ou 2 nucleotídeos perdidos ou adicionados → seqüência de AAs altera radicalmente.

Componentes da Tradução • AAs: – Dieta → AAs essenciais; • RNAt ou moléculas adaptadoras: – Em humanos existem em torno de 50 espécies de RNAt, enquanto bactérias possuem em torno de 30 -40 espécies; – Sítios de ligação ao AA – extremidade 3’ do RNAt se liga ao grupo carboxila do AA; – Anticódon → seqüência de 3 nucleotídeos que reconhece o códon específico do RNAm; – Pode estar carregado ou descarregado.

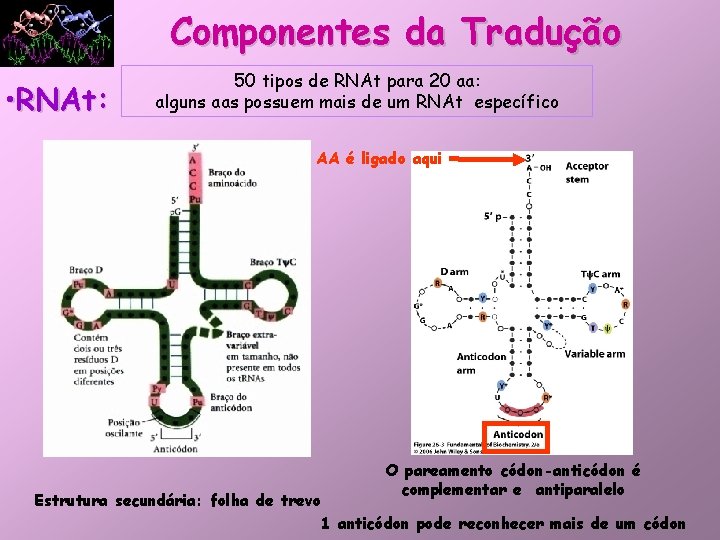
Componentes da Tradução • RNAt: 50 tipos de RNAt para 20 aa: alguns aas possuem mais de um RNAt específico AA é ligado aqui O pareamento códon-anticódon é complementar e antiparalelo Estrutura secundária: folha de trevo 1 anticódon pode reconhecer mais de um códon

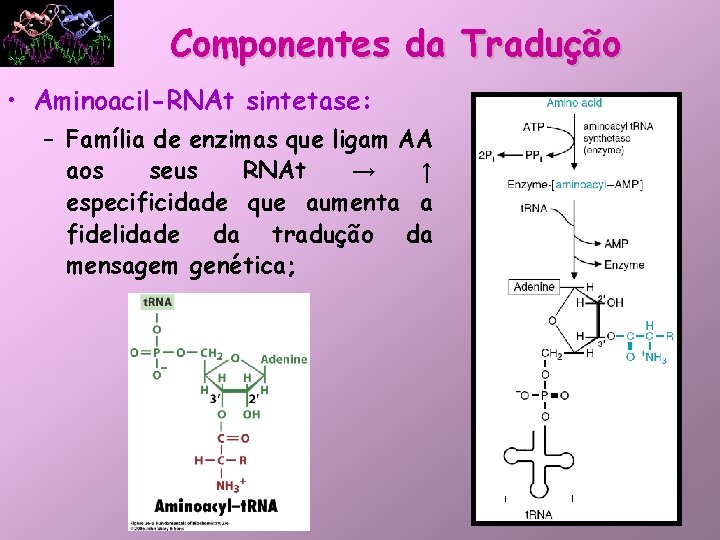
Componentes da Tradução • Aminoacil-RNAt sintetase: – Família de enzimas que ligam AA aos seus RNAt → ↑ especificidade que aumenta a fidelidade da tradução da mensagem genética;

Componentes da Tradução • RNAm (molde); • Ribossomos: grandes complexos de RNAr proteínas compostos por duas subunidades. e São as estruturas responsáveis pela síntese protéica (local da síntese). Livres ou no RER. Ribossomo de eucariotos: subunidades 60 S (5 S, 5. 8 S e 28 S/49 proteínas) + 40 S (18 S/33 proteínas) Em procariotos: subunidades 50 S (5 S e 23 S/36 proteínas) + 30 S (16 S/21 proteínas) RNAr: responsáveis pela estabilização do complexo de iniciação e dos demais participantes da tradução

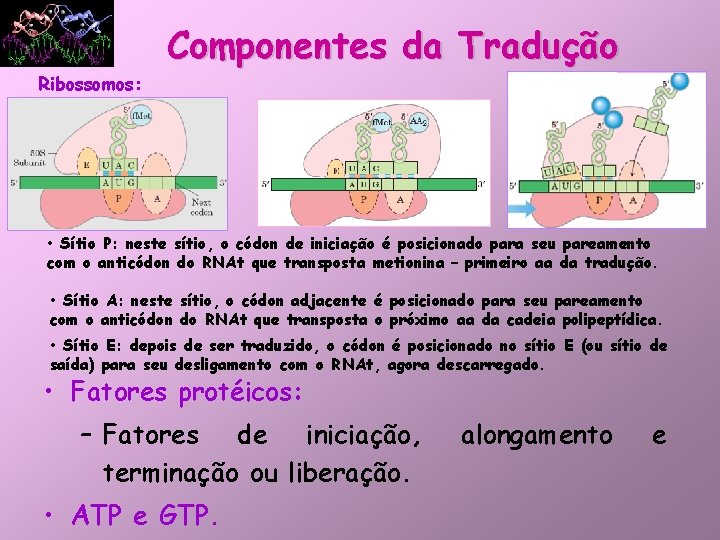
Componentes da Tradução Ribossomos: • Sítio P: neste sítio, o códon de iniciação é posicionado para seu pareamento com o anticódon do RNAt que transposta metionina – primeiro aa da tradução. • Sítio A: neste sítio, o códon adjacente é posicionado para seu pareamento com o anticódon do RNAt que transposta o próximo aa da cadeia polipeptídica. • Sítio E: depois de ser traduzido, o códon é posicionado no sítio E (ou sítio de saída) para seu desligamento com o RNAt, agora descarregado. • Fatores protéicos: – Fatores de iniciação, terminação ou liberação. • ATP e GTP. alongamento e

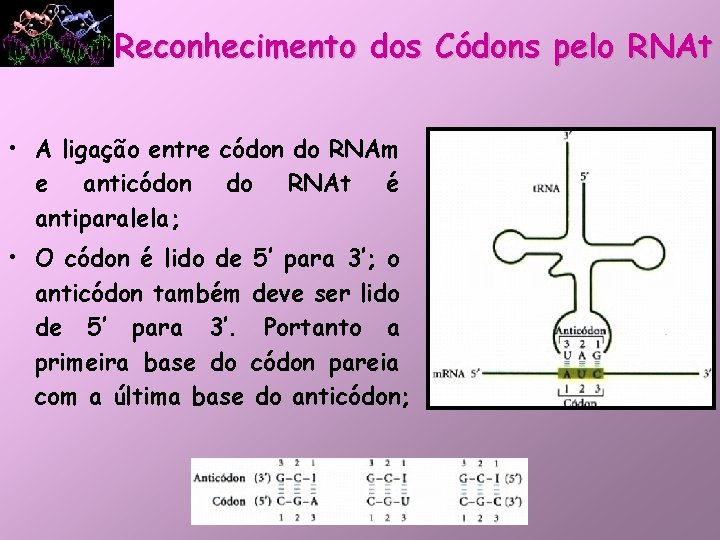
Reconhecimento dos Códons pelo RNAt • A ligação entre códon do RNAm e anticódon do RNAt é antiparalela; • O códon é lido de 5’ para 3’; o anticódon também deve ser lido de 5’ para 3’. Portanto a primeira base do códon pareia com a última base do anticódon;

Reconhecimento dos Códons pelo RNAt • Hipótese da Oscilação: – Se a trinca do anticódon reconhecesse apenas 1 trinca do códon por pareamento, as células deveriam ter 1 RNAt para cada códon de AA → NÃO É O QUE OCORRE!; – As 2 primeiras bases do códon formam pares de bases Watson-Crick com fortes pontes de hidrogênio → dão especificidade da codificação; – A terceira base do códon que pareia com a primeira base do anticódon forma pontes de hidrogênio mais fracas e a primeira base do anticódon pode parear com mais de 1 base.

Hipótese da Oscilação 1 anticódon pareia com mais de 1 códon! Lig. + específica Lig. - específica OBS: I (inosina) contém base hipoxantina pode ser encontrado como a primeira base do anticódon → base oscilante que pareia com mais de 1 base As interações códon-anticódon otimizam tanto a exatidão quanto a velocidade de síntese protéica

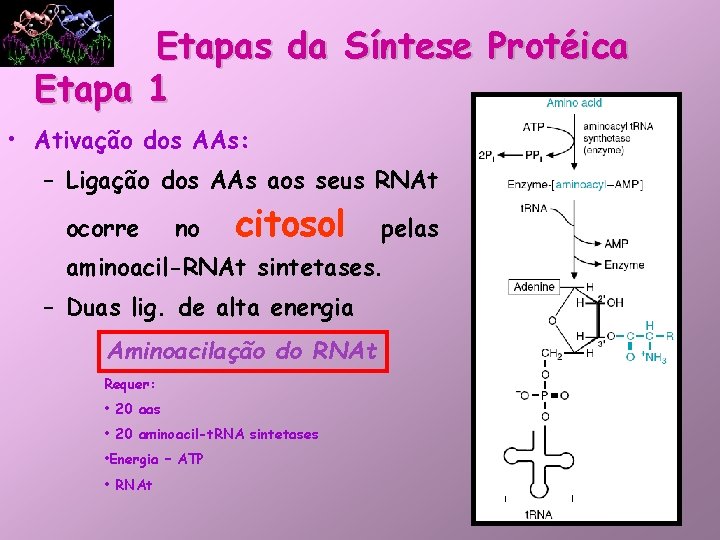
Etapas da Síntese Protéica Etapa 1 • Ativação dos AAs: – Ligação dos AAs aos seus RNAt ocorre no citosol pelas aminoacil-RNAt sintetases. – Duas lig. de alta energia Aminoacilação do RNAt Requer: • 20 aas • 20 aminoacil-t. RNA sintetases • Energia – ATP • RNAt

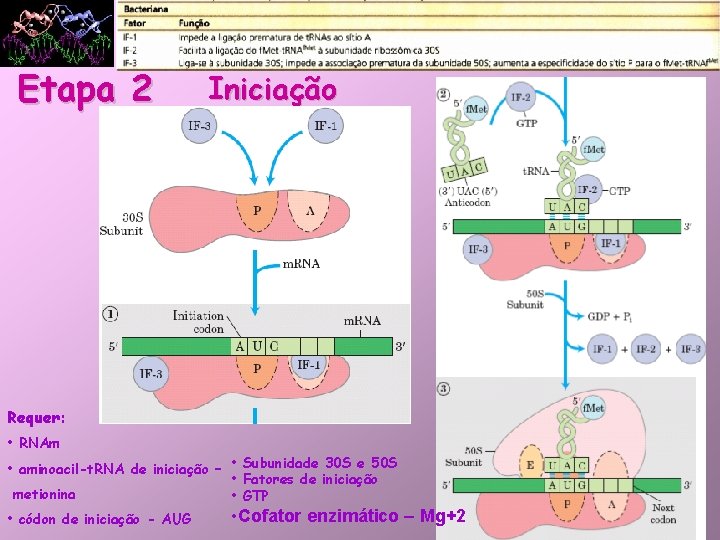
Etapas da Síntese Protéica Etapa 2 • Iniciação: – O RNAm liga-se a menor das 2 subunidades ribossômicas e ao aminoacil-RNAt de iniciação; – Na E. coli, a seqüência reconhecida no RNAm pelo ribossomo é chamada de seqüência de Shine -Dalgarno (nos eucariotos o “quepe” do RNAm é reconhecido pelo ribossomo) → 6 a 10 bases longe do códon de iniciação AUG;

Etapas da Síntese Protéica Etapa 2 • Iniciação: – O aminoacil-RNAt de iniciação pareia com o códon AUG, que é o códon que sinaliza o início da proteína a ser sintetizada; – Em bactérias e na mitocôndria, esse RNAt de iniciação carrega uma metionina N-formilada (grupo formila é adicionado pela enzima transformilase). Nos eucariotos, a metionina não está formilada;

Etapa 2 Iniciação Requer: • RNAm • aminoacil-t. RNA de iniciação – • Subunidade 30 S e 50 S • Fatores de iniciação metionina • GTP • códon de iniciação - AUG • Cofator enzimático – Mg+2

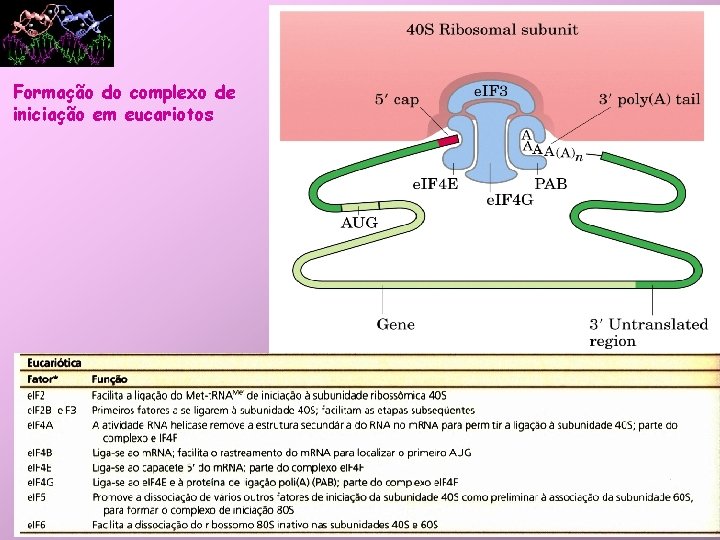
Formação do complexo de iniciação em eucariotos

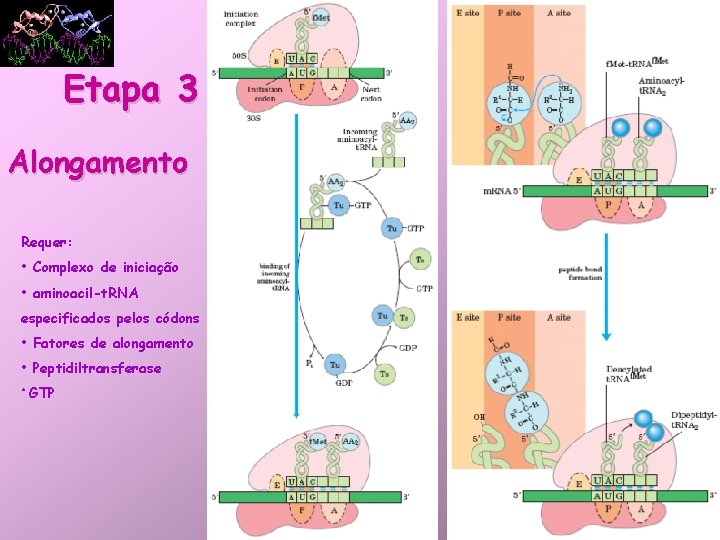
Etapas da Síntese Protéica Etapa 3 • Alongamento: – Fatores de alongamento são necessários (EF-Tu, EF-Ts, EF-G); – Peptidiltransferase (ribozima) → liga o peptídeo em formação e o AA a ser adicionado; – Após a ligação peptídica se formar, o ribossomo avança 3 nucleotídeos na direção 3’→Translocação (requer energia, GTP). – O RNAt não-carregado vai para o sítio E antes de ser liberado e o RNAt carregando o peptídeo vai para o sítio P.

Etapa 3 Alongamento Requer: • Complexo de iniciação • aminoacil-t. RNA especificados pelos códons • Fatores de alongamento • Peptidiltransferase • GTP

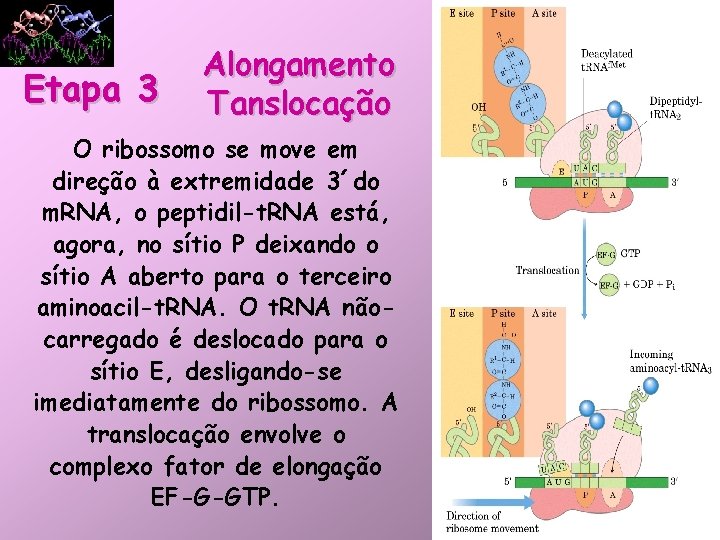
Etapa 3 Alongamento Tanslocação O ribossomo se move em direção à extremidade 3´do m. RNA, o peptidil-t. RNA está, agora, no sítio P deixando o sítio A aberto para o terceiro aminoacil-t. RNA. O t. RNA nãocarregado é deslocado para o sítio E, desligando-se imediatamente do ribossomo. A translocação envolve o complexo fator de elongação EF-G-GTP.

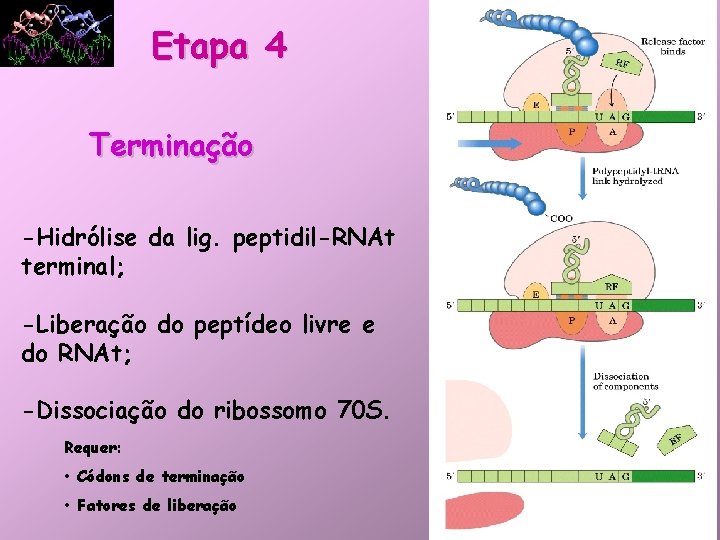
Etapa 4 • Terminação: – Ocorre quando 1 dos 3 códons (UAA, UAG, UGA) de terminação é “colocado” no sítio A; – Na E. coli, os fatores de terminação ou liberação reconhecem esses códons e ocorre a liberação do complexo ribossomal.

Etapa 4 Terminação -Hidrólise da lig. peptidil-RNAt terminal; -Liberação do peptídeo livre e do RNAt; -Dissociação do ribossomo 70 S. Requer: • Códons de terminação • Fatores de liberação

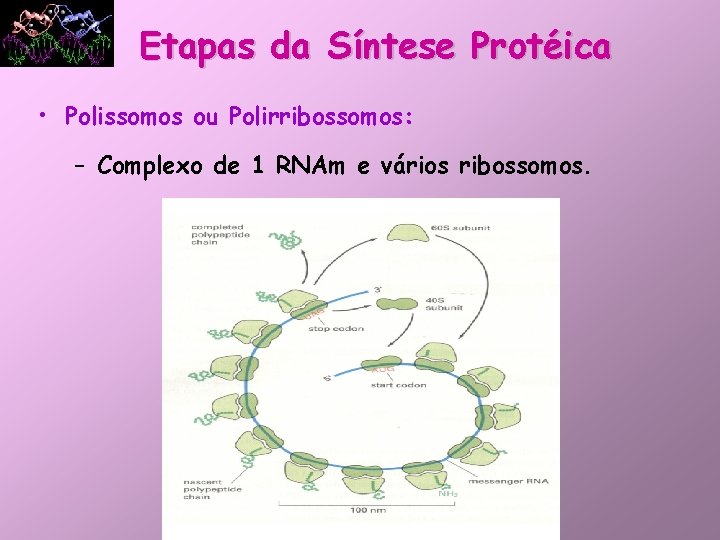
Etapas da Síntese Protéica • Polissomos ou Polirribossomos: – Complexo de 1 RNAm e vários ribossomos.

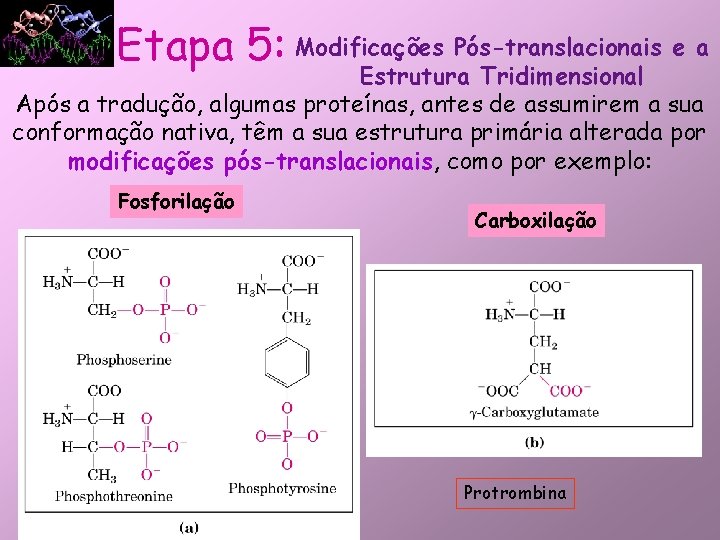
Etapa 5: Modificações Pós-translacionais e a Estrutura Tridimensional Após a tradução, algumas proteínas, antes de assumirem a sua conformação nativa, têm a sua estrutura primária alterada por modificações pós-translacionais, como por exemplo: Fosforilação Carboxilação Protrombina

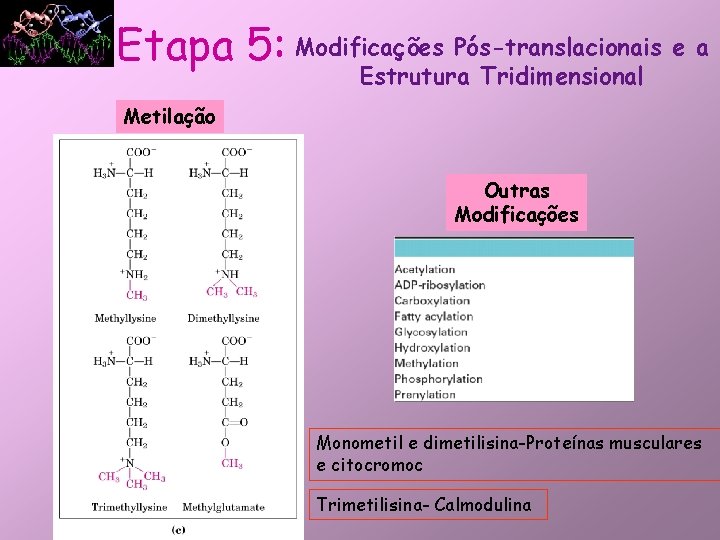
Etapa 5: Modificações Pós-translacionais e a Estrutura Tridimensional Metilação Outras Modificações Monometil e dimetilisina-Proteínas musculares e citocromoc Trimetilisina- Calmodulina

Etapa 5: Modificações Pós-translacionais e a Estrutura Tridimensional As proteínas assumem a sua conformação nativa com o auxílio das chaperonas ou proteínas do estresse ou proteínas do choque térmico (heat shock proteins). Ajudam as proteínas a se moldar, associar a outras proteínas de maneira estável e tornarem-se estruturas ativas, evitando a associação de proteínas ainda não dobradas corretamente Conformação Desnaturada Conformação Nativa

Diferenças entre procariotos e eucariotos EUCARIOTOS Ligação do m. RNA à subunidade menor ribossomal Primeiro Aminoácido O 5´-CAP do m. RNA liga-se aos fatores de iniciação e à subunidade 40 S. O m. RNA é lido a partir do códon de iniciação Metionina (não formilada) Fatores de iniciação e. IFs (8 ou mais) Fatores de terminação e. RF Fatores de alongamento EF 1 a (EF-Tu) EFbg (EF-Ts) EF 2 (EF-G) Ribossomo · 80 S (40 S + 60 S) · ausência de sítio E (exit · tradução não simultânea com transcrição

Diferenças e semelhanças entre eucariotos e procariotos O esquema geral é o mesmo, e a síntese em si ocorre em estruturas similares: os ribossomos Mas, em eucariotos a transcrição está separada da síntese de proteínas (tradução) pela membrana nuclear

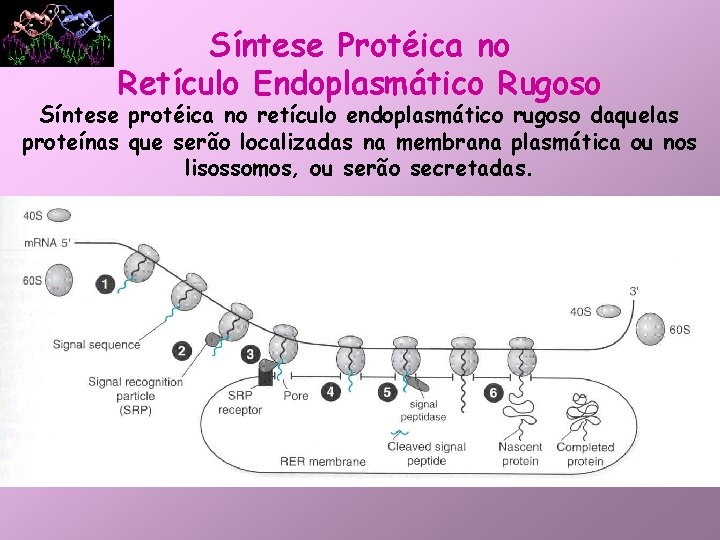
Síntese Protéica no Retículo Endoplasmático Rugoso Síntese protéica no retículo endoplasmático rugoso daquelas proteínas que serão localizadas na membrana plasmática ou nos lisossomos, ou serão secretadas.

Síntese proteíca no reticulo endoplasmático rugoso daquelas proteínas que serão localizadas na membrana plasmática ou nos lisossomos, ou serão secretadas Síntese Protéica no RER

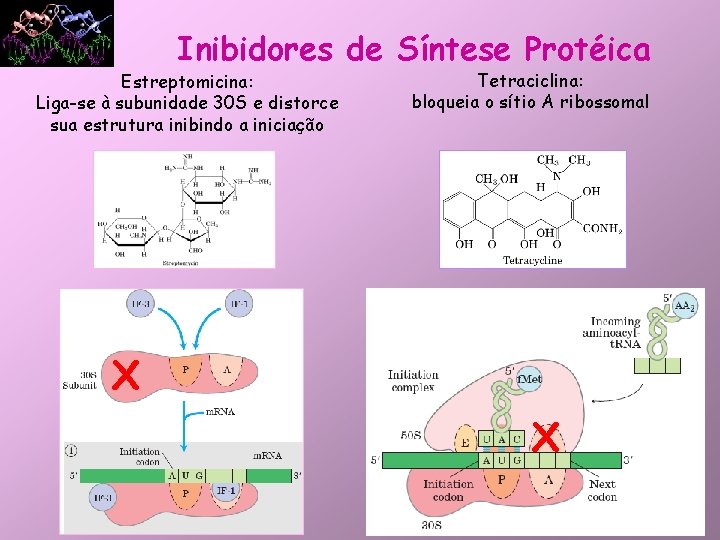
Inibidores de Síntese Protéica Estreptomicina: Liga-se à subunidade 30 S e distorce sua estrutura inibindo a iniciação Tetraciclina: bloqueia o sítio A ribossomal X X

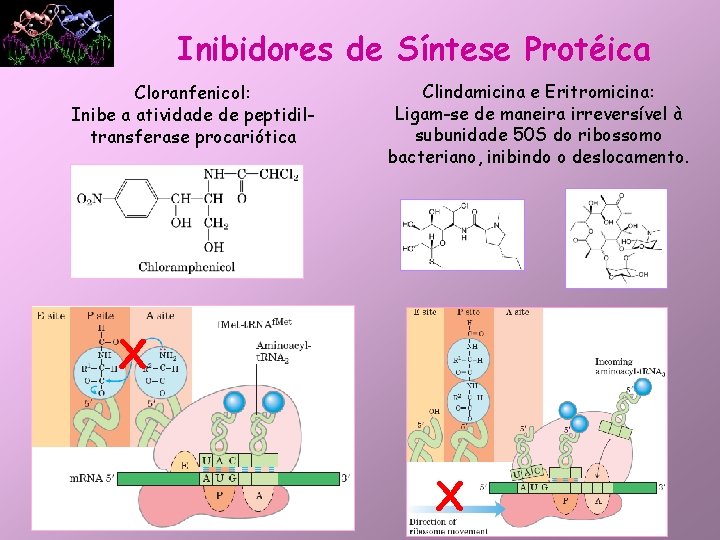
Inibidores de Síntese Protéica Cloranfenicol: Inibe a atividade de peptidiltransferase procariótica Clindamicina e Eritromicina: Ligam-se de maneira irreversível à subunidade 50 S do ribossomo bacteriano, inibindo o deslocamento. X X

Bibliografia: Voet, D. , Voet, J. G. , Pratt, C. W. Fundamentos de Bioquímica (2000). Champe, P. C. ; Harvey, R. A. ; Ferrier, D. R. Bioquímica Ilustrada (3 a ed, 2006). Nelson, D. L. , Cox, M. M. Lehninger, Princípios de Bioquímica. Quarta edição (2004).
 Tradutor
Tradutor Codigo gentico
Codigo gentico Diagrama de fluxo de dados
Diagrama de fluxo de dados Exemplos de truncação
Exemplos de truncação Humor
Humor Sntese
Sntese Neurnio
Neurnio Sntese
Sntese Cdigo ff
Cdigo ff Acronym of ascii
Acronym of ascii Ascii
Ascii Fibosoma
Fibosoma Cdigo ascii
Cdigo ascii Cdigo ascii
Cdigo ascii Cdigo de comercio
Cdigo de comercio Cdigo de colores
Cdigo de colores Qr
Qr American standard code for information interchange
American standard code for information interchange Cdigo ascii
Cdigo ascii Grupo prostetico de la hemoglobina
Grupo prostetico de la hemoglobina La unión peptídica
La unión peptídica Ejemplo de cromoproteinas
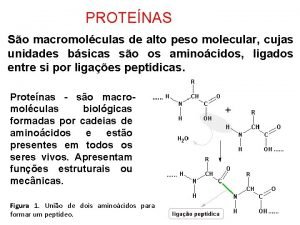
Ejemplo de cromoproteinas Protenas
Protenas Protenas
Protenas Protenas
Protenas Protenas
Protenas Proteinas monomericas
Proteinas monomericas Protenas
Protenas Protenas
Protenas Funciones de la proteina
Funciones de la proteina Proteinas como estan formadas
Proteinas como estan formadas