Reparacin del DNA Dao al DNA puede ocasionar
















- Slides: 55

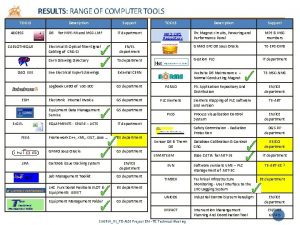
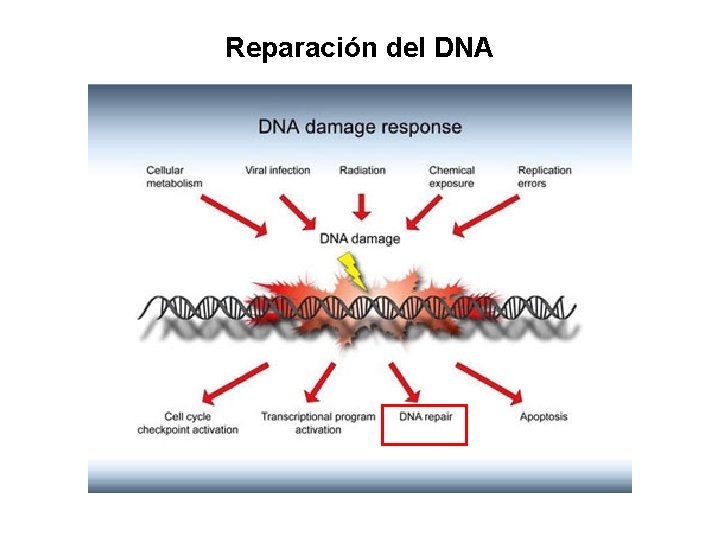
Reparación del DNA

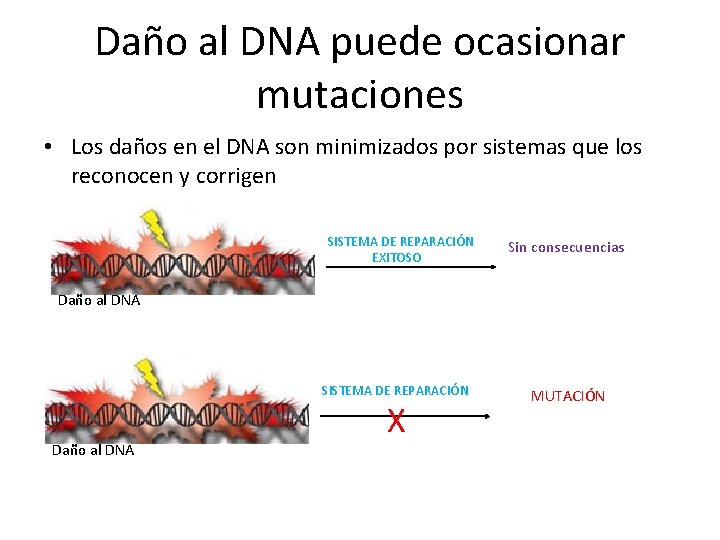
Daño al DNA puede ocasionar mutaciones • Los daños en el DNA son minimizados por sistemas que los reconocen y corrigen SISTEMA DE REPARACIÓN EXITOSO Sin consecuencias Daño al DNA SISTEMA DE REPARACIÓN Daño al DNA X MUTACIÓN

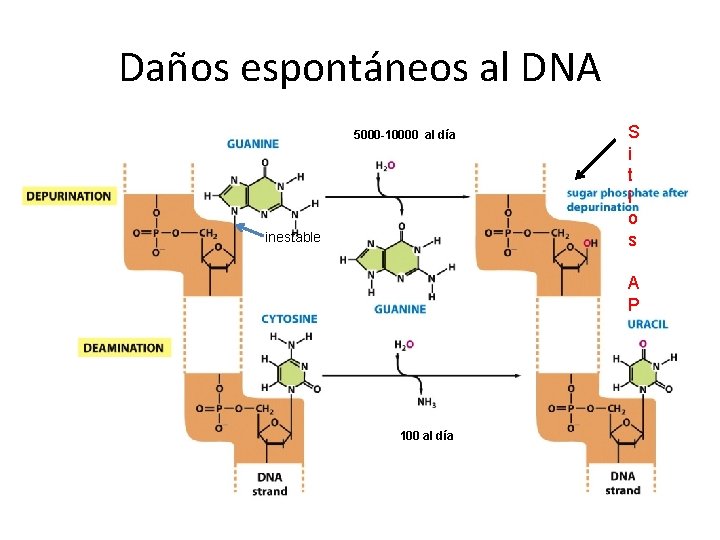
Daños espontáneos al DNA 5000 -10000 al día inestable S i t i o s A P 100 al día

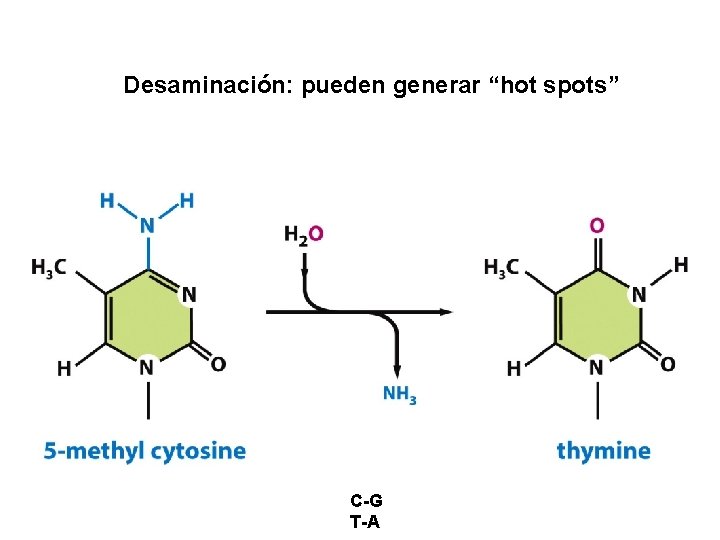
Desaminación: pueden generar “hot spots” C-G T-A

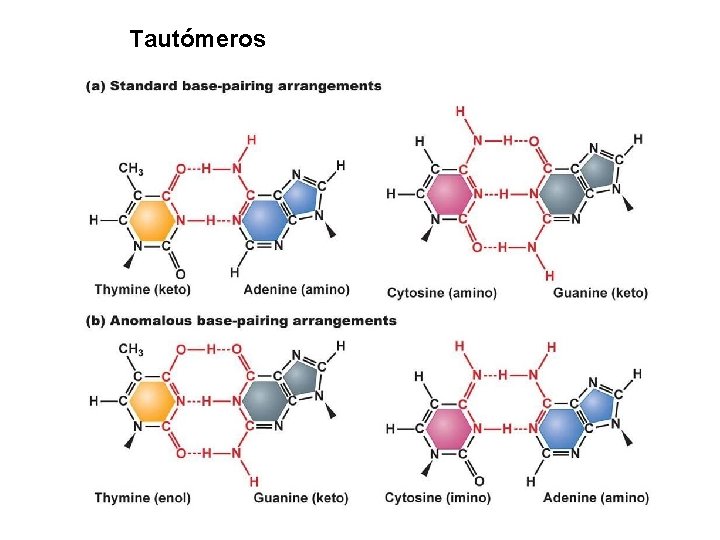
Tautómeros


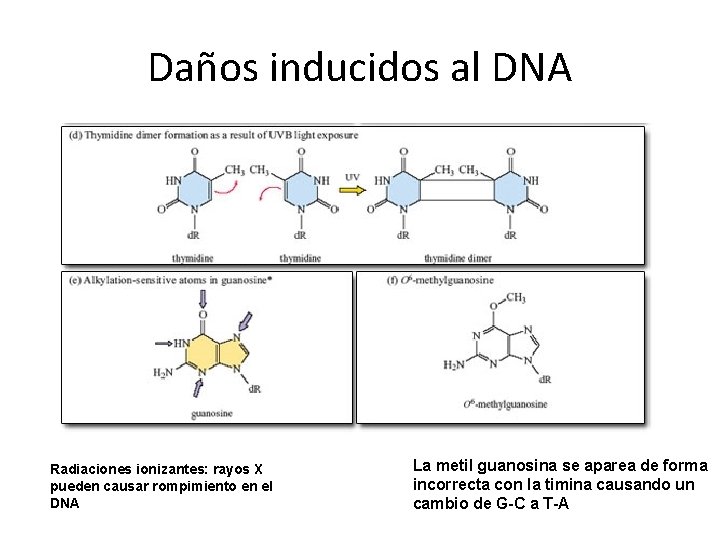
Daños inducidos al DNA Radiaciones ionizantes: rayos X pueden causar rompimiento en el DNA La metil guanosina se aparea de forma incorrecta con la timina causando un cambio de G-C a T-A

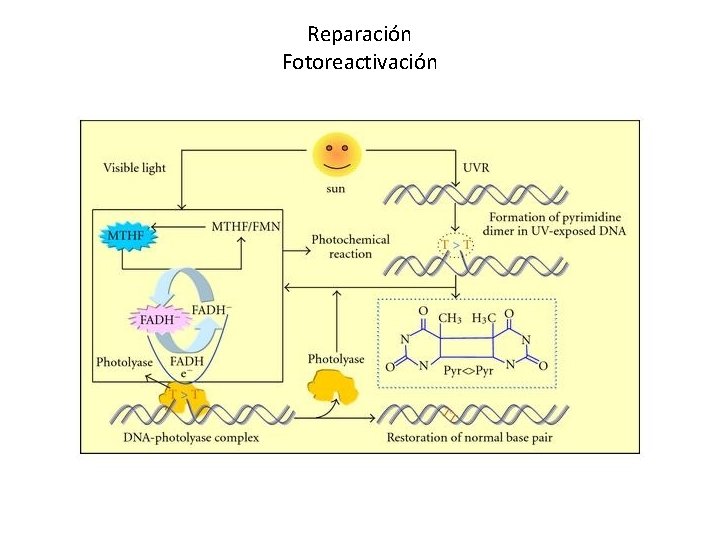
MECANISMOS DE REPARACION 1 - Sistemas de Reparación directos • Enzimas que revierten directamente el daño. • Por estos mecanismos se reparan: metilación de guanina, y en algunos vertebrados dímeros de pirimidína. No intervienen nucleasas ni ADN-polimerasas. • Fotoreactivación (ruptura de los dímeros de pirimidinas por acción de una fotoliasa (phr) activada mediante luz visible).


Reparación Fotoreactivación

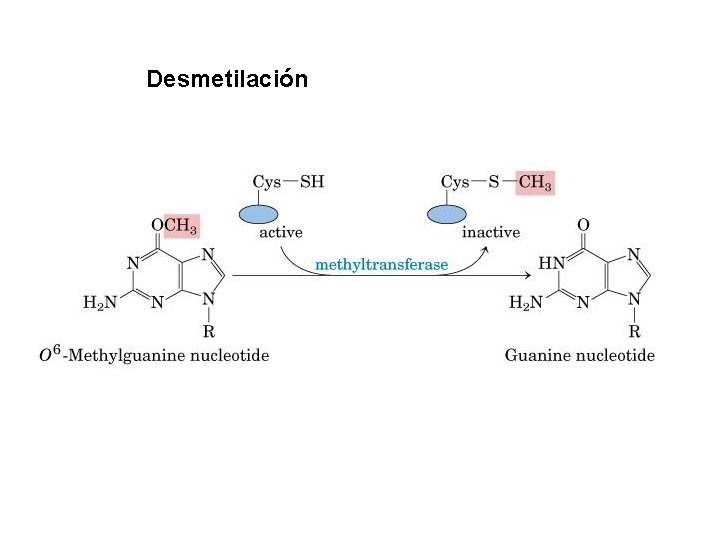
Desmetilación

2 - Sistemas de Reparación Indirecta Hay intervención de nucleasas y ADN-polimerasas. Se necesita de la hebra “molde” perteneciente al mismo cromosoma o al homólogo. Reparación por Escisio n (BER , NER, MMR) • Reparación de nucleótidos (NER) aislados por lesión UV: necesita la otra hebra como templado (hasta 30 bp). Intervienen las endonucleasas uvr. A, B, C y la helicasa uvr. D. ü Además de foto productos, repara lesiones voluminosas (bulky) que distorsionan la conformación del dúplex y que obstaculizarían la transcripción y replicación. • Reparación de bases modificadas (BER). - Repara casos de alteraciones puntuales en bases nitrogenadas (lesiones NO voluminosas) producidas por alquilación, oxidación o desaminación. Se origina un “sitio AP” y luego se retira el nucleótido “AP” y se re sintetiza la hebra)

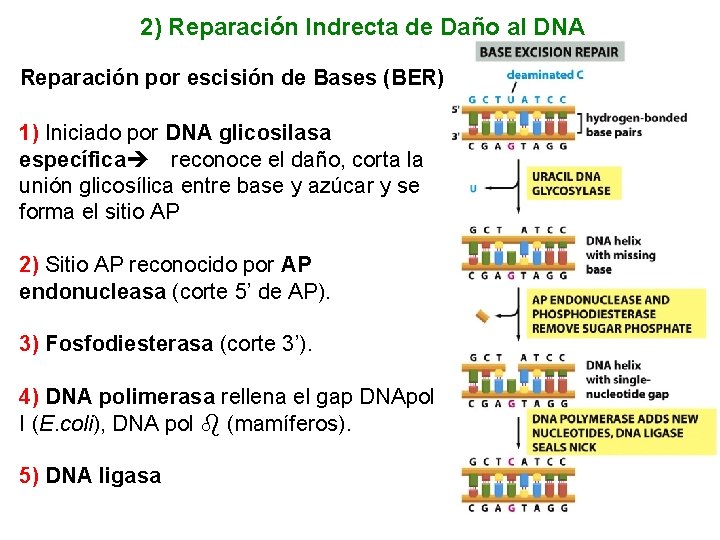
2) Reparación Indrecta de Daño al DNA Reparación por escisión de Bases (BER) 1) Iniciado por DNA glicosilasa específica reconoce el daño, corta la unión glicosílica entre base y azúcar y se forma el sitio AP 2) Sitio AP reconocido por AP endonucleasa (corte 5’ de AP). 3) Fosfodiesterasa (corte 3’). 4) DNA polimerasa rellena el gap DNApol I (E. coli), DNA pol (mamíferos). 5) DNA ligasa

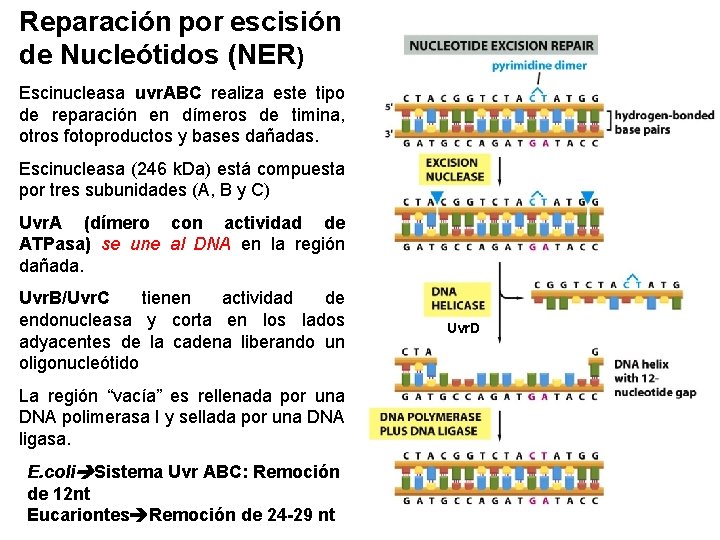
Reparación por escisión de Nucleótidos (NER) Escinucleasa uvr. ABC realiza este tipo de reparación en dímeros de timina, otros fotoproductos y bases dañadas. Escinucleasa (246 k. Da) está compuesta por tres subunidades (A, B y C) Uvr. A (dímero con actividad de ATPasa) se une al DNA en la región dañada. Uvr. B/Uvr. C tienen actividad de endonucleasa y corta en los lados adyacentes de la cadena liberando un oligonucleótido La región “vacía” es rellenada por una DNA polimerasa I y sellada por una DNA ligasa. E. coli Sistema Uvr ABC: Remoción de 12 nt Eucariontes Remoción de 24 -29 nt Uvr. D

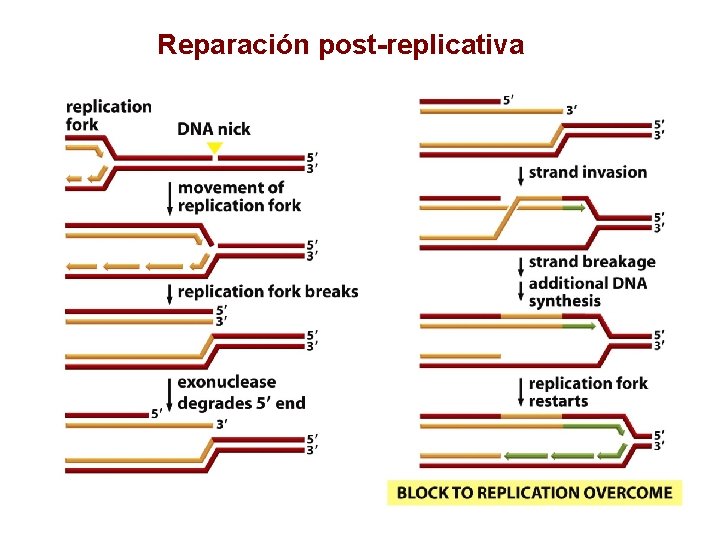
Reparación post- replicativa • Reparación del apareamiento (MMR) (“mismatch repair”): ü Su principal tarea es remover bases mal aparadas y pequeños “loops” introducidos por inserciones / deleciones durante la replicación ü Reduce los errores de replicación de 10 -7 a 10 -10 pb / replicación

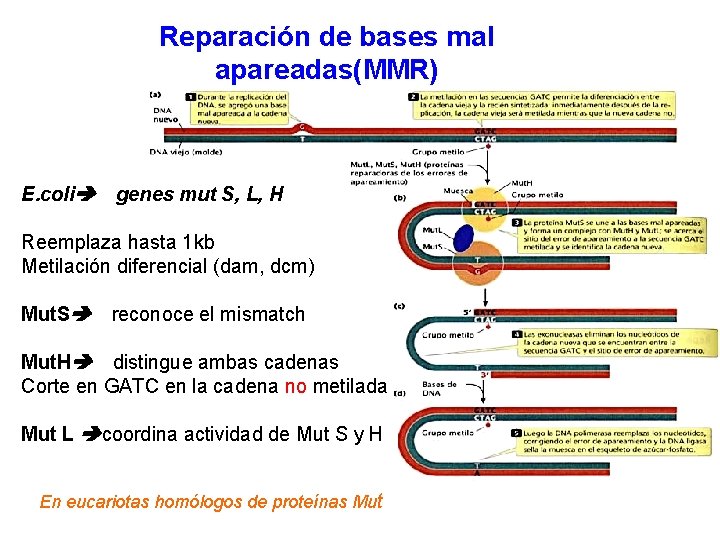
Reparación de bases mal apareadas(MMR) E. coli genes mut S, L, H Reemplaza hasta 1 kb Metilación diferencial (dam, dcm) Mut. S reconoce el mismatch Mut. H distingue ambas cadenas Corte en GATC en la cadena no metilada Mut L coordina actividad de Mut S y H En eucariotas homólogos de proteínas Mut

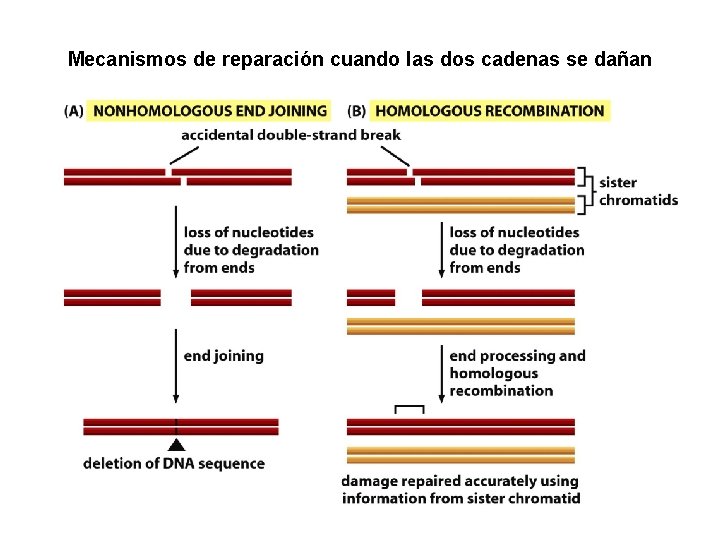
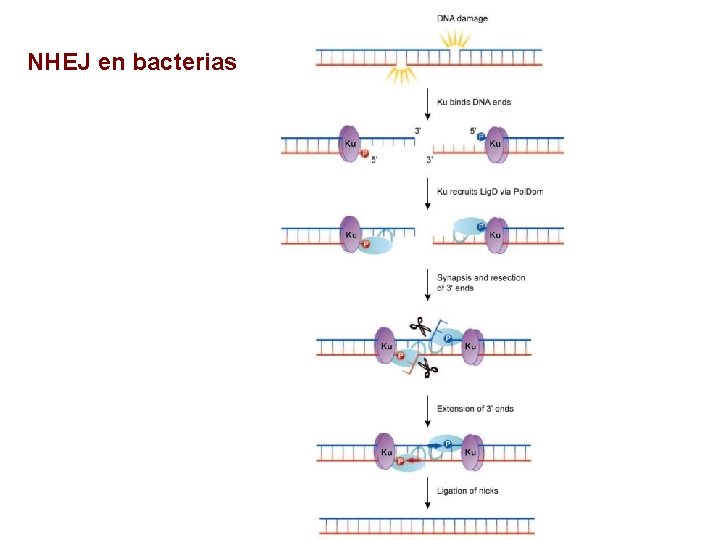
… Reparación post- replicativa • Recombinación Homóloga (HR) ü Reparación de ambas cadenas ü Usa ADN homólogo como templado y es altamente exacto ü Más activo durante la Fase S y G 2 ü Unión de extremos no homólogos (NHEJ) ü Reparación de ambas cadenas ü No usa ADN templado y generalmente se pierden algunos nucleótidos. ü Más activo en la Fase G 1

Mecanismos de reparación cuando las dos cadenas se dañan

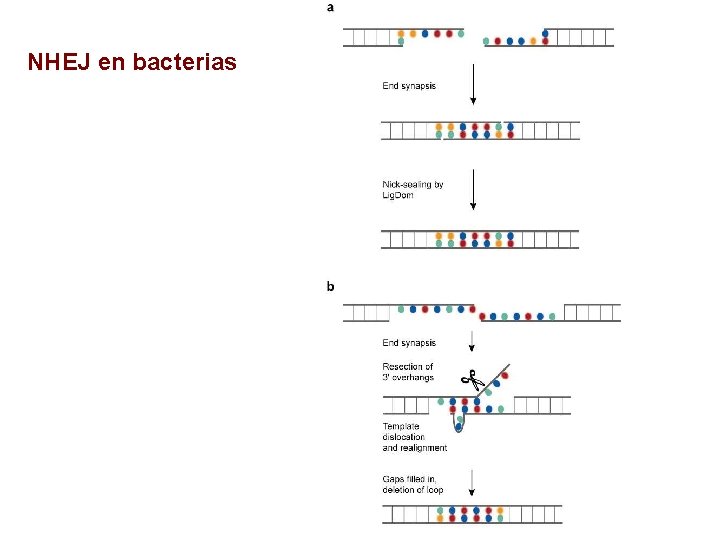
NHEJ en bacterias


NHEJ en bacterias

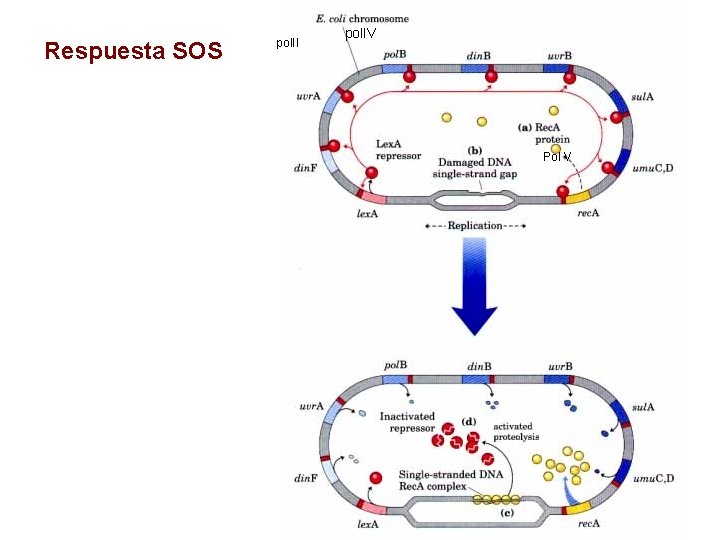
Respuesta SOS pol. II pol. IV Pol V


Mecanismos de Reparación en Eucariontes

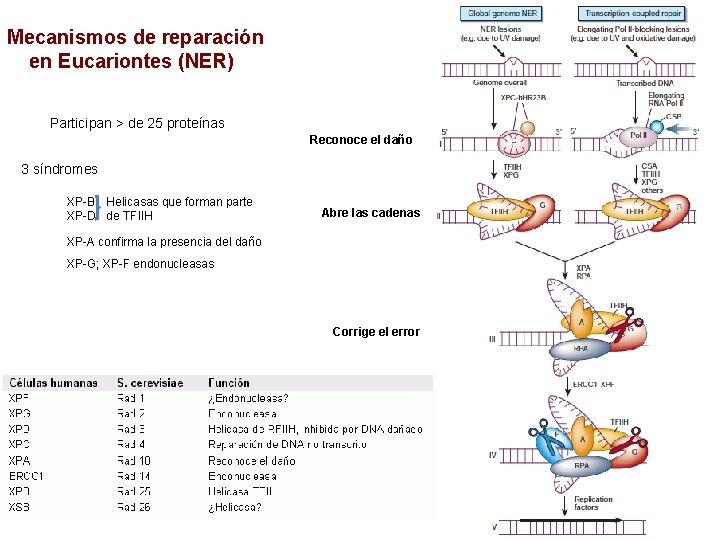
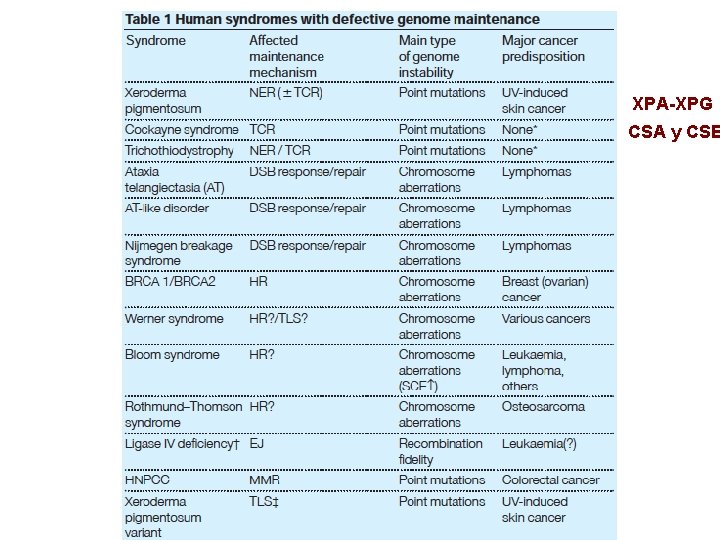
Mecanismos de reparación en Eucariontes (NER) Participan > de 25 proteínas Reconoce el daño 3 síndromes XP-B Helicasas que forman parte XP-D de TFIIH Abre las cadenas XP-A confirma la presencia del daño XP-G; XP-F endonucleasas Corrige el error

XPA-XPG CSA y CSB

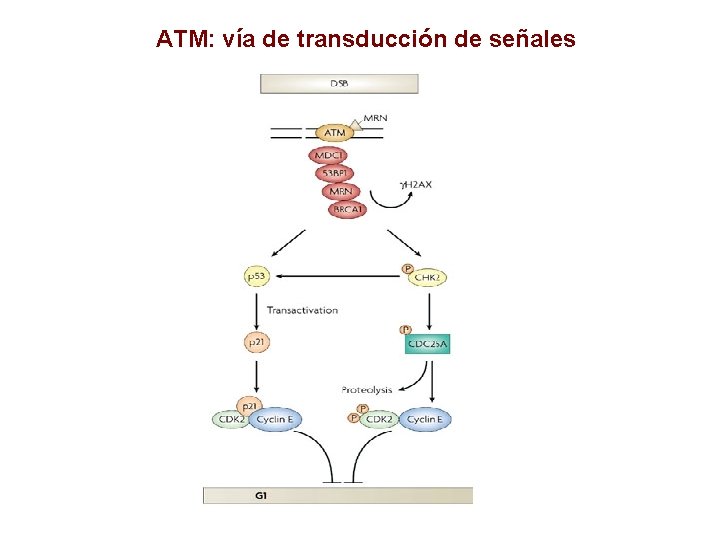
ATM: vía de transducción de señales


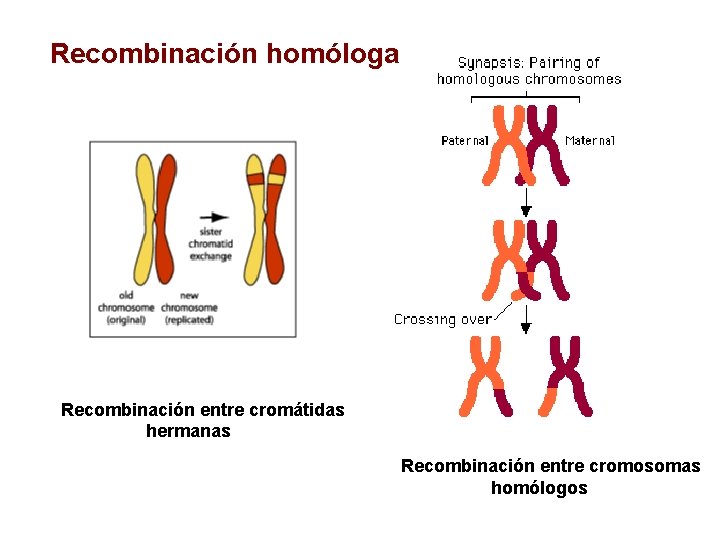
RECOMBINACIÓN Recombinación homóloga Recombinación no homóloga

Recombinación homóloga Recombinación entre cromátidas hermanas Recombinación entre cromosomas homólogos

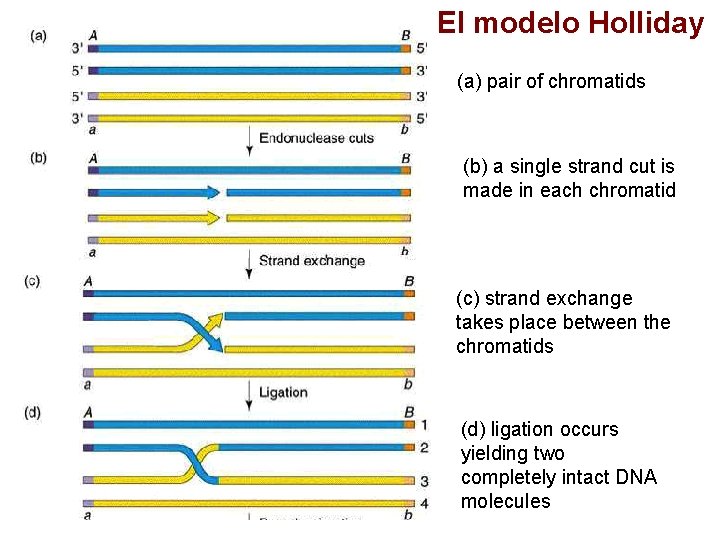
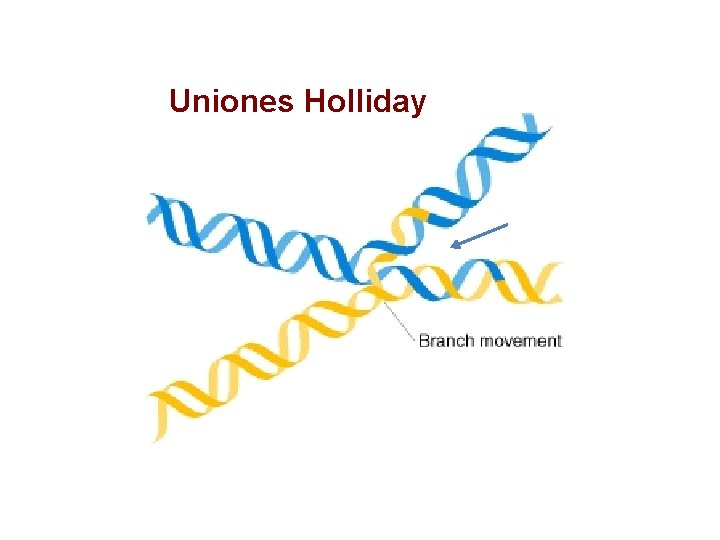
El modelo Holliday (a) pair of chromatids (b) a single strand cut is made in each chromatid (c) strand exchange takes place between the chromatids (d) ligation occurs yielding two completely intact DNA molecules

Uniones Holliday

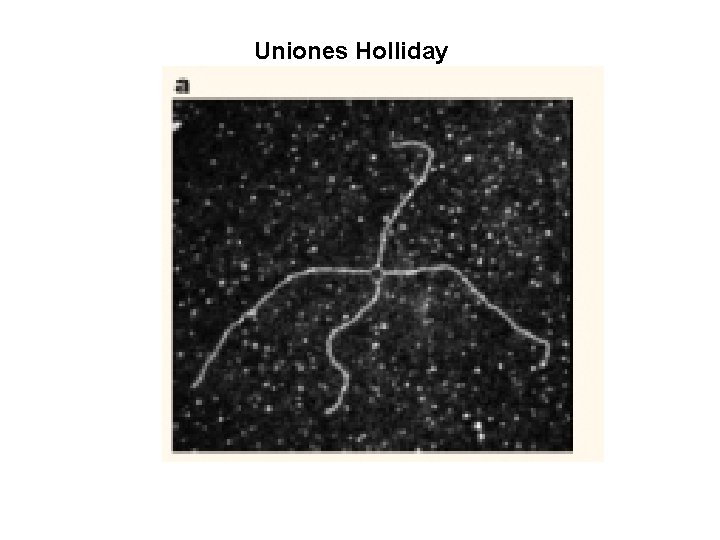
Uniones Holliday

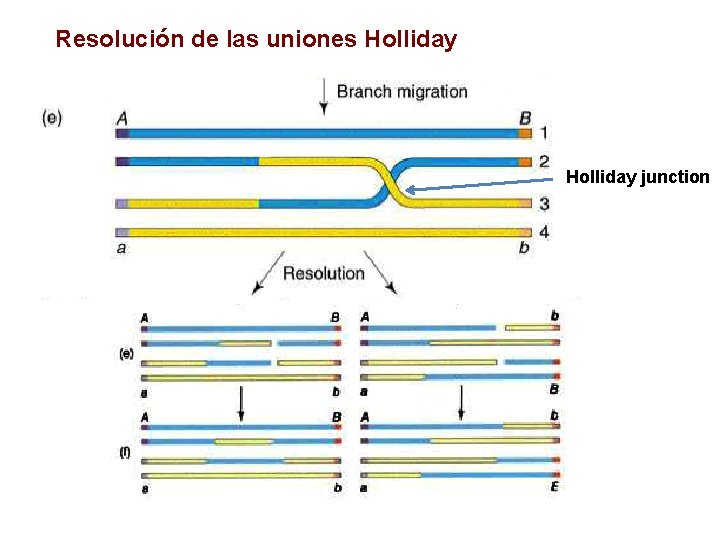
Resolución de las uniones Holliday junction


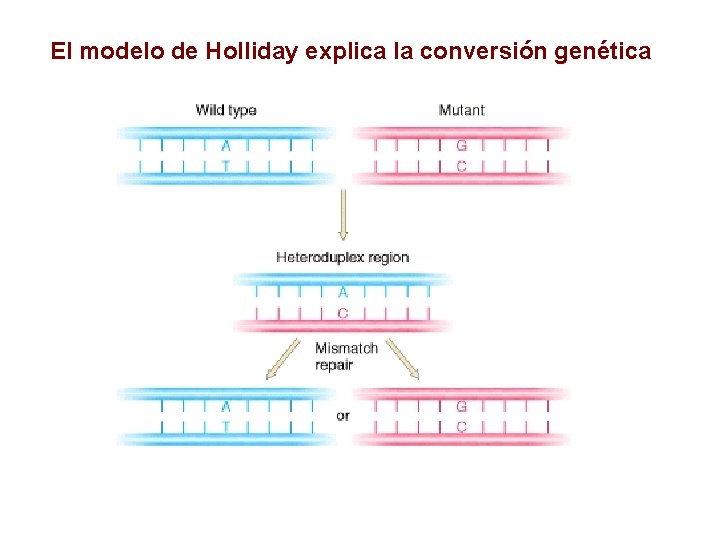
El modelo de Holliday explica la conversión genética

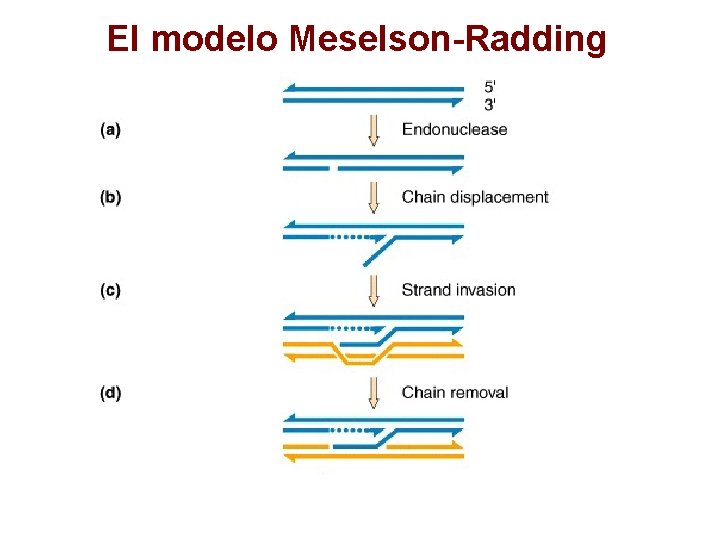
El modelo Meselson-Radding

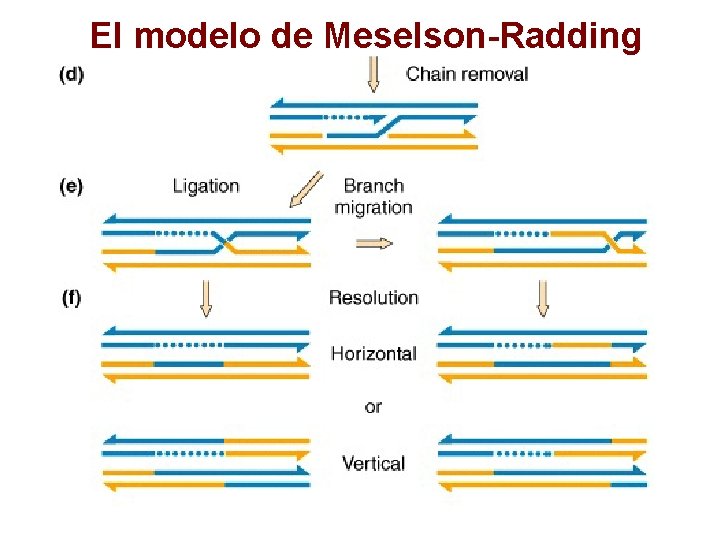
El modelo de Meselson-Radding

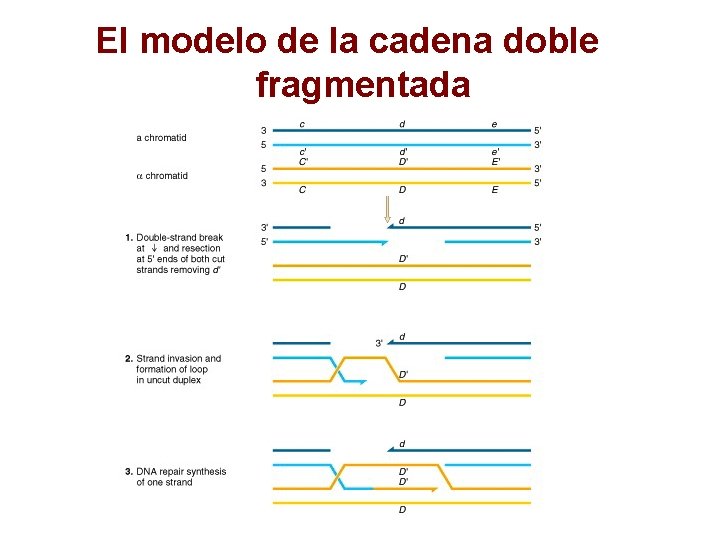
El modelo de la cadena doble fragmentada



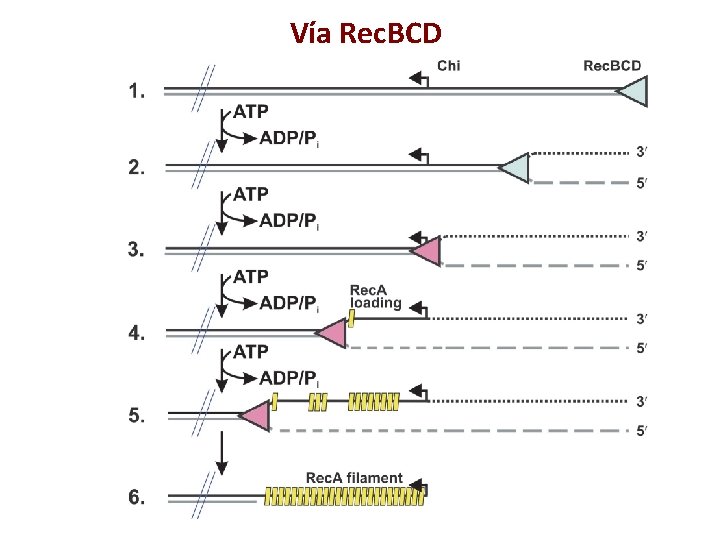
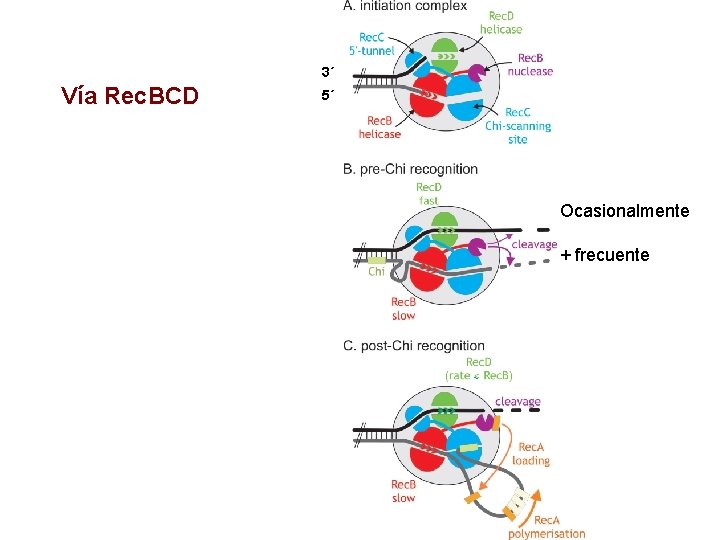
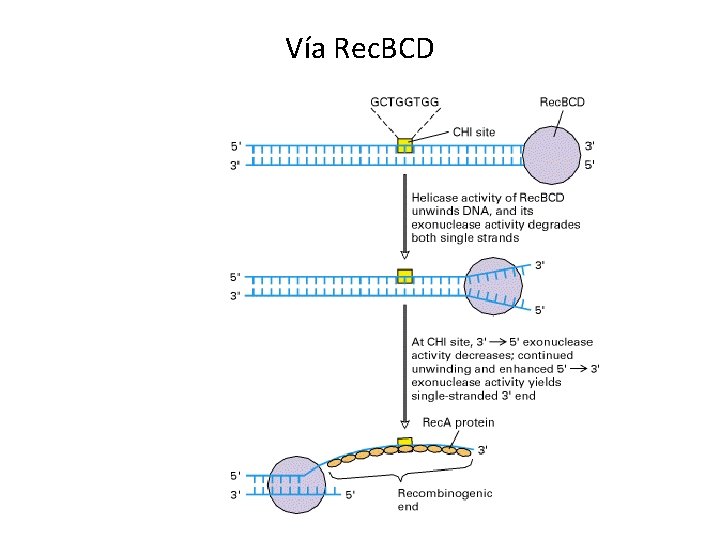
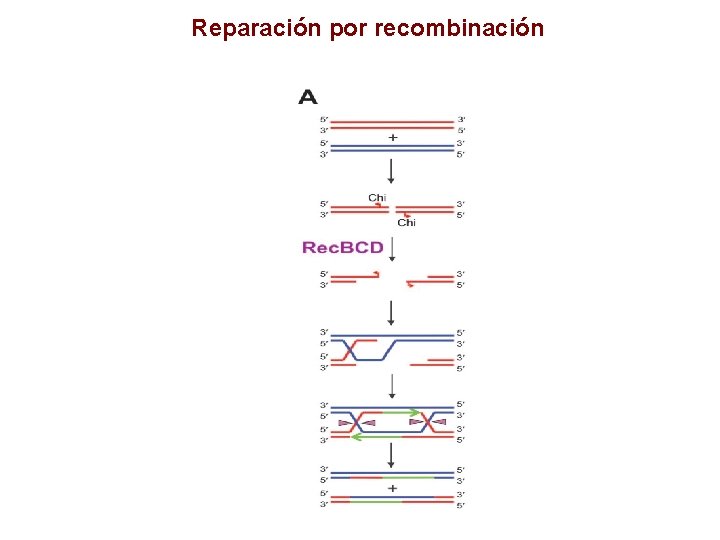
Vía Rec. BCD

Secuencia Chi • • • Estimula la recombinación en forma direccional 5´-GCTGGTGG-3´ Octámero sobre-representado en el genoma de E. coli. Existen aprox 1, 008 sitios Chi. Aparecen en promedio cada 4. 5 kpb. 75% de los sitios Chi están orientados hacia el origen de la replicación

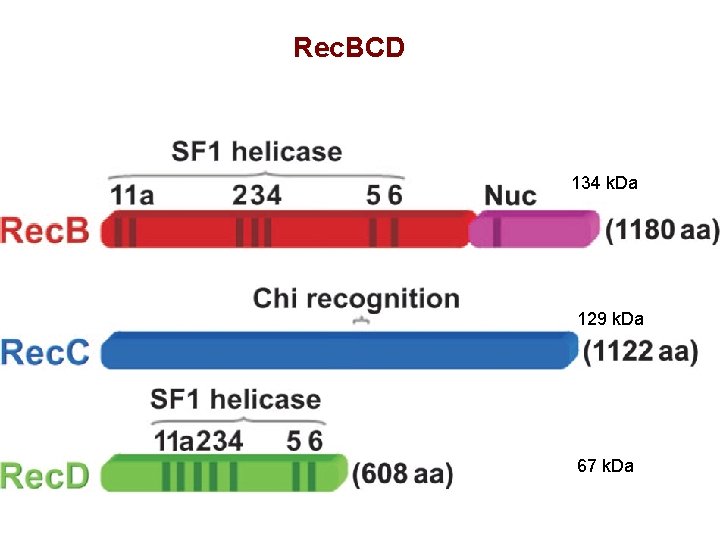
Rec. BCD 134 k. Da 129 k. Da 67 k. Da

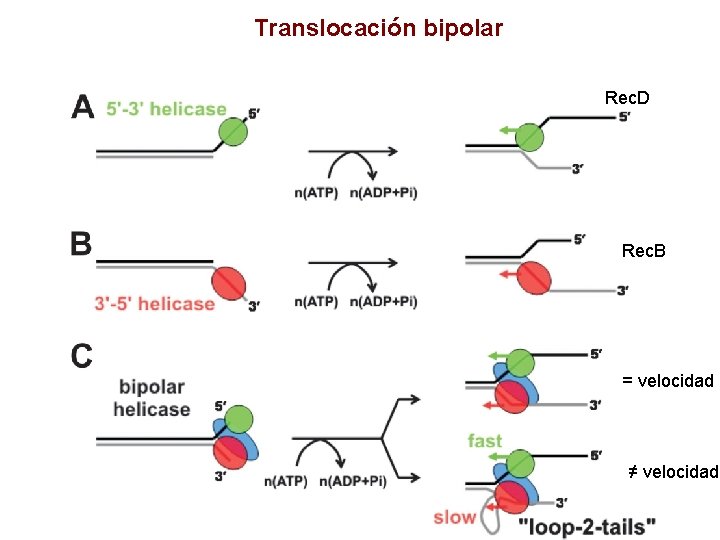
Translocación bipolar Rec. D Rec. B = velocidad ≠ velocidad

3´ Vía Rec. BCD 5´ Ocasionalmente + frecuente

Vía Rec. BCD

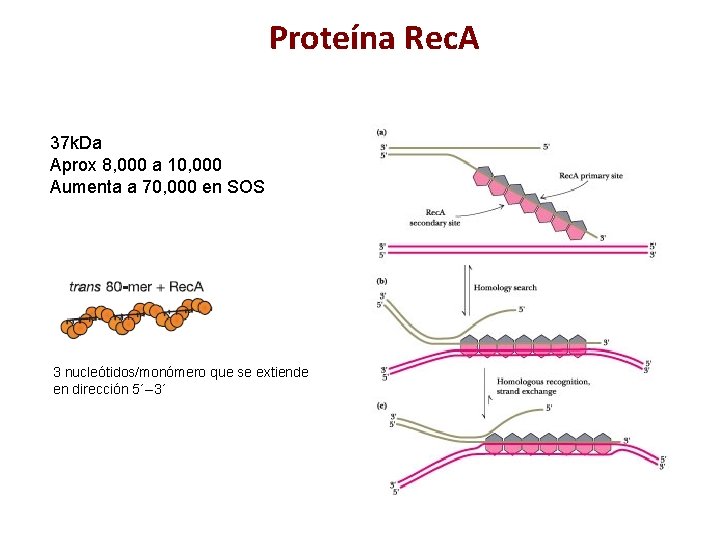
Proteína Rec. A 37 k. Da Aprox 8, 000 a 10, 000 Aumenta a 70, 000 en SOS 3 nucleótidos/monómero que se extiende en dirección 5´--3´


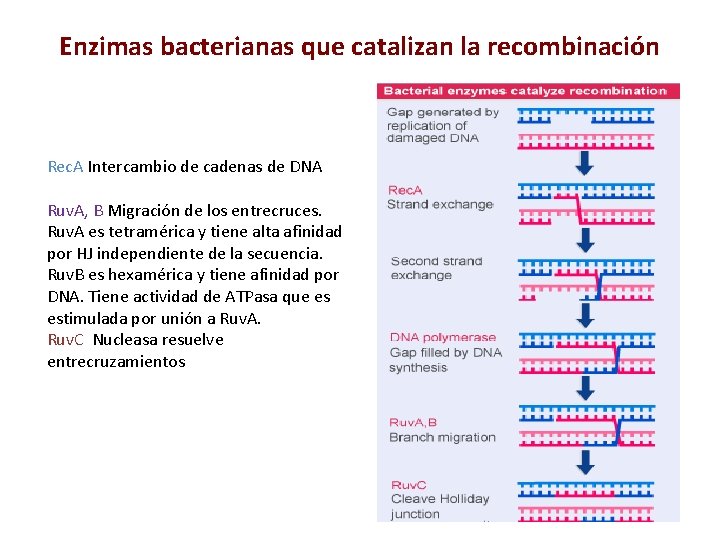
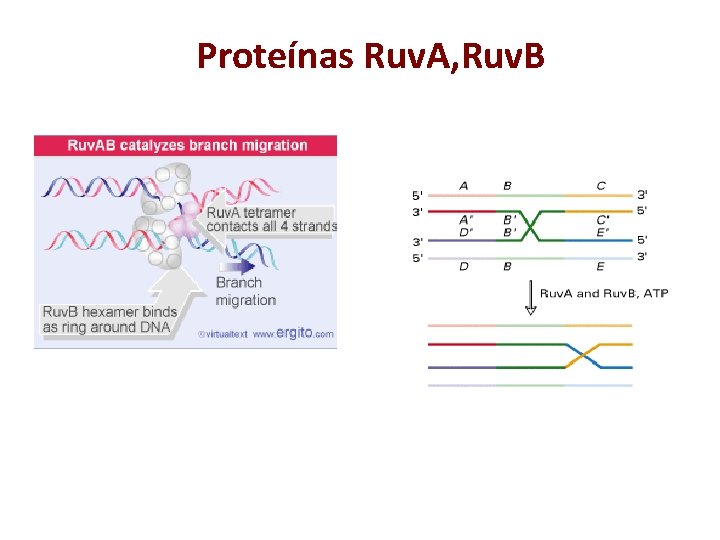
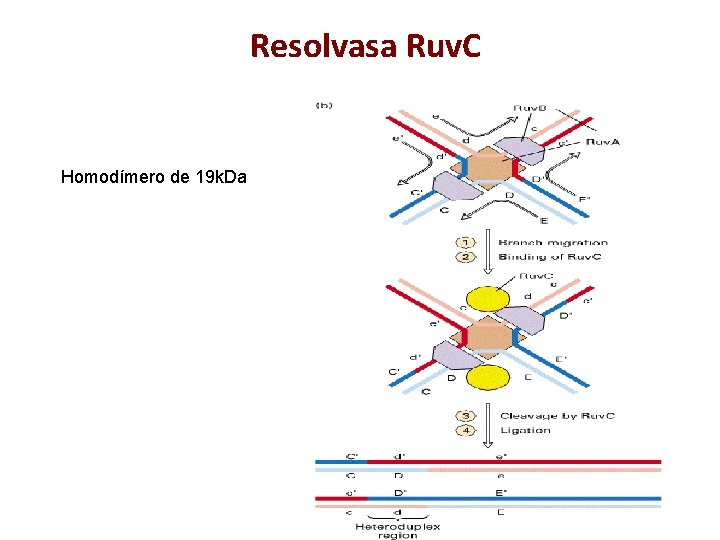
Enzimas bacterianas que catalizan la recombinación Rec. A Intercambio de cadenas de DNA Ruv. A, B Migración de los entrecruces. Ruv. A es tetramérica y tiene alta afinidad por HJ independiente de la secuencia. Ruv. B es hexamérica y tiene afinidad por DNA. Tiene actividad de ATPasa que es estimulada por unión a Ruv. A. Ruv. C Nucleasa resuelve entrecruzamientos

Proteínas Ruv. A, Ruv. B

Resolvasa Ruv. C Homodímero de 19 k. Da


Reparación por recombinación

Reparación post-replicativa
 Reparacin
Reparacin La distancia no causa el olvido pero si la nostalgia
La distancia no causa el olvido pero si la nostalgia Ti dao si nam čvrstu riječ
Ti dao si nam čvrstu riječ Kathryn dao
Kathryn dao Dao deficiency
Dao deficiency Daj nam svima duha svog
Daj nam svima duha svog Data access object pattern
Data access object pattern điện giật dùi đâm dao cắt
điện giật dùi đâm dao cắt Verse dao
Verse dao Mc.163.com download
Mc.163.com download Character traits of winnie the pooh
Character traits of winnie the pooh What do you mean by duty cycle
What do you mean by duty cycle Irene zhao
Irene zhao Tko je uveo izraelce u obećanu zemlju
Tko je uveo izraelce u obećanu zemlju Dao dto design pattern
Dao dto design pattern O mestre o mar se revolta
O mestre o mar se revolta Boga slavimo mi aleluja radosno srcem svim
Boga slavimo mi aleluja radosno srcem svim Ti dao si nam čvrstu riječ
Ti dao si nam čvrstu riječ Vba bof
Vba bof Decentralized autonomous organization (dao)
Decentralized autonomous organization (dao) Lateranska bazilika
Lateranska bazilika Qps jira
Qps jira Tko je isusu dao rubac
Tko je isusu dao rubac Dao moral
Dao moral Có công mài sắt có ngày nên kim
Có công mài sắt có ngày nên kim Rs10156191
Rs10156191 Dao as a service
Dao as a service Ni zhao
Ni zhao Replication
Replication Bioflix activity dna replication dna replication diagram
Bioflix activity dna replication dna replication diagram Coding dna and non coding dna
Coding dna and non coding dna What are the enzymes involved in dna replication
What are the enzymes involved in dna replication Dna rna protein synthesis homework #2 dna replication
Dna rna protein synthesis homework #2 dna replication Yo soy la puerta apocalipsis
Yo soy la puerta apocalipsis 20 corren uno pita dos detienen muchos gritan
20 corren uno pita dos detienen muchos gritan Una instruccion incrustada no puede ser una declaracion c#
Una instruccion incrustada no puede ser una declaracion c# Consolidación escritura ley pierri
Consolidación escritura ley pierri Puede un sacerdote perdonar pecados
Puede un sacerdote perdonar pecados 1
1 Donde se puede tomar el pulso
Donde se puede tomar el pulso Lo que puede el dinero analisi
Lo que puede el dinero analisi Contacto y distancia
Contacto y distancia La luna se puede tomar a cucharadas
La luna se puede tomar a cucharadas Donde tomar el pulso
Donde tomar el pulso Se puede romper un alambre estirandolo con las manos
Se puede romper un alambre estirandolo con las manos El ser humano puede vivir aislado de los demás
El ser humano puede vivir aislado de los demás Conjuntos por comprensión ejemplos
Conjuntos por comprensión ejemplos Acido folico beneficios
Acido folico beneficios Baile es diptongo o hiato
Baile es diptongo o hiato Modelo entidad relacion herencia
Modelo entidad relacion herencia Signo que indica una pausa corta
Signo que indica una pausa corta Sitrared dge
Sitrared dge El hiato puede romper triptongos
El hiato puede romper triptongos Como encontrar a dios
Como encontrar a dios Cómo localizaría un lugar en la superficie terrestre
Cómo localizaría un lugar en la superficie terrestre No se engañen de dios nadie se burla
No se engañen de dios nadie se burla