FARMACOGENOMICA FARMACOGENETICA Disciplina cientfica que estudia las variantes


















- Slides: 34

FARMACOGENOMICA

FARMACOGENETICA Disciplina científica que estudia las variantes genéticas de los individuos en respuesta a las drogas Genética aplicada a la optimización de la terapia Farmacológica

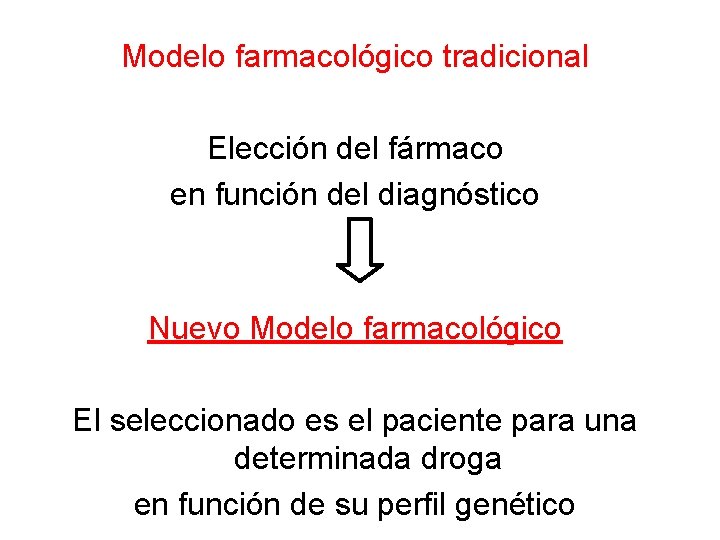
Modelo farmacológico tradicional Elección del fármaco en función del diagnóstico Nuevo Modelo farmacológico El seleccionado es el paciente para una determinada droga en función de su perfil genético

FARMACOGENETICA Muchos factores no genéticos influyen en los efectos de la medicación: v Edad v Sexo v Función orgánica v Terapias concomitantes v Severidad de la enfermedad v Interacción con drogas, etc. Los factores genéticos permanecen estables a lo largo de toda la vida de una persona

¿Cuáles son estos factores genéticos?

• Polimorfismos de secuencia: Genes HLA • Polimorfismos de longitud: Mini y Microsatélites • Polimorfismos de nucleótido único (SNPs) Son los más frecuentes y pueden asociarse a determinadas enfermedades o respuestas a fármacos.

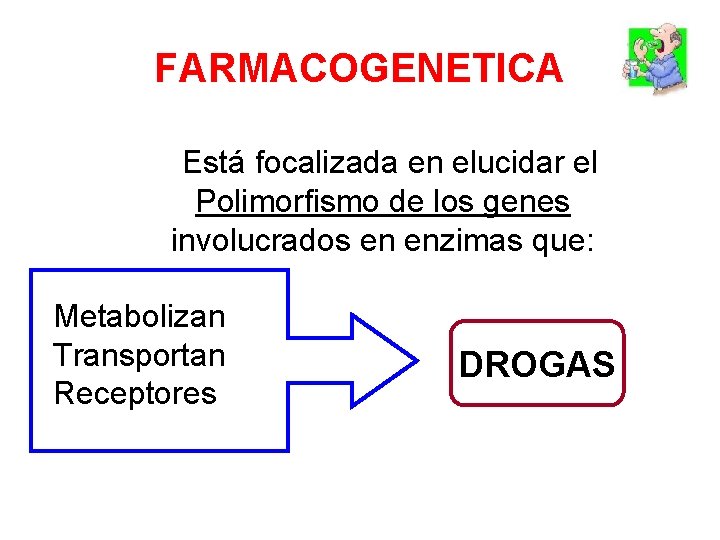
FARMACOGENETICA Está focalizada en elucidar el Polimorfismo de los genes involucrados en enzimas que: Metabolizan Transportan Receptores DROGAS

Pirámide farmacogenomica SNPs que modifican desenlace clínico SNPs que cambian respuesta a drogas SNPs que cambian FC SNPs que cambian actividad in vitro Cambios no conservadores en Aa SNPs no sinónimos en exones Cambios en exones Todos los SNPs

¿Cómo se descubre el SNP asociado?

Previo al proyecto genoma humano … HYPOTHESIS-DRIVEN APPROACH Después del proyecto genoma humano … GWAS

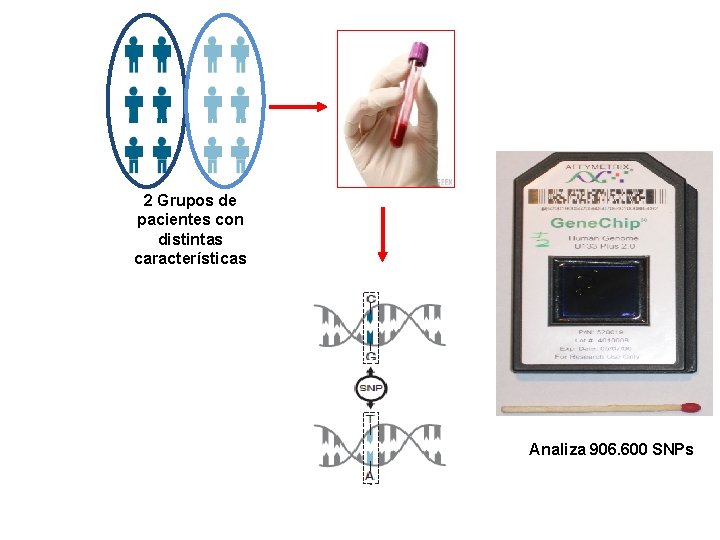
FARMACOGENETICA ESTRATEGIA GWAS Comparar la secuencia del genoma de 2 poblaciones Buena respuesta al tratamiento Mala respuesta al tratamiento (controles) (casos) Identificar los genes asociados con la causa de la variabilidad al tratamiento

2 Grupos de pacientes con distintas características Analiza 906. 600 SNPs


¿Qué técnicas se emplean para determinar los SNPs? PCR + RFLP PCR alelo específica PCR + secuenciación Real time PCR con gradientes de temperatura

FARMACOGENETICA n Mejorar la concepción del ensayo clínico n Desarrollar tratamientos más seguros y eficaces n Identificar nuevas indicaciones terapéuticas para los tratamientos existentes

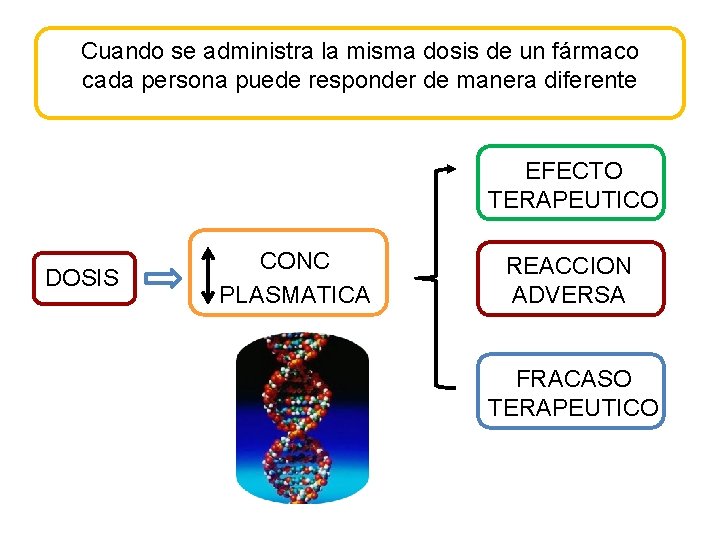
Cuando se administra la misma dosis de un fármaco cada persona puede responder de manera diferente EFECTO TERAPEUTICO DOSIS CONC PLASMATICA REACCION ADVERSA FRACASO TERAPEUTICO

CITOCROMO P 450 CYP • Superfamilia muy diversa de hemoproteínas encontrada en bacterias, plantas y eucariontes. • Involucradas en el metabolismo de enorme cantidad de compuestos endógenos y exógenos. • Introducen cambios de reducción, oxidación o peroxidación sobre moléculas de diferente estructura química • Contienen un núcleo hemo unido a la proteína a través de un ligando tiolato derivado de cisteína • La unión del sustrato inicia una cadena de transporte de electrones y unión de oxígeno • Otras proteínas (CYP 450 reductasa, ferredoxina, Citocromo B 5) proveen electrones para le reducción del hierro • El oxígeno molecular es captado y separado, un átomo se consume en la monooxigenación del sustrato y el otro es reducido a agua.

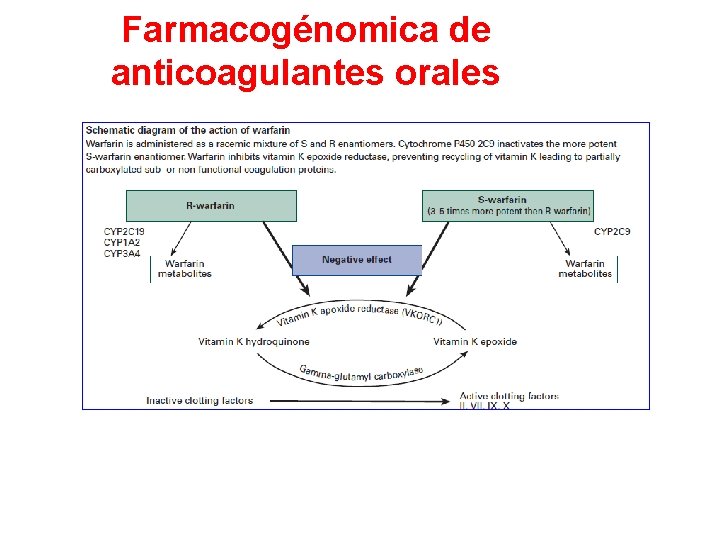
Farmacogénomica de anticoagulantes orales

Polimorfismos de CYP 2 C 9 • Alelo*1 (WT) metabolizadores normales. • Alelo *2: actividad enzimática del 12% • Alelo *3: actividad enzimática del 5% disminución del metabolismo de la warfarina requiere dosis menores Polimorfismos de la Vitamina K epóxido reductasa-VKORC 1 Polimorfismo 1639 (G>A) en región promotora del intrón 1 Alelo G: WT Alelo A: disminuye su expresión

Farmacogénomica de Inmunosupresores CYP 3 A 5 • Citocromo P 450, familia 3, subfamilia A, polipéptido 5 • Ubicado en el cromosoma 7 q 21. 1. Proteína monoxigenasa que cataliza reacciones involucradas en el metabolismo de drogas, síntesis de colesterol, esteroides y otros lípidos Esta localizada en RE. Metaboliza inmunosupresores.

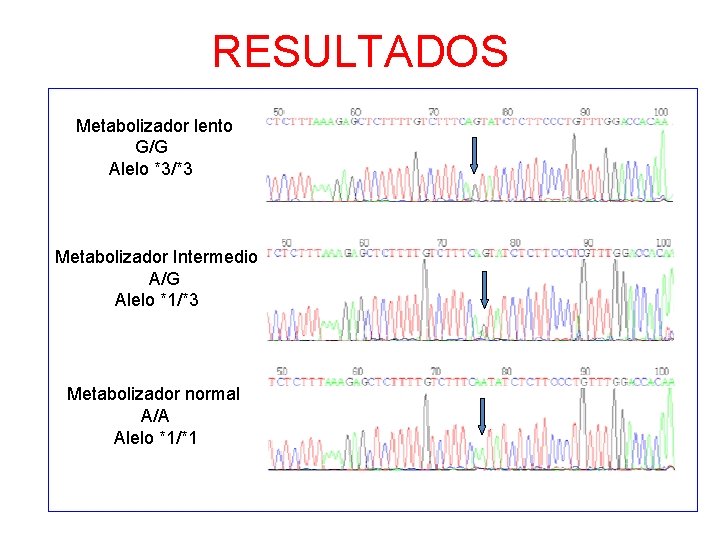
Polimorfismos de CYP 3 A 5 • Alelo *1 expresan la proteína normal (wild type). Pacientes *1/*1 METABOLIZADORES NORMALES (A/A) • SNP intrón 3: A G (6986 A>G). Alelo *3 proteína truncada y no funcional. METABOLIZADORES INTERMEDIOS Pacientes *1/*3 (A/G) METABOLIZADORES LENTOS Pacientes *3/*3 (G/G)

RESULTADOS Metabolizador lento G/G Alelo *3/*3 Metabolizador Intermedio A/G Alelo *1/*3 Metabolizador normal A/A Alelo *1/*1

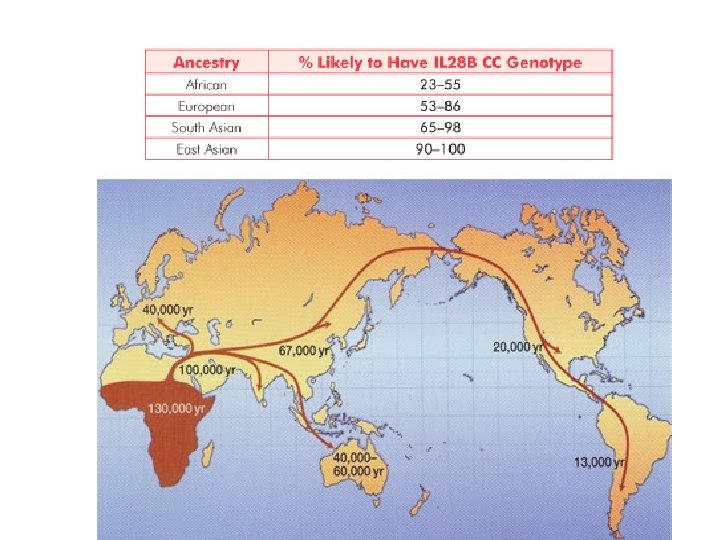
SNPs EN EL GEN DEL INTERFERON LAMBDA Ge et al. 2009; Thompson et al. 2010; Rauch et al. 2010; Tanaka et al. 2009; Suppiah et al. 2009; Montes-Cano et al. 2010; Prokunina-Olsson et al. 2013.

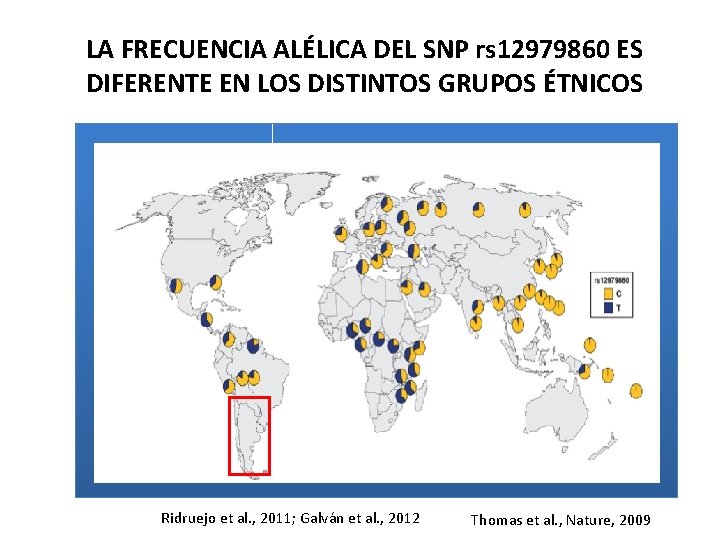
LA FRECUENCIA ALÉLICA DEL SNP rs 12979860 ES DIFERENTE EN LOS DISTINTOS GRUPOS ÉTNICOS Ridruejo et al. , 2011; Galván et al. , 2012 Thomas et al. , Nature, 2009

ORIGINAL POPULATION SELECTION PRESSURE AFTER SELECTION FINAL POPULATION

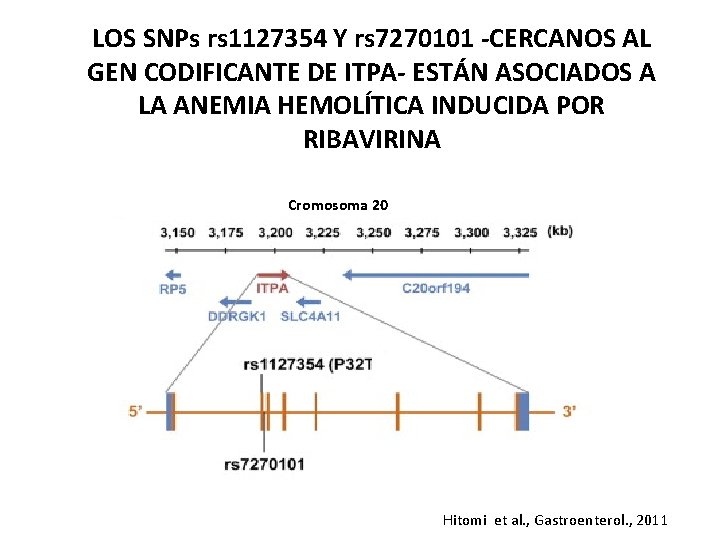
LOS SNPs rs 1127354 Y rs 7270101 -CERCANOS AL GEN CODIFICANTE DE ITPA- ESTÁN ASOCIADOS A LA ANEMIA HEMOLÍTICA INDUCIDA POR RIBAVIRINA Cromosoma 20 Hitomi et al. , Gastroenterol. , 2011

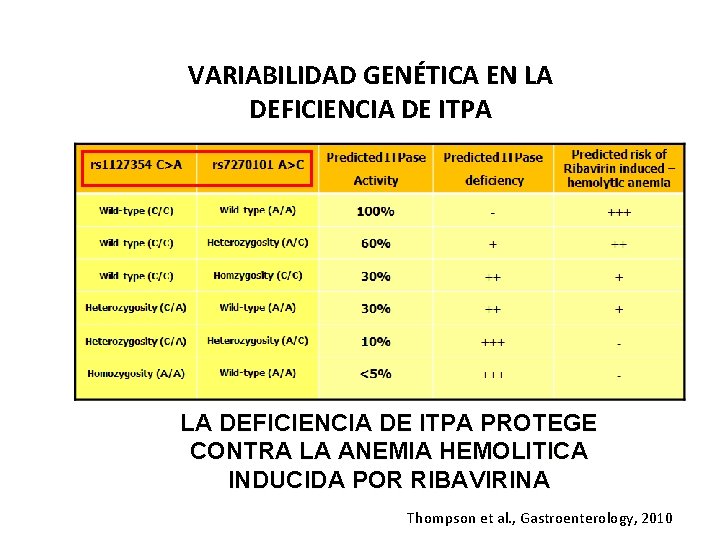
VARIABILIDAD GENÉTICA EN LA DEFICIENCIA DE ITPA PROTEGE CONTRA LA ANEMIA HEMOLITICA INDUCIDA POR RIBAVIRINA Thompson et al. , Gastroenterology, 2010

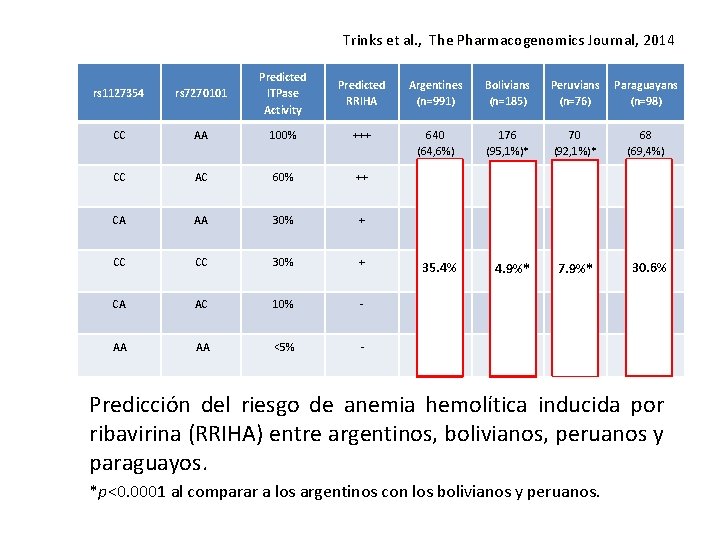
Trinks et al. , The Pharmacogenomics Journal, 2014 rs 1127354 rs 7270101 Predicted ITPase Activity CC AA 100% +++ 640 (64, 6%) 176 (95, 1%)* 70 (92, 1%)* 68 (69, 4%) CC AC 60% ++ 199 (20. 1%) 6 (3. 2%) 3 (4. 0%) 23 (23. 5%) CA AA 30% + 102 (10. 3%) 2 (1. 1%) 2 (2. 6%) 6 (6. 1%) CC CC 30% + 18 35. 4% 1 4. 9%* 0 7. 9%* 1 30. 6% Predicted RRIHA Argentines (n=991) Bolivians (n=185) Peruvians (n=76) Paraguayans (n=98) (1. 8%) (0. 6%) (1. 0%) CA AC 10% - 14 (1. 4%) 0 1 (1. 3%) 0 AA AA <5% - 18 (1. 8%) 0 0 0 Predicción del riesgo de anemia hemolítica inducida por ribavirina (RRIHA) entre argentinos, bolivianos, peruanos y paraguayos. *p<0. 0001 al comparar a los argentinos con los bolivianos y peruanos.

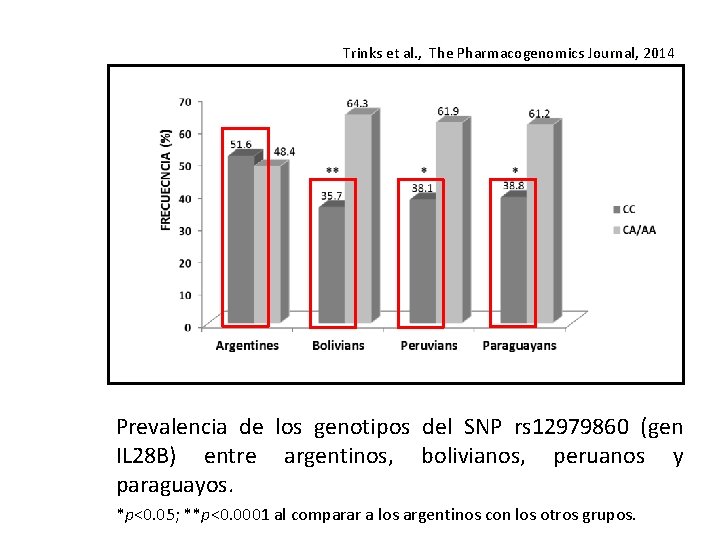
Trinks et al. , The Pharmacogenomics Journal, 2014 Prevalencia de los genotipos del SNP rs 12979860 (gen IL 28 B) entre argentinos, bolivianos, peruanos y paraguayos. *p<0. 05; **p<0. 0001 al comparar a los argentinos con los otros grupos.

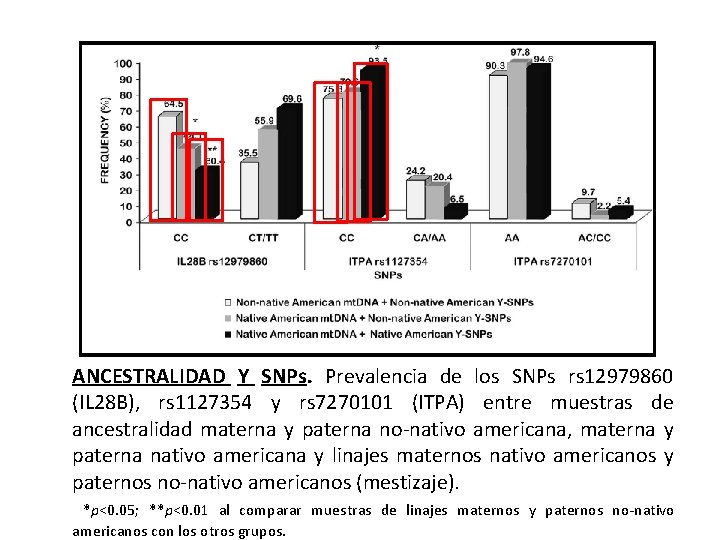
ANCESTRALIDAD Y SNPs. Prevalencia de los SNPs rs 12979860 (IL 28 B), rs 1127354 y rs 7270101 (ITPA) entre muestras de ancestralidad materna y paterna no-nativo americana, materna y paterna nativo americana y linajes maternos nativo americanos y paternos no-nativo americanos (mestizaje). *p<0. 05; **p<0. 01 al comparar muestras de linajes maternos y paternos no-nativo americanos con los otros grupos.

Conclusiones La farmacogenómica es una herramienta que abre un campo de acción hacia la individualización terapéutica Ayuda en la selección de fármacos y combinaciones Impacto potencial sobre eficacia y prevención de toxicidades

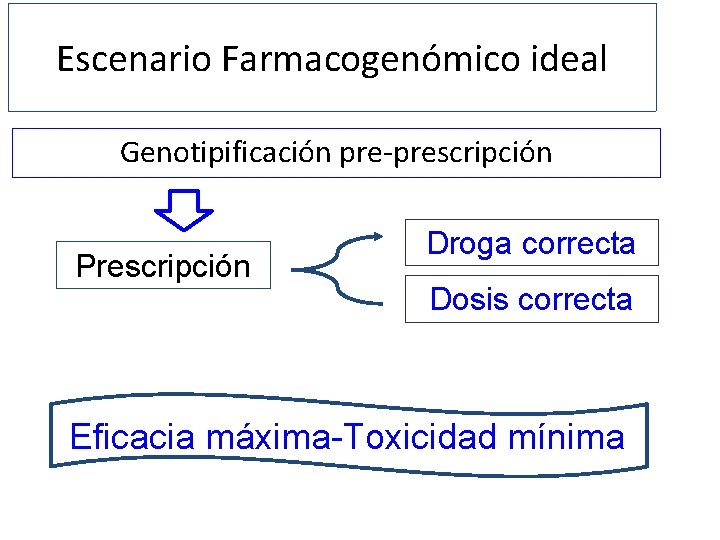
Escenario Farmacogenómico ideal Genotipificación pre-prescripción Prescripción Droga correcta Dosis correcta Eficacia máxima-Toxicidad mínima

CONCLUSION Los polimorfismos genéticos pueden determinar la respuesta a drogas y el diagnóstico molecular debe ser trasladado a la práctica clínica sirviendo como guía de selección de medicamentos y dosis de drogas que serán las óptima para cada individuo.

Medicina personalizada
 Farmacogenetica
Farmacogenetica Paradigma interpretativo
Paradigma interpretativo Para que se investiga
Para que se investiga Transposicion didáctica
Transposicion didáctica La ciencia que estudia las bacterias
La ciencia que estudia las bacterias Contabilidad es la disciplina que enseña las normas
Contabilidad es la disciplina que enseña las normas Variedades diafasicas ejemplos
Variedades diafasicas ejemplos Variantes tipograficas
Variantes tipograficas Alofonos de r
Alofonos de r Variantes anatomicas del esternon
Variantes anatomicas del esternon Procidence carotidienne sinus sphénoïdale
Procidence carotidienne sinus sphénoïdale Ejemplos de variable diatopica
Ejemplos de variable diatopica Variante anatomique à risque chirurgical
Variante anatomique à risque chirurgical O variantes
O variantes Leyes de la quinta disciplina ejemplos
Leyes de la quinta disciplina ejemplos Que estudia la microeconomía
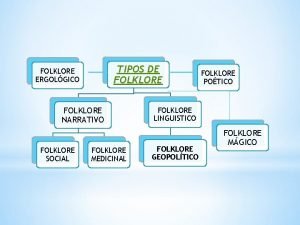
Que estudia la microeconomía Folklore narrativo
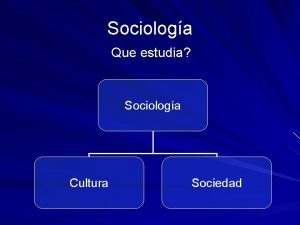
Folklore narrativo Que estudia la sociologia
Que estudia la sociologia Propiedades extensivas
Propiedades extensivas Demoecologia
Demoecologia Epinisio
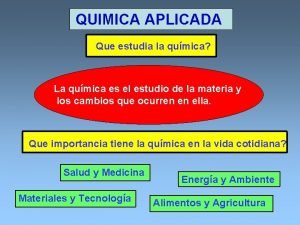
Epinisio Cosas basicas de la quimica
Cosas basicas de la quimica Metanomologico
Metanomologico Parenquima acuifero funcion
Parenquima acuifero funcion Partes del electroscopio
Partes del electroscopio Cuál es la ciencia que estudia el suelo
Cuál es la ciencia que estudia el suelo Sinecologia concepto
Sinecologia concepto Qué estudia la gramática
Qué estudia la gramática Trabajo social y antropologia
Trabajo social y antropologia Clases de parrafo
Clases de parrafo Que estudia la hidrostática
Que estudia la hidrostática Cinco magnitudes escalares
Cinco magnitudes escalares Macroperspectiva del comportamiento organizacional
Macroperspectiva del comportamiento organizacional Como se llama la carrera que estudia la biblia
Como se llama la carrera que estudia la biblia Que es la silvicultura
Que es la silvicultura