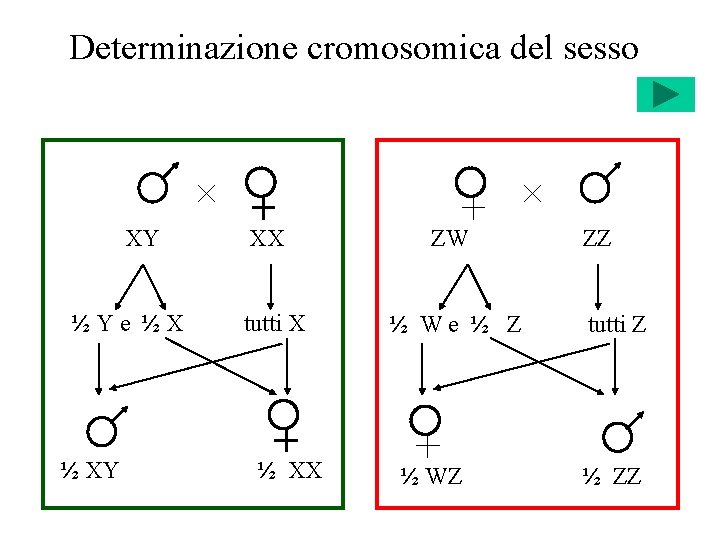
Determinazione cromosomica del sesso XY Ye X XY

- Slides: 33

Determinazione cromosomica del sesso XY ½Ye ½X ½ XY XX tutti X ½ XX ZW ½ We ½ Z ½ WZ ZZ tutti Z ½ ZZ

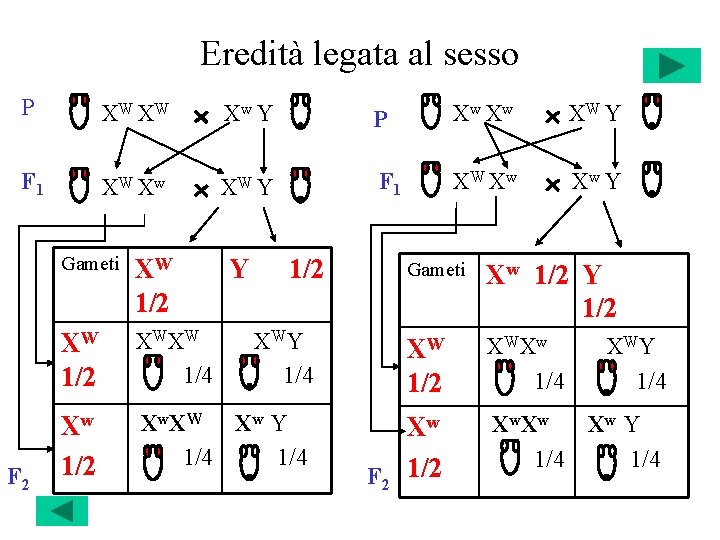
Eredità legata al sesso P XW XW Xw Y P Xw Xw XW Y F 1 XW Xw Xw Y F 2 Gameti XW 1/2 XW XW 1/4 Xw 1/2 Y 1/2 Gameti Xw 1/2 Y 1/2 XW Y 1/4 XW 1/2 XW Xw 1/4 X w. X W X w Y 1/4 Xw F 2 1/2 XW Y 1/4 X w Xw Y 1/4

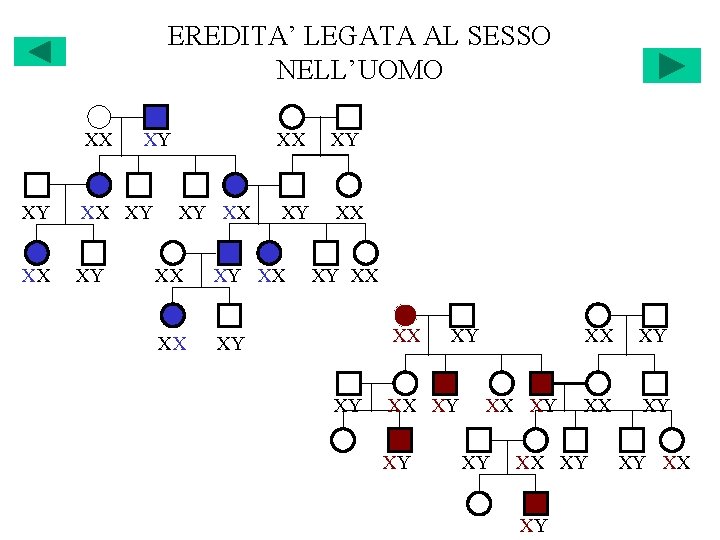
EREDITA’ LEGATA AL SESSO NELL’UOMO XX XY XY XX XX XY XY XX XY XX XY XY XY XX

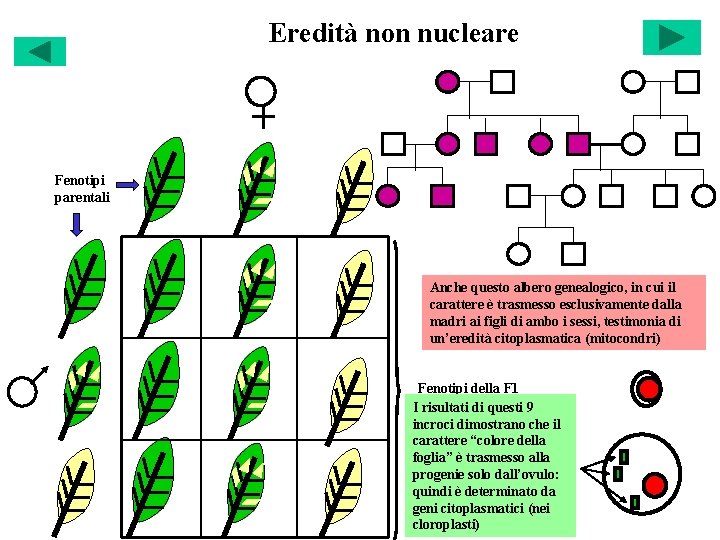
Eredità non nucleare Fenotipi parentali Anche questo albero genealogico, in cui il carattere è trasmesso esclusivamente dalla madri ai figli di ambo i sessi, testimonia di un’eredità citoplasmatica (mitocondri) Fenotipi della F 1 I risultati di questi 9 incroci dimostrano che il carattere “colore della foglia” è trasmesso alla progenie solo dall’ovulo: quindi è determinato da geni citoplasmatici (nei cloroplasti)

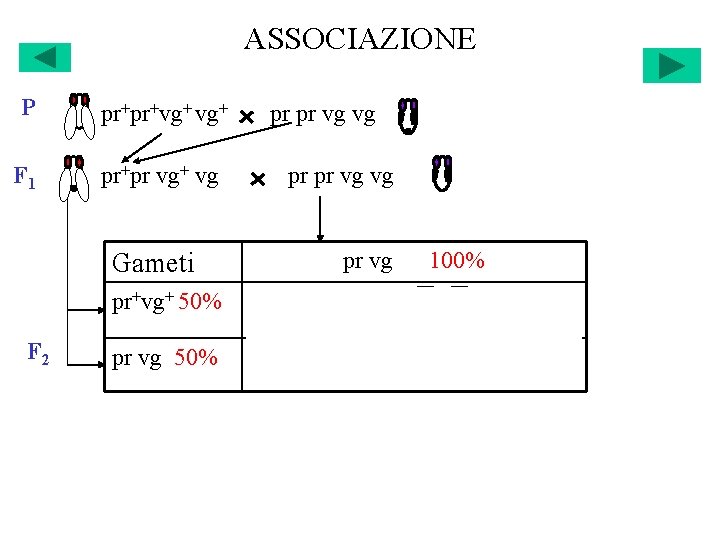
ASSOCIAZIONE P pr+pr+vg+ F 1 pr+pr vg+ vg Gameti F 2 pr pr vg vg pr vg 100% pr+vg+ 50% pr+ pr vg+ vg 25% +25% pr vg 50% pr pr vg vg 25% +25%

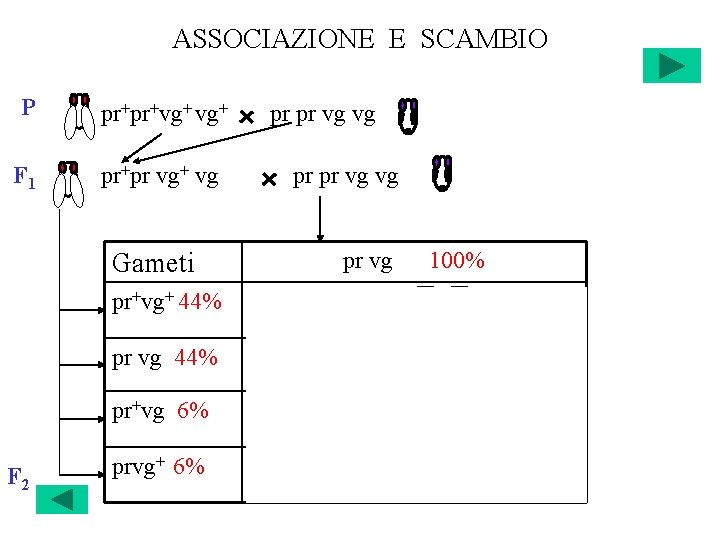
ASSOCIAZIONE E SCAMBIO P pr+pr+vg+ F 1 pr+pr vg+ vg Gameti F 2 pr pr vg vg pr vg 100% pr+vg+ 44% pr+ pr vg+ vg 22% +22% pr vg 44% pr pr vg vg 22% +22% pr+vg 6% pr+ pr vg vg 3% +3% prvg+ 6% pr pr vg+ vg 3% +3%

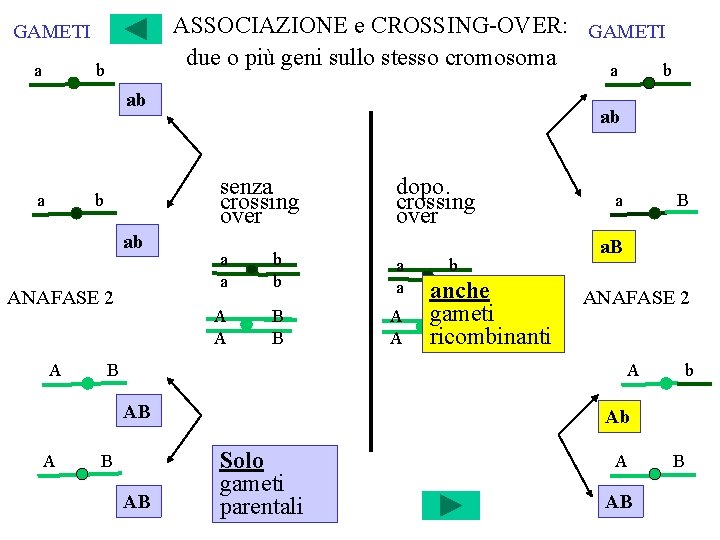
ASSOCIAZIONE e CROSSING-OVER: GAMETI due o più geni sullo stesso cromosoma a b GAMETI a b ab a senza crossing over b ab ANAFASE 2 A ab a a b b a a A A B B A A B b B anche gameti b B ricombinanti a B a. B ANAFASE 2 A AB A dopo crossing over B AB b Ab Solo gameti parentali A AB B

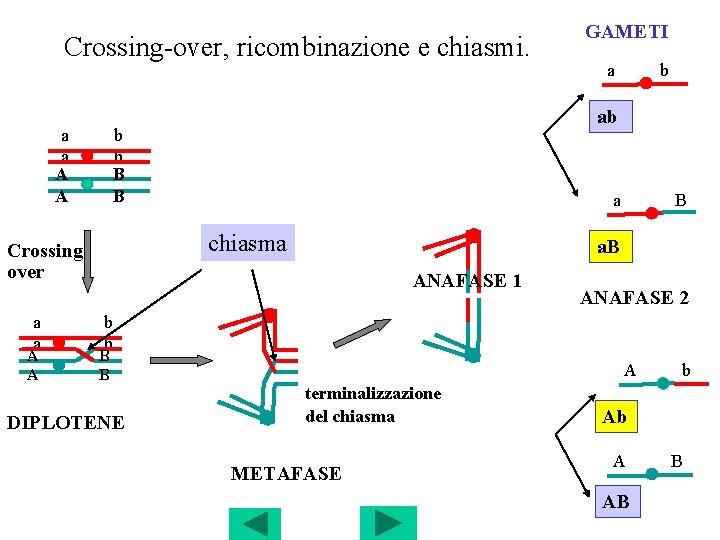
Crossing-over, ricombinazione e chiasmi. a a A A a b ab b b B B a chiasma Crossing over GAMETI B a. B ANAFASE 1 ANAFASE 2 b b B B DIPLOTENE A terminalizzazione del chiasma METAFASE b Ab A AB B

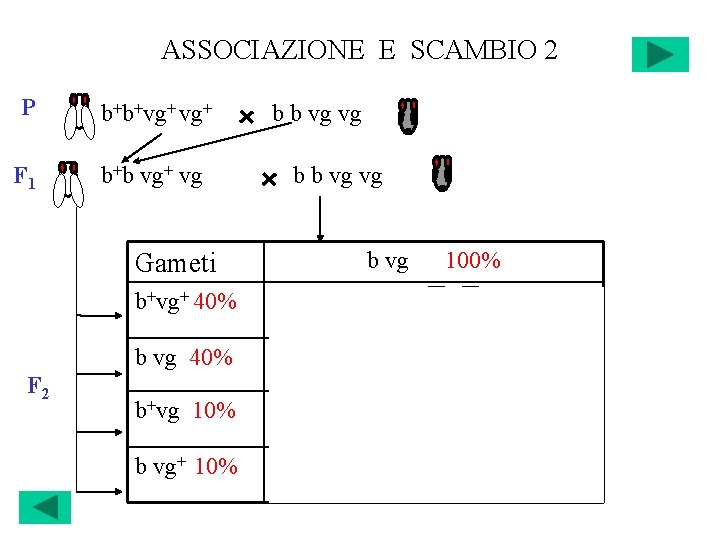
ASSOCIAZIONE E SCAMBIO 2 P b+b+vg+ F 1 b+b vg+ vg b b vg vg b vg Gameti F 2 100% b+vg+ 40% b+ b vg+ vg b vg 40% b b vg vg 20% +20% b+vg 10% b+ b vg vg 5% +5% b vg+ 10% b b vg+ vg 5% +5% 20% +20%

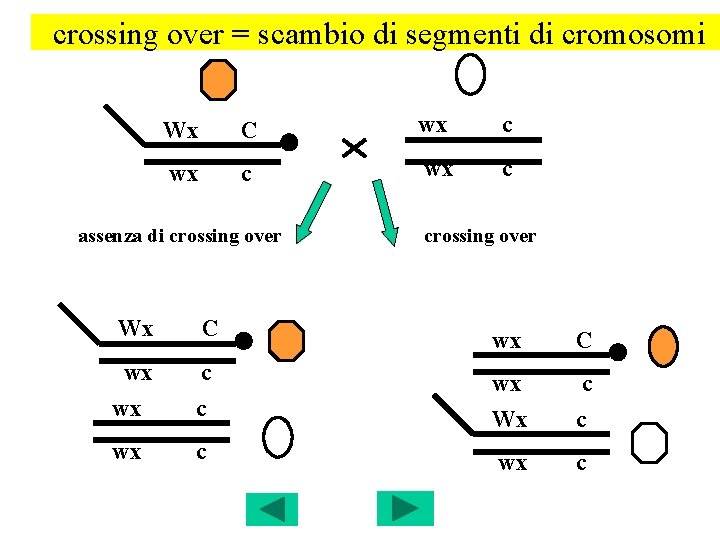
crossing over = scambio di segmenti di cromosomi Wx C wx c assenza di crossing over Wx C wx c Wx c wx c

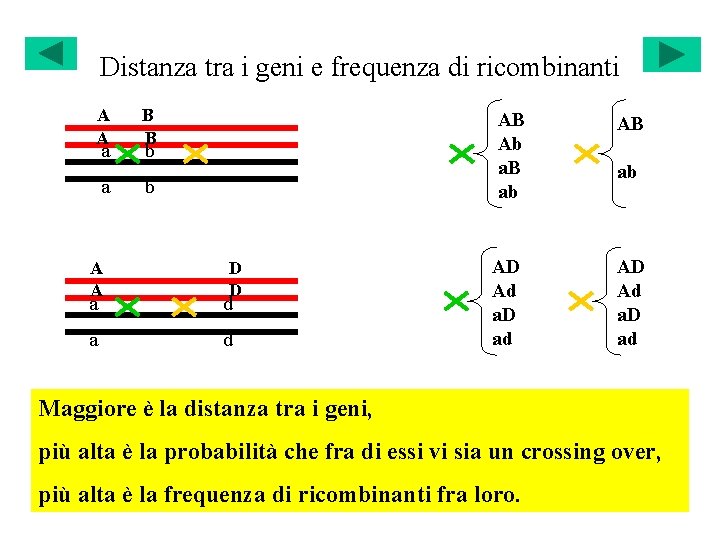
Distanza tra i geni e frequenza di ricombinanti A A a a A A B B b b D D a d AB Ab a. B ab AB AD Ad a. D ad ab Maggiore è la distanza tra i geni, più alta è la probabilità che fra di essi vi sia un crossing over, più alta è la frequenza di ricombinanti fra loro.

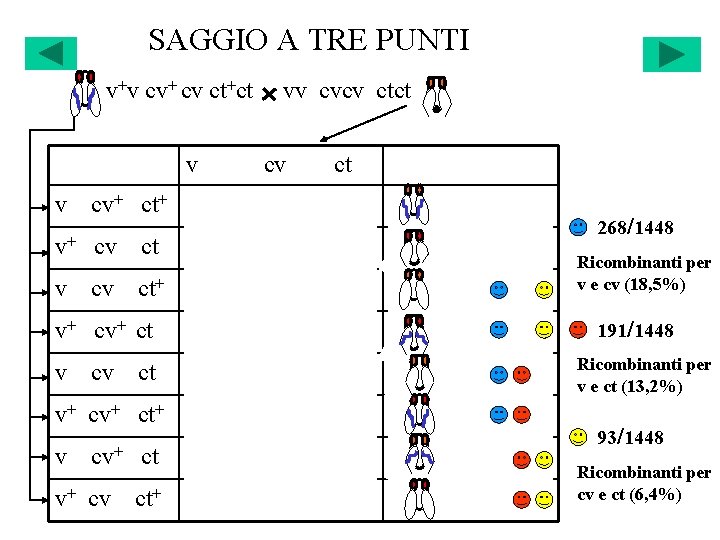
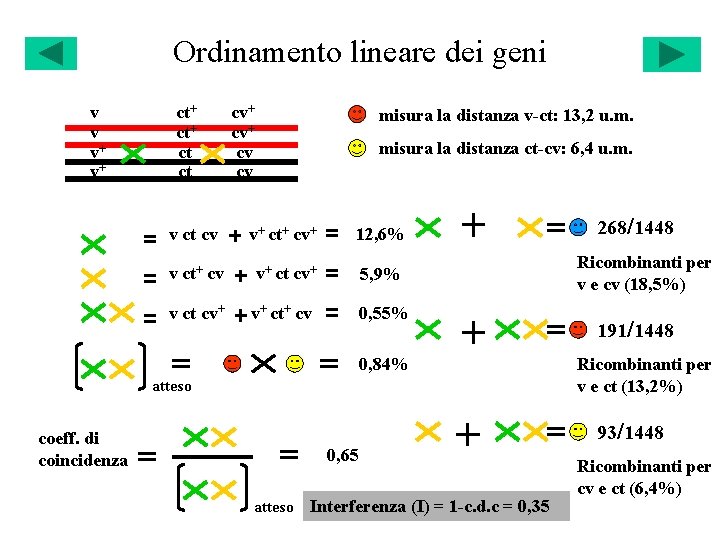
SAGGIO A TRE PUNTI v+v cv+ cv ct+ct v v cv+ ct+ vv v+ v+v cv ct+ vv vv cvcv ctct cv+cv ct+ ct 580 cvcv ct ct 592 cvcv ct+ ct 45 v+ ct v+v cv+cv ct ct 40 v vv cvcv ct ct 89 cv+cv ct+ct 94 ct ct 3 ct+ ct 5 cv ct v+ ct+ v+v v cv+ v+ cv ct vv cv+cv ct+ v+v cvcv 268/1448 Ricombinanti per v e cv (18, 5%) 191/1448 Ricombinanti per v e ct (13, 2%) 93/1448 Ricombinanti per cv e ct (6, 4%)

Ordinamento lineare dei geni v v v+ v+ ct+ ct ct cv+ cv cv misura la distanza v-ct: 13, 2 u. m. misura la distanza ct-cv: 6, 4 u. m. v ct cv v+ ct+ cv+ 12, 6% v ct+ cv v+ ct cv+ 5, 9% v ct cv+ v+ ct+ cv 0, 55% 0, 84% atteso 268/1448 Ricombinanti per v e cv (18, 5%) 191/1448 Ricombinanti per v e ct (13, 2%) 93/1448 coeff. di coincidenza 0, 65 atteso Interferenza (I) = 1 -c. d. c = 0, 35 Ricombinanti per cv e ct (6, 4%)

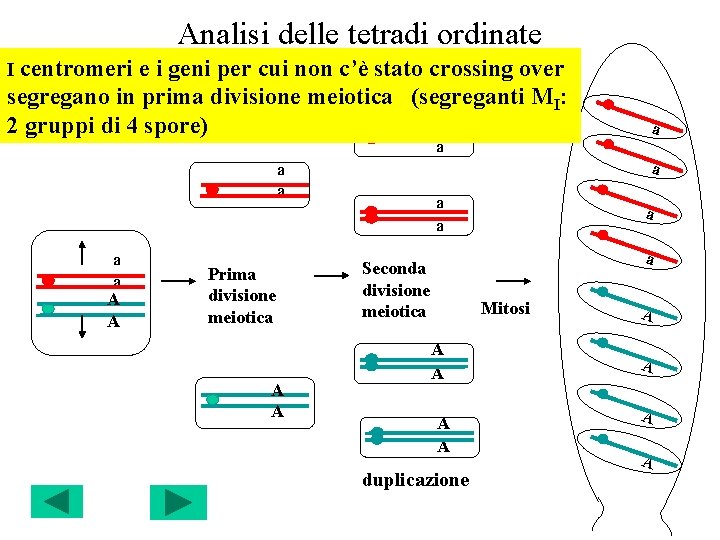
Analisi delle tetradi ordinate I centromeri e i geni per cui non c’è stato crossing over segregano in prima divisione meiotica (segreganti MI: a 2 gruppi di 4 spore) a a a A A Prima divisione meiotica A A a a a Seconda divisione meiotica Mitosi A A duplicazione A A

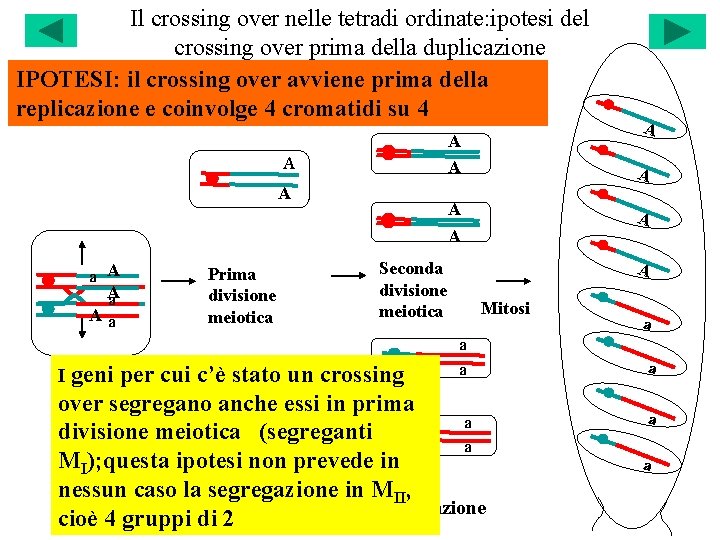
Il crossing over nelle tetradi ordinate: ipotesi del crossing over prima della duplicazione IPOTESI: il crossing over avviene prima della replicazione e coinvolge 4 cromatidi su 4 A A a Aa I geni Prima divisione meiotica A A A Seconda divisione meiotica A A A Mitosi a a per cui c’è stato aun crossing over segregano anche aessi in prima a divisione meiotica (segreganti a MI); questa ipotesi non prevede in nessun caso la segregazione in MII, duplicazione cioè 4 gruppi di 2 a a

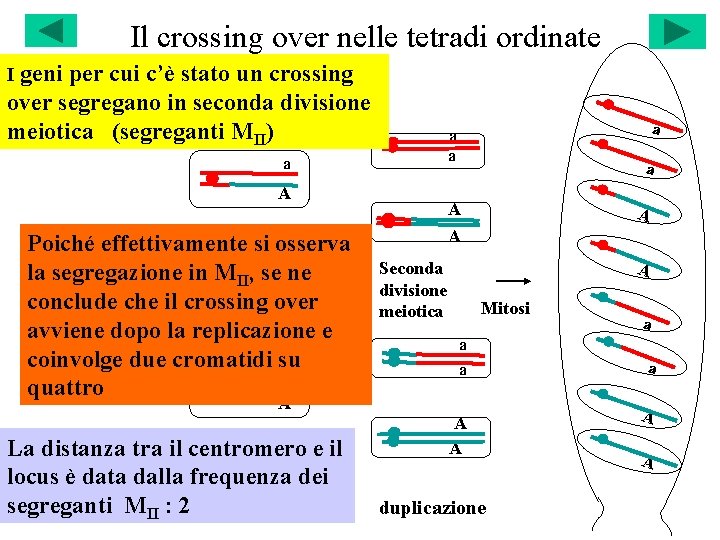
Il crossing over nelle tetradi ordinate I geni per cui c’è stato un crossing over segregano in seconda divisione meiotica (segreganti MII) a A Poiché effettivamente si osserva a la segregazione in. Prima MII, se ne a divisioneover conclude. A che il crossing meiotica A avviene dopo la replicazione e coinvolge due cromatidi su a quattro A La distanza tra il centromero e il locus è data dalla frequenza dei segreganti MII : 2 a a A A Seconda divisione meiotica A A Mitosi a a A A duplicazione a a A A

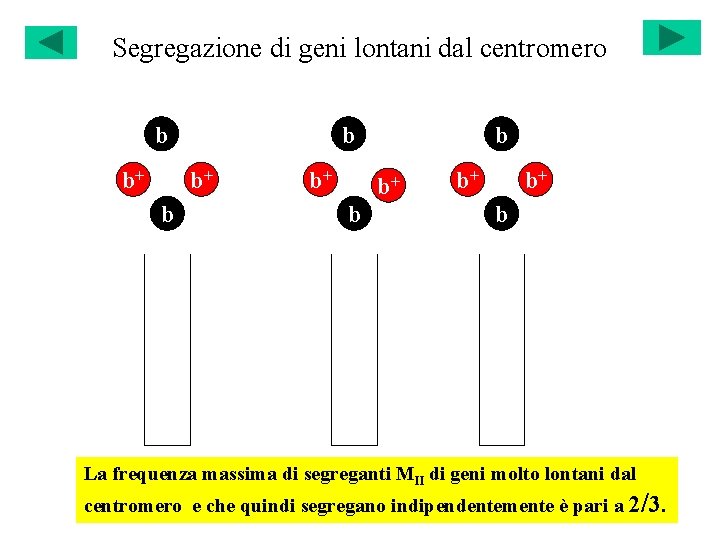
Segregazione di geni lontani dal centromero b b+ b b+ b+ b La frequenza massima di segreganti MII di geni molto lontani dal centromero e che quindi segregano indipendentemente è pari a 2/3.

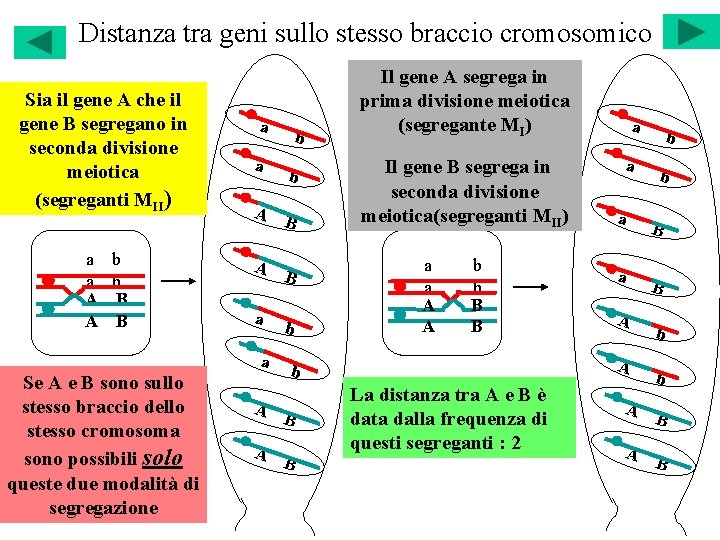
Distanza tra geni sullo stesso braccio cromosomico Sia il gene A che il gene B segregano in seconda divisione meiotica (segreganti MII) a b A B a a A A a a Se A e B sono sullo stesso braccio dello stesso cromosoma sono possibili solo queste due modalità di segregazione A A b b B B b Il gene A segrega in prima divisione meiotica (segregante MI) Il gene B segrega in seconda divisione meiotica(segreganti MII) a a A A b b B B B a a a A A b B a La distanza tra A e B è data dalla frequenza di questi segreganti : 2 A A b b B B

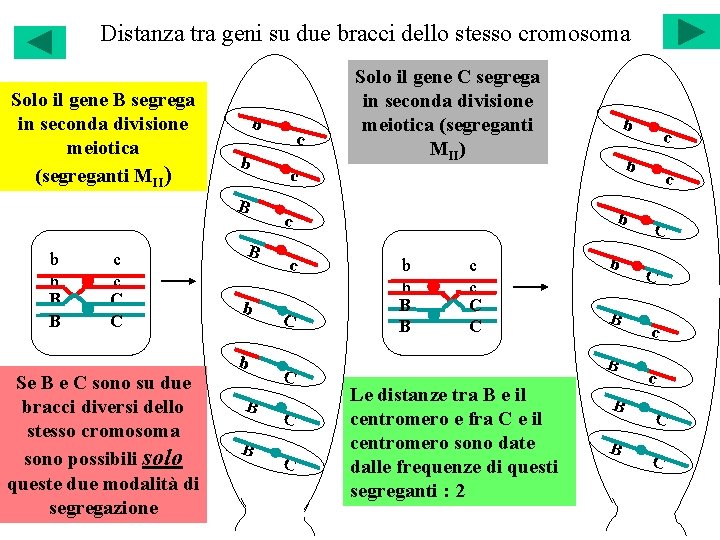
Distanza tra geni su due bracci dello stesso cromosoma Solo il gene B segrega in seconda divisione meiotica (segreganti MII) b b B B c c C C B b b Se B e C sono su due bracci diversi dello stesso cromosoma sono possibili solo queste due modalità di segregazione B B c Solo il gene C segrega in seconda divisione meiotica (segreganti MII) b b c c C C b b B B c c C C b B B Le distanze tra B e il centromero e fra C e il centromero sono date dalle frequenze di questi segreganti : 2 c B B c C C c c C C

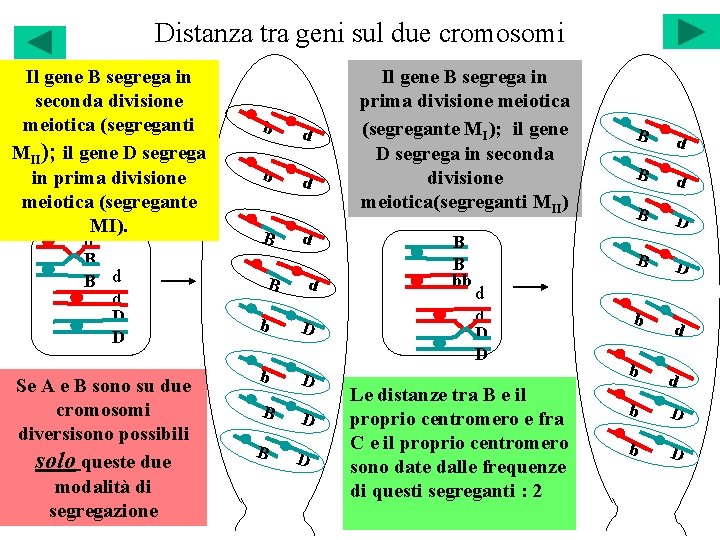
Distanza tra geni sul due cromosomi Il gene B segrega in seconda divisione meiotica (segreganti MII); il gene D segrega in prima divisione meiotica (segregante b. MI). b B B d d D D Se A e B sono su due cromosomi diversisono possibili solo queste due modalità di segregazione b d B B d d b D B D Il gene B segrega in prima divisione meiotica (segregante MI); il gene D segrega in seconda divisione meiotica(segreganti MII) B d B D B B bb B D d d D D Le distanze tra B e il proprio centromero e fra C e il proprio centromero sono date dalle frequenze di questi segreganti : 2 b b d d b D

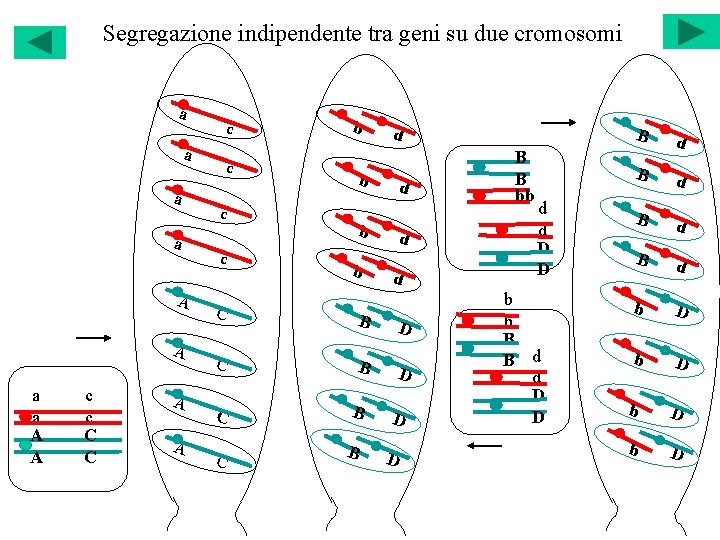
Segregazione indipendente tra geni su due cromosomi a a A A c c C C A A c c b b d d C B D C C B B D D B B bb d d D D b b B B d d D D B d B d b D b D

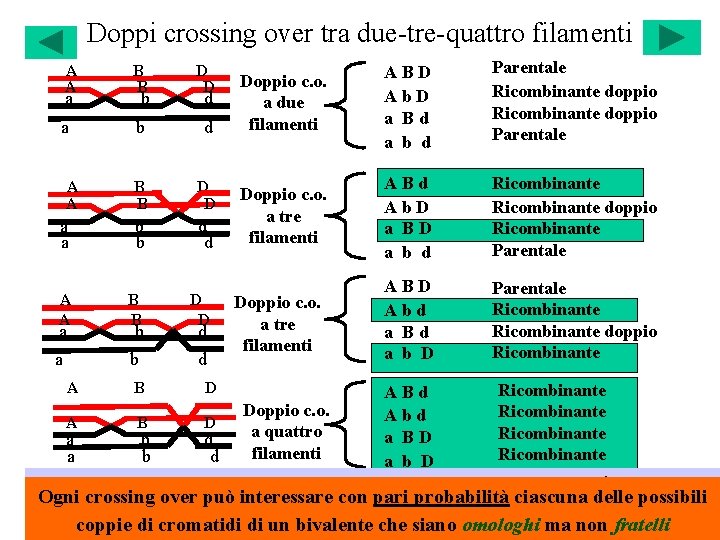
Doppi crossing over tra due-tre-quattro filamenti A A B B D D a b d B B D D a a b b d d A A B B D D a b d a A A a A b b B d d D Doppio c. o. a due filamenti ABD Ab. D a Bd a b d Parentale Ricombinante doppio Parentale Doppio c. o. a tre filamenti ABd Ab. D a BD a b d Ricombinante doppio Ricombinante Parentale ABD Abd a Bd a b D Parentale Ricombinante doppio Ricombinante Doppio c. o. a tre filamenti Ricombinante ABd Doppio c. o. Ricombinante Abd A B D a quattro Ricombinante a BD a b d filamenti Ricombinante a b d a b D Le quattro combinazioni di doppi crossing over sono equiprobabili (non c’è interferenza Ogni crossing over può interessare confra pari ciascuna delle cromatidica) la frequenza ricombinanti A eprobabilità D è del 50 %, come dopo unpossibili crossing coppie di cromatidi di un bivalente che siano omologhi ma non fratelli over singolo

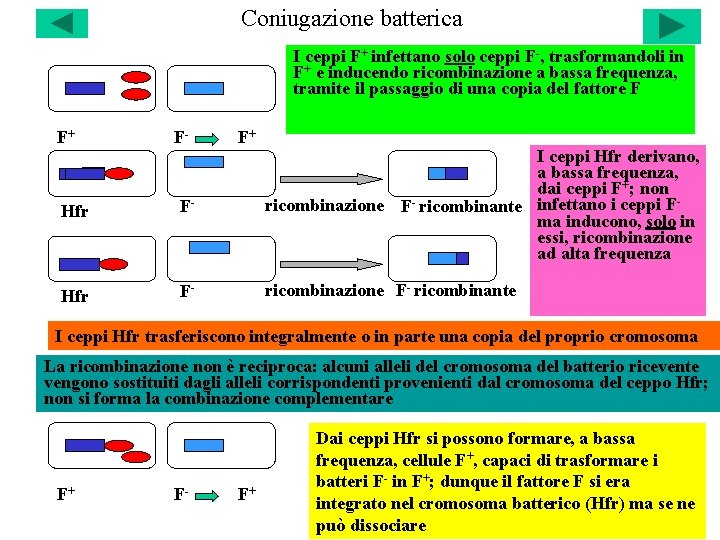
Coniugazione batterica I ceppi F+ infettano solo ceppi F-, trasformandoli in F+ e inducendo ricombinazione a bassa frequenza, tramite il passaggio di una copia del fattore F F+ F- F+ Hfr F- I ceppi Hfr derivano, a bassa frequenza, dai ceppi F+; non ricombinazione F- ricombinante infettano i ceppi Fma inducono, solo in essi, ricombinazione ad alta frequenza Hfr F- ricombinazione F- ricombinante I ceppi Hfr trasferiscono integralmente o in parte una copia del proprio cromosoma La ricombinazione non è reciproca: alcuni alleli del cromosoma del batterio ricevente vengono sostituiti dagli alleli corrispondenti provenienti dal cromosoma del ceppo Hfr; non si forma la combinazione complementare F+ F- F+ Dai ceppi Hfr si possono formare, a bassa frequenza, cellule F+, capaci di trasformare i batteri F- in F+; dunque il fattore F si era integrato nel cromosoma batterico (Hfr) ma se ne può dissociare

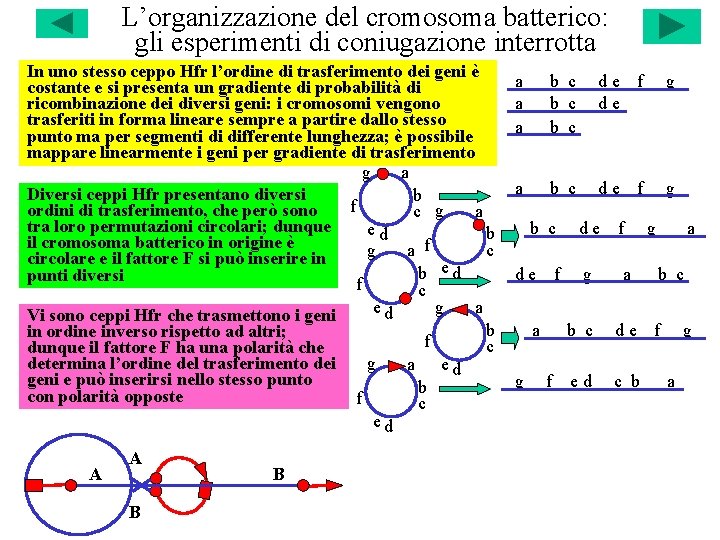
L’organizzazione del cromosoma batterico: gli esperimenti di coniugazione interrotta In uno stesso ceppo Hfr l’ordine di trasferimento dei geni è costante e si presenta un gradiente di probabilità di ricombinazione dei diversi geni: i cromosomi vengono trasferiti in forma lineare sempre a partire dallo stesso punto ma per segmenti di differente lunghezza; è possibile mappare linearmente i geni per gradiente di trasferimento g a Diversi ceppi Hfr presentano diversi b f ordini di trasferimento, che però sono a c g tra loro permutazioni circolari; dunque ed b il cromosoma batterico in origine è f c g a circolare e il fattore F si può inserire in b ed punti diversi f c g a ed Vi sono ceppi Hfr che trasmettono i geni b in ordine inverso rispetto ad altri; f c dunque il fattore F ha una polarità che determina l’ordine del trasferimento dei g a ed geni e può inserirsi nello stesso punto b con polarità opposte f c ed A A B B a a a b c b c de de f g a b c de f g b c de f a g f de f g a b c de ed c b g a b c f g a

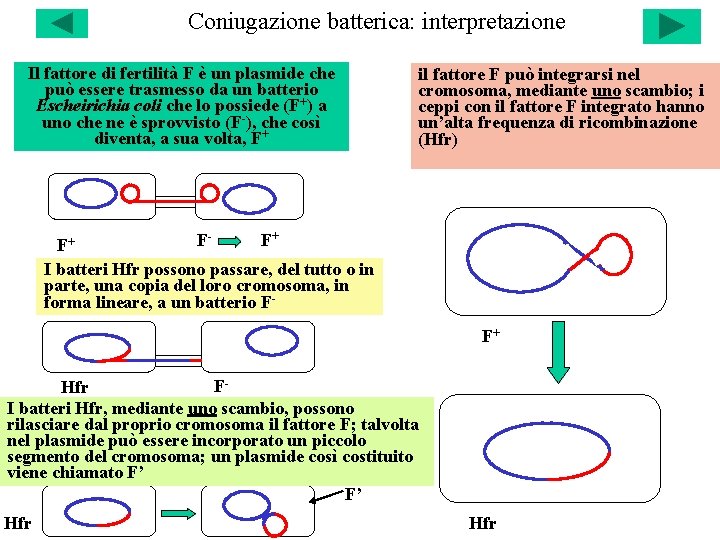
Coniugazione batterica: interpretazione Il fattore di fertilità F è un plasmide che può essere trasmesso da un batterio Escheirichia coli che lo possiede (F+) a uno che ne è sprovvisto (F-), che così diventa, a sua volta, F+ il fattore F può integrarsi nel cromosoma, mediante uno scambio; i ceppi con il fattore F integrato hanno un’alta frequenza di ricombinazione (Hfr) FF+ F+ I batteri Hfr possono passare, del tutto o in parte, una copia del loro cromosoma, in forma lineare, a un batterio FF+ FHfr I batteri Hfr, mediante uno scambio, possono rilasciare dal proprio cromosoma il fattore F; talvolta nel plasmide può essere incorporato un piccolo segmento del cromosoma; un plasmide così costituito viene chiamato F’ F’ Hfr

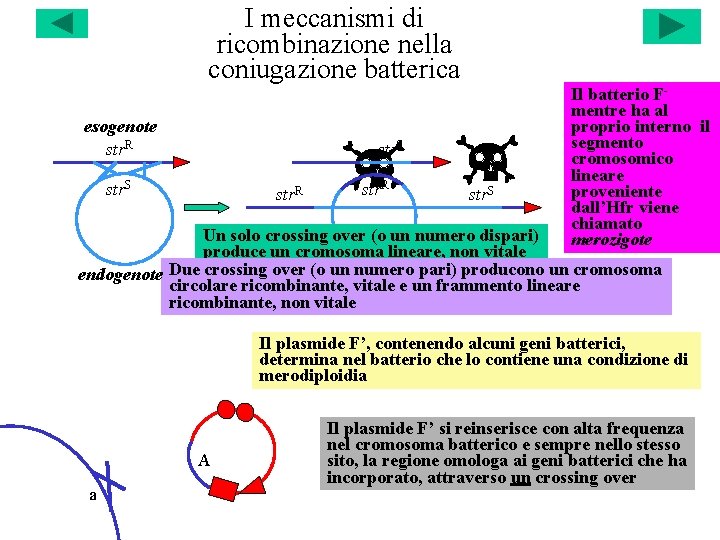
I meccanismi di ricombinazione nella coniugazione batterica esogenote str. R str. S Il batterio Fmentre ha al proprio interno il segmento cromosomico lineare proveniente dall’Hfr viene chiamato merozigote Un solo crossing over (o un numero dispari) produce un cromosoma lineare, non vitale endogenote Due crossing over (o un numero pari) producono un cromosoma circolare ricombinante, vitale e un frammento lineare ricombinante, non vitale Il plasmide F’, contenendo alcuni geni batterici, determina nel batterio che lo contiene una condizione di merodiploidia A a Il plasmide F’ si reinserisce con alta frequenza nel cromosoma batterico e sempre nello stesso sito, la regione omologa ai geni batterici che ha incorporato, attraverso un crossing over

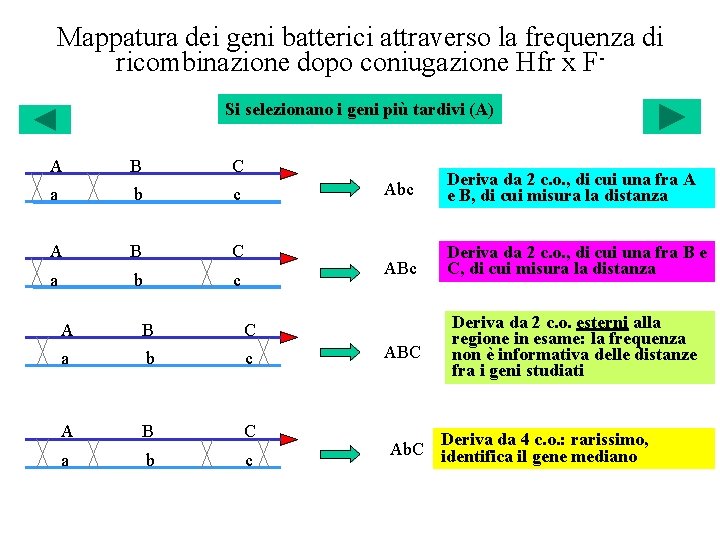
Mappatura dei geni batterici attraverso la frequenza di ricombinazione dopo coniugazione Hfr x FSi selezionano i geni più tardivi (A) A B C a b c Abc Deriva da 2 c. o. , di cui una fra A e B, di cui misura la distanza ABc Deriva da 2 c. o. , di cui una fra B e C, di cui misura la distanza ABC Deriva da 2 c. o. esterni alla regione in esame: la frequenza non è informativa delle distanze fra i geni studiati Deriva da 4 c. o. : rarissimo, Ab. C identifica il gene mediano

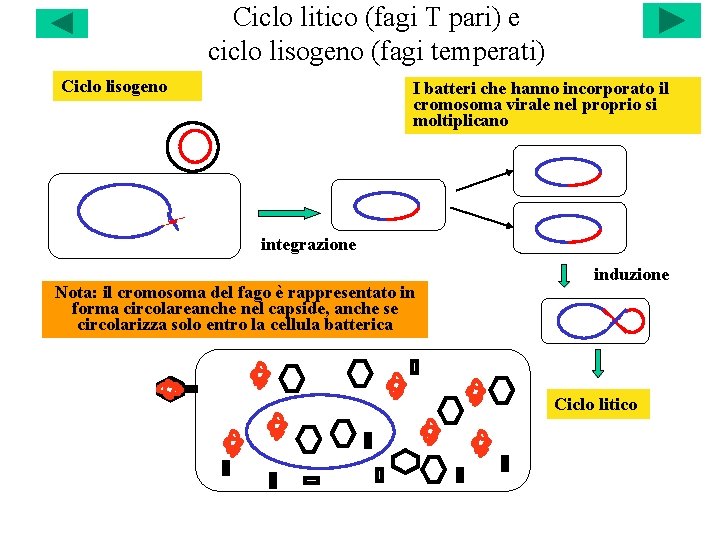
Ciclo litico (fagi T pari) e ciclo lisogeno (fagi temperati) Ciclo lisogeno I batteri che hanno incorporato il cromosoma virale nel proprio si moltiplicano integrazione Nota: il cromosoma del fago è rappresentato in forma circolareanche nel capside, anche se circolarizza solo entro la cellula batterica induzione Ciclo litico

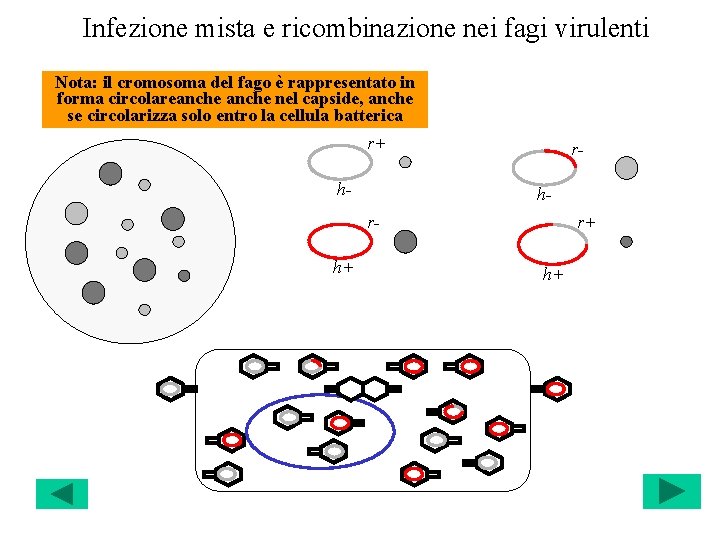
Infezione mista e ricombinazione nei fagi virulenti Nota: il cromosoma del fago è rappresentato in forma circolareanche nel capside, anche se circolarizza solo entro la cellula batterica r+ h- rhr+ rh+ h+

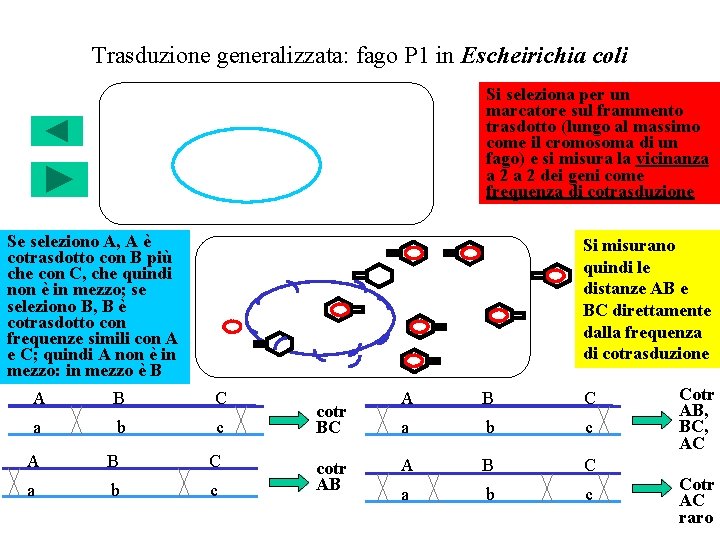
Trasduzione generalizzata: fago P 1 in Escheirichia coli Si seleziona per un marcatore sul frammento trasdotto (lungo al massimo come il cromosoma di un fago) e si misura la vicinanza a 2 dei geni come frequenza di cotrasduzione Se seleziono A, A è cotrasdotto con B più che con C, che quindi non è in mezzo; se seleziono B, B è cotrasdotto con frequenze simili con A e C; quindi A non è in mezzo: in mezzo è B Si misurano quindi le distanze AB e BC direttamente dalla frequenza di cotrasduzione A B C a b c cotr BC cotr AB A B C a b c Cotr AB, BC, AC Cotr AC raro

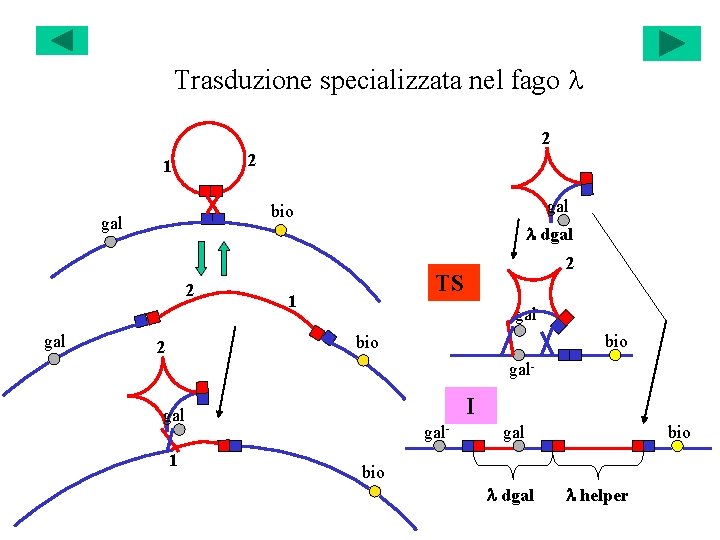
Trasduzione specializzata nel fago l 2 2 1 gal l dgal 2 gal bio 2 TS 1 gal bio 2 gal- I gal 1 gal- gal bio l dgal l helper

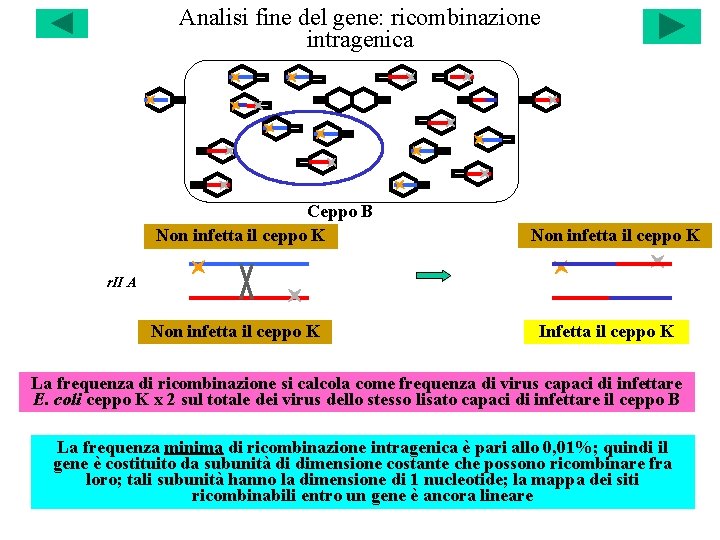
Analisi fine del gene: ricombinazione intragenica Ceppo B Non infetta il ceppo K r. II A Non infetta il ceppo K Infetta il ceppo K La frequenza di ricombinazione si calcola come frequenza di virus capaci di infettare E. coli ceppo K x 2 sul totale dei virus dello stesso lisato capaci di infettare il ceppo B La frequenza minima di ricombinazione intragenica è pari allo 0, 01%; quindi il gene è costituito da subunità di dimensione costante che possono ricombinare fra loro; tali subunità hanno la dimensione di 1 nucleotide; la mappa dei siti ricombinabili entro un gene è ancora lineare

Mappatura per delezione di siti mutabili Esistono mutazioni che non possono ricombinare con altre mutazioni nello stesso gene: si tratta di delezioni Cromosoma normale 4 3 5 1 2 6 7 Cromosoma con delezione del segmento che contiene i siti mutabili 3, 4 e 5; per questi siti non vi può essere ricombinazione con il cromosoma normale Se si dispone di un numero adeguato di delezioni si può suddividere il gene in regioni caratterizzate dalle sovrapposizioni di diverse delezioni, cui si possono assegnare i siti mutabili sulla base delle delezioni con cui non ricombinano I II III V VI VIII a b c d 1 IV Il gene è costituito da un numero molto elevato di siti mutabili; tali subunità hanno la dimensione di 1 nucleotide; la mappa dei siti mutabili entro un gene è ancora lineare; alcuni siti hanno una frequenza di mutazione molto più alta di quella attesa per caso: sono stati chiamati punti caldi e 2 3 4 5 6 7 8 9 10 11 12 È possibile mettere in sequenza le delezioni a seconda delle regioni delete che condividono, che ne inibiscono la ricombinazione Si può localizzare ogni nuova mutazione in una delle regioni in cui è suddiviso il gene sulla base delle delezioni con cui ricombinao meno
 Peso specifico latte
Peso specifico latte Titolazione dell'aceto
Titolazione dell'aceto Determinazione ferro nelle acque
Determinazione ferro nelle acque Determinazione del prezzo
Determinazione del prezzo Determinazione del prezzo
Determinazione del prezzo Determinazione del prezzo
Determinazione del prezzo Sesso a 90
Sesso a 90 Sesso cinema
Sesso cinema Spotting ciclo
Spotting ciclo Calorie orgasmo
Calorie orgasmo Un dibujo que represente
Un dibujo que represente Genes alelos
Genes alelos Permutacion cromosomica
Permutacion cromosomica Dotacion cromosomica
Dotacion cromosomica Dotacion cromosomica de las celulas hijas en la meiosis
Dotacion cromosomica de las celulas hijas en la meiosis Dotacion
Dotacion Cromosomas homologos
Cromosomas homologos Mutación cromosómica
Mutación cromosómica Simone signorelli
Simone signorelli Determinazione attività enzimatica spettrofotometro
Determinazione attività enzimatica spettrofotometro Tabella criteri di determinazione ore sostegno
Tabella criteri di determinazione ore sostegno Ecos del rocío yo soy del sur
Ecos del rocío yo soy del sur Alineacion de ejes intubacion
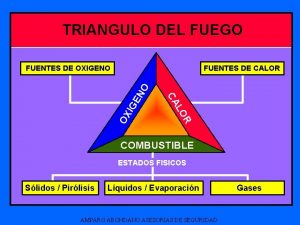
Alineacion de ejes intubacion Triangulo de fuego definicion
Triangulo de fuego definicion El calor es uno de los elementos del triángulo del fuego
El calor es uno de los elementos del triángulo del fuego Ejemplos de teatro del absurdo
Ejemplos de teatro del absurdo Funcion del sentido del tacto
Funcion del sentido del tacto Colores bandera sena
Colores bandera sena Planteamiento del problema y formulacion del problema
Planteamiento del problema y formulacion del problema Letanias del santo rosario
Letanias del santo rosario Manual de sanidad del alma
Manual de sanidad del alma Integralidad mais
Integralidad mais Principios del pacto del matrimonio
Principios del pacto del matrimonio Andalucía es mi tierra yo soy del sur
Andalucía es mi tierra yo soy del sur