CRISTALOGRAFIA DE PROTENAS Determinao da Estrutura tridimensional de










- Slides: 18

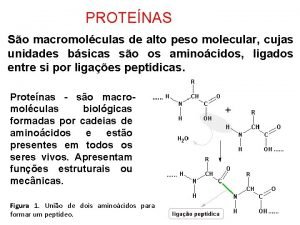
CRISTALOGRAFIA DE PROTEÍNAS Determinação da Estrutura tri-dimensional de Proteínas utilizando difração de Raios-X http: //www. cbi. cnpia. embrapa. br Paula Kuser Falcão

CRISTALOGRAFIA DE PROTEÍNAS O processo completo consiste em produzir cristais de proteínas (>0. 1 mm na sua menor dimensão), nos quais as moléculas estejam razoavelmente bem ordenadas para que o feixe de raios-X incidente sobre esse cristal seja difratado gerando um padrão de reflexões que, através da análise dos dados cristalográficos, produzam um mapa tri-dimensional de densidade eletrônica da molécula. Um modelo da proteína com sequência conhecida deve então ser modelado neste mapa. http: //www. cbi. cnpia. embrapa. br Paula Kuser Falcão

POR QUE RAIOS-X ? Adaptado de http: //www. ipac. caltech. edu/Outreach/Multiwave/ O uso de radiação eletromagnética para visualizar objetos requer que a radiação tenha um comprimento de onda comparável às menores características que desejamos resolver. Os raios-X tem um comprimento de onda de 1. 54Å. Este valor é próximo à distância entre átomos de carbono numa ligação peptídica, sendo então ideal para estudar estruturas de proteínas. http: //www. cbi. cnpia. embrapa. br Paula Kuser Falcão

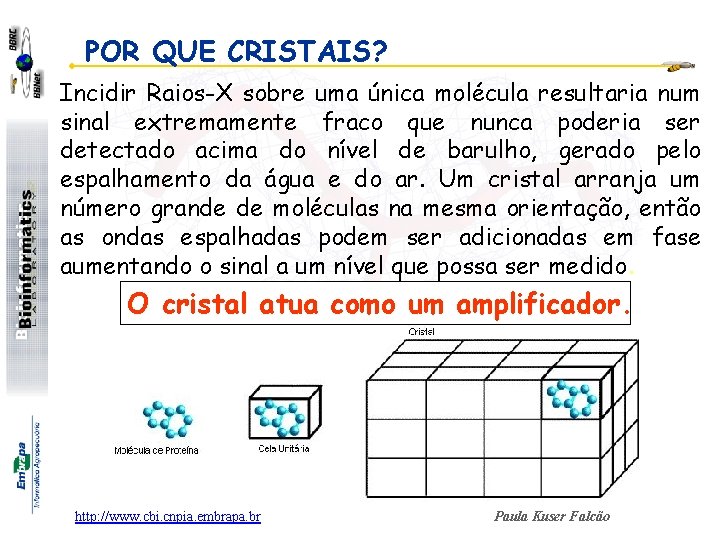
POR QUE CRISTAIS? Incidir Raios-X sobre uma única molécula resultaria num sinal extremamente fraco que nunca poderia ser detectado acima do nível de barulho, gerado pelo espalhamento da água e do ar. Um cristal arranja um número grande de moléculas na mesma orientação, então as ondas espalhadas podem ser adicionadas em fase aumentando o sinal a um nível que possa ser medido. O cristal atua como um amplificador. http: //www. cbi. cnpia. embrapa. br Paula Kuser Falcão

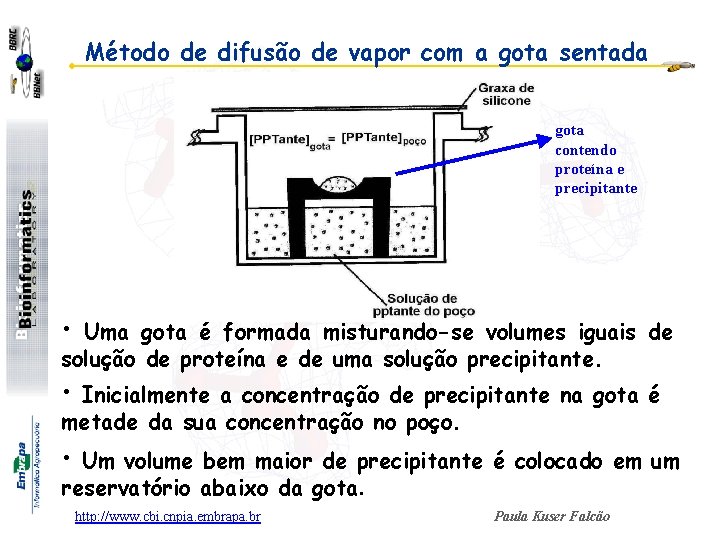
Método de difusão de vapor com a gota sentada gota contendo proteína e precipitante • Uma gota é formada misturando-se volumes iguais de solução de proteína e de uma solução precipitante. • Inicialmente a concentração de precipitante na gota é metade da sua concentração no poço. • Um volume bem maior de precipitante é colocado em um reservatório abaixo da gota. http: //www. cbi. cnpia. embrapa. br Paula Kuser Falcão

Exemplos de cristais de proteínas já estudados Proteína que liga quitina Tjaard Pijning RU Groninger Biliverdina-IX redustase complexada com NADP Rosa Perez-Luque IBMB-CSIC Barcelona Dihidrolipoamida desidrogenase Tassie & Roeber NASA GP 120 - uma glicoproteína do envelope exterior do vírus HIVtipo 1 Universidade de Columbia http: //www. cbi. cnpia. embrapa. br TIM Aparício LNLS Lisozima Eddie Snell NASA Paula Kuser Falcão

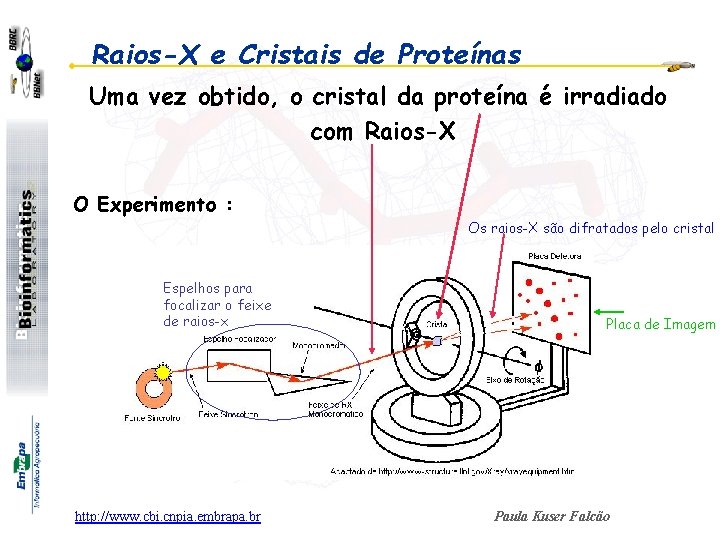
Raios-X e Cristais de Proteínas Uma vez obtido, o cristal da proteína é irradiado com Raios-X O Experimento : Os raios-X são difratados pelo cristal Espelhos para focalizar o feixe de raios-x http: //www. cbi. cnpia. embrapa. br Placa de Imagem Paula Kuser Falcão

Padrão de Difração de Raios-X ØO espalhamento do feixe de raios-X pelo cristal produz o padrão de difração; ØA organização dos pontos no padrão está relacionada com a distribuição de moléculas no cristal; Ø Diferenças de intensidade relacionam-se com a distribuição dos átomos na molécula. http: //www. cbi. cnpia. embrapa. br Paula Kuser Falcão

Mapa de densidade eletrônica O resultado do experimento não é a figura dos átomos , mas um mapa de distribuição dos elétrons na molécula, isto é, um mapa de densidade eletrônica. No entanto, uma vez que os elétrons são mais localizados ao redor do núcleo, o mapa de densidade eletrônica nos dá uma boa idéia da molécula. http: //www. cbi. cnpia. embrapa. br Paula Kuser Falcão

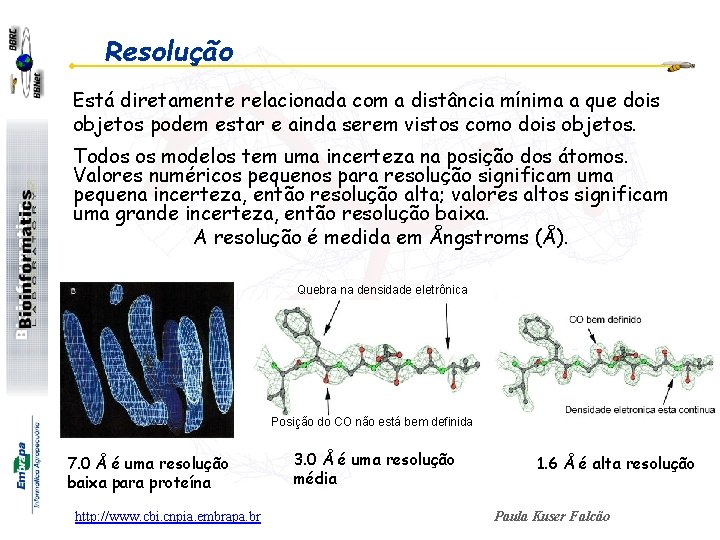
Resolução Está diretamente relacionada com a distância mínima a que dois objetos podem estar e ainda serem vistos como dois objetos. Todos os modelos tem uma incerteza na posição dos átomos. Valores numéricos pequenos para resolução significam uma pequena incerteza, então resolução alta; valores altos significam uma grande incerteza, então resolução baixa. A resolução é medida em Ångstroms (Å). Quebra na densidade eletrônica Posição do CO não está bem definida 7. 0 Å é uma resolução baixa para proteína http: //www. cbi. cnpia. embrapa. br 3. 0 Å é uma resolução média 1. 6 Å é alta resolução Paula Kuser Falcão

Ajustando o modelo na densidade eletrônica Mapa de densidade eletrônica http: //www. cbi. cnpia. embrapa. br Átomos são inseridos na densidade para construir o modelo da proteína Paula Kuser Falcão

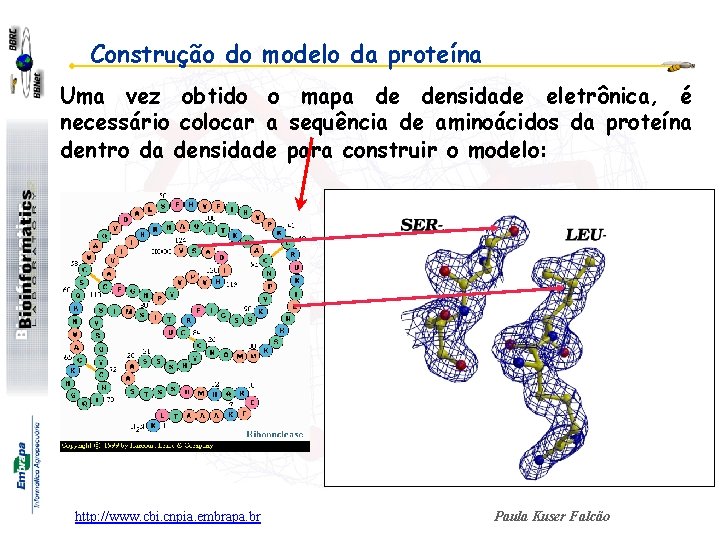
Construção do modelo da proteína Uma vez obtido o mapa de densidade eletrônica, é necessário colocar a sequência de aminoácidos da proteína dentro da densidade para construir o modelo: http: //www. cbi. cnpia. embrapa. br Paula Kuser Falcão

Análise do modelo para estudo da função da proteína Uma vez obtida a estrutura tri-dimensional da proteína podemos utilizá-la para: entender seu funcionamento. . . http: //www. cbi. cnpia. embrapa. br Paula Kuser Falcão

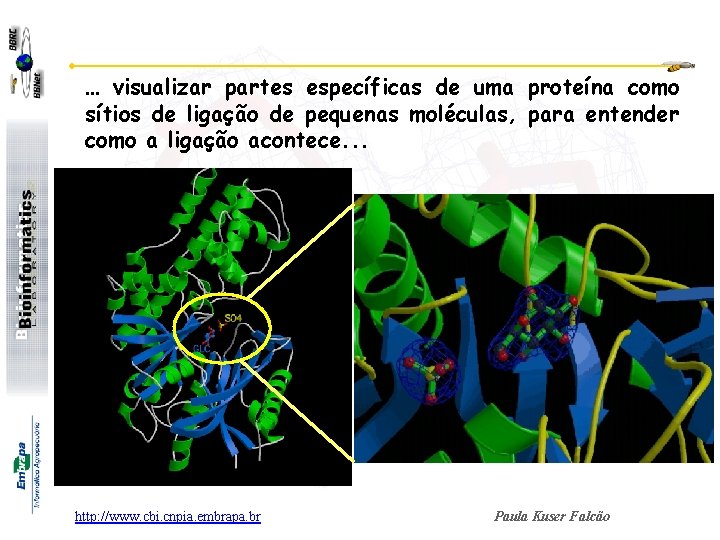
… visualizar partes específicas de uma proteína como sítios de ligação de pequenas moléculas, para entender como a ligação acontece. . . http: //www. cbi. cnpia. embrapa. br Paula Kuser Falcão

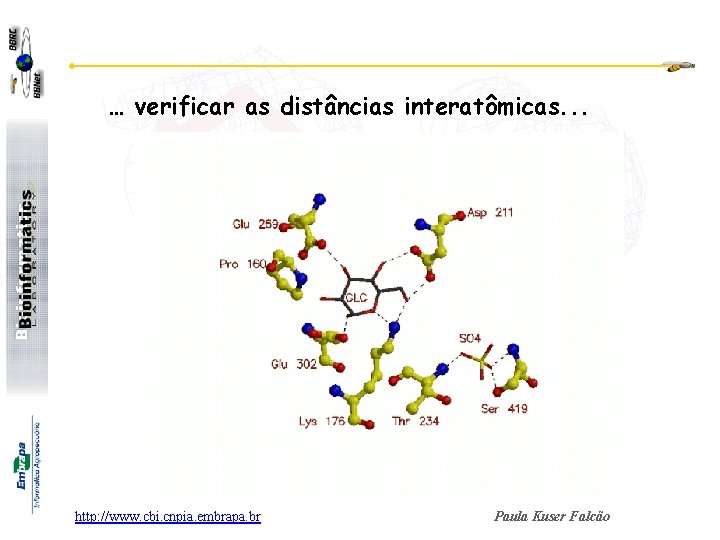
… verificar as distâncias interatômicas. . . http: //www. cbi. cnpia. embrapa. br Paula Kuser Falcão

… fazer comparações entre estruturas de proteínas, . . . http: //www. cbi. cnpia. embrapa. br Paula Kuser Falcão

… visualizar a distribuição de cargas na superfície da proteína, etc. Vermelho: cargas negativas Azul: cargas positivas http: //www. cbi. cnpia. embrapa. br Paula Kuser Falcão

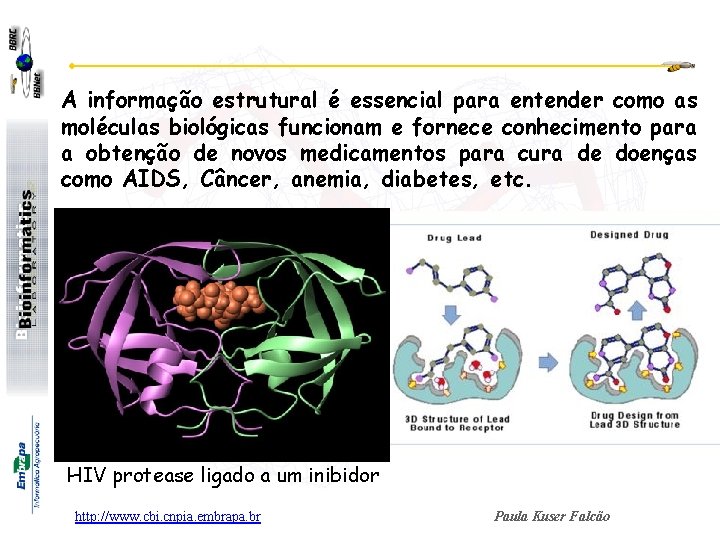
A informação estrutural é essencial para entender como as moléculas biológicas funcionam e fornece conhecimento para a obtenção de novos medicamentos para cura de doenças como AIDS, Câncer, anemia, diabetes, etc. HIV protease ligado a um inibidor http: //www. cbi. cnpia. embrapa. br Paula Kuser Falcão