Chapter 8 SDSPAGE Analysis and Molecular Weight Determination












![RESULT [Fig. 1] [Fig. 2] Rf = migration distance of the protein/ migration distance RESULT [Fig. 1] [Fig. 2] Rf = migration distance of the protein/ migration distance](https://slidetodoc.com/presentation_image_h/b7ab05211a88a5222d9c962cbda5afdf/image-16.jpg)





- Slides: 24

Chapter 8. SDS-PAGE Analysis and Molecular Weight Determination 담당조교 : 김보은(S 329), 오혜경(S 338)

Part 1. SDS-PAGE Analysis

Gel electrophoresis • 전기영동(Electrophoresis)이란 a charged molecule이 전기장 (electric field)에서 +극 또는 –극 쪽으로 이동하는 현상. • A technique used for the separation of DNA(deoxyribonucleic acid), RNA(ribonucleic acid), or protein molecules using an electric field applied to a gel matrix.

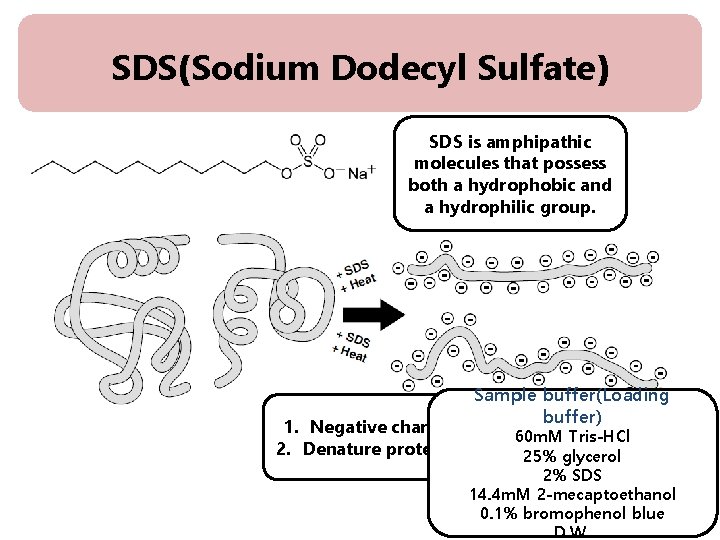
SDS(Sodium Dodecyl Sulfate) SDS is amphipathic molecules that possess both a hydrophobic and a hydrophilic group. 1. Negative charge 2. Denature proteins Sample buffer(Loading buffer) 60 m. M Tris-HCl 25% glycerol 2% SDS 14. 4 m. M 2 -mecaptoethanol 0. 1% bromophenol blue D. W.

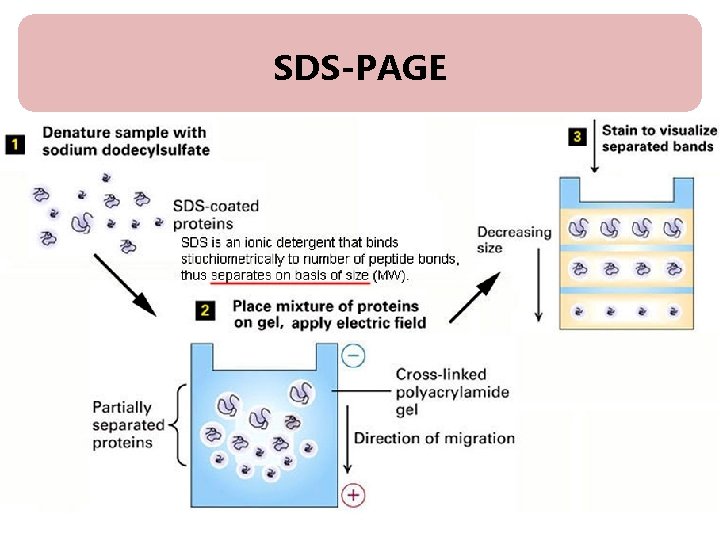
SDS-PAGE

Polyacrylamide Gels (PAG) Polyacrylamide gel은 거르는 체 역할을 하여, molecule을 charge나 weight 따라서 분리. * Advantage of polyacrylamide gels 1) DNA, RNA 그리고 protein의 분리 수행 2) Gel의 농도와 조성을 변화시켜 목적에 따른 분리가 용이 * The Starting Materials Crosslinking agents Ammonium persulfate (APS) : “initiator” of the free radical polymerization TEMED (tetramethylenediamine) : “catalyst” of the gel formation reaction

The Polymerization Reaction • • Ammonium persulfate forms radicals in solution These radicals react with acrylamide to form acrylamide radicals Acrylamide radicals react with acrylamide to form long polymer chains (No branch) Solutions of this polymer are viscous but do not form gels The polymers must be crosslinked to form a gel This is accomplished by polymerizing in the presence of a “crosslinker”N, N’-methylene-bis (acrylamide) The product is a large network of linked acrylamide chains

Varying the Size of the Net • The size of the pores in the acrylamide net depends on two parameters: – the amount of acrylamide per volume – the degree of crosslinking • Acrylamide와 bis-acrylamide의 %중량과 crosslinker인 bis-acrylamide의 %중량이 커지면 그만큼 미세통로의 크기는 작아지게 되어 작은 물질의 분 리에 이용할 수 있게 됨. Acrylamide concentration(%) Protein size (kd) 15. 0 15~45 12. 0 15~60 10. 0 18~75 7. 5 30~100 5. 0 60~212 *p 53 -His size : 53~55 KDa

Composition Running gel (10%) : 10 ml Stacking gel (5%) : 4 ml H 2 O 4 ml 2. 7 ml 30% acrylamide/bis mix 3. 3 ml 0. 67 ml 1. 5 M Tris (p. H 8. 8) 2. 5 ml 0. 5 ml 1 M Tris (p. H 6. 8) 10% SDS 0. 1 ml 0. 04 ml 10% ammonium peroxidisulfate solution(APS) 0. 1 ml 0. 04 ml TEMED 0. 01 ml 0. 008 ml Stacking gel 2 ml Running gel 4. 5 ml

SDS-PAGE : Procedure 1. SDS-PAGE 조성에 따라 각solution을 섞는다. 우선 Running gel을 만든다. (APS와 TEMED는mixture를 붓기 전에 넣는다. ) 2. Running gel을 적정선까지 넣고, 증류수로 채워준다. 3. Running gel이 굳고나면, Stacking gel을 만든다. 증류수를 제거하고 Stacking gel을 부어준다. 4. Comb을 꽂고 stacking gel을 굳힌다. 5. Comb을 제거하고, gel을 electrophoresis kit에 casting하고 running buffer 를 채워준다. 6. size-marker와 sample을 loading하고, 전기영동 한다. 7. Gel running이 끝나면, caster에서 gel만 분리하여 CBB로 staining한다. 8. Destaining solution으로 destaining하여 band만 나타나게 한다. 9. Gel 사진을 찍는다.

10% running gel 1. SDS-PAGE 조성에 따라 Running gel solution을 섞는다. (APS와 TEMED는 mixture를 붓기 전에 넣는다. ) 2. Running gel을 적정선까지 넣고, 증류수로 채워준다 Running gel 4. 5 ml

stacking gel 3. Running gel이 굳고나면, Stacking gel solution을 섞는다. 4. 물을 제거하고 stacking gel을 부 어준다. (APS와 TEMED는 mixture를 붓기 전에 넣는다. ) 5. Comb을 꽂고 stacking gel을 굳힌다. Stacking gel 2 ml Running gel 4. 5 ml

6. Comb을 제거하고, gel을 electrophoresis kit에 casting한다. Protein Sample 100μg Lysate : 7. 2μl pc. DNA plasmid: 5. 9μl pc. DNA-p 53 -His: 6. 4μl Marker: 5μl 7. running buffer를 채워준다. 8. size-marker와 sample을 loading하고, 전기영동 한다.

9. Gel running이 끝 나면, caster에서 gel만 분리하여 CBB로 staining 한다. 10. Destaining solution으로 destaining하여 band만 나타나게 한다. Staining SDS-polyacrylamide gels with Coomassie brilliant blue(CBB) : PAGE에 의해 분리된 protein은 CBB나 silver salt로 staining하여 detection한 다. CBB는 gel이 아닌 protein에 nonspecific binding을 하여 blue band 로 나타난다. CBB는 aminotriarylmethanedye이며, protein과 covalent complex가 아닌 주 로 van der Waals force, electrostatic interaction을 NH 3+ group과 강하게 형 성한다. Dye uptake는 protein 양에 비례한다.

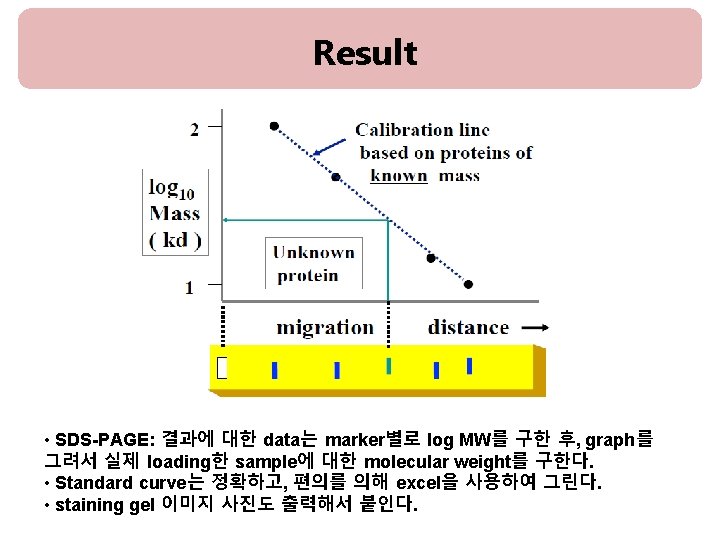
![RESULT Fig 1 Fig 2 Rf migration distance of the protein migration distance RESULT [Fig. 1] [Fig. 2] Rf = migration distance of the protein/ migration distance](https://slidetodoc.com/presentation_image_h/b7ab05211a88a5222d9c962cbda5afdf/image-16.jpg)
RESULT [Fig. 1] [Fig. 2] Rf = migration distance of the protein/ migration distance of the dye front

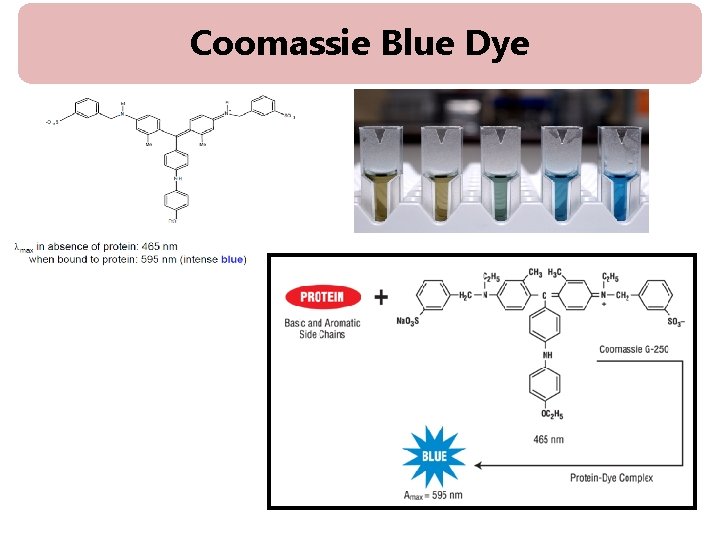
Part 2. Bradford Assay Coomassie Blue Dye

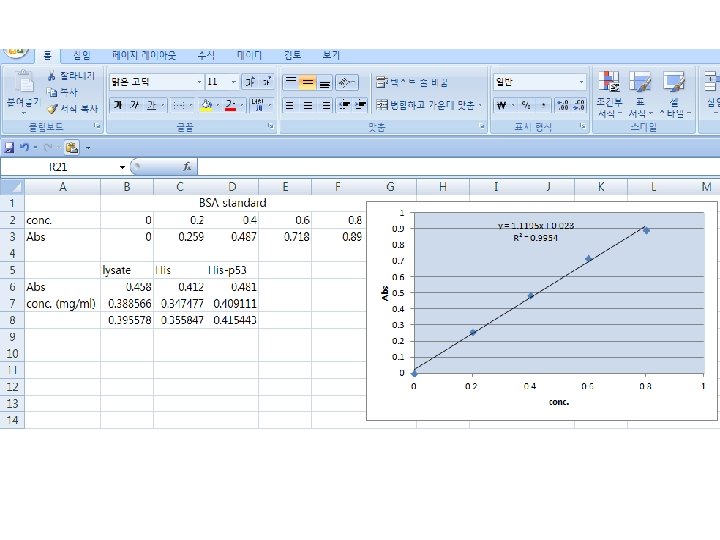
• Bradford assay란? - UV-visible spectrophotometer를 이용, 흡광도를 측정하여 standard물질인 BSA(bovine serum albumin)나 BGG(Bovine gamma globulin)를 기준으로 자신의 시 료에 단백질이 얼마만큼 들어있는지 측정하는 방법 중 하나. 신속, 간편하게 단백질 정 량 가능하여 널리 사용. Dye로는 Coomassie Blue G-250를 사용하며, 이 dye가 단백질과 결합해 생기는 파장 의 변화 측정. • A max of CB G-250 shifts from 465 to 595 nm when bound to protein - dye reacts primarily with Arg - lesser extent with His, Lys, Tyr, Trp, Phe • • sensitivity is 1 -100 mg/ml depending on circumstances single step and few interfering substances protein concentration extrapolated from standard curve sample not recoverable

Coomassie Blue Dye

Procedure 1. Warm up the spectrophotometer 15 min. before use. 2. Dilute samples with buffer to an estimated concentration. 3. Prepare standards containing a range of 5 to 20 mg/ml protein to a standard volume 1 ml. 4. Add 1 ml dye reagent. 5. Measure the absorbance at 595 nm. 0 BSA (2 mg/ml) D. W 100μl 0. 2 mg/ ml 0. 4 mg/ ml 0. 6 mg/ ml 0. 8 mg/ ml 10μl 20μl 30μl 40μl 90μl 80μl 70μl 60μl 10μl Bradford dye 1 ml


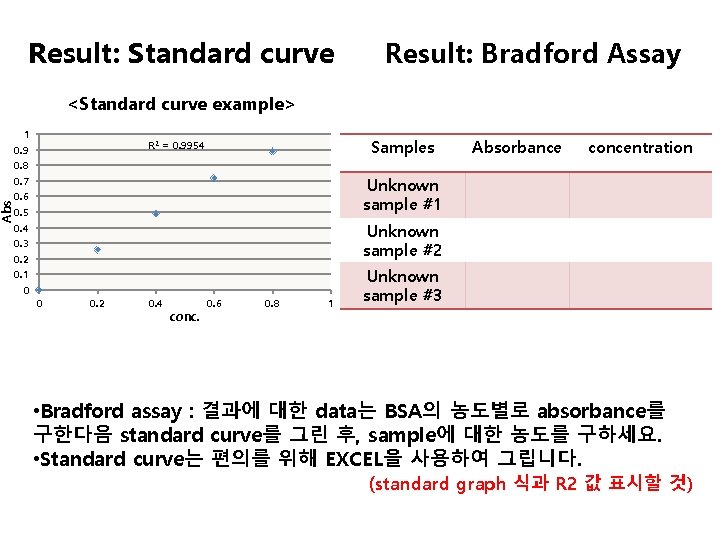
Abs Result: Standard curve Result: Bradford Assay <Standard curve example> 1 0. 9 0. 8 0. 7 0. 6 0. 5 0. 4 0. 3 0. 2 0. 1 0 Samples R 2 = 0. 9954 Absorbance concentration Unknown sample #1 Unknown sample #2 0 0. 2 0. 4 conc. 0. 6 0. 8 1 Unknown sample #3 • Bradford assay : 결과에 대한 data는 BSA의 농도별로 absorbance를 구한다음 standard curve를 그린 후, sample에 대한 농도를 구하세요. • Standard curve는 편의를 위해 EXCEL을 사용하여 그립니다. (standard graph 식과 R 2 값 표시할 것)

Further Study • Protein Quantification : Lowry (copper and Folin-Ciocalteau. Phenol) : Bicinchoninicacid method : Bradford method (Coomassie brilliant blue G 250) : Biuretmethod (copper and tartrate) → 원리와 방법에 대한 장점 및 단점
