Marcadores moleculares PCR RAPD Profa Dra Sandra Regina

















- Slides: 21

Marcadores moleculares PCR RAPD • Profa. Dra. Sandra Regina Ceccato Antonini

Marcador molecular é todo e qualquer fenótipo molecular oriundo de um gene expresso, como no caso das isoenzimas, ou de um segmento específico de DNA

Diversas técnicas de biologia molecular estão hoje disponíveis para a detecção de variabilidade genética ao nível de sequência de DNA, ou seja, para a detecção de polimorfismo genético.

A tecnologia de DNA recombinante e o desenvolvimento da amplificação de segmentos de DNA via PCR abriram o caminho para uma mudança no paradigma genético: da inferência do genótipo a partir do fenótipo, para a análise genética direta da variação na sequência de DNA

PCR Polymerase Chain Reaction Reação de polimerização em cadeia ou Polimerização em cadeia do DNA PCR - consiste em fazer cópias de DNA “in vitro”, usando os elementos básicos do processo de replicação natural do DNA.

PCR se constitui hoje no método de rotina para isolar rapidamente sequências específicas a partir de uma mistura complexa de seqüências genômicas ou de c. DNAs.

1984 - PCR foi apresentada pela primeira em um encontro científico Publicação: Mullis and Faloona, 1987 Saiki at al. 1988 1976: Thomas D. Brock descobriu a DNA polimerase termoestável de Thermus aquaticus. 1988: Esta DNA polimerase foi usada por Saiki para fazer PCR.

Thomas D. Brock no lago do “Yellowstone National Park”

PCR requer 7 componentes essenciais: - DNA polimerase termoestável - Um par de oligonucleotídeos para iniciar a síntese de DNA - Deoxinucleosídeos trifosfato (d. NTPs) - Cátion divalente : toda DNA polimerase requer cátion divalente, usualmente Mg 2+ - Tampão para manter o p. H - Cátions monovalentes, usualmente K+ (KCl) - DNA molde

Virtualmente, qualquer seqüência alvo de DNA pode ser amplificada por PCR A localização da seqüência alvo é feita pelos “primers”. Oligonucleotídeos com 20 “mers” é usualmente seletivo o suficiente para localizar um sítio único em um genoma de alta complexidade, tal como o genoma humano. O pareamento dos “primers” com a seqüência alvo na template se faz pelo estabelecimento de pontes de hidrogênio, segundo o pareamento convencional descrito por Watson e Crick: A T C G

Especificidade dos iniciadores: É possível calcular de ocorrência aleatória da seqüência: n = 2 NK n = número de sítios complementares ao iniciador N = tamanho do genoma (humano: 3 x 109) K = [g/2]G+C x [1 -g]A+T g conteúdo relativo de G+C Este cálculo mostra que um oligonucleotídeo de 15 nts está representado uma única vez no genoma. Devido às seqüências repetidas e às famílias de proteínas, menos de 85% do genoma de mamíferos pode ser encontrado com precisão, mesmo com iniciadores de 20 nts ou mais.

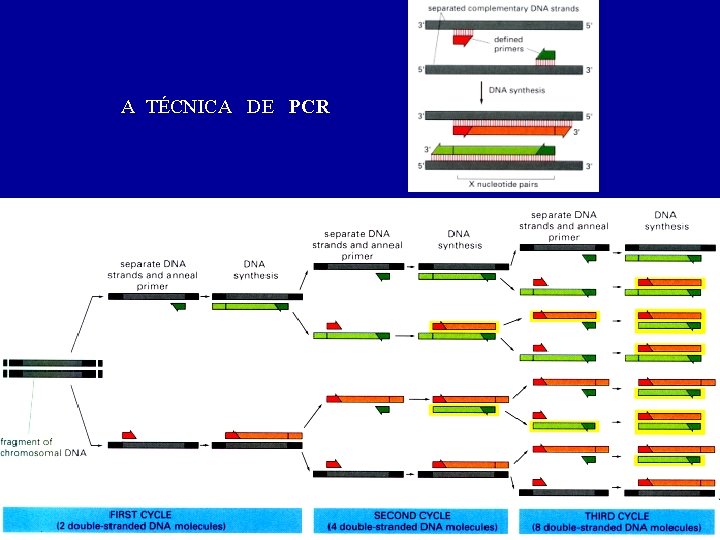
PCR é um processo iterativo, consistindo de três elementos: - Desnaturação da template por aquecimento - Pareamento dos iníciadores à seqüência alvo fita única - Extensão dos iniciadores pela DNA polimerase termoestável

A TÉCNICA DE PCR



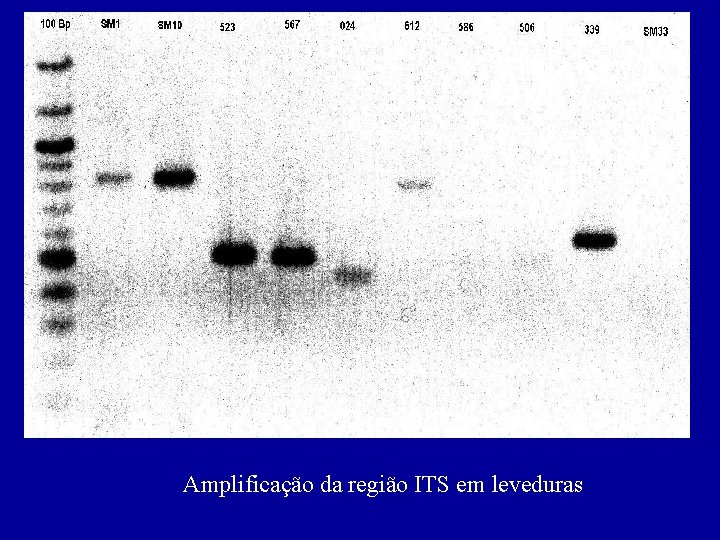
Amplificação da região ITS em leveduras

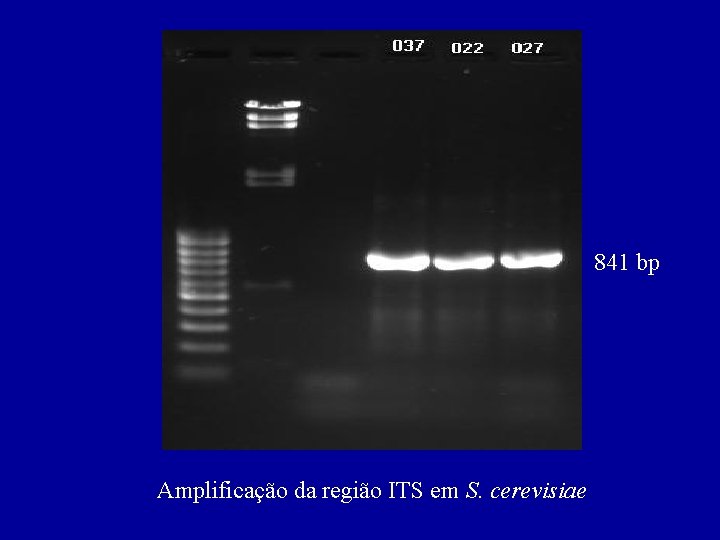
841 bp Amplificação da região ITS em S. cerevisiae

RAPD Random Amplified Polymorphic DNA É uma variação do protocolo de PCR, com duas características distintivas: utiliza um primer único, e o primer tem sequência arbitrária, isto é, sua sequência alvo é desconhecida

Aplicações do RAPD: • Obtenção de ´fingerprints´ genômicos de indivíduos, variedades e populações; • Análise da estrutura e diversidade genética em populações naturais, de melhoramento e bancos de germoplasma; • Estabelecimento de relacionamentos filogenéticos em diferentes espécies; • Construção de mapas genéticos e localização de genes de interesse econômico.

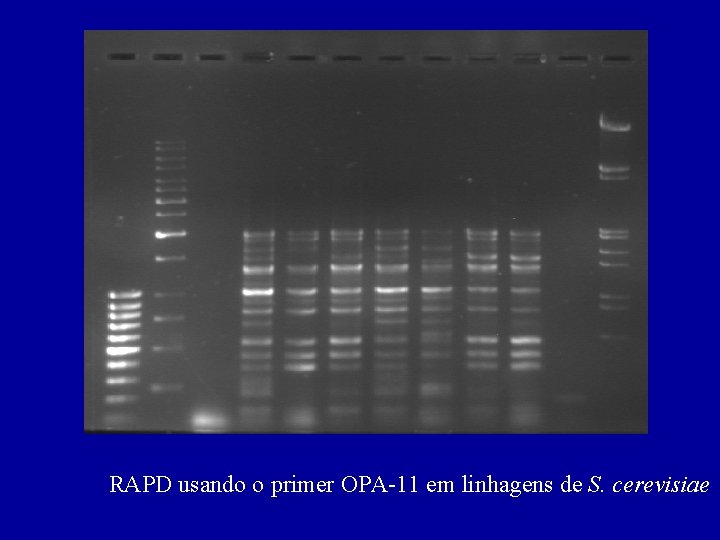
RAPD usando o primer OPA-11 em linhagens de S. cerevisiae

RAPD de 3 linhagens de S. cerevisiae
 Dra iva brito
Dra iva brito Grupo funcional nitrogenado
Grupo funcional nitrogenado Internet profa
Internet profa Valor de verdad
Valor de verdad Analizador de sector magnetico
Analizador de sector magnetico Basta de multiplicaciones
Basta de multiplicaciones Profa ma
Profa ma Momento resultante
Momento resultante Profa campis
Profa campis Internet profa
Internet profa Www.profa
Www.profa Diagrama do orbital molecular
Diagrama do orbital molecular Difference between rflp and rapd
Difference between rflp and rapd Restriction fragment length polymorphism
Restriction fragment length polymorphism Professor helen danesh-meyer
Professor helen danesh-meyer Similar images
Similar images Papilloedema fundoscopy
Papilloedema fundoscopy Tipos de debreagem
Tipos de debreagem Marcadores sericos
Marcadores sericos Texto expositivos
Texto expositivos Dr jose wilson zangirolami
Dr jose wilson zangirolami Que es la coherencia en un texto
Que es la coherencia en un texto