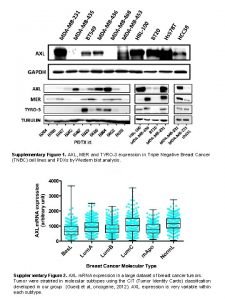
Flersekvenssammenstillinger vil alltid vre mer informative enn parvise















![Neighbour joining Grenlengdene S(AU) og S(BU) beregnes så: S(AU) =d(AB) / 2 + [r(A)-r(B)] Neighbour joining Grenlengdene S(AU) og S(BU) beregnes så: S(AU) =d(AB) / 2 + [r(A)-r(B)]](https://slidetodoc.com/presentation_image_h2/daf5aa32d5a5284057a797d5886f4a02/image-25.jpg)








- Slides: 40

Flersekvenssammenstillinger vil alltid være mer informative enn parvise sammenstillinger: Hva er konservert, hvor er gapene?

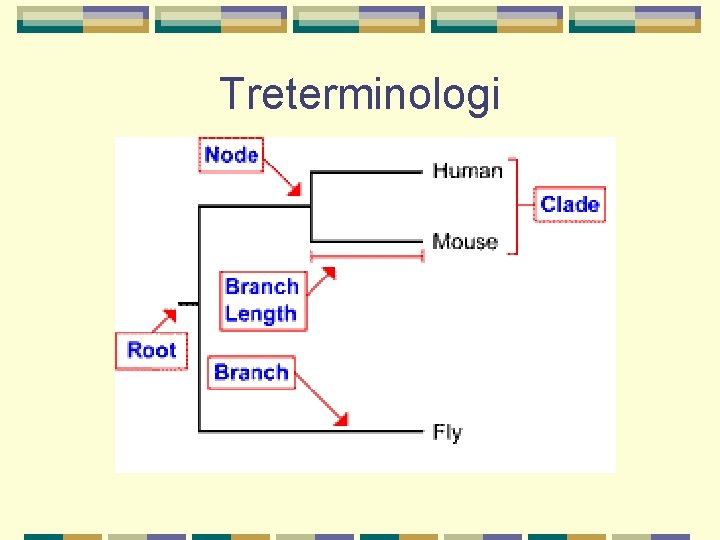
Treterminologi

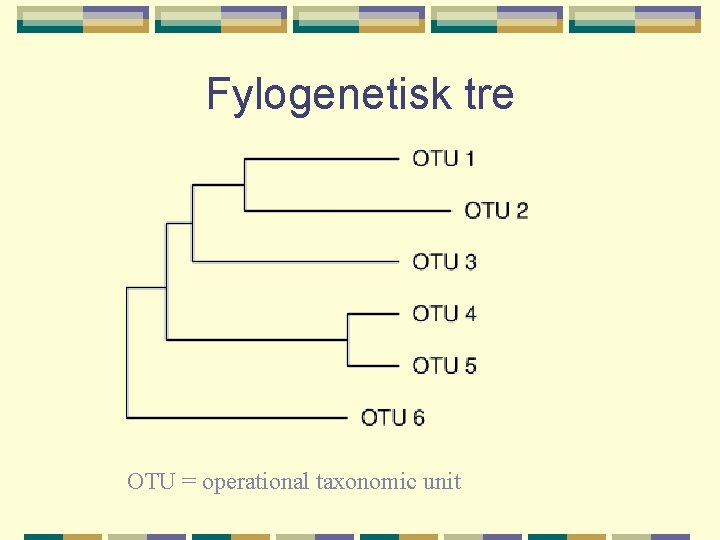
Fylogenetisk tre OTU = operational taxonomic unit

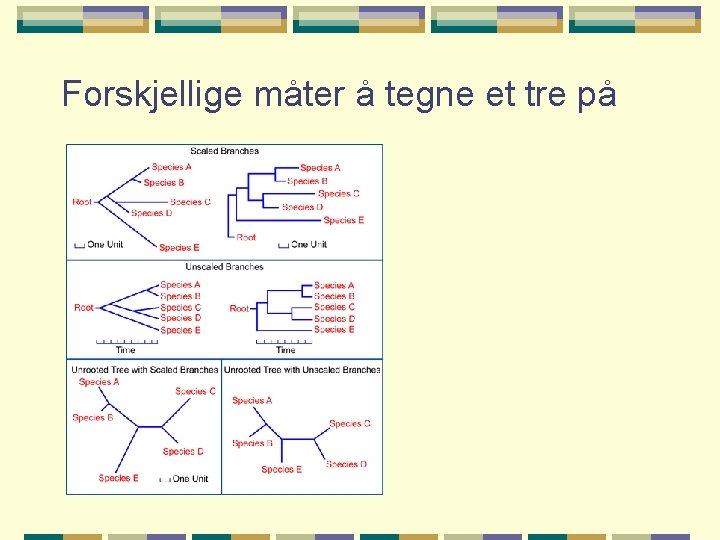
Forskjellige måter å tegne et tre på

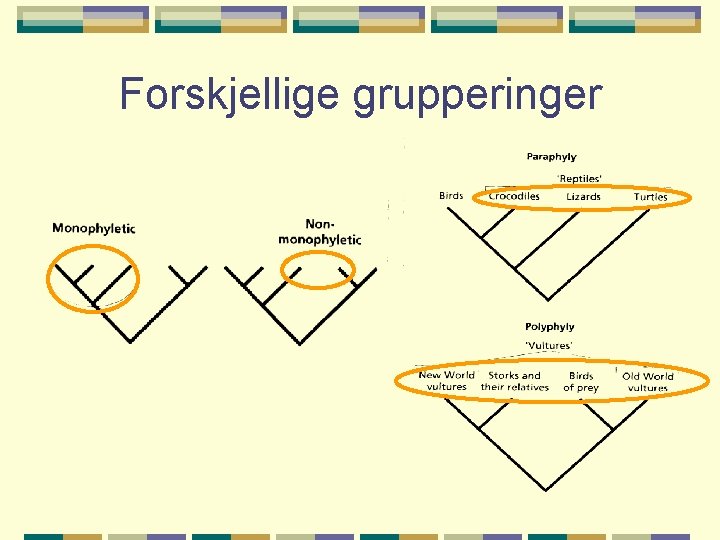
Forskjellige grupperinger

Antall mulige trær øker raskt Number of OTUs Number of unrooted trees Number of rooted trees 2 1 1 3 4 3 15 5 15 105 6 105 945 7 954 10, 395 8 10, 395 135, 135 9 135, 135 34, 459, 425 10 34, 459, 425 2. 13 E 15 15 2. 13 E 15 8. E 21

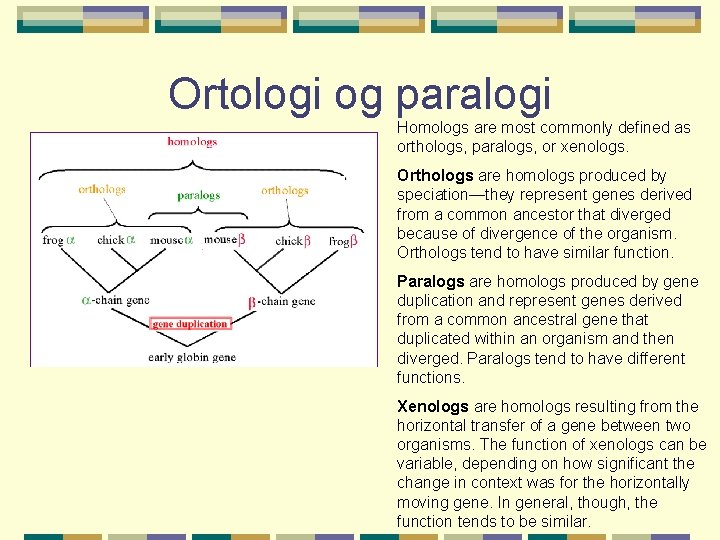
Ortologi og paralogi Homologs are most commonly defined as orthologs, paralogs, or xenologs. Orthologs are homologs produced by speciation—they represent genes derived from a common ancestor that diverged because of divergence of the organism. Orthologs tend to have similar function. Paralogs are homologs produced by gene duplication and represent genes derived from a common ancestral gene that duplicated within an organism and then diverged. Paralogs tend to have different functions. Xenologs are homologs resulting from the horizontal transfer of a gene between two organisms. The function of xenologs can be variable, depending on how significant the change in context was for the horizontally moving gene. In general, though, the function tends to be similar.

Fenetikk vs. cladistikk Fenetikk: Studiet av sammenhenger mellom organismer basert på graden av likhet mellom dem. Fenogram: Et trelignende nettverk som viser fenetiske sammmenhenger Cladistikk: Studiet av evolusjonære veier. Cladogram: Fylogenetisk tre med rot

Hva slags sekvenser bør sammenstilles? r. RNA l l best for very long term evolutionary studies spanning biological kingdoms most consistent with an evolutionary clock. Selective processes constraining sequence evolution should be roughly the same across species boundaries DNA/RNA l l contains more evolutionary information than protein high rate of base substitution makes DNA best for very short term studies eg. closely-related species Protein l l l more reliable alignment than DNA fewer homoplasies than DNA lower rate of substitution than DNA; better for wide species comparisons

Flersekvenssammenstilling I den fylogenetiske analysen vil man ofte velge å se bort fra posisjoner med gap

Hvordan komme fra flersekvenssammenstilling til fylogenetisk tre? Distansebaserte metoder UPGMA (unweighted pair-group method with arithmetic mean l NJ (neighbour joining) l Karakterbaserte metoder MP (maximum parsimony, ”maksimal gjerrighet”) l ML (maximum likelihood, maksimal sannsynlighet) l

Distanse (avstand) mellom sekvenser Hamming-distansen: Antall posisjoner med mismatch Levenshtein-distansen eller edit-distansen: antall redigeringsoperasjoner (delesjoner, insersjoner, utbyttinger) som skal til for å overføre den ene sekvensen til den andre agtc cgta Hamming-distanse = 2 ag-tcc cgctca Levenshtein-distanse = 3

Distanse mellom sekvenser p-distansen: Andelen av seter hvor de to sekvensene er forskjellige t 1 aaaaaccg t 2 tgca-gtt t 3 tgcaagtt t 1 og t 3: 6 av 8 forskjellige, distanse = 6/8 = 0. 75 t 1 og t 2: 6 av 7 forskjellige, distanse = 6/7 = 0. 85714

Distansebaserte metoder Det tas utgangspunkt i en distansematrise som viser distansen mellom de enkelte sekvensene A B C D B 2 C 4 4 D 6 6 6 E 6 6 6 4 F 8 8 E 8 Her har jeg for enkelthets skyld brukt antall forskjeller mellom sekvensene, snarere enn den relative mengden av forskjeller

Distansebaserte metoder: UPGMA A B C D B 2 C 4 4 D 6 6 6 E 6 6 6 4 F 8 8 E 8 1. Vi begynner med å finne det sekvenspar som er likest hverandre (minst distanse), i dette tilfellet A og B (avstand 2)

Distansebaserte metoder: UPGMA 2. Vi lager et cluster med A og B, separert av en avstand på 2. Forgreningspunktet ligger i en avstand på 2/2 = 1 fra de to nodene. A, B C D E C 4 D 6 6 E 6 6 4 F 8 8 8 3. Det lages en ny distansematrise hvor A og B betraktes som en enkelt, sammensatt OTU. dist(A, B), C dist(A, B), D dist(A, B), E dist(A, B), F 8 = = (dist. AC (dist. AD (dist. AE (dist. AF + + dist. BC) dist. BD) dist. BE) dist. BF) / / 2 2 = = 4 6 6 8

Distansebaserte metoder: UPGMA A, B C C 4 D, E 6 6 F 8 8 D, E 8 4. Etter at vi igjen har funnet det OTU-par som er likest hverandre ( i dette tilfelle D og E, avstand 4) clustres disse, og det lages en ny distansetabell

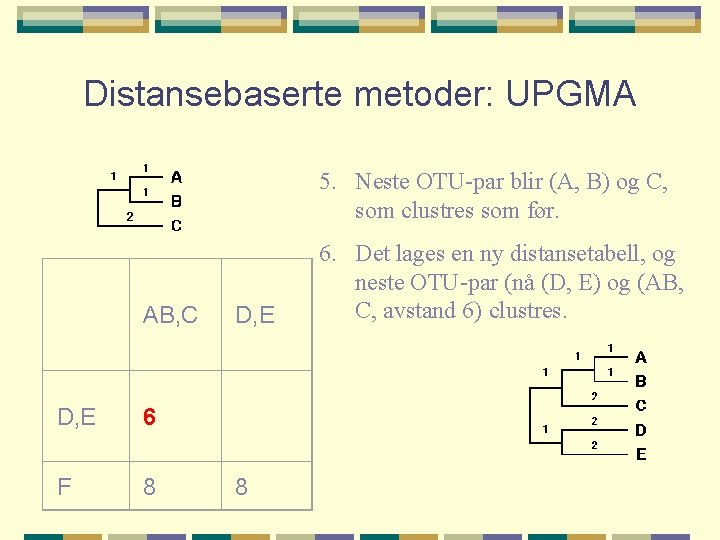
Distansebaserte metoder: UPGMA 5. Neste OTU-par blir (A, B) og C, som clustres som før. AB, C D, E 6 F 8 D, E 8 6. Det lages en ny distansetabell, og neste OTU-par (nå (D, E) og (AB, C, avstand 6) clustres.

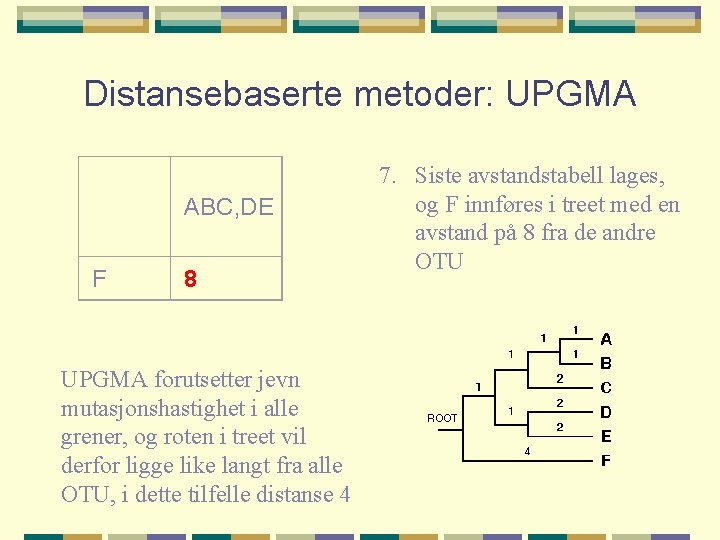
Distansebaserte metoder: UPGMA ABC, DE F 8 UPGMA forutsetter jevn mutasjonshastighet i alle grener, og roten i treet vil derfor ligge like langt fra alle OTU, i dette tilfelle distanse 4 7. Siste avstandstabell lages, og F innføres i treet med en avstand på 8 fra de andre OTU

Fallgruber med UPGMA er svært følsom for ulik mutasjonshastighet i de forskjellige grenene. Dersom for eksempel det egentlige treet ser ut som til venstre vil UPGMA gi det feilaktige treet til høyre

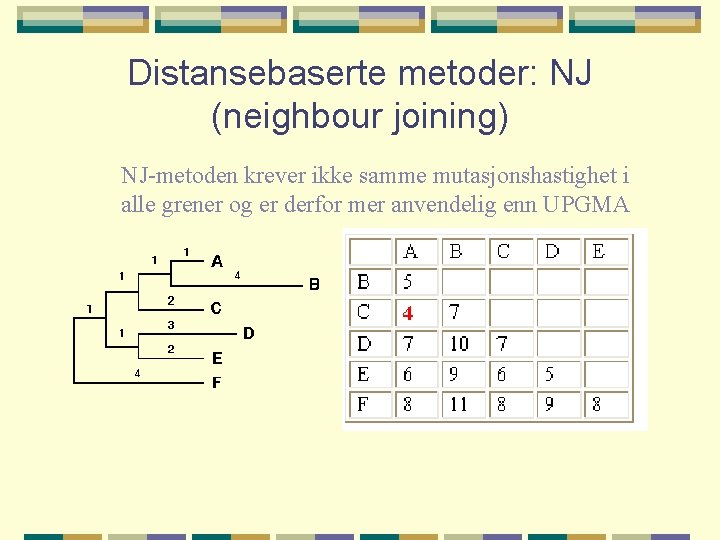
Distansebaserte metoder: NJ (neighbour joining) NJ-metoden krever ikke samme mutasjonshastighet i alle grener og er derfor mer anvendelig enn UPGMA

Neighbour joining Beregn netto divergens r(i) for hver OTU fra alle andre OTU r(A) = 5+4+7+6+8=30 r(B) = 42 r(C) = 32 r(D) = 38 r(E) = 34 r(F) = 44

Neighbour joining Beregn ny distansetabell for hvert OTU-par ved å bruke formelen M(ij)=d(ij) - [r(i) + r(j)]/(N-2) eller for paret A, B: M(AB)=d(AB) -[(r(A) + r(B)]/(N-2) =5 -(30+42)/4=5 -18 = -13

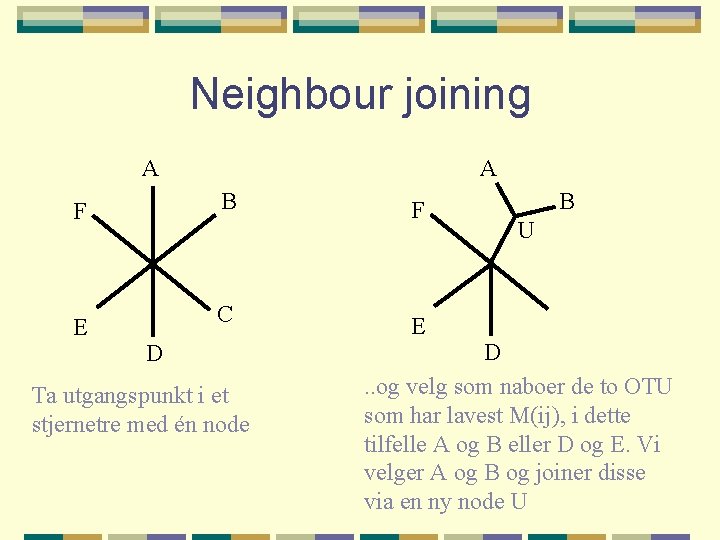
Neighbour joining A A B F E C D Ta utgangspunkt i et stjernetre med én node F E B U D. . og velg som naboer de to OTU som har lavest M(ij), i dette tilfelle A og B eller D og E. Vi velger A og B og joiner disse via en ny node U
![Neighbour joining Grenlengdene SAU og SBU beregnes så SAU dAB 2 rArB Neighbour joining Grenlengdene S(AU) og S(BU) beregnes så: S(AU) =d(AB) / 2 + [r(A)-r(B)]](https://slidetodoc.com/presentation_image_h2/daf5aa32d5a5284057a797d5886f4a02/image-25.jpg)
Neighbour joining Grenlengdene S(AU) og S(BU) beregnes så: S(AU) =d(AB) / 2 + [r(A)-r(B)] / 2(N-2)=5/2 -12/8 = 1 S(BU) =d(AB) -S(AU) = 4 A F B E C D A F E 1 U 4 C D B

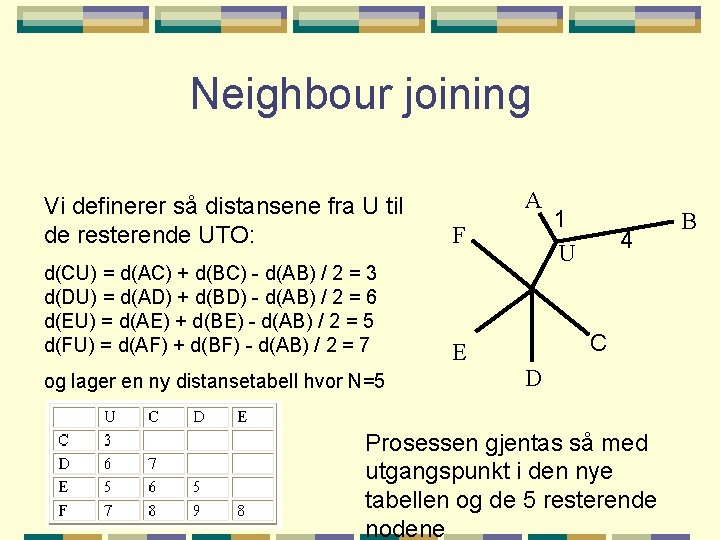
Neighbour joining A Vi definerer så distansene fra U til de resterende UTO: F d(CU) = d(AC) + d(BC) - d(AB) / 2 = 3 d(DU) = d(AD) + d(BD) - d(AB) / 2 = 6 d(EU) = d(AE) + d(BE) - d(AB) / 2 = 5 d(FU) = d(AF) + d(BF) - d(AB) / 2 = 7 E og lager en ny distansetabell hvor N=5 1 U 4 C D Prosessen gjentas så med utgangspunkt i den nye tabellen og de 5 resterende nodene B

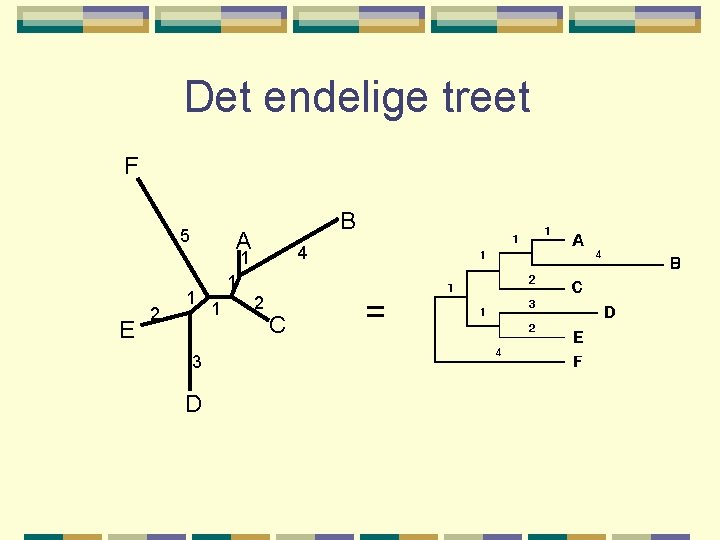
Det endelige treet F 5 B A 4 1 E 2 1 3 D 1 1 2 C =

Karakterbaserte metoder: Maximum parsimony (MP) Man ser på flersekvenssammenstillingene, ikke distansetabeller Alle mulige trær tas i betraktning. Ancestrale sekvenser estimeres, det tre som gir det laveste antall mutasjoner utvelges

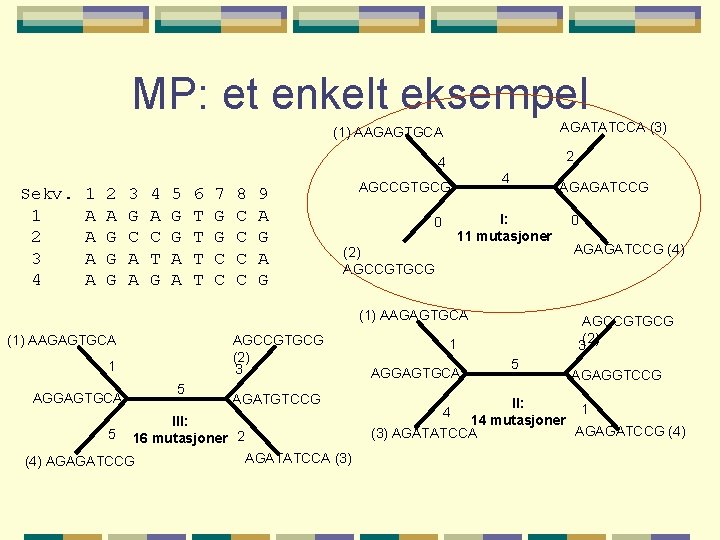
MP: et enkelt eksempel AGATATCCA (3) (1) AAGAGTGCA 2 4 Sekv. 1 2 3 4 1 A A 2 A G G G 3 G C A A 4 A C T G 5 G G A A 6 T T 7 G G C C 8 C C 9 A G 4 AGCCGTGCG AGAGATCCG I: 11 mutasjoner 0 (2) AGCCGTGCG (1) AAGAGTGCA AGCCGTGCG (2) 3 1 5 AGGAGTGCA 5 AGATGTCCG III: 16 mutasjoner 2 (4) AGAGATCCG AGATATCCA (3) AGAGATCCG (4) AGCCGTGCG 3(2) 1 AGGAGTGCA 0 5 AGAGGTCCG II: 1 14 mutasjoner AGAGATCCG (4) (3) AGATATCCA 4

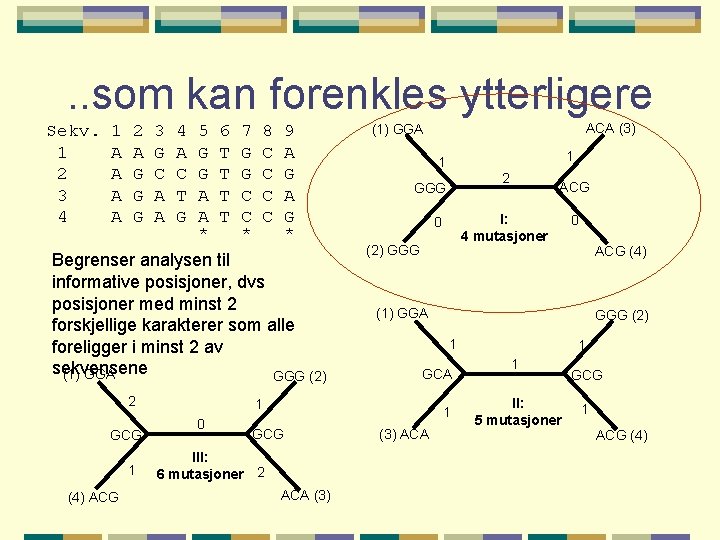
. . som kan forenkles ytterligere Sekv. 1 2 3 4 1 A A 2 A G G G 3 G C A A 4 A C T G 5 G G A A * 6 T T 7 G G C C * 8 C C 9 A G * Begrenser analysen til informative posisjoner, dvs posisjoner med minst 2 forskjellige karakterer som alle foreligger i minst 2 av sekvensene (1) GGA GGG (2) 2 GCG 1 (4) ACG 1 1 2 GGG (2) GGG III: 6 mutasjoner 2 ACA (3) 0 ACG (4) (1) GGA GGG (2) 1 GCA 1 GCG ACG I: 4 mutasjoner 0 1 0 ACA (3) (1) GGA (3) ACA 1 1 II: 5 mutasjoner GCG 1 ACG (4)

MP - oppsummering • Maximum Parsimony (positive punkter): • Bygger på felles og avledede karakterer, er derfor en cladistisk snarere enn en fenetisk metode • reduserer ikke sekvensinformasjonen til et enkelt tall • prøver å utlede informasjon om de ancestrale sekvensene • evaluerer flere trær • Maximum Parsimony (negative punkter): • langsom sammenlignet med distansemetoder • bruker ikke all sekvensinformasjon (kun informative seter benyttes) • korrigerer ikke for flere mutasjoner i samme sete (bygger ikke på en gitt evolusjonsmodell • gir ikke informasjon om grenlengder • beryktet for å være følsom for kodon-bias

Karakterbaserte metoder: Maximum likelihood (ML) ML tar utgangspunkt i en flersekvenssammenstilling og en evolusjonær modell ML tar for seg alle mulige trær og regner ut hvilke(t) som mest sannsynlig vil gi de observerte sekvensene

Evolusjonære modeller

Hvordan sette rot på treet? De fleste metodene for utledning av fylogenetiske trær gir trær uten rot. Treet gir derfor ikke informasjon om hvilken OTU som først skilte lag med de andre Man kan sette rot på treet ved å innføre en ”utgruppe” i sekvenssammenstilligen

Hvordan sette rot på treet? Utgruppen bør ikke være for fjernt beslektet med de andre sekvensene Utgruppen bør ikke være for nært beslektet med de andre sekvensene Tretopologien forbedres ved bruk av flere utgrupper I fravær av en god utgruppe kan roten plasseres midtveis på den lengste vei mellom to OTU, idet antas tilnærmet lik evolusjonshastighet. Mid-point rooting

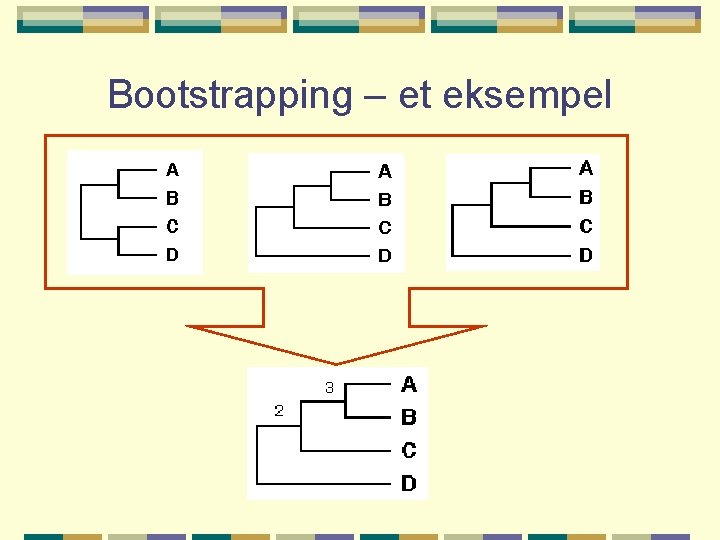
Bootstrapping …eller hvordan kan vi få et mål på hvor pålitelig treet er? Ved bootstrapping lages nye datasett (”flersekvenssammenstillinger”) ved å velge ut et antall tilfeldige posisjoner fra vår opprinnelige flersekvenssammenstilling. Samme posisjon kan utvelges flere ganger i samme datasett. Det velges nye tilfeldige posisjoner inntil de nye datasettene er like store som det opprinnelige. Hvert av de nye datasettene sendes så gjennom samme trekonstruksjonsmetode, slik at hvert sett gir opphav til et nytt tre. Ved å sammenligne de forskjellige treene kan sannsynligheten for hver clade bestemmes. Antall nye datasett: minst like stort som antall posisjoner i sekvenssammenstillingen!

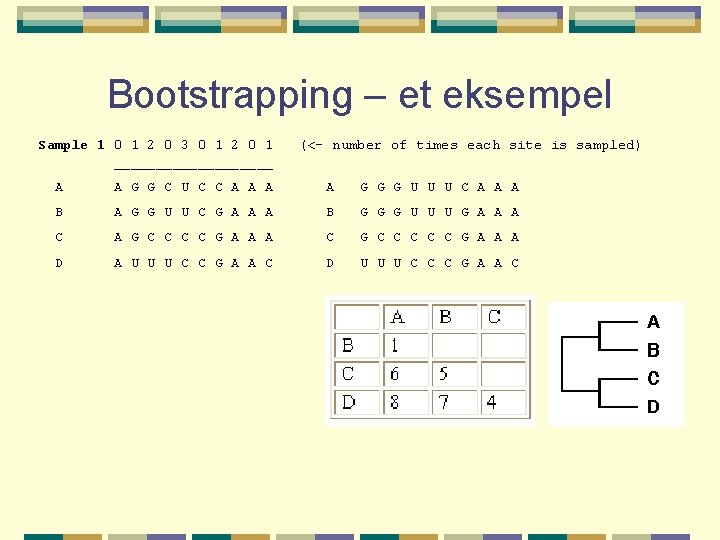
Bootstrapping – et eksempel Sample 1 0 1 2 0 3 0 1 2 0 1 __________ (<- number of times each site is sampled) A A G G C U C C A A G G G U U U C A A A B A G G U U C G A A A B G G G U U U G A A A C A G C C G A A A C G C C C G A A A D A U U U C C G A A C D U U U C C C G A A C

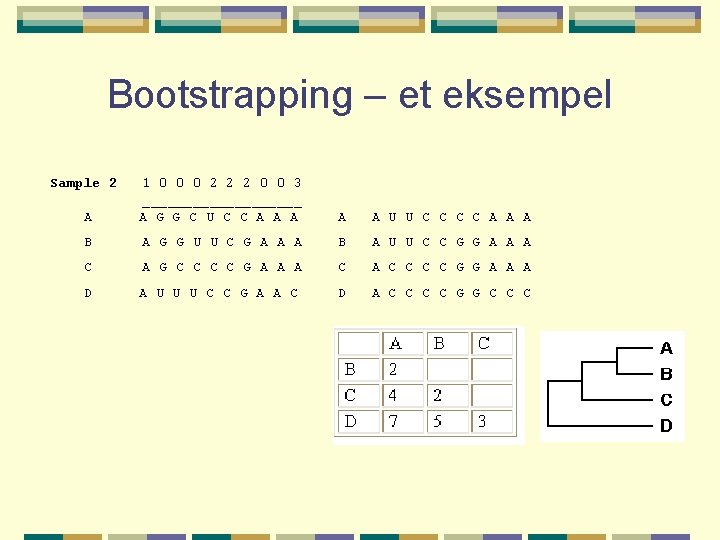
Bootstrapping – et eksempel Sample 2 A 1 0 0 0 2 2 2 0 0 3 __________ A G G C U C C A A A U U C C A A A B A G G U U C G A A A B A U U C C G G A A A C A G C C G A A A C C C C G G A A A D A U U U C C G A A C D A C C G G C C C

Bootstrapping – et eksempel Sample 3 1 0 0 0 2 2 2 0 0 3 __________ A A G G C U C C A A A B A G G U U C G A A A C A G C C G A A A D A U U U C C G A A C A B C D A A U U C C C C G G G A A A C

Bootstrapping – et eksempel
 Om du gör som du alltid har gjort
Om du gör som du alltid har gjort Enn root word
Enn root word Redesim sp
Redesim sp Mrsa prenos
Mrsa prenos Vre erreger
Vre erreger Redesin sp
Redesin sp Vi vil gi dig ære
Vi vil gi dig ære Vil bert
Vil bert Vi vil leve
Vi vil leve Ordleter
Ordleter Gi os lyset tilbage tekst
Gi os lyset tilbage tekst Cmos inverter cross sectional view
Cmos inverter cross sectional view La statue regarde la mer
La statue regarde la mer Chien de mer roussette
Chien de mer roussette Les toiles de mer
Les toiles de mer Axl mer
Axl mer Le vieil homme et la mer
Le vieil homme et la mer Dr mer
Dr mer Centre marceau batz sur mer
Centre marceau batz sur mer La secuencia 13 mer del origen (oric) se desnaturaliza
La secuencia 13 mer del origen (oric) se desnaturaliza Symbole du vin dans la bible
Symbole du vin dans la bible Isomeria fac mer
Isomeria fac mer Dr mer
Dr mer Mer banco de dados
Mer banco de dados Mer kunnskap bedre barnevern
Mer kunnskap bedre barnevern Kai wed game
Kai wed game Le moine au bord de la mer
Le moine au bord de la mer Mer d'aral
Mer d'aral Dr mer
Dr mer Association pleine mer
Association pleine mer Lucien simon peintre
Lucien simon peintre Sjednocení měr a vah marie terezie
Sjednocení měr a vah marie terezie Dr mer
Dr mer Gondrand fos sur mer
Gondrand fos sur mer Törzslapnyilvántartás
Törzslapnyilvántartás Forbrenner man mer når man har mensen
Forbrenner man mer når man har mensen Friedrich le voyageur contemplant une mer de nuages analyse
Friedrich le voyageur contemplant une mer de nuages analyse Durmio es una palabra aguda grave o esdrujula
Durmio es una palabra aguda grave o esdrujula Agregacion mer
Agregacion mer Discordance jard sur mer
Discordance jard sur mer Mer locadora de veiculos
Mer locadora de veiculos