Specjalizowane jzyki programowania dr in Maciej Miostan Harmonogram


























- Slides: 27

Specjalizowane języki programowania dr inż. Maciej Miłostan

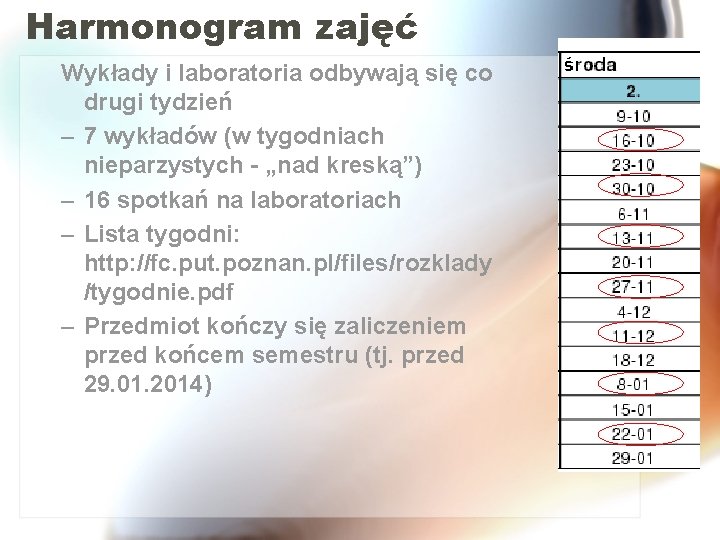
Harmonogram zajęć Wykłady i laboratoria odbywają się co drugi tydzień – 7 wykładów (w tygodniach nieparzystych - „nad kreską”) – 16 spotkań na laboratoriach – Lista tygodni: http: //fc. put. poznan. pl/files/rozklady /tygodnie. pdf – Przedmiot kończy się zaliczeniem przed końcem semestru (tj. przed 29. 01. 2014)

Zasady zaliczenia • Zgodnie z regulaminem studiów: 1. „Nieobecność studenta, nawet usprawiedliwiona, na więcej niż 1/3 zajęć, może być podstawą do niezaliczenia tych zajęć. Niewykonanie ćwiczeń określonych regulaminem laboratorium uniemożliwia zaliczenie zajęć laboratoryjnych. ” 2. „Podstawą do zaliczenia wszystkich rodzajów ćwiczeń i wykładów niekończących się egzaminem są pozytywne wyniki bieżącej kontroli wiadomości. Formę tej kontroli określa prowadzący zajęcia i wystawia ocenę do końca semestru. ” 3. „Studentowi, który w wyniku bieżącej kontroli wiadomości otrzymał ocenę niedostateczną, przysługuje prawo do jednego zaliczenia poprawkowego. ” • Ergo: – – Można opuścić maksimum pięć laboratoriów, ale sprawozdania należy oddać ze wszystkich Na laboratoriach będą obowiązywały regularne sprawozdania z zajęć jako forma sprawdzania wiedzy, możliwe wejściówki

Konsultacje i kontakt • Gdzie? – Centrum Wykładowo-Konferencyjne Politechniki Poznańskiej – Pokój 122 (na I piętrze) • Kiedy? – Czwartki od 8. 15 do 9. 40 Lub inny umówiony termin (np. w tygodniu parzystym przed laboratorium) • Informacje kontaktowe: Maciej. Milostan@cs. put. poznan. pl Tel. : +48 -61 -665 -29 -78

Plan przedmiotu • Treści kształcenia Języki programowania ukierunkowane na specyficzne zastosowania, ze szczególnym uwzględnieniem przetwarzania ciągów znaków. Języki skryptowe. Perl. Python. Przetwarzanie danych tekstowych. • Efekty kształcenia - umiejętności i kompetencje Student zna wybrane specjalizowane języki programowania w stopniu umożliwiającym swobodne pisanie programów, ze szczególnym uwzględnieniem przetwarzania danych tekstowych, w tym sekwencji nukleotydowych i aminokwasowych.

Karta ETCS • Pokaż kartę ETCS

Plan zajęć • Informacje organizacyjne i wprowadzenie do przedmiotu • Języki skryptowe i przetwarzanie tekstów (1 x) • Perl i Bio. Perl (3 x) • Python i Bio. Python (2 x + 1/2 x) • Zaliczenie (1/2 x)

Motywacja • Współczesne eksperymenty generują duże ilości danych, które wymagają zautomatyzowanego przetwarzania i gromadzenia w rozmaitych bazach danych

Motywacja • Istnieje wiele formatów przechowywania podobnych danych – Np. formaty sekwencji aminokwasowych i nukleotydowych: • PIR • FASTA • Sama sekwencja (bare sequence) • Sekwencja z numeracją i spacjami

Motywacja • Informacje z baz danych, w celu dalszej analizy, często muszą zostać pobrane i przekonwertowane do formatu kompatybilnego z wykorzystywaną aplikacją • Z plików danych musi zostać wyekstraktowana tylko część danych np. pojedynczy model białka z pliku PDB • Dane generowane przez różne aplikacje muszą zostać zagregowane

Rodzaje danych biologicznych • • Sekwencje, np. dane genomowe Struktury przestrzenne Drzewa filogenetyczne Sieci / grafy – np. mapy interakcji białek, modelowanie oddziaływań • Szlaki metaboliczne • Dane z mikromacierzy i ekspresja genów • Dane obrazowe

Źródło danych – rodzaje baz • Pochodzenie – Dane pochodzące bezpośrednio z eksperymentu – Dane wywiedzione z danych eksperymentalnych – Dane zagregowane • Jakość – Dane deponowane bezpośrednio – Dane deponowane przez „kuratorów” – Zarządzanie błędami – usuwanie błędnych danych lub ich znakowanie – Sprawdzanie błędów – Spójność, aktualizacje

Organizacja danych • Pliki płaskie • Relacyjne bazy danych • Obiektowe bazy danych

Dostępność danych • Dostępne (ang. available) publicznie bez restrykcji • Dostępne (ang. available), ale chronione prawami autorskimi • Dostępne (ang. accessible), ale bez możliwość pobrania • Dostępne dla środowiska akademickiego • Komercyjne

Opiekunowie/kuratorzy baz • Duże instytucje publiczne (NCBI, EMBL) • Instytucje kwasi-akademickie (Swiss Institute of Bioinformatics) • Grupa akademicka lub naukowiec • Firma komercyjna

Algorytmy i analizy 1) Proste operacje – wyszukiwanie ciągów znaków, zliczanie, itp. 2) Porównywanie sekwencji 3) Konstrukcja drzew (filogenetycznych) 4) Detekcja wzorców w sekwencjach 5) Modelowanie struktur 3 D z sekwencji 6) Wnioskowanie dot. mechanizmów regulacji komórkowej 7) Przewidywanie lub determinacja funkcji białek i szlaków metabolicznych 8) Asemblacja fragmentów DNA

Języki skryptowe • • Języki interpretowane Zwykle nie wymagają kompilacji Często stosują automatyczne typowanie Przykłady: – Powłoka systemu linux/unix np. język skryptów Bash – Pliki wsadowe. bat – AWK (Zobacz: http: //www. cs. put. poznan. pl/jnawrocki/ics/0203/ 02 wpr 5 -awk. ppt) – Perl – Python – Ruby

Języki skryptowe • Bio. Perl jest zbiorem modułów Perl-a • Trzy paradygmaty projektowe w Bio. Perlu – Separacja interfejsów od implementacji – Dostarczenie bazowego wzorca (framework-u) dla odpowiednich operacji poprzez generalizacię typowych procedur do pojedynczego modułu – Wykorzystanie wzorców projektowych opracowanych przez Ericha Gamma: metoda wytwórcza i wzorzec strategii

Języki skryptowe • Biopython jest zbiorem modułów Python • Kluczowe elementy projektu Bio. Python – tworzenie parserów do danych biologicznych – Projektowanie interfejsów użytecznych w przetwarzaniu sekwencji

Języki skryptowe • Bio. PHP dawniej Gene. PHP, „seeks to encourage the use of PHP as a “glue” language to bind web-based bioinformatics applications and databases” • Funkcje zaimplementowane w Bio. PHP – Odczyt danych biologicznych w formatach Gen. Bank-u, Swissprot-a, Fasta, alignment-ów Clustal-a (ALN) – Proste analizy sekwencji

Języki skryptowe • Ruby jest obiektowym językiem skryptowym • Projekt Bio. Ruby jest wspierany i finansowany przez – Human Genome Center na Uniwersytecie w Tokyo – Bioinformatics Center na Uniwersytecie w Kyoto

XML i składowanie danych • XML jest uniwersalnym i roszerzalnym formatem przechowywania i wymiany danych oraz dokumentów ustrukturalizowanych • Dwie inicjatywy godne odnotowania: – Bioinformatics Sequence Markup Language (BSML) – BIOpolymer Markup Language (Bio. ML)

Programowanie deklaratywne i funkcyjne • Przykład czysto funkcyjnego języka • Pod nazwa Biohaskell zbierane są wszelkie programy i biblioteki związane z zastosowaniem haskell-a w biologii.

Programowanie deklaratywne i funkcyjne • Biomedical Logic Programming (Blip) – Zbiór modułów stworzonych z myślą o zastosowaniach bioinformatycznych i biomedycznych – Zintregrowany system zapytań – Zaimplementowane w SWI-Prolog

Programowanie deklaratywne i funkcyjne • Bio. Bike jest interaktywnym (z interfejsem web) środowiskiem programistycznym, które umożliwia biologom analizę systemów biologicznych poprzez połączenie wiedzy i danych poprzez bezpośrednie programowanie dokonywane przez użytkownika końcowego • Zaimplementowano przy użyciu Bio. Lisp (Lisp z dodatkiem funkcjonalności biologicznej)

AWK - PRZYPOMNIENIE

• Przypomnienie wykładu prof. J. Nawrockiego: http: //www. cs. put. poznan. pl/jnawrocki/i cs/0203/02 wpr 5 -awk. ppt
 Samochody niskotonażowe
Samochody niskotonażowe Co to jest magazyn
Co to jest magazyn Programowanie deklaratywne
Programowanie deklaratywne Zasady programowania strukturalnego
Zasady programowania strukturalnego Paradygmaty programowania obiektowego
Paradygmaty programowania obiektowego Język drabinkowy symbole
Język drabinkowy symbole Pascal język programowania
Pascal język programowania Czy warto uczyć się programowania
Czy warto uczyć się programowania Język programowania logo
Język programowania logo Harmonogram rzeczowo finansowy wzór
Harmonogram rzeczowo finansowy wzór Esf muni harmonogram
Esf muni harmonogram Zasady sporządzania harmonogramu prac wykonawczych
Zasady sporządzania harmonogramu prac wykonawczych Harmonogram liniowy
Harmonogram liniowy Harmonogram pf upol
Harmonogram pf upol Econ muni harmonogram
Econ muni harmonogram Fappz harmonogram
Fappz harmonogram Główny harmonogram produkcji przykład
Główny harmonogram produkcji przykład Uis czu přihlášení
Uis czu přihlášení Co je harmonogram
Co je harmonogram Harmonogram pf upol
Harmonogram pf upol Fhs harmonogram 2020/21
Fhs harmonogram 2020/21 Podanie o praktyki
Podanie o praktyki Sekcja toku studiów wdib
Sekcja toku studiów wdib Pf upol stag
Pf upol stag Mrp przykład
Mrp przykład Wariant siła argumentacji przed prawem
Wariant siła argumentacji przed prawem Marcin tryba
Marcin tryba Abcde schemat
Abcde schemat



















































