Expression du Gnome Le transcriptome Dchiffrage de lInformation










- Slides: 20

Expression du Génome Le transcriptome

Déchiffrage de l’Information Génique q L’information est encodée dans la séquence de nucléotides contenus dans des unités discrètes q Les gènes q L’information contenue dans les gènes est transcrite pour générer les ARNs puis décodée pour générer les protéines

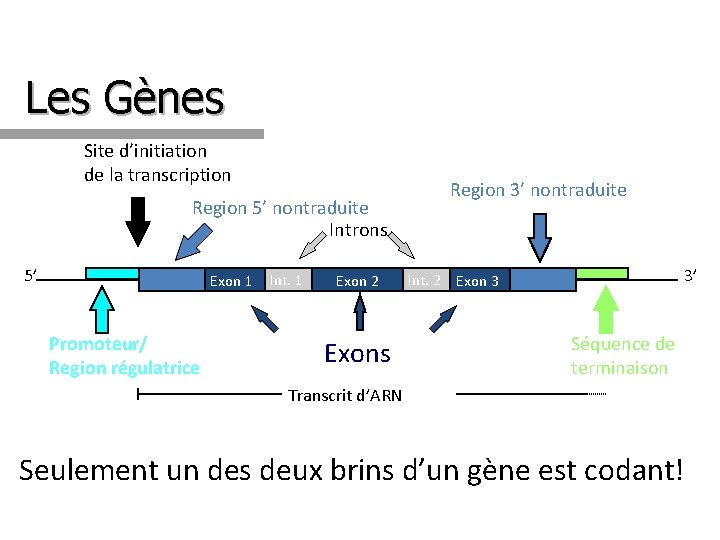
Les Gènes Site d’initiation de la transcription Region 5’ nontraduite Introns 5’ Exon 1 Promoteur/ Region régulatrice Int. 1 Exon 2 Exons Region 3’ nontraduite 3’ Int. 2 Exon 3 Séquence de terminaison Transcrit d’ARN Seulement un des deux brins d’un gène est codant!

Codant q Brin positif q Brin sens q Brin complémentaire à la matrice q Brin dont la séquence est la même que celle de l’ARN transcrit q Brin sur lequel se retrouve le promoteur 4

Non Codant q Brin négatif q Brin anti sens q Brin matrice q Brin dont la séquence est complémentaire à celle de l’ARN transcrit 5

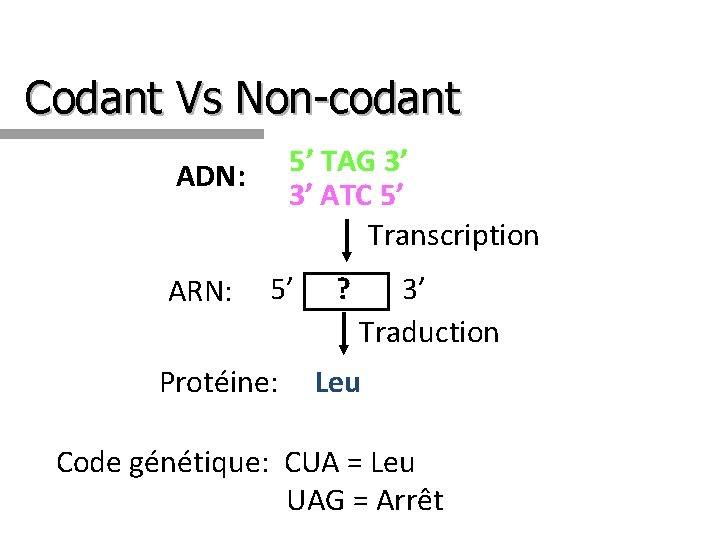
Codant Vs Non-codant 5’ TAG 3’ 3’ ATC 5’ Transcription ADN: ARN: 5’ Protéine: ? 3’ Traduction Leu Code génétique: CUA = Leu UAG = Arrêt

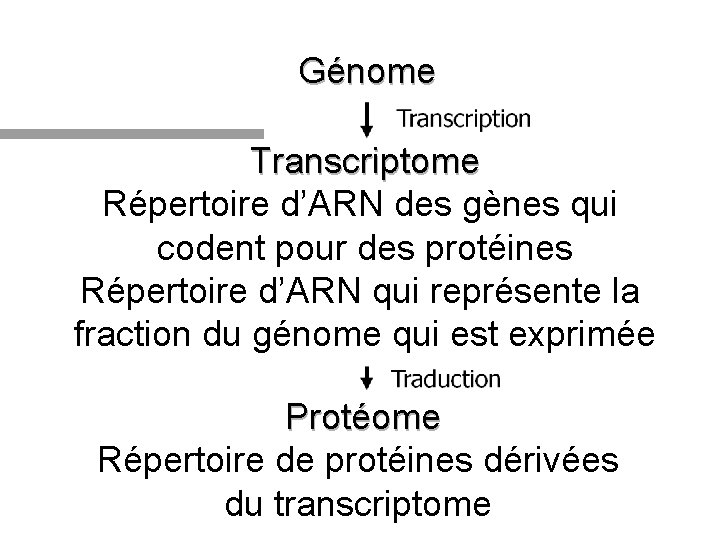
Génome Transcriptome Répertoire d’ARN des gènes qui codent pour des protéines Répertoire d’ARN qui représente la fraction du génome qui est exprimée Protéome Répertoire de protéines dérivées du transcriptome

Un Génome q. Le transcriptome est-il le même dans toutes les cellules d’un organisme? q. Le transcriptome est-il toujours le même dans une cellule donnée?

Est-ce qu’une Séquence Code pour un Transcrit? q Analyse d’hybridations Northern : q RT-PCR 9

Comparaison des Méthodes Séquence doit être connue Northern Non RT-PCR Oui Présence ou absence d’un transcrit Oui Permets de déterminer la taille Oui Non Sensibilité Faible Élevée Comparer l’abondance relative Oui Obtenir la séquence du transcrit Non Oui Déterminer quel brin d’ADN est transcrit Oui Déterminer combien de transcrits sont fait à partir d’une séquence LA SÉQUENCE DOIT ÊTRE EXPRIMÉE Oui Non OUI 10

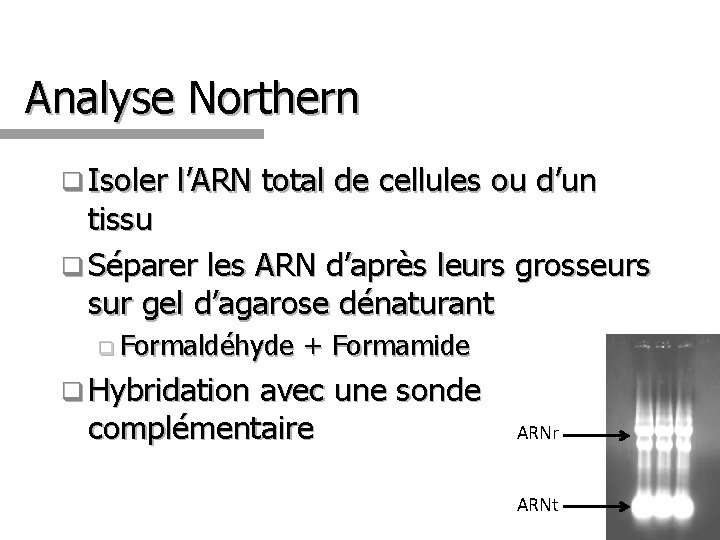
Analyse Northern q Isoler l’ARN total de cellules ou d’un tissu q Séparer les ARN d’après leurs grosseurs sur gel d’agarose dénaturant q Formaldéhyde + Formamide q Hybridation avec une sonde complémentaire ARNr ARNt

Hybridation Northern q Nécessite une sonde q Hybridation = la sonde possède des séquences d’un gène q La séquence est exprimée q Intensité du signal d’hybridation = abondance relative q Nombre de signaux d’hybridation = nombre de transcrit q Possiblement nombre de gènes 12

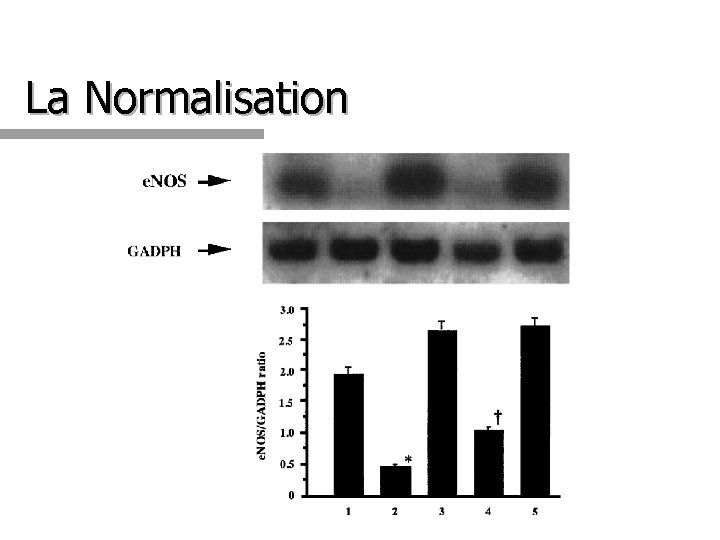
Hybridation Northern q Permet de comparer la quantité relative d’un transcrit q Sensibilité faible q Requiert un témoin interne q Gène dont l’abondance est constante sous les différentes conditions examinées – Témoin pour les variations de la quantité d’ARN chargé – Utilise des gènes domestiques : q Gènes qui assurent les fonctions indispensables à la vie de tous les types de cellules 13 q Expression constitutive

La Normalisation 14

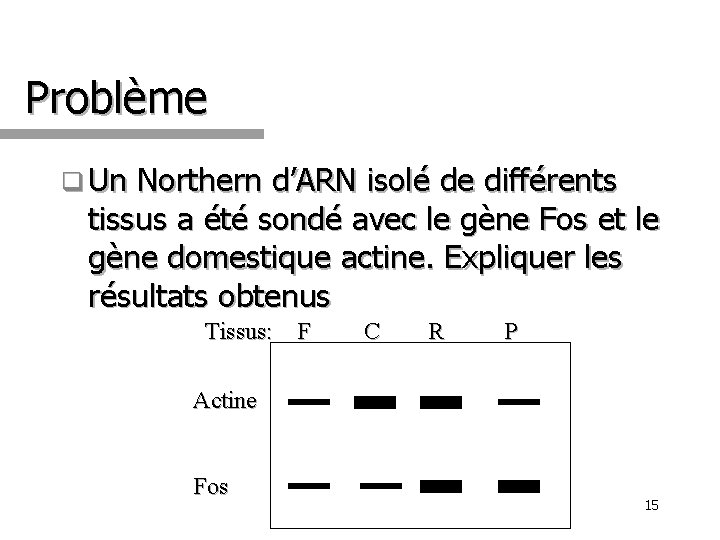
Problème q Un Northern d’ARN isolé de différents tissus a été sondé avec le gène Fos et le gène domestique actine. Expliquer les résultats obtenus Tissus: F C R P Actine Fos 15

RT-PCR q Permets d’amplifier une séquence d’ARN q Isoler l’ARN total de cellules ou d’un tissu q Transcrire les ARN en ADNc avec transcriptase inverse q Amplifier par PCR la séquence d’intérêt 16

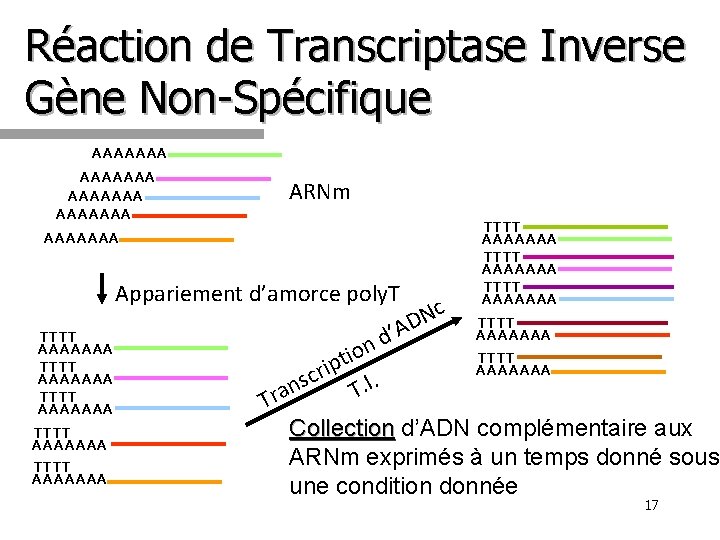
Réaction de Transcriptase Inverse Gène Non-Spécifique AAAAAAA ARNm AAAAAAA Appariement d’amorce poly. T TTTT AAAAAAA TTTT AAAAAAA Nc D ’A Tr d n tio p i r c s T. I. an TTTT AAAAAAA TTTT AAAAAAA Collection d’ADN complémentaire aux ARNm exprimés à un temps donné sous une condition donnée 17

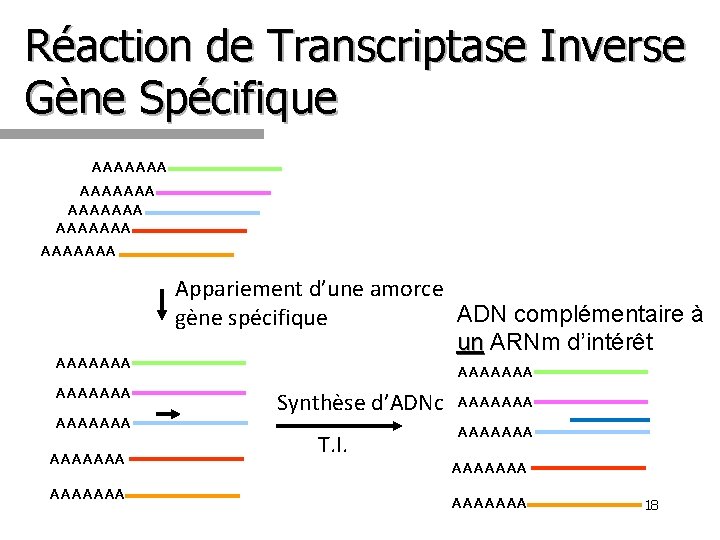
Réaction de Transcriptase Inverse Gène Spécifique AAAAAAA AAAAAAA AAAAAAA Appariement d’une amorce ADN complémentaire à gène spécifique un ARNm d’intérêt AAAAAAA Synthèse d’ADNc T. I. AAAAAAA 18

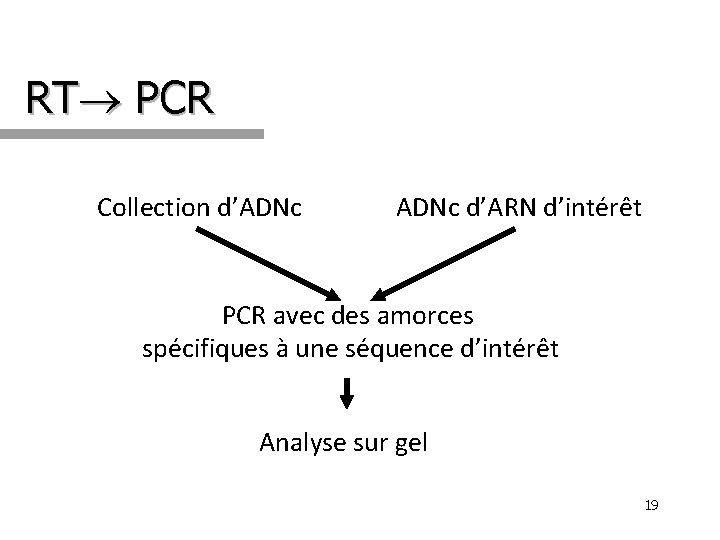
RT PCR Collection d’ADNc d’ARN d’intérêt PCR avec des amorces spécifiques à une séquence d’intérêt Analyse sur gel 19

RT-PCR q La séquence doit être connue pour la conception d’amorces q Produit d’amplification = q Les séquences des amorces font partie d’un gène q La séquence est exprimée q Intensité du produit d’amplification = abondance relative q La taille du produit d’amplification n’est pas égale à la taille du transcrit 20