Deteccin de Secuencias Reguladoras en el Genoma Karinna










- Slides: 17

Detección de Secuencias Reguladoras en el Genoma Karinna Rubio Peña

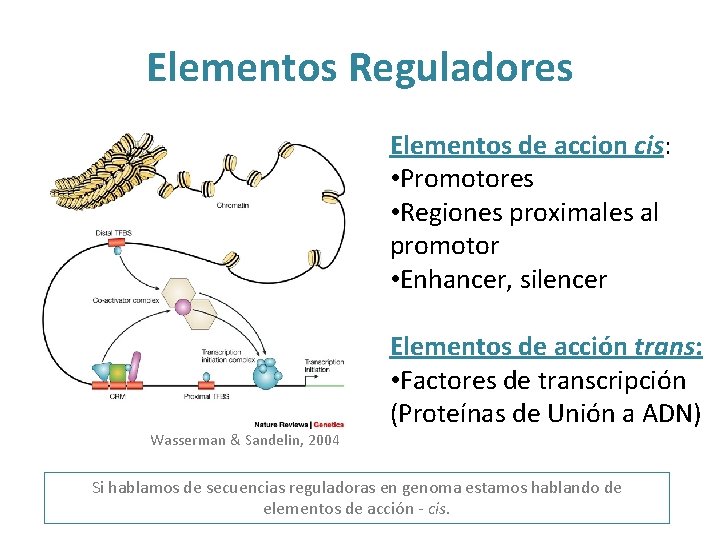
Elementos Reguladores Elementos de accion cis: • Promotores • Regiones proximales al promotor • Enhancer, silencer Elementos de acción trans: • Factores de transcripción (Proteínas de Unión a ADN) Wasserman & Sandelin, 2004 Si hablamos de secuencias reguladoras en genoma estamos hablando de elementos de acción - cis.

¿Porqué es importante conocer los elementos reguladores? • La regulación de la expresión génica esta implicada en: – Procesos del desarrollo – Diferenciación celular – Especificidad de los tejidos – Producción de hormonas y enzimas – Respuesta celular a estrés (enfermedades o condiciones físicas adversas)

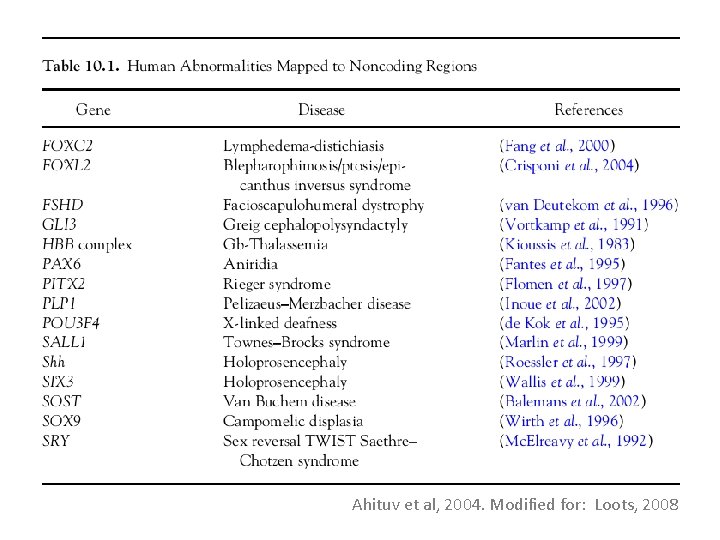
Ahituv et al, 2004. Modified for: Loots, 2008

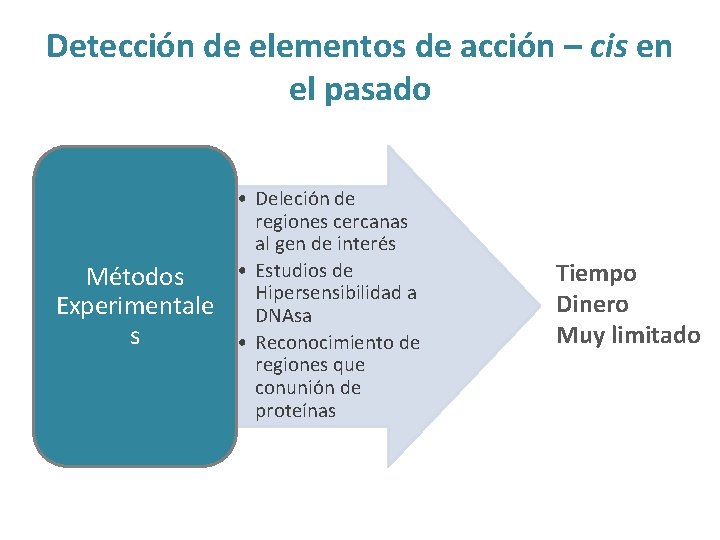
Detección de elementos de acción – cis en el pasado Métodos Experimentale s • Deleción de regiones cercanas al gen de interés • Estudios de Hipersensibilidad a DNAsa • Reconocimiento de regiones que conunión de proteínas Tiempo Dinero Muy limitado

La identificación de nuevos elementos de acción – cis se esta dando gracias a que ahora contamos con secuencias de genomas completos.

Para tener en cuenta… • Sitios de Unión a Factores de Transcripción (TFBS) cortos (4 -6 bp) • Elementos cis se encuentran entre 10010000 bp del sitio de inicio de transcripción. • No suelen actuar solos. • La verificación de las predicciones son complicadas.

Métodos de predicción de secuencias reguladoras en el genoma • Comparación de secuencias inter-especies. • Localización de motifs (Perfiles de Expresión). • Métodos completamente computacionales.

Comparacion de secuencias inter-especies Cualquier región conservada identificada de esta manera podría ser un elemento regulación de acción - cis. Pero que especies debo comparar?

Comparación se secuencias inter especies Genómica comparativa de múltiples especies Entonces las especies que se comparen deben estar lo suficientemente relacionadas para compartir genes con patrones de expresión similares PERO lo suficiente distantes como para que la región del genoma no codante tenga cierta divergencia. Pennacchio & Rubin, 2001

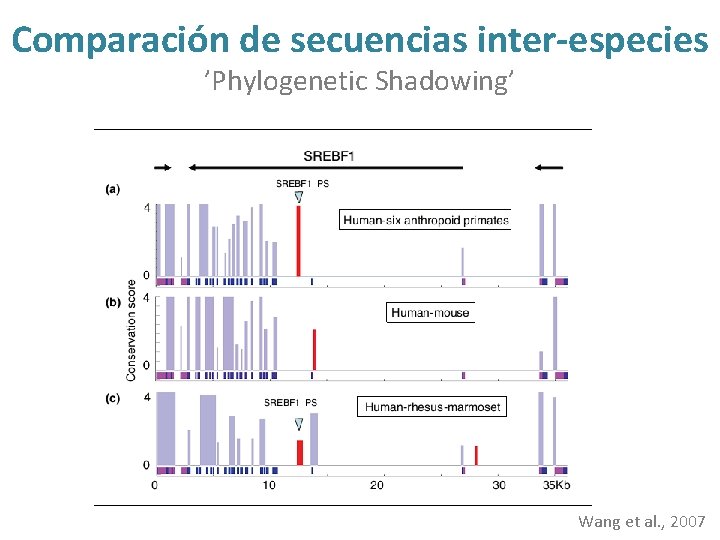
Comparación de secuencias inter-especies ’Phylogenetic Shadowing’ Wang et al. , 2007

¿Qué especies debo comparar? “No importa que especies compraremos, de una forma u otra alguna fracción de elementos reguladores va a ser pasada por alto. ” Pennacchio & Rubien. 2001

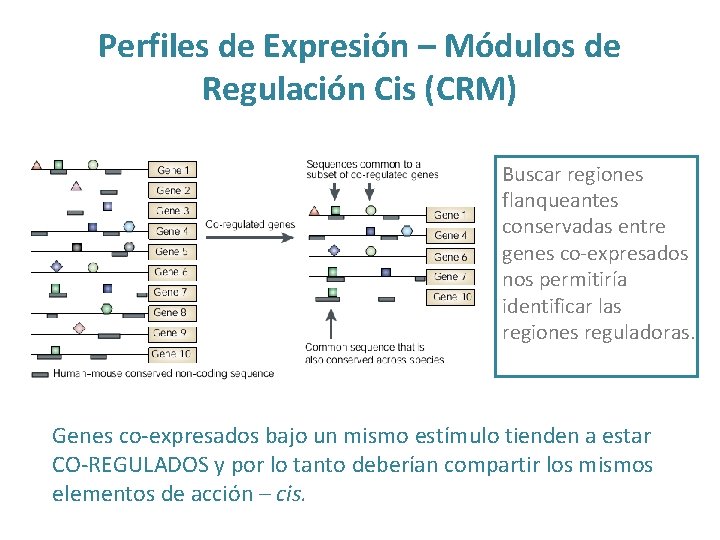
Perfiles de Expresión – Módulos de Regulación Cis (CRM) Buscar regiones flanqueantes conservadas entre genes co-expresados nos permitiría identificar las regiones reguladoras. Genes co-expresados bajo un mismo estímulo tienden a estar CO-REGULADOS y por lo tanto deberían compartir los mismos elementos de acción – cis.

Método computacional • Algoritmos que buscan reconocer secuencias en el genoma cuya presencia indique la detección de una putativa región reguladora. – Ej: Islas de Cp. G, caja. TATA. o p s o s Fal s o v i t i s Fal sos ne gat ivo s

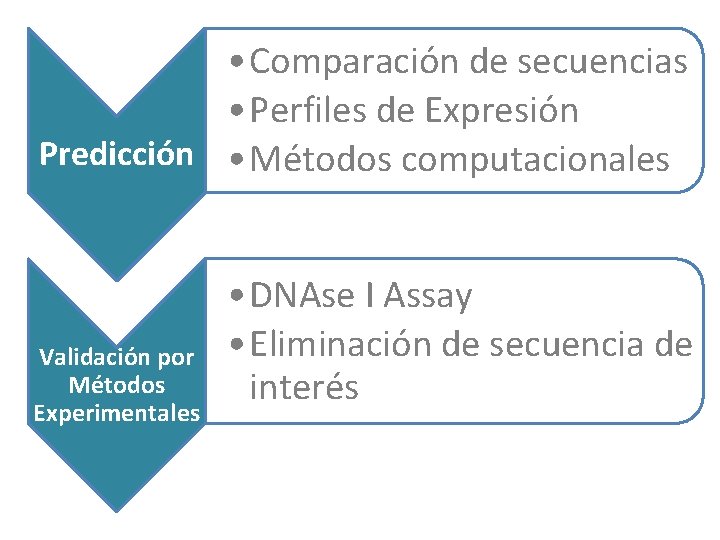
• Comparación de secuencias • Perfiles de Expresión Predicción • Métodos computacionales Validación por Métodos Experimentales • DNAse I Assay • Eliminación de secuencia de interés


Bibliografía • Riethoven JJ. , 2010. Regulatory regions in DNA: promoters, enhancers, silencers, and insulators. Methods Mol Biol. 74: 33 -42. • Griffiths AJF, Miller JH, Suzuki DT, et al. , 2000. An Introduction to Genetic Analysis. 7 th edition. New York: W. H. Freeman. • Loots GG. Genomic Identification of Regulatory Elements by Evolutionary Sequence Comparison and Functional Analysis. • Wang et al. , 2007. Detection of weakly conserved ancestral mammalian regulatory sequences by primate comparisons. Genome Biology. 8: R 1 • Pennacchio & Rubin. 2001. Genomic strategies to identify mammalian regulatory sequences. Net. Rev. Genetics. Vol 1 • Wasserman & Sandelin. 2004. Applied bioinformatics for the identification of regulatory elements. Nat. Rev. genetics vol 5 • Mishra M. Regulation of Gene Expression: Methods for Finding • Regulatory Regions in DNA Sequences
 Secuencias reguladoras
Secuencias reguladoras Lineas reguladoras
Lineas reguladoras Sustancias reguladoras
Sustancias reguladoras Proteinas reguladoras
Proteinas reguladoras Nrm 19
Nrm 19 Estructura gen eucariota
Estructura gen eucariota Que es genoma humana
Que es genoma humana Trasposones
Trasposones Genoma humano
Genoma humano Noticia del genoma humano
Noticia del genoma humano Cariótipo
Cariótipo Historia del proyecto genoma humano
Historia del proyecto genoma humano Genoma
Genoma Que es el proyecto genoma humano
Que es el proyecto genoma humano Que es el genoma humano
Que es el genoma humano La secuencia formativa
La secuencia formativa Secuencia gradiente de eco
Secuencia gradiente de eco Estructura de control de secuencia
Estructura de control de secuencia