Rle des transferts horizontaux dans lmergence du complexe




- Slides: 18

Rôle des transferts horizontaux dans l’émergence du complexe Mycobacterium tuberculosis Jennifer Becq 1, C. Gutierrez 3, V. Rosas Magallanes 2, J. Rauzier 2, B. Gicquel 2, O. Neyrolles 3, 4 & P. Deschavanne 1 1 Equipe de Bioinformatique Génomique et Moléculaire, Inserm U 726, Paris 7 2 Département Infection et Epidémiologie, Institut Pasteur, Paris 3 Unité de Génétique Mycobactérienne, Institut Pasteur, Paris 4 CNRS URA 2172, Institut Pasteur, Paris

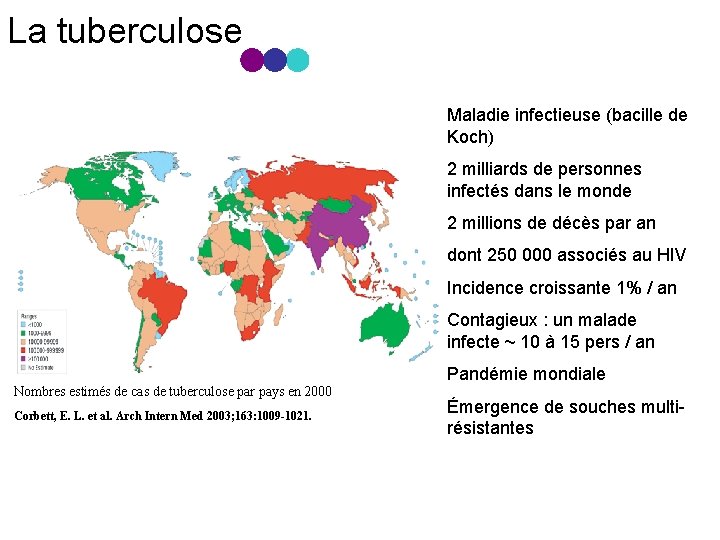
La tuberculose Maladie infectieuse (bacille de Koch) 2 milliards de personnes infectés dans le monde 2 millions de décès par an dont 250 000 associés au HIV Incidence croissante 1% / an Contagieux : un malade infecte ~ 10 à 15 pers / an Pandémie mondiale Nombres estimés de cas de tuberculose par pays en 2000 Corbett, E. L. et al. Arch Intern Med 2003; 163: 1009 -1021. Émergence de souches multi résistantes

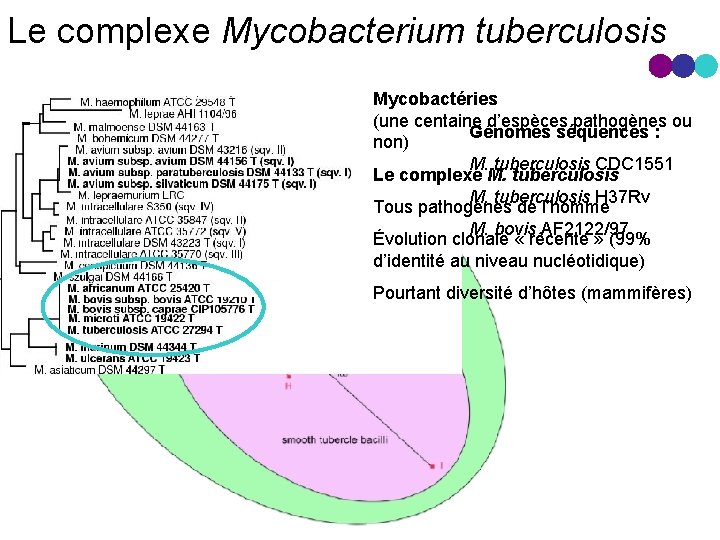
Le complexe Mycobacterium tuberculosis Mycobactéries (une centaine d’espèces pathogènes ou Génomes séquencés : non) M. tuberculosis CDC 1551 Le complexe M. tuberculosis H 37 Rv Tous pathogènes de l’homme M. bovis AF 2122/97 Évolution clonale « récente » (99% d’identité au niveau nucléotidique) Pourtant diversité d’hôtes (mammifères)

Les transferts horizontaux Facteurs d’évolution des génomes « îlots génomiques » « îlots de pathogénicité » Transfert de résistance aux antibiotiques de Doolittle 1999 Science ~ 4. 4 Mb ~ 66% G+C Pas de transferts horizontaux décrits Pas d’îlots de pathogénicité décrits Peu de gènes de virulence connus (mce, plc…) de Cole et al. 1998 Nature

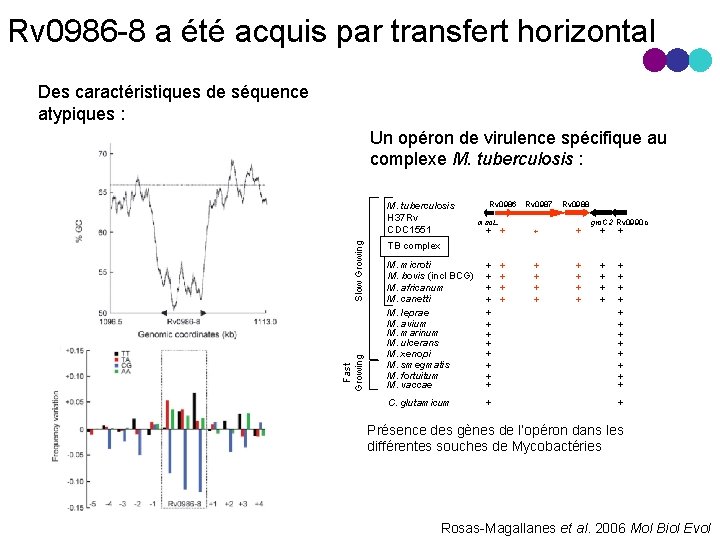
Rv 0986 8 a été acquis par transfert horizontal Des caractéristiques de séquence atypiques : Un opéron de virulence spécifique au complexe M. tuberculosis : Fast Growing Slow Growing M. tuberculosis H 37 Rv CDC 1551 Rv 0986 Rv 0987 msc. L Rv 0988 + + M. microti M. bovis (incl BCG) M. africanum M. canetti M. leprae M. avium M. marinum M. ulcerans M. xenopi M. smegmatis M. fortuitum M. vaccae + + + + + C. glutamicum + grc. C 2 Rv 0990 c + + + + + TB complex + + + Présence des gènes de l’opéron dans les différentes souches de Mycobactéries Rosas Magallanes et al. 2006 Mol Biol Evol

Recherche systématique de transferts horizontaux Les méthodes paramétriques : critère d’anormalité des TH Diversité des méthodes paramétriques Diversité des résultats (Ragan 2001, Dufraigne 2005). Élaboration d’un consensus de méthodes spécificité ou sensibilité 3 méthodes différentes : % GC position 1 et 3 usage des codons signature génomique Spécificité Consensus 2 / 3

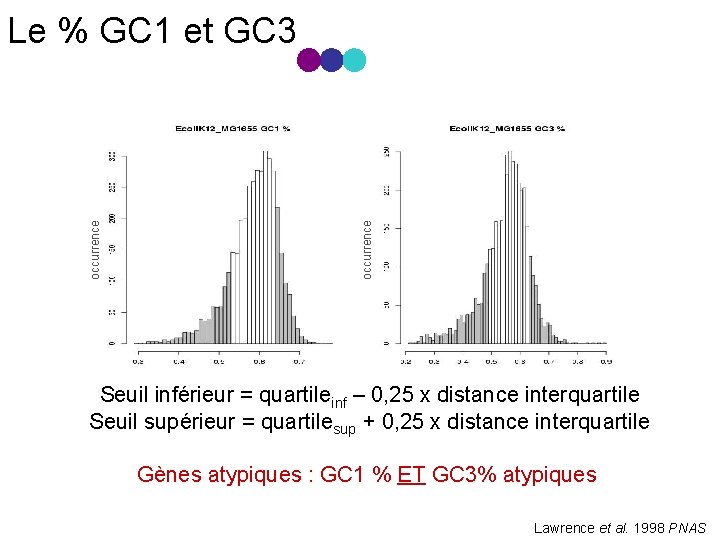
occurrence Le % GC 1 et GC 3 Seuil inférieur = quartileinf – 0, 25 x distance interquartile Seuil supérieur = quartilesup + 0, 25 x distance interquartile Gènes atypiques : GC 1 % ET GC 3% atypiques Lawrence et al. 1998 PNAS

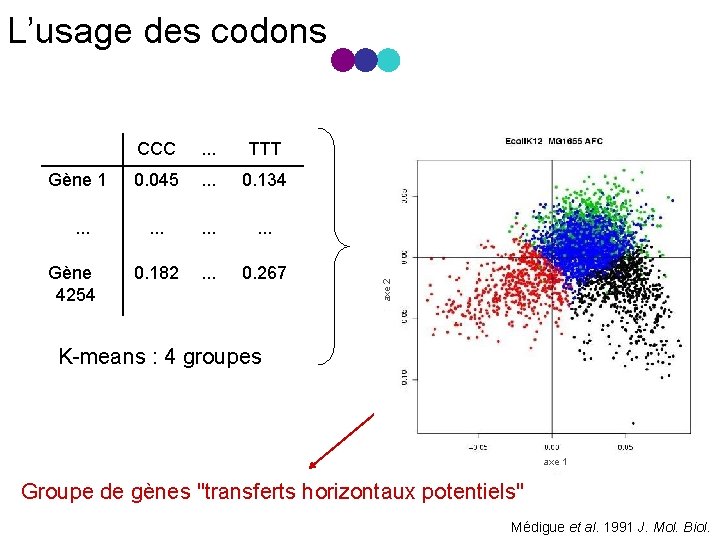
CCC . . . TTT Gène 1 0. 045 . . . 0. 134 . . . 0. 182 . . . 0. 267 Gène 4254 axe 2 L’usage des codons K means : 4 groupes axe 1 Groupe de gènes "transferts horizontaux potentiels" Médigue et al. 1991 J. Mol. Biol.

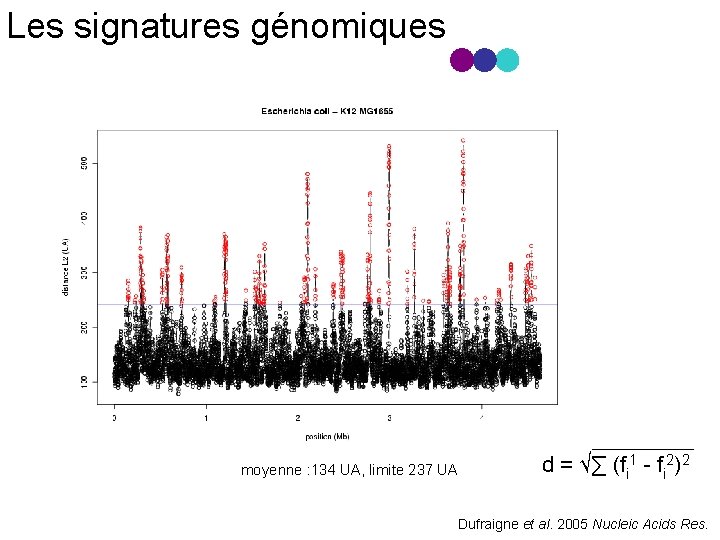
Les signatures génomiques Génome de E. coli K 12 MG 1655 mots CCCC AAAA 1 4, 6 Mb Position moyenne : 134 UA, limite 237 UA d = √∑ (fi 1 fi 2)2 Dufraigne et al. 2005 Nucleic Acids Res.

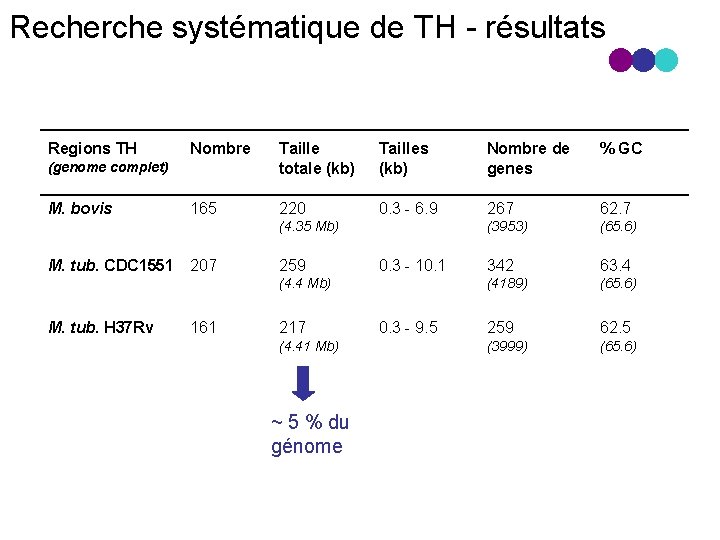
Recherche systématique de TH résultats Regions TH Nombre Taille totale (kb) Tailles (kb) Nombre de genes % GC 165 220 0. 3 6. 9 267 62. 7 (3953) (65. 6) 342 63. 4 (4189) (65. 6) 259 62. 5 (3999) (65. 6) (genome complet) M. bovis (4. 35 Mb) M. tub. CDC 1551 207 259 0. 3 10. 1 (4. 4 Mb) M. tub. H 37 Rv 161 217 (4. 41 Mb) ~ 5 % du génome 0. 3 9. 5

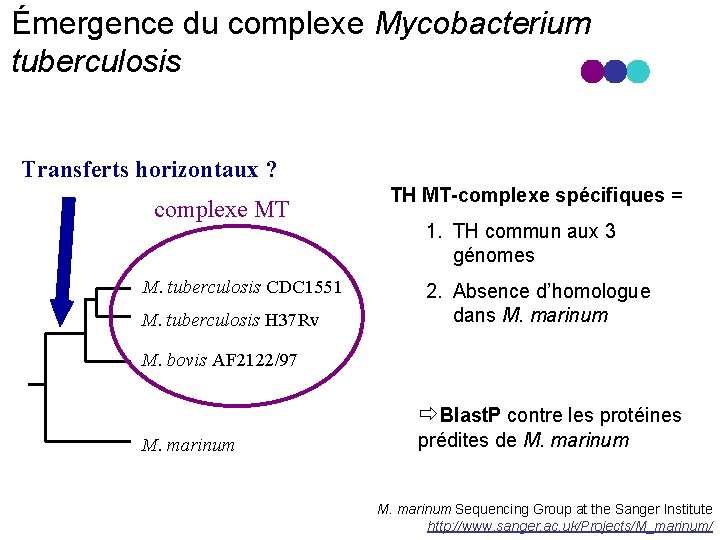
Émergence du complexe Mycobacterium tuberculosis Transferts horizontaux ? complexe MT M. tuberculosis CDC 1551 M. tuberculosis H 37 Rv TH MT-complexe spécifiques = 1. TH commun aux 3 génomes 2. Absence d’homologue dans M. marinum M. bovis AF 2122/97 Blast. P contre les protéines M. marinum prédites de M. marinum Sequencing Group at the Sanger Institute http: //www. sanger. ac. uk/Projects/M_marinum/

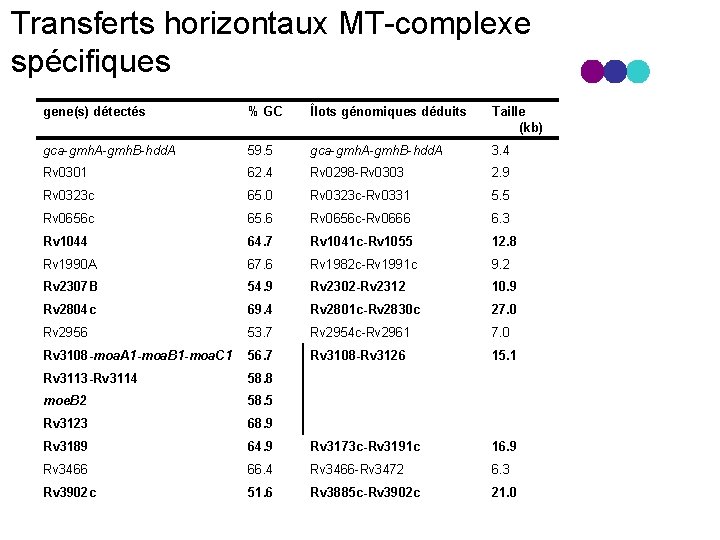
Transferts horizontaux MT complexe spécifiques gene(s) détectés % GC Îlots génomiques déduits Taille (kb) gca gmh. A gmh. B hdd. A 59. 5 gca gmh. A gmh. B hdd. A 3. 4 Rv 0301 62. 4 Rv 0298 Rv 0303 2. 9 Rv 0323 c 65. 0 Rv 0323 c Rv 0331 5. 5 Rv 0656 c 65. 6 Rv 0656 c Rv 0666 6. 3 Rv 1044 64. 7 Rv 1041 c-Rv 1055 12. 8 Rv 1990 A 67. 6 Rv 1982 c Rv 1991 c 9. 2 Rv 2307 B 54. 9 Rv 2302 -Rv 2312 10. 9 Rv 2804 c 69. 4 Rv 2801 c-Rv 2830 c 27. 0 Rv 2956 53. 7 Rv 2954 c Rv 2961 7. 0 Rv 3108 -moa. A 1 -moa. B 1 -moa. C 1 56. 7 Rv 3108 -Rv 3126 15. 1 Rv 3113 -Rv 3114 58. 8 moe. B 2 58. 5 Rv 3123 68. 9 Rv 3189 64. 9 Rv 3173 c-Rv 3191 c 16. 9 Rv 3466 66. 4 Rv 3466 Rv 3472 6. 3 Rv 3902 c 51. 6 Rv 3885 c-Rv 3902 c 21. 0

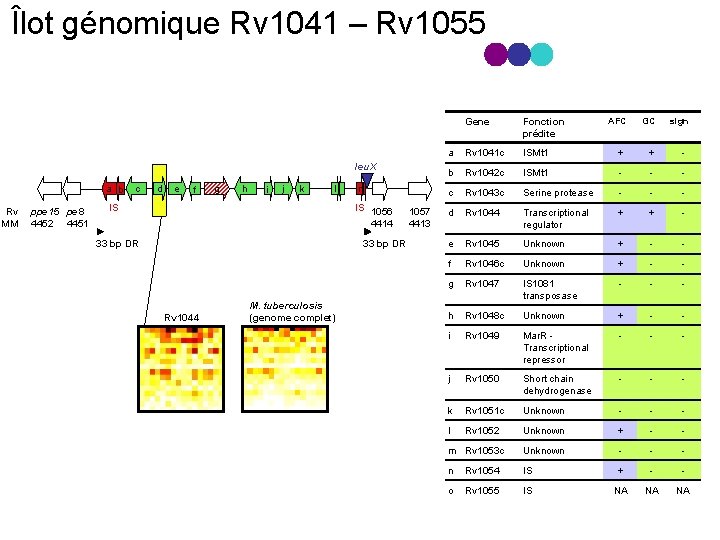
Îlot génomique Rv 1041 – Rv 1055 leu. X a b Rv MM ppe 15 pe 8 4452 4451 c d e f g h i j k l IS n IS 1056 4414 33 bp DR Rv 1044 M. tuberculosis (genome complet) 1057 4413 AFC GC sign Gene Fonction prédite a Rv 1041 c ISMt 1 + + b Rv 1042 c ISMt 1 c Rv 1043 c Serine protease d Rv 1044 Transcriptional regulator + + e Rv 1045 Unknown + f Rv 1046 c Unknown + g Rv 1047 IS 1081 transposase h Rv 1048 c Unknown + i Rv 1049 Mar. R Transcriptional repressor j Rv 1050 Short chain dehydrogenase k Rv 1051 c Unknown l Rv 1052 Unknown + m Rv 1053 c Unknown n Rv 1054 IS + o Rv 1055 IS NA NA NA

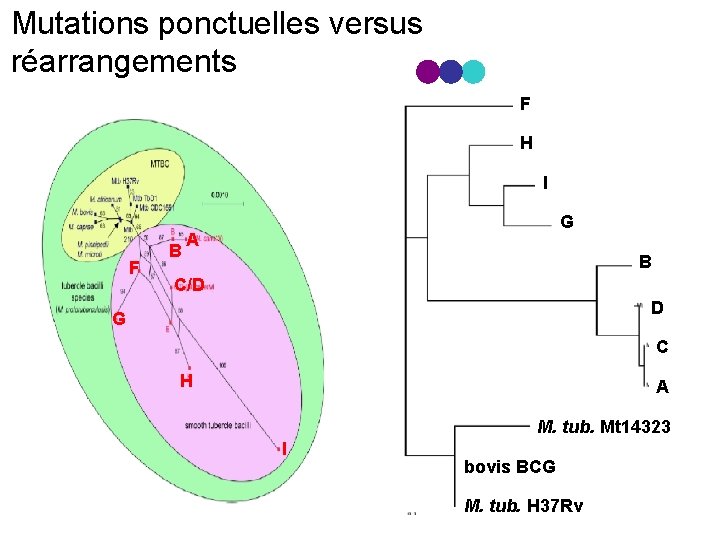
Rv 0301 Rv 0323 c Rv 0656 c Rv 1044 Rv 1053 c Rv 1990 A Rv 2307 B Rv 2804 c Rv 2819 c Rv 2956 Rv 3108 Rv 3123 Rv 3178 Rv 3189 Rv 3466 Présence / absence chez les prototuberculosis (Southern blot) A + + + B + + + C + + + D + + + F + + + + G + + + H + + + + I + + + + M. tub. H 37 Rv + + + + M. bovis BCG + + + + M. tub. Mt 14323 + + + + groupes « M. prototub. »

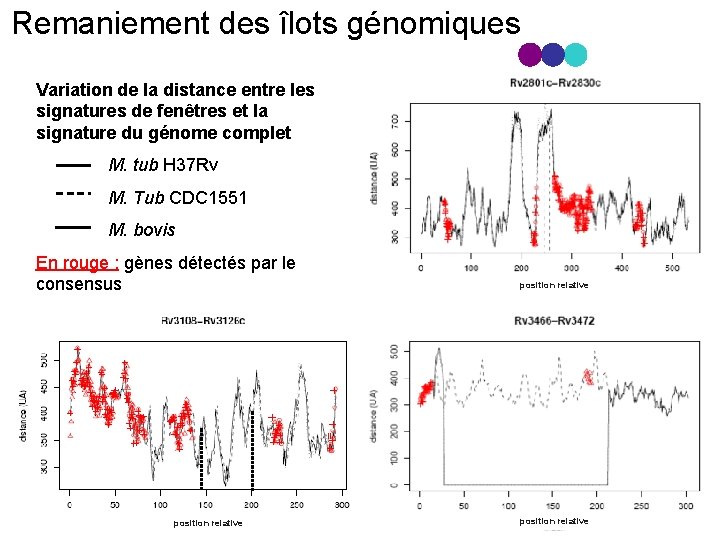
Remaniement des îlots génomiques Variation de la distance entre les signatures de fenêtres et la signature du génome complet M. tub H 37 Rv M. Tub CDC 1551 M. bovis En rouge : gènes détectés par le consensus position relative

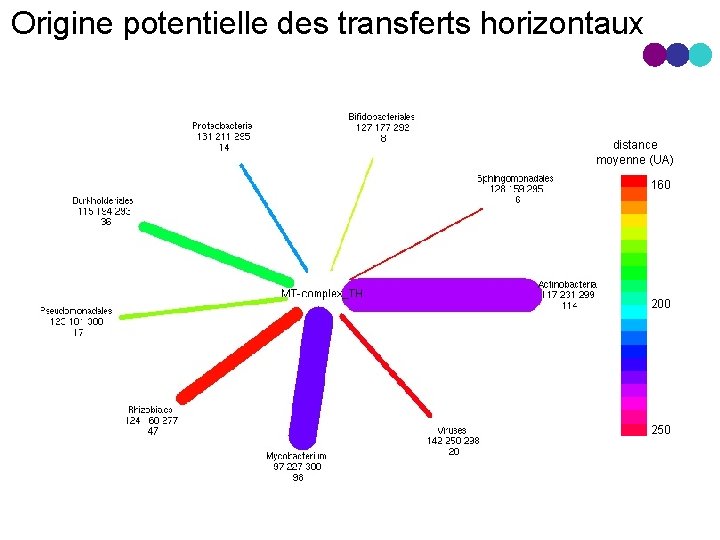
Origine potentielle des transferts horizontaux distance moyenne (UA) 160 200 250

Conclusions et perspectives Détection des transferts horizontaux 1. Critères paramétriques anormaux 2. Environnement génomique propice aux transferts (IS, régions répétées, PE_PGRS/PPE, ARNt) 3. Remaniement des régions Importance des transferts horizontaux dans l’évolution du complexe Mycobacterium tuberculosis Perspectives Étude fonctionnelle des gènes impliqués dans les transferts horizontaux (gènes de virulence ? ) Caractérisation des TH propres à chaque génome (spécificité de chacune des souches/espèces ? ) Extension à des souches cliniques de M. tuberculosis

Mutations ponctuelles versus réarrangements F H I F B G A B C/D D G C H A M. tub. Mt 14323 I bovis BCG M. tub. H 37 Rv