QJ 1210253 Vyuit metod molekulrn genetiky jako nstroje





- Slides: 12

QJ 1210253 Využití metod molekulární genetiky jako nástroje pro efektivní plemenářskou práci v malé populaci prasat C 001 Detekce a monitoring molekulárně genetické struktury C 002 Implementace genomických markerů do šlechtitelské práce Řešitel Laboratoř agrogenomiky, ÚMFGZ, Mendelu Brno Ing. Irena Vrtková, Ph. D. Poznáte “přeštíka”? - genetika ano A B C

Identifikace a dohledatelnost u PC – MS Zaveden panel tetranukleodtidových MS pro identifikaci, parentitu a dohledatelnost. V rámci projektu udělena LAG Mendelu mezinárodní akreditace ČIA dle normy ČSN 17025. Následně implementován do ověřování parentit a stanovování genetických typů všech plemen prasat v ČR. U plemene Přeštické černostrakaté – genový zdroj – zaznamenány nové, u jiných plemen neznámé, alely v markerech 387 A 12 F, S 0655, SBH 2, SBH 10, SBH 18, SBH 19. Nejdůležitější populační parametry u PC – ukazatele pro plemenářskou práci Skutečná heterozygotnost – HO = 0. 68 Polymorfní informační obsah – PIC = 0. 66 Fixační index – F = 0. 03 (molekulární inbreeding) – parametry pro dohledatelnost Pravděpodobnost shodných genotypů u nepříbuzných jedinců – PI = 4. 037· 10 -11 Pravděpodobnost shodných genotypů u příbuzných jedinců – PISibs = 8. 315· 10 -5 – hodnoty všech parametrů ukazují vhodnost pro forenzní použití Doporučení: po výběru nových prasnic a kanců do plemenitby (více jak 30 ks) provést korekci citovaných parametrů Vrtková, I. , Vrtek, Š. , Falková, L. 2016: Efficiency of tetrameric Short Tandem Repeats in Prestice Black-Pied pig for traceability and parentity testing, Acta Universitatis Agriculturae et Silviculturae Mendelianae Brunensis, 64, 2, 557 -565. Vrtková, I. 2015: Genetic Admixture Analysis in Prestice Black Pied Pig. Archiv für Tierzucht (Archives of Animal Breeding), 58, 2015, 1, 115 -121 Genové zdroje stejného zbarvení z předchozího obrázku: A – Cinta Senese, B – Přeštické černostrakaté, C – Schwäbisch-Hällisches

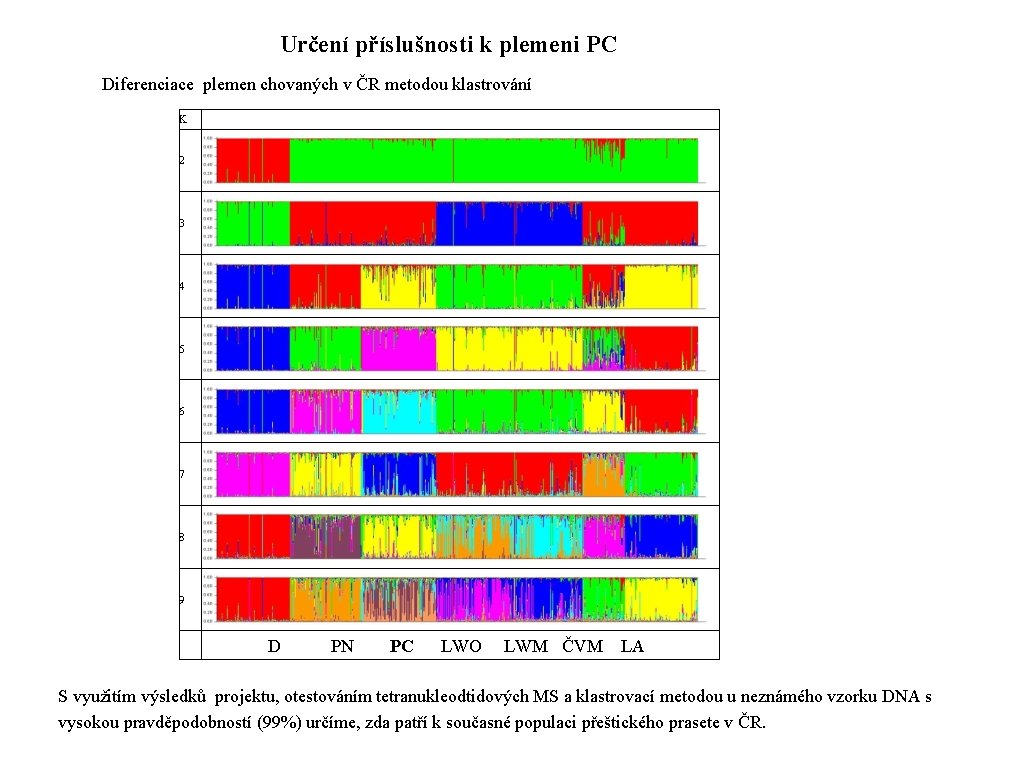
Určení příslušnosti k plemeni PC Diferenciace plemen chovaných v ČR metodou klastrování K 2 3 4 5 6 7 8 9 D PN PC LWO LWM ČVM LA S využitím výsledků projektu, otestováním tetranukleodtidových MS a klastrovací metodou u neznámého vzorku DNA s vysokou pravděpodobností (99%) určíme, zda patří k současné populaci přeštického prasete v ČR.

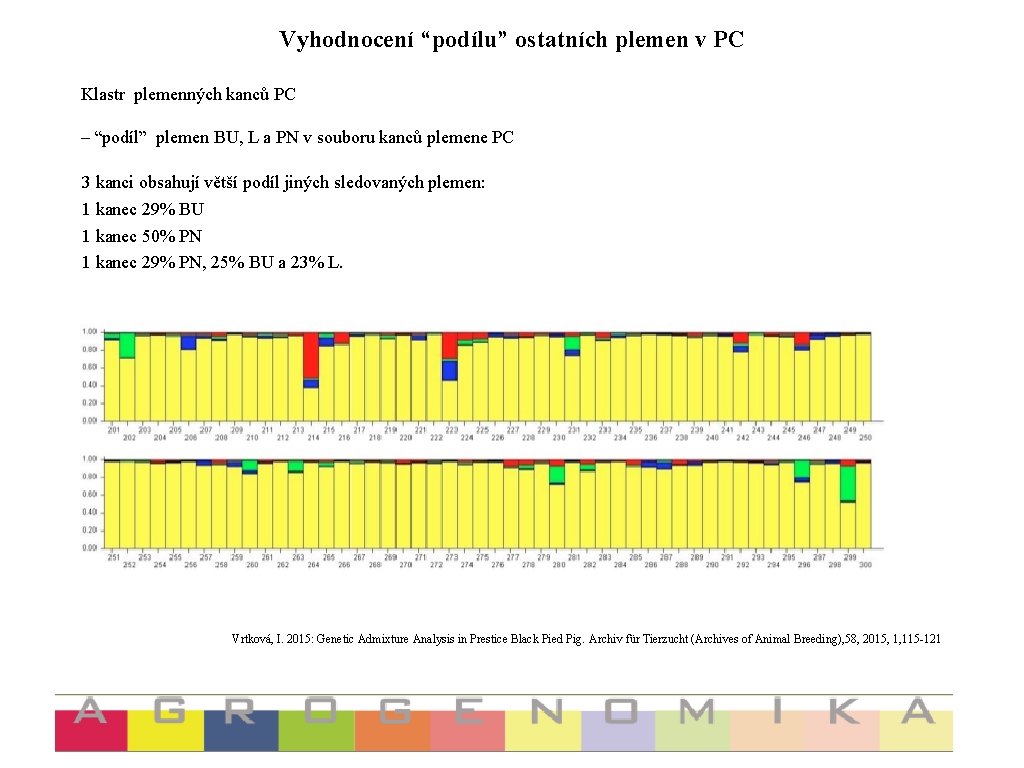
Vyhodnocení “podílu” ostatních plemen v PC Klastr plemenných kanců PC – “podíl” plemen BU, L a PN v souboru kanců plemene PC 3 kanci obsahují větší podíl jiných sledovaných plemen: 1 kanec 29% BU 1 kanec 50% PN 1 kanec 29% PN, 25% BU a 23% L. Vrtková, I. 2015: Genetic Admixture Analysis in Prestice Black Pied Pig. Archiv für Tierzucht (Archives of Animal Breeding), 58, 2015, 1, 115 -121

Identifikace a dohledatelnost u PC – SNP Marker MC 1 R – zbarvení Fontanesi, L. 2016: Authentication of “mono-breed” pork products: Identification of a coat colour gene marker in Cinta Senese pigs useful to this purpose, Livestock Science, 184, 71 -77 Fontanesi, L. 2014: Differentiation of meat from European wild boars and domestic pigs using polymorphisms in the MC 1 R and NR 6 A 1 genes, Meat Science, 98, 781784 Frekvence alel a genotypů MC 1 R u PC ED 2 ED 1 ED 2 /ED 1 0. 81 0. 19 0. 62 0. 38 Určení původu masa z PC lze vedle pomocí klastrování MS doplnit detekcí polymorfismů genu MC 1 R metodou PCR-RFLP. U masa ze současné populace PC v ČR musí být přítomna alela ED 2 Detekcí polymorfismů v kodonech 121 a 240 genu MC 1 R lze v masných výrobcích identifikovat přítomnost masa z divokého prasete.

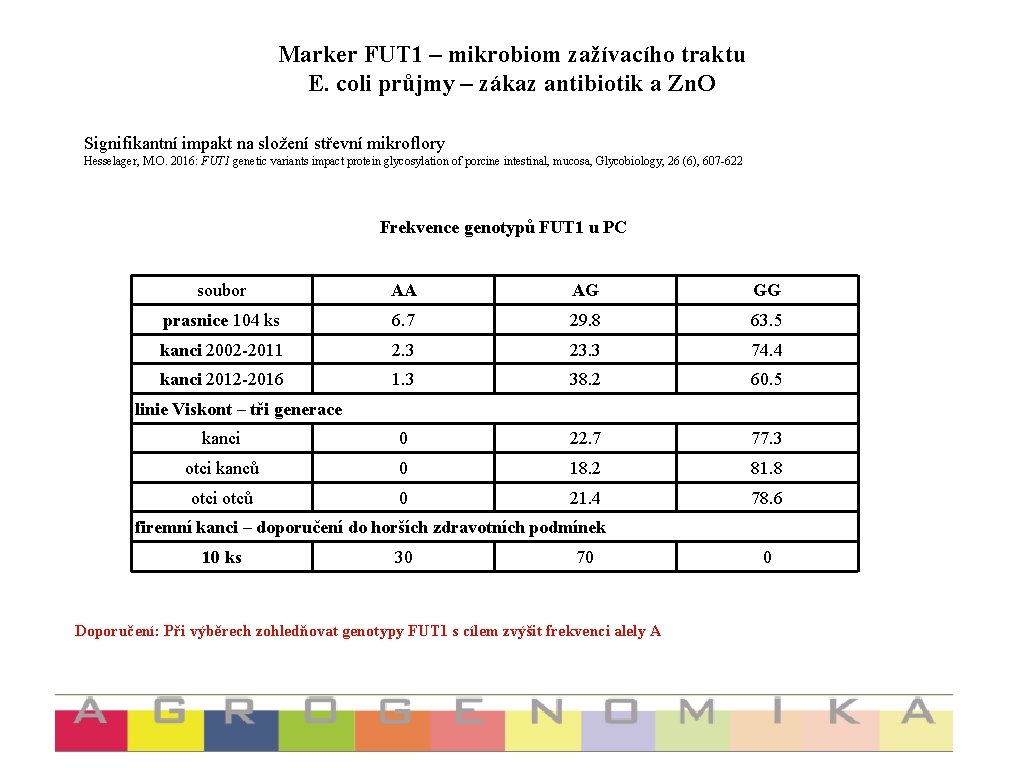
Marker FUT 1 – mikrobiom zažívacího traktu E. coli průjmy – zákaz antibiotik a Zn. O Signifikantní impakt na složení střevní mikroflory Hesselager, M. O. 2016: FUT 1 genetic variants impact protein glycosylation of porcine intestinal, mucosa, Glycobiology, 26 (6), 607 -622 Frekvence genotypů FUT 1 u PC soubor AA AG GG prasnice 104 ks 6. 7 29. 8 63. 5 kanci 2002 -2011 2. 3 23. 3 74. 4 kanci 2012 -2016 1. 3 38. 2 60. 5 kanci 0 22. 7 77. 3 otci kanců 0 18. 2 81. 8 otci otců 0 21. 4 78. 6 linie Viskont – tři generace firemní kanci – doporučení do horších zdravotních podmínek 10 ks 30 70 Doporučení: Při výběrech zohledňovat genotypy FUT 1 s cílem zvýšit frekvenci alely A 0

Ukázka působení patogenní E. coli

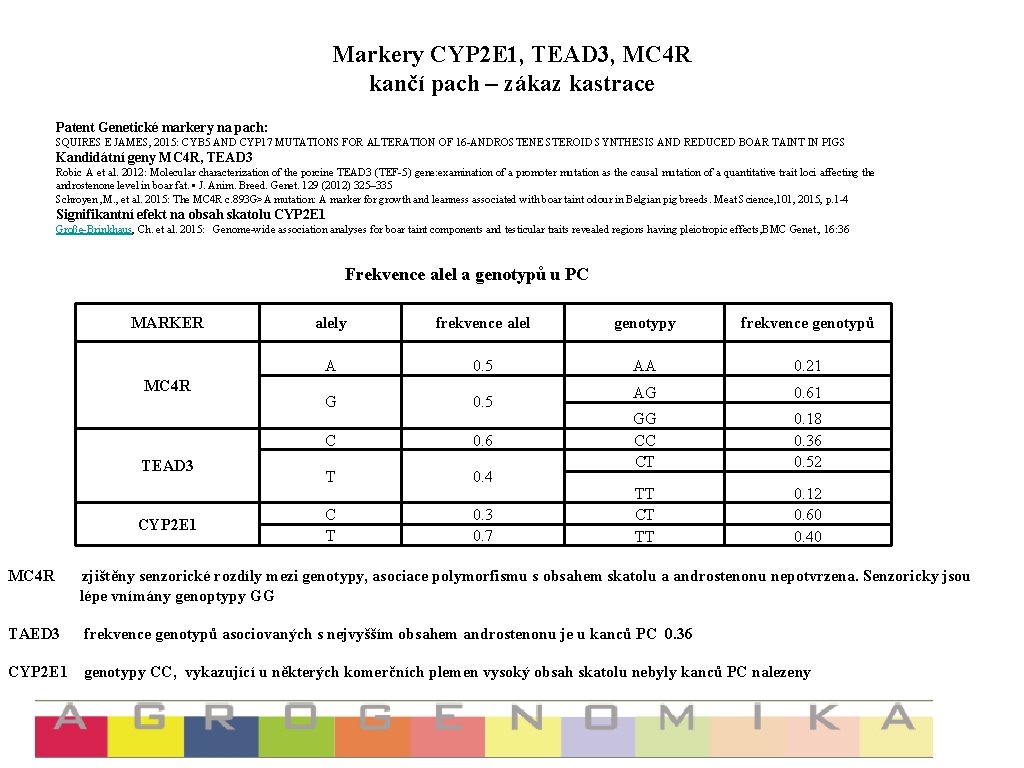
Markery CYP 2 E 1, TEAD 3, MC 4 R kančí pach – zákaz kastrace Patent Genetické markery na pach: SQUIRES E JAMES, 2015: CYB 5 AND CYP 17 MUTATIONS FOR ALTERATION OF 16 -ANDROSTENE STEROID SYNTHESIS AND REDUCED BOAR TAINT IN PIGS Kandidátní geny MC 4 R, TEAD 3 Robic A et al. 2012: Molecular characterization of the porcine TEAD 3 (TEF-5) gene: examination of a promoter mutation as the causal mutation of a quantitative trait loci affecting the androstenone level in boar fat. • J. Anim. Breed. Genet. 129 (2012) 325– 335 Schroyen , M. , et al. 2015: The MC 4 R c. 893 G>A mutation: A marker for growth and leanness associated with boar taint odour in Belgian pig breeds. Meat Science, 101, 2015, p. 1 -4 Signifikantní efekt na obsah skatolu CYP 2 E 1 Große-Brinkhaus, Ch. et al. 2015: Genome-wide association analyses for boar taint components and testicular traits revealed regions having pleiotropic effects, BMC Genet. , 16: 36 Frekvence alel a genotypů u PC MARKER MC 4 R TEAD 3 CYP 2 E 1 alely frekvence alel genotypy frekvence genotypů A 0. 5 AA 0. 21 G 0. 5 AG 0. 61 C 0. 6 T 0. 4 GG CC CT 0. 18 0. 36 0. 52 C T 0. 3 0. 7 TT CT TT 0. 12 0. 60 0. 40 MC 4 R zjištěny senzorické rozdíly mezi genotypy, asociace polymorfismu s obsahem skatolu a androstenonu nepotvrzena. Senzoricky jsou lépe vnímány genoptypy GG TAED 3 frekvence genotypů asociovaných s nejvyšším obsahem androstenonu je u kanců PC 0. 36 CYP 2 E 1 genotypy CC, vykazující u některých komerčních plemen vysoký obsah skatolu nebyly kanců PC nalezeny

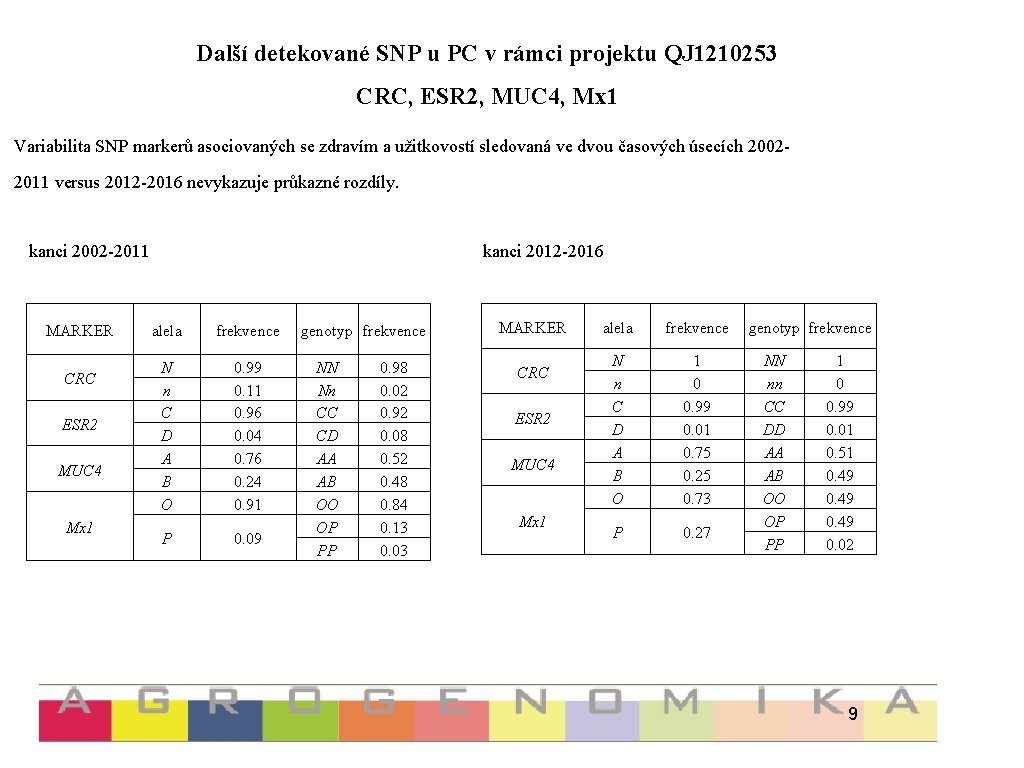
Další detekované SNP u PC v rámci projektu QJ 1210253 CRC, ESR 2, MUC 4, Mx 1 Variabilita SNP markerů asociovaných se zdravím a užitkovostí sledovaná ve dvou časových úsecích 20022011 versus 2012 -2016 nevykazuje průkazné rozdíly. kanci 2002 -2011 MARKER CRC ESR 2 MUC 4 Mx 1 kanci 2012 -2016 alela frekvence N n C D A B O 0. 99 0. 11 0. 96 0. 04 0. 76 0. 24 0. 91 P 0. 09 genotyp frekvence NN Nn CC CD AA AB OO OP PP 0. 98 0. 02 0. 92 0. 08 0. 52 0. 48 0. 84 0. 13 0. 03 MARKER CRC ESR 2 MUC 4 Mx 1 alela frekvence N n C D A B O 1 0 0. 99 0. 01 0. 75 0. 25 0. 73 P 0. 27 genotyp frekvence NN nn CC DD AA AB OO OP PP 1 0 0. 99 0. 01 0. 51 0. 49 0. 02 9

Závěr Dílčí cíle “Detekce a monitoring molekulárně genetické struktury” “Implementace genomických markerů do šlechtitelské práce” byly, jak vyplývá z předcházejících údajů, splněny v celém rozsahu. Na základě našich výsledků (8900 genotypů MS, 1650 genotypů SNP) navrhujeme pro plemeno PC: 1. U všech plemenných kanců a prasnic určovat DNA profily Využívat je k: – ověřování parentit – hodnocení inbreedingu – příslušnosti zvířat k plemeni – dohledatenosti a kontrole původu masa z PC – forenzní účely 2. U všech kanců a vybraných prasnic určovat genotypy markerů FUT 1, CYP 2 E 1 a MC 1 R – genotypy využívat při hodnocení zvířat Jsme přesvědčeni, že výsledky z projektu a obecně poznatky genomiky by měly být v udržitelném programu chovu přeštického prasete v ČR zakotveny.

Přehled publikací dedikovaných výhradně k projektu QJ 1210253 Jimp Vrtková, I. 2015: Genetic Admixture Analysis in Prestice Black Pied Pig. Archiv für Tierzucht (Archives of Animal Breeding), 58, 2015, 1, 115 -121 Vrtková, I. , Filistowicz, A. , Buczyński, J. T. , Vrtek, Š. , Falková, L. : Parameters of variability and diversity of tetrameric STRs for practical use at Zlotnicka White and Zlotnicka Spotted pigs. Animal Science Papers and Reports, v oponentním řízení Jsc Vrtková, I. , Vrtek, Š. , Falková, L. 2016: Efficiency of tetrameric Short Tandem Repeats in Prestice Black-Pied pig for traceability and parentity testing, Acta Universitatis Agriculturae et Silviculturae Mendelianae Brunensis, 64, 2, 557 -565. Jrec Vrtková I. , Stehlík L. , Putnová L. , Kratochvílová L. , Falková L 2012. : GENETIC STRUCTURE IN THREE BREEDS OF PIGS POPULATIONS USING MICROSA- TELLITE MARKERS IN THE CZECH REPUBLIC, Research in Pig Breeding, 6, 2012 (2): 83 -87. ISSN 1802 -7547 Vrtková I. , Stehlík L. , Putnová L. 2013: Research of variability in Prestice Black Pied pig using dinucleotide and tetranucleotide microsatellite markers. Research in Pig Breeding, 7, 2013 (1): 9 -14. ISSN 1802 -7547 Falková, L. , Vrtková, I. , Kratochvílová, L. 2014: Boar SNP variability in genetic resource Přeštice Black-Pied pig. Research in Pig Breeding, 8, 2014 (2): 4 -7. ISSN 1802 -7547 článek ve sborníku Vrtková I. , Stehlík L. , Putnová L. , Kratochvílová L. , Falková L. : Validace biomarkerů pro monitoring vývoje genetické diverzity Přeštického černostrakatého prasete. In šlechtění na masnou užitkovost a aktuální otázky produkce jatečných zvířat. 1. vyd. Brno: Mendelova univerzita v Brně, 2012, s. 182 -185. ISBN 978 -80 -7375 -645 -1. abstrakt ve sborníku Vrtková I. , Stehlík L. , Putnová L. , Kratochvílová L. , Falková L. : Genetic structure of Czech pig populations for traceability usage. Research in Pig Breeding – workshop. Kostelec nad Orlicí–Vrbice: VÚŽV, 2012. s. 31 ISBN 978 -80 -7403 -101 -4. Vrtková I. , Stehlík L. , Putnová L. , Kratochvilova L. , Falkova L. : Genetic structure in four selected pig populations of Czech Republic using mcrosatellite markers. Book of Abstracts of the 64 th Annual Meeting of EAAP No. 19, 2013, 336, ISBN 978 -90 -8686 -228 -3 Falková, L. , Vrtková, I. , Kratochvílová, L. : Boar SNP variability in genetic resource Přeštice Black-Pied pig. Workshop Research in Pig Breeding. Kostelec nad Orlicí - Vrbice. 2014, ISBN 978 -80 -7403129 -8, s. 9 Falková L. , Vrtková I. , Kratochvílová, L. : Next alleles describe in tetranucleotide STR markers in the Czech genetic resource Prestice Black-Pied pig. VIth International Scientific Symposium for Ph. D Students and Students of Agriculture Colleges “Innovative researches for the future of agriculture and rural areas development” Bydgoszcz – Ciechocinek, 17 -19 September 2015, první vydání, 200 výtisků, s. 43 Falková L. , Vrtková I. : Comparison of SNP variability between two over time clusters of Prestice Black-Pied pig boars. Workshop Research in Pig Breeding. Kostelec nad Orlicí - Vrbice. 2015, ISBN 978 -80 -7403 -143 -4, s. 14 Falková, L. , Vrtková, I. 2016: Genetic variability in eight genes related to meat quality and health traits in genetic resource pig breed- Přeštice Black-Pied pig. VIIth International Scientific Symposium for Young Scientists, Ph. D Students and Students of Agriculture Colleges “Innovative researches for the future of agriculture and rural areas development” Bydgoszcz, 15 -17 September 2016, první vydání, 150 výtisků, s. 43 Falková, L. , Vrtková, I. 2016: A null allele and next alleles detected in STR markers in Prestice Black-Pied pig. Workshop Research in Pig Breeding. Kostelec nad Orlicí - Vrbice. 2016, ISBN 978 -807403 -154 -0, s. 12 informační materiály Vrtková, I. : Kančí pach. Materiály pro chovatelskou veřejnost, 2012 informace genetické diverzity v databázi EFABIS databáze EFABIS – genetic features: www. efabis. net – breeds – breed data sheets – czech republic – pig – presticke cernostrakate prase – submit – breed morphology information – genetic features.

Děkuji za pozornost Genom prasete Prezentace zveřejněna na www. lamgen. cz
 Dejiny genetiky
Dejiny genetiky Základní dogma molekulární genetiky vyvrácení
Základní dogma molekulární genetiky vyvrácení Wady pkb jako miernika
Wady pkb jako miernika Siła jako przyczyna zmian ruchu wzory
Siła jako przyczyna zmian ruchu wzory Rozčepýřený jako
Rozčepýřený jako Polarita rozpouštědel
Polarita rozpouštědel Maud adams jako johanka z arku
Maud adams jako johanka z arku Rodzaje oddziaływań w przyrodzie
Rodzaje oddziaływań w przyrodzie Gatunek
Gatunek Hawaiian alphabet
Hawaiian alphabet Obserwacja standaryzowana i niestandaryzowana
Obserwacja standaryzowana i niestandaryzowana Zasady zdrowego stylu życia prezentacja
Zasady zdrowego stylu życia prezentacja Ziemia jako kondensator
Ziemia jako kondensator