Giacomo Monticelli Universit degli studi del Piemonte Orientale





- Slides: 10

Giacomo Monticelli Università degli studi del Piemonte Orientale «A. Avogadro» Laboratorio di Biologia Molecolare Dipartimento di Scienze della Salute – Novara Caratterizzazione del ruolo di USP 10 all’interno della risposta al danno al DNA

Introduzione • Le cellule vengono continuamente esposte ad una moltitudine di lesioni a livello del DNA; per combattere le minacce e riparare il proprio materiale genetico, esse hanno sviluppato un complesso meccanismo di sorveglianza chiamato Risposta al Danno al DNA (DDR – DNA Damage Response). • RNF 168 è una proteina nucleare che, grazie ai suoi domini che legano l’ubiquitina è in grado di riconoscere l’ubiquitina stessa ed essere in questo modo reclutata ai foci di danno, in cui ubiquitina gli istoni H 2 A ed H 2 A. X. • Inizialmente l’ubiquitinazione fu caratterizzata come processo di marcatura per l’indirizzamento delle proteine verso la degradazione proteolitica. Più recentemente, invece, è emerso un ruolo di tipo non degradativo a carico di questa modificazione post-traduzionale. L’aggiunta, da parte di ubiquitin-ligasi, di monomeri di ubiquitina sulla lisina 63 (K 63), della prima ubiquitina aggiunta al substrato, è largamente associato con la riparazione del danno al DNA. • Il laboratorio in cui ho svolto la mia tesi è focalizzato sulla caratterizzazione di RNF 168 all’interno del DDR. Grazie ad una analisi di spettrometria di massa, sono stati individuati diversi interattori tra cui USP 10 è una deubiquitinasi (DUB) capace di indurre la stabilizzazione di p 53. • Il mio lavoro di tesi ha contribuito a caratterizzare il ruolo di USP 10 nella risposta al danno al DNA.

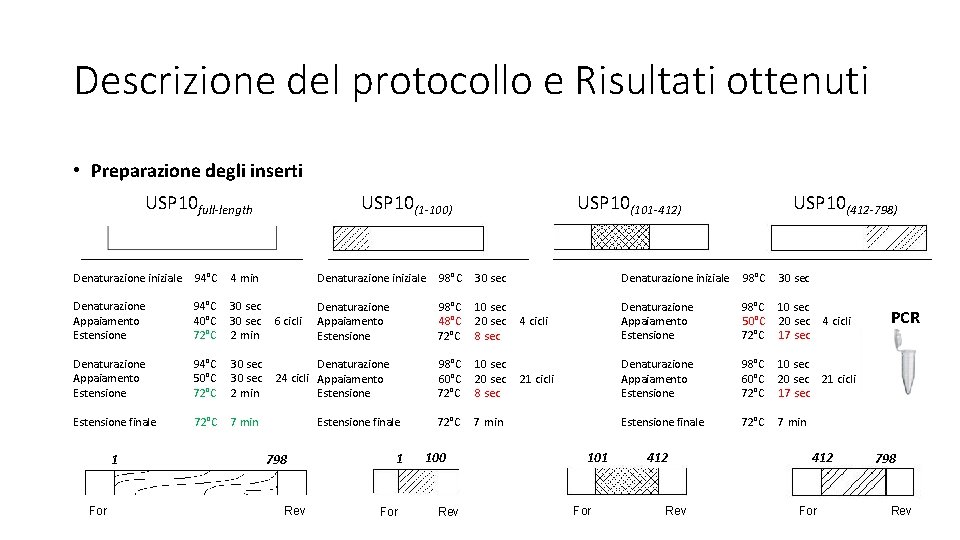
Descrizione del protocollo e Risultati ottenuti • Preparazione degli inserti USP 10 full-length USP 10(1 -100) USP 10(101 -412) USP 10(412 -798) Denaturazione iniziale 94°C 4 min Denaturazione iniziale 98°C 30 sec Denaturazione Appaiamento Estensione 94°C 40°C 72°C 30 sec 2 min 6 cicli Denaturazione Appaiamento Estensione 98°C 48°C 72°C 10 sec 20 sec 8 sec 4 cicli Denaturazione Appaiamento Estensione 98°C 50°C 72°C 10 sec 20 sec 17 sec 4 cicli Denaturazione Appaiamento Estensione 94°C 50°C 72°C 30 sec 2 min Denaturazione 24 cicli Appaiamento Estensione 98°C 60°C 72°C 10 sec 20 sec 8 sec 21 cicli Denaturazione Appaiamento Estensione 98°C 60°C 72°C 10 sec 20 sec 17 sec 21 cicli Estensione finale 72°C 7 min 1 For Estensione finale 798 Rev 1 For 100 Rev 101 For 412 Rev 412 For PCR 798 Rev

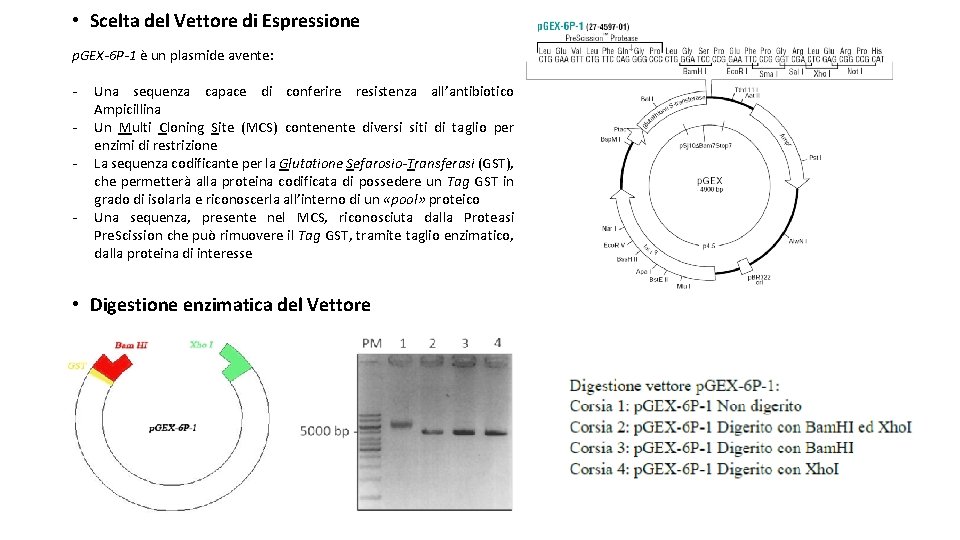
• Scelta del Vettore di Espressione p. GEX-6 P-1 è un plasmide avente: - Una sequenza capace di conferire resistenza all’antibiotico Ampicillina Un Multi Cloning Site (MCS) contenente diversi siti di taglio per enzimi di restrizione La sequenza codificante per la Glutatione Sefarosio-Transferasi (GST), che permetterà alla proteina codificata di possedere un Tag GST in grado di isolarla e riconoscerla all’interno di un «pool» proteico Una sequenza, presente nel MCS, riconosciuta dalla Proteasi Pre. Scission che può rimuovere il Tag GST, tramite taglio enzimatico, dalla proteina di interesse • Digestione enzimatica del Vettore

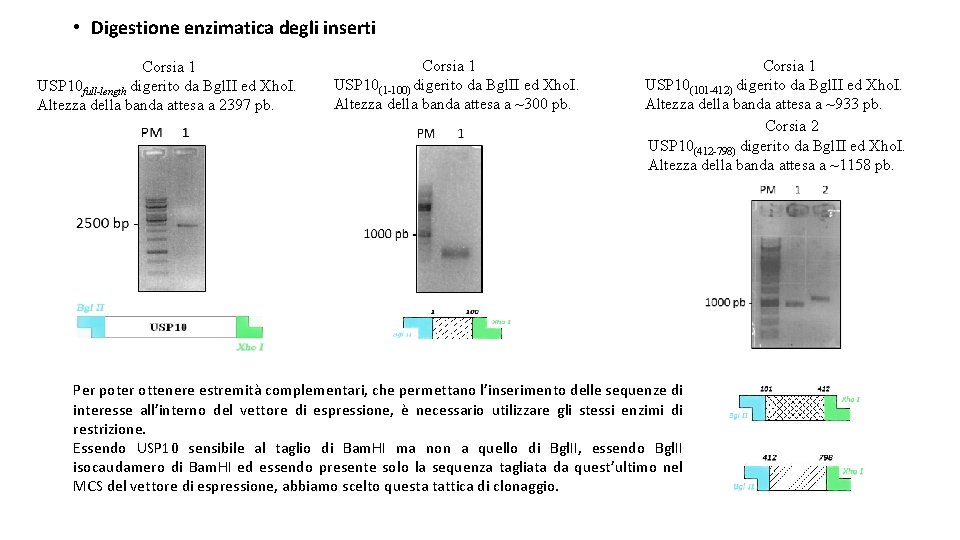
• Digestione enzimatica degli inserti Corsia 1 USP 10 full-length digerito da Bgl. II ed Xho. I. Altezza della banda attesa a 2397 pb. Corsia 1 USP 10(1 -100) digerito da Bgl. II ed Xho. I. Altezza della banda attesa a ~300 pb. Corsia 1 USP 10(101 -412) digerito da Bgl. II ed Xho. I. Altezza della banda attesa a ~933 pb. Corsia 2 USP 10(412 -798) digerito da Bgl. II ed Xho. I. Altezza della banda attesa a ~1158 pb. Per poter ottenere estremità complementari, che permettano l’inserimento delle sequenze di interesse all’interno del vettore di espressione, è necessario utilizzare gli stessi enzimi di restrizione. Essendo USP 10 sensibile al taglio di Bam. HI ma non a quello di Bgl. II, essendo Bgl. II isocaudamero di Bam. HI ed essendo presente solo la sequenza tagliata da quest’ultimo nel MCS del vettore di espressione, abbiamo scelto questa tattica di clonaggio.

• Defosforilazione del vettore di espressione Attraverso la defosforilazione è possibile limitare l’eventualità che il vettore si possa richiudere su se stesso senza incorporare l’inserto. La defosforilazione è ad opera dell’enzima CIP, una fosfatasi alcalina che stacca il fosfato dalle basi delle estremità tagliate. • Purificazione e quantificazione di p. GEX-6 P-1 e degli inserti Il vettore e gli inserti sono stati purificati attraverso appositi kit per eliminare interferenti che potrebbero falsare il risultato e quantificati mediante l’utilizzo di uno spettrofluorimentro (Qubit) per conoscere la concentrazione effettiva di cloni ottenuti. • Reazione di clonaggio e trasformazione dei batteri competenti Processo di ligazione: USP 10(full-length) USP 10(1 -100) USP 10(101 -412) USP 10(412 -798) V: I 1: 1 1: 4 1: 2 1: 1 V: I 1: 2 1: 8 1: 4 1: 2 Una volta terminata la reazione, abbiamo proceduto con la trasformazione dei costrutti nei batteri competenti JM 109; un ceppo Escherichia coli caratterizzato da una buona efficienza di trasformazione. Infine, usando un’ansa sterile, ho piastrato i batteri trasformati su piastre di LB agar contenente Ampicillina e li ho fatti crescere in agitazione a 37°C per tutta la notte.

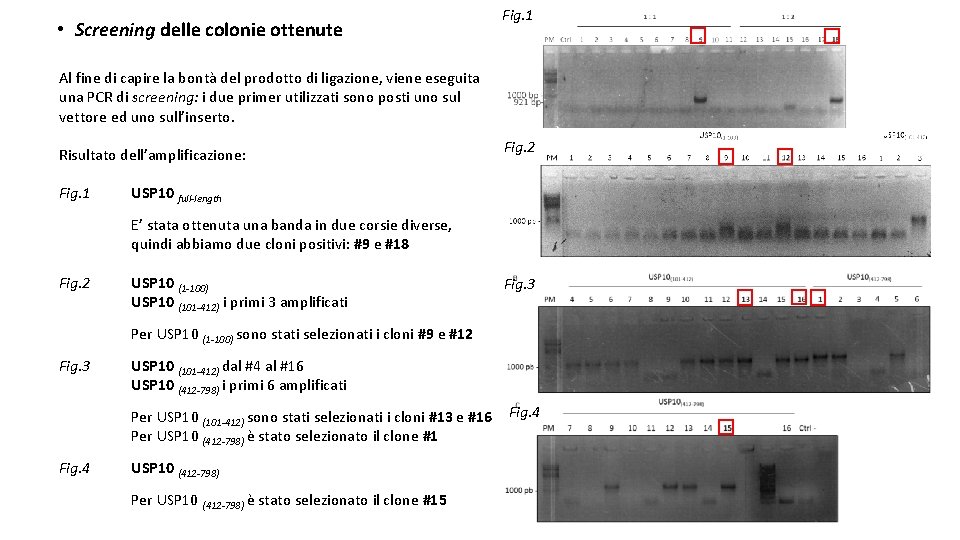
• Screening delle colonie ottenute Fig. 1 Al fine di capire la bontà del prodotto di ligazione, viene eseguita una PCR di screening: i due primer utilizzati sono posti uno sul vettore ed uno sull’inserto. Risultato dell’amplificazione: Fig. 1 Fig. 2 USP 10 full-length E’ stata ottenuta una banda in due corsie diverse, quindi abbiamo due cloni positivi: #9 e #18 Fig. 2 USP 10 (1 -100) USP 10 (101 -412) i primi 3 amplificati Fig. 3 Per USP 10 (1 -100) sono stati selezionati i cloni #9 e #12 Fig. 3 USP 10 (101 -412) dal #4 al #16 USP 10 (412 -798) i primi 6 amplificati Per USP 10 (101 -412) sono stati selezionati i cloni #13 e #16 Per USP 10 (412 -798) è stato selezionato il clone #1 Fig. 4 USP 10 (412 -798) Per USP 10 (412 -798) è stato selezionato il clone #15 Fig. 4

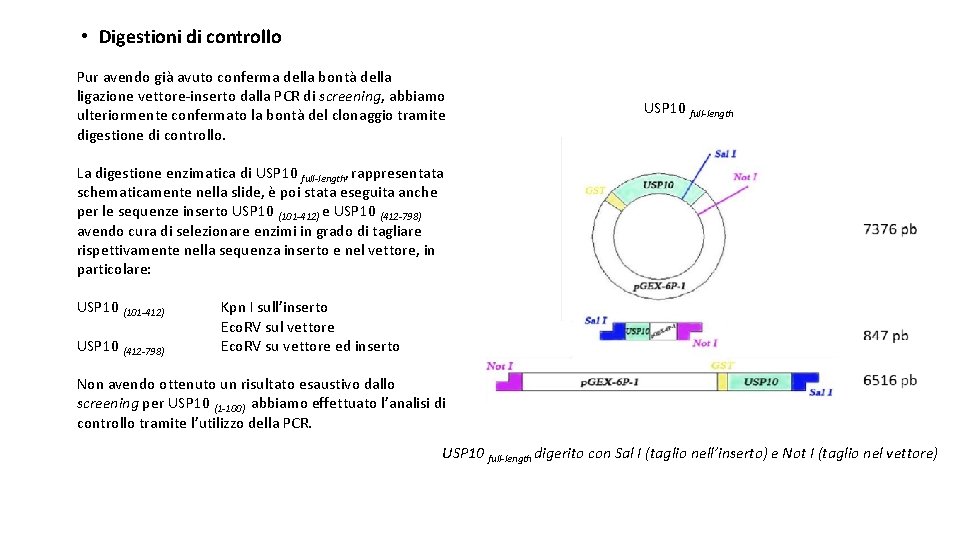
• Digestioni di controllo Pur avendo già avuto conferma della bontà della ligazione vettore-inserto dalla PCR di screening, abbiamo ulteriormente confermato la bontà del clonaggio tramite digestione di controllo. USP 10 full-length La digestione enzimatica di USP 10 full-length, rappresentata schematicamente nella slide, è poi stata eseguita anche per le sequenze inserto USP 10 (101 -412) e USP 10 (412 -798) avendo cura di selezionare enzimi in grado di tagliare rispettivamente nella sequenza inserto e nel vettore, in particolare: USP 10 (101 -412) USP 10 (412 -798) Kpn I sull’inserto Eco. RV sul vettore Eco. RV su vettore ed inserto Non avendo ottenuto un risultato esaustivo dallo screening per USP 10 (1 -100) abbiamo effettuato l’analisi di controllo tramite l’utilizzo della PCR. USP 10 full-length digerito con Sal I (taglio nell’inserto) e Not I (taglio nel vettore)

Conclusioni • Con l’obiettivo di caratterizzare il ruolo di USP 10 all’interno della risposta al danno al DNA abbiamo effettuato una serie di clonaggi volti all’ottenimento di sequenze da impiegare in studi in vitro. • Lo scopo degli esperimenti in vitro è quello dell’individuazione e mappatura di possibili UBD (Ubiquitin Binding Domain) all’interno della sequenza di USP 10. • L’identificazione di tali domini permetterebbe una migliore comprensione, della capacità funzionale della DUB nella riparazione del DNA.

Grazie per l’attenzione!