POSTGENOMICA O GENOMICA FUNZIONALE Analisi della sequenza genomica






- Slides: 15

POSTGENOMICA O GENOMICA FUNZIONALE Analisi della sequenza genomica per identificare geni, sequenze di controllo, e tutti gli altri elementi che contribuiscono all’espressione di geni specifici e del genoma nel suo insieme e Studio del trascrittoma: studio dei trascritti di una cellula per comprendere lo schema di espressione del genoma nel suo insieme Tecnica: microarray di espressione Studio della variabilità cellulare dell’espressione genica Tecnica: PCR quantitativa (Real. Time PCR) Studio del proteoma: studio del contenuto proteico di una cellula per comprendere tutte le sue capacità biochimiche Tecnica: microarray di proteine

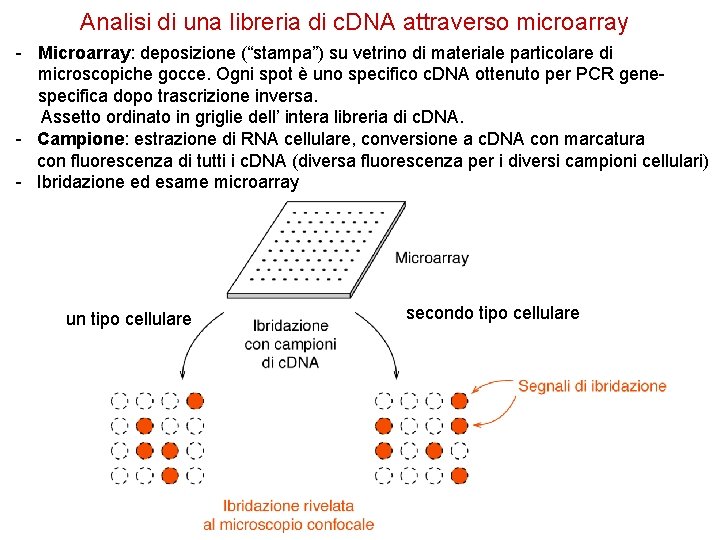
Analisi di una libreria di c. DNA attraverso microarray - Microarray: deposizione (“stampa”) su vetrino di materiale particolare di microscopiche gocce. Ogni spot è uno specifico c. DNA ottenuto per PCR genespecifica dopo trascrizione inversa. Assetto ordinato in griglie dell’ intera libreria di c. DNA. - Campione: estrazione di RNA cellulare, conversione a c. DNA con marcatura con fluorescenza di tutti i c. DNA (diversa fluorescenza per i diversi campioni cellulari) - Ibridazione ed esame microarray un tipo cellulare secondo tipo cellulare

Studio del trascrittoma attraverso microarray con c. DNA Esempio: valutazione dell’ espressione genica nel campione sperimentale trattato Esperimento Es: trattamento farmaco controllo Esperimento: trattamento farmaco Immagine combinata

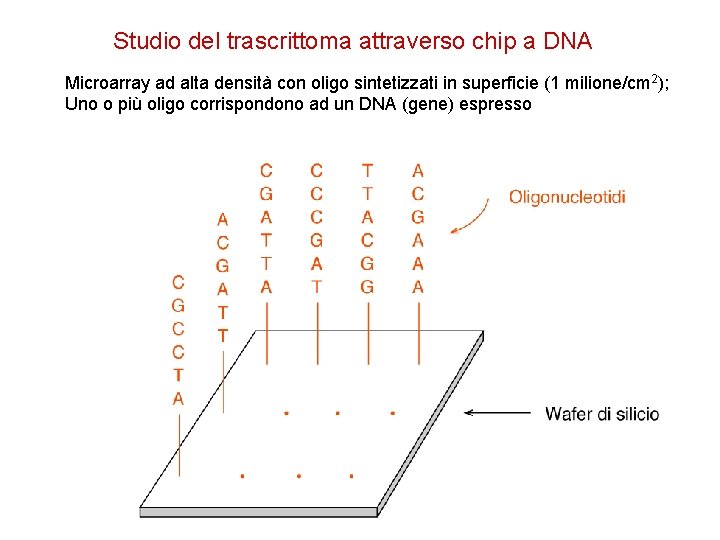
Studio del trascrittoma attraverso chip a DNA Microarray ad alta densità con oligo sintetizzati in superficie (1 milione/cm 2); Uno o più oligo corrispondono ad un DNA (gene) espresso

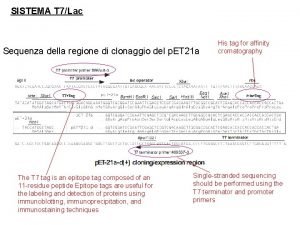
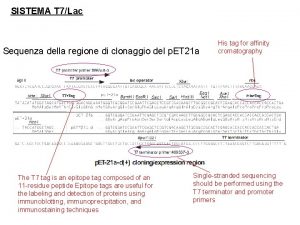
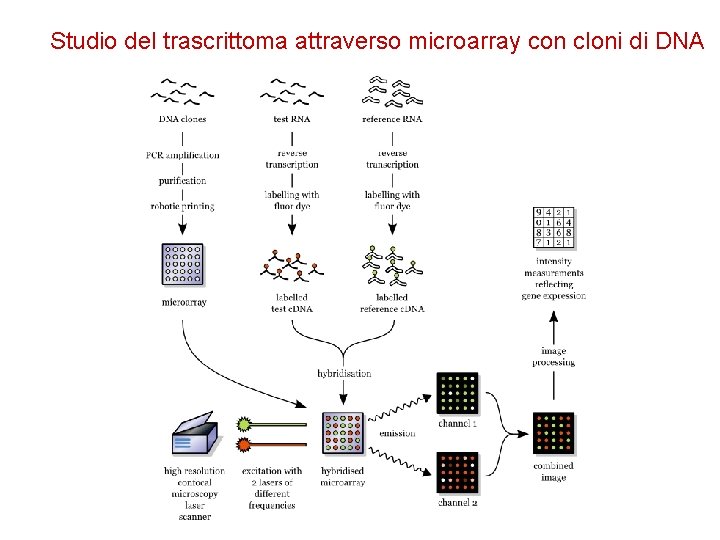
Studio del trascrittoma attraverso microarray con cloni di DNA

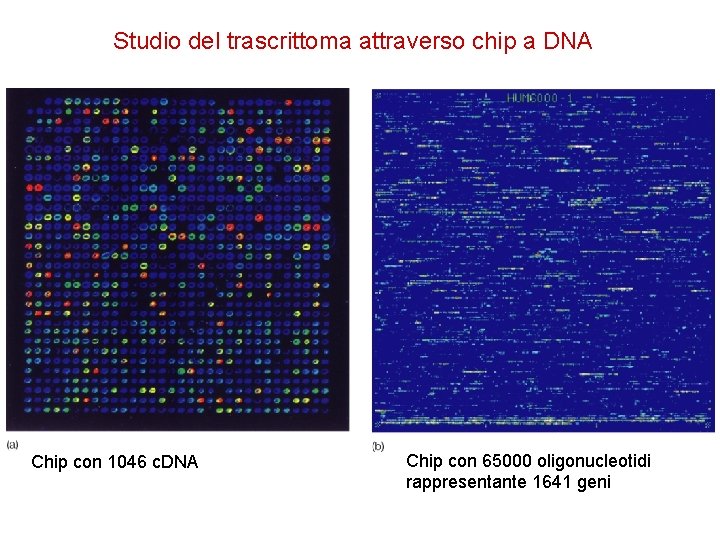
Studio del trascrittoma attraverso chip a DNA

Studio del trascrittoma attraverso chip a DNA Chip con 1046 c. DNA Chip con 65000 oligonucleotidi rappresentante 1641 geni

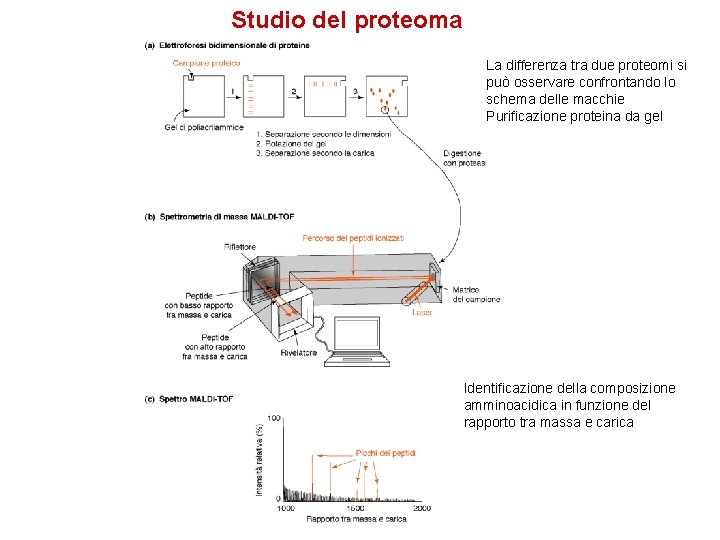
Studio del proteoma La differenza tra due proteomi si può osservare confrontando lo schema delle macchie Purificazione proteina da gel Identificazione della composizione amminoacidica in funzione del rapporto tra massa e carica


Real-Time PCR o PCR Quantitativa Valutazione dell’espressione di un gene: Retrotrascrizione dell’RNA totale in c. DNA • Amplificazione gene specifica del c. DNA con Real-Time PCR • Valutazione del prodotto PCR ottenuto durante la fase esponenziale Log [DNA] • Plateau Lineare Esponenziale Prodotto variabile N° cicli

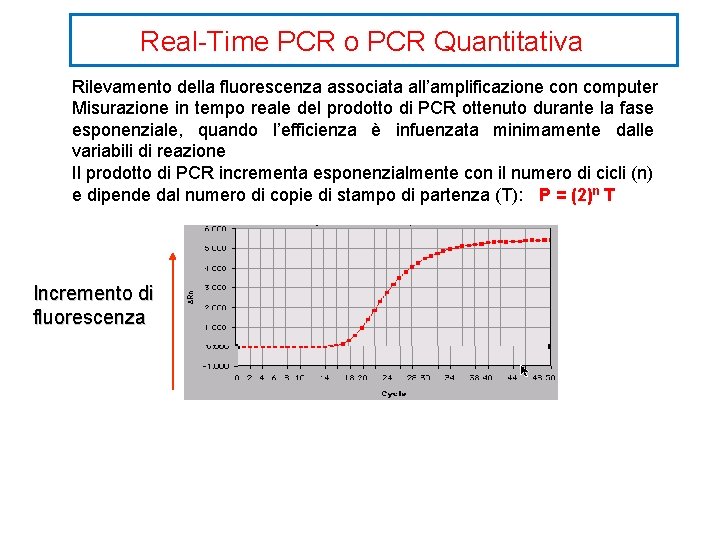
Real-Time PCR o PCR Quantitativa Rilevamento della fluorescenza associata all’amplificazione con computer Misurazione in tempo reale del prodotto di PCR ottenuto durante la fase esponenziale, quando l’efficienza è infuenzata minimamente dalle variabili di reazione Il prodotto di PCR incrementa esponenzialmente con il numero di cicli (n) e dipende dal numero di copie di stampo di partenza (T): P = (2)n T Incremento di fluorescenza

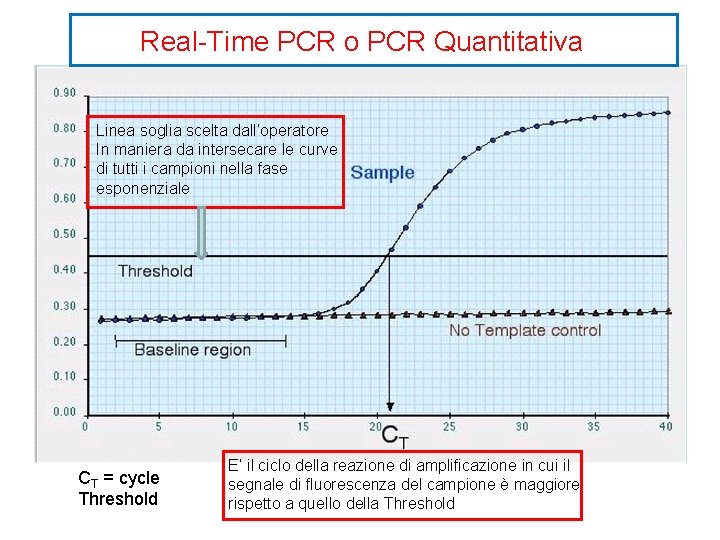
Real-Time PCR o PCR Quantitativa Linea soglia scelta dall’operatore In maniera da intersecare le curve di tutti i campioni nella fase esponenziale CT = cycle Threshold E’ il ciclo della reazione di amplificazione in cui il segnale di fluorescenza del campione è maggiore rispetto a quello della Threshold

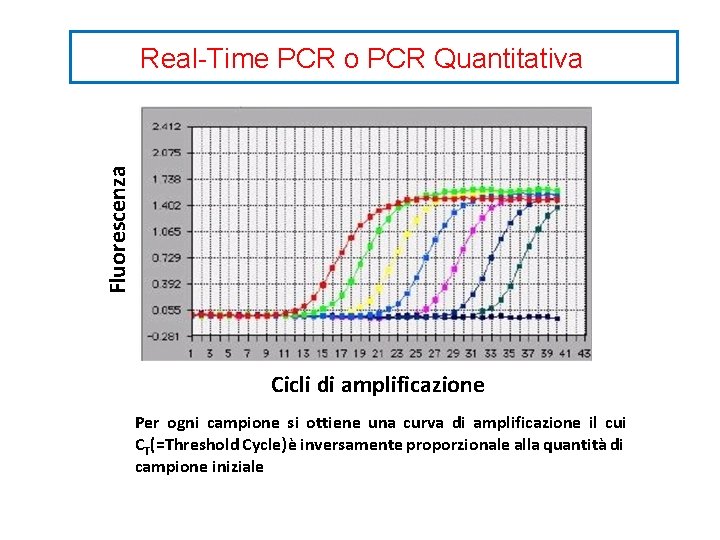
Fluorescenza Real-Time PCR o PCR Quantitativa Cicli di amplificazione Per ogni campione si ottiene una curva di amplificazione il cui CT(=Threshold Cycle) è inversamente proporzionale alla quantità di campione iniziale

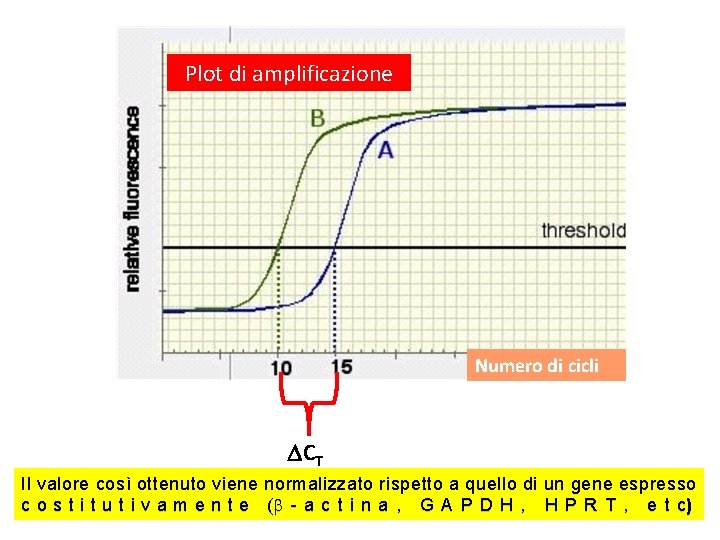
Plot di amplificazione Numero di cicli CT Il valore così ottenuto viene normalizzato rispetto a quello di un gene espresso c o s t i t u t i v a m e n t e ( - a c t i n a , G A P D H , H P R T , e t c)

Real-Time PCR o PCR Quantitativa La fluorescenza si genera durante la PCR mediante l’impiego: • La fluorescenza si genera durante la - coloranti intercalanti ( es. SYBR green), che si legano in maniera PCR per effetto di diverse aspecifica a tutto il DNA possibili reazioni chimiche - sonde ad ibridazione, specifiche per il frammento di interesse, marcate con molecole fluorescenti • Le chimiche principali sono basate sia Quantificazione assoluta: i campioni sono quantificati in modo sul legame di coloranti fluorescenti che si intercalano nella doppia elica di DNA, assoluto, occorre una curva standard come il SYBR Green, sia sull'ibridazione d i s o n d e s p e c i f i c h e. Quantificazione relativa: Confronto dei CT dei diversi campioni. Il valore così ottenuto permette di determinare la concentrazione relativa del target
 Analisi funzionale del comportamento
Analisi funzionale del comportamento Dieta genomica
Dieta genomica Lalign
Lalign Fiori fibonacci
Fiori fibonacci Vieni santo spirito di dio come vento soffia sulla chiesa
Vieni santo spirito di dio come vento soffia sulla chiesa Algoritmo blast
Algoritmo blast Sequenza fibonacci
Sequenza fibonacci Segnale di localizzazione nucleare
Segnale di localizzazione nucleare Progressione geometrica e aritmetica
Progressione geometrica e aritmetica Disegni fiabe in sequenza
Disegni fiabe in sequenza Riconoscitore di sequenza
Riconoscitore di sequenza Kdel sequenza
Kdel sequenza Vieni santo spirito
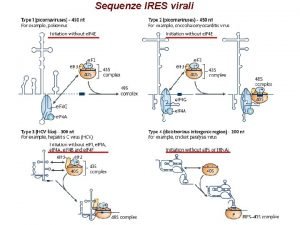
Vieni santo spirito Sequenze ires
Sequenze ires Contatore johnson
Contatore johnson Sequenze pierino e il lupo
Sequenze pierino e il lupo