De ADN a protena Los genes se expresan







- Slides: 56

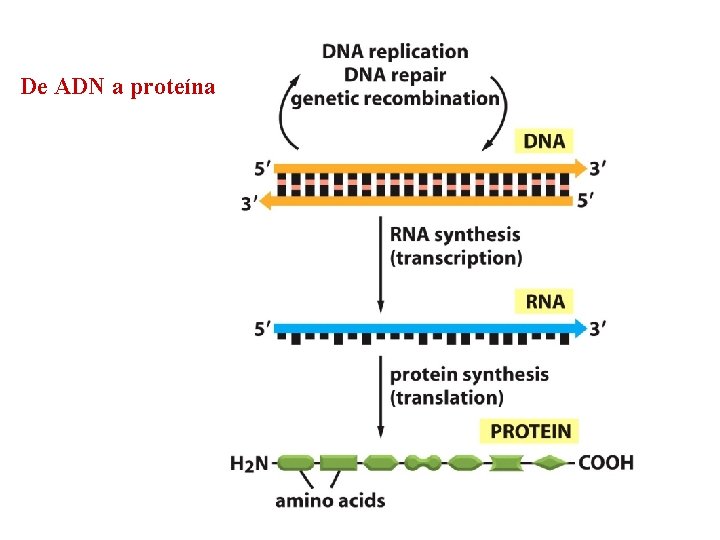
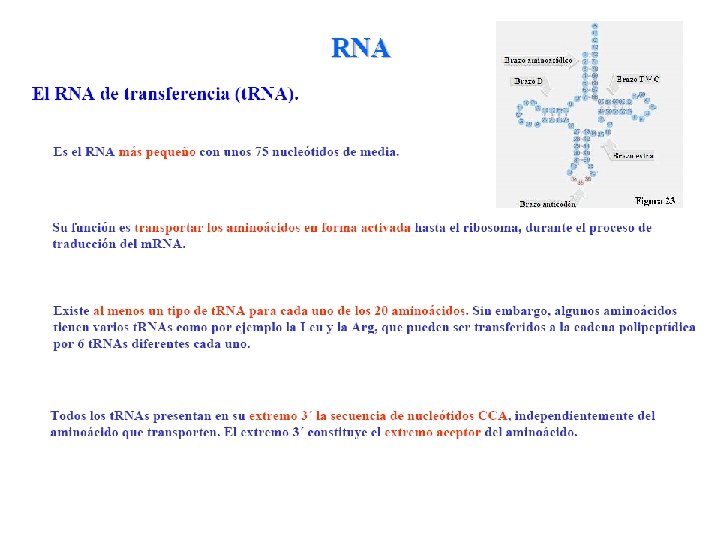
De ADN a proteína

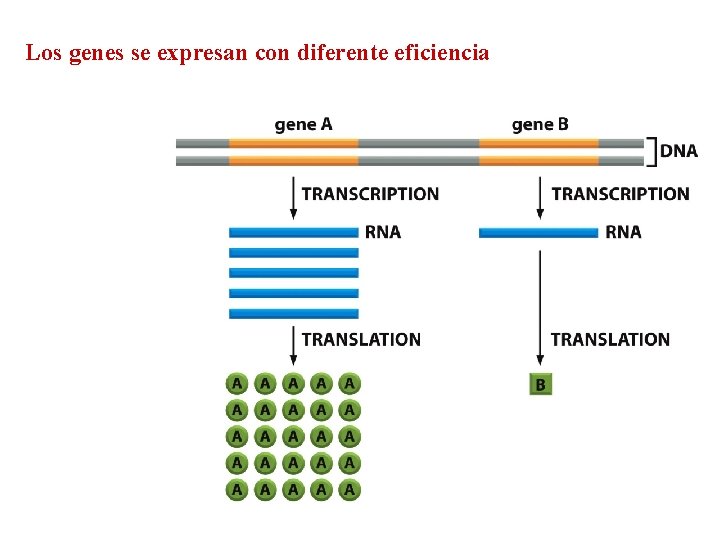
Los genes se expresan con diferente eficiencia

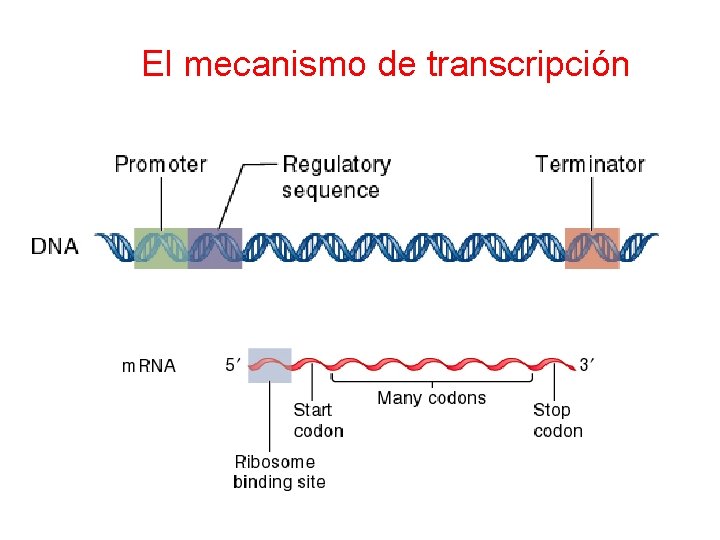
El mecanismo de transcripción

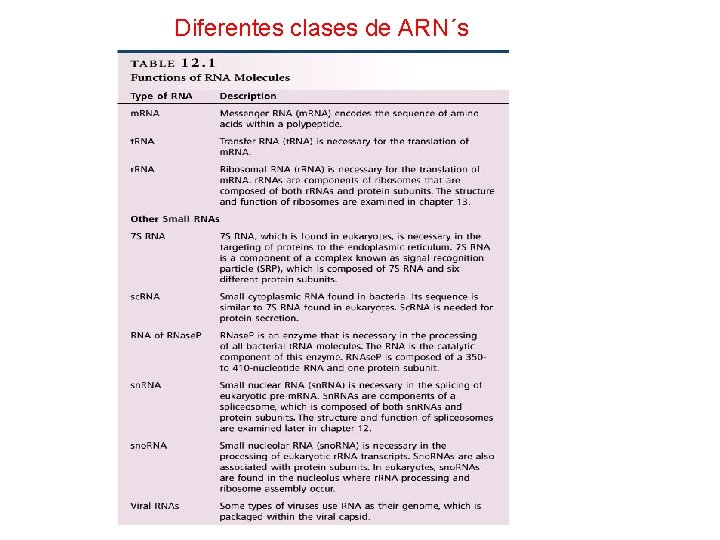
Diferentes clases de ARN´s

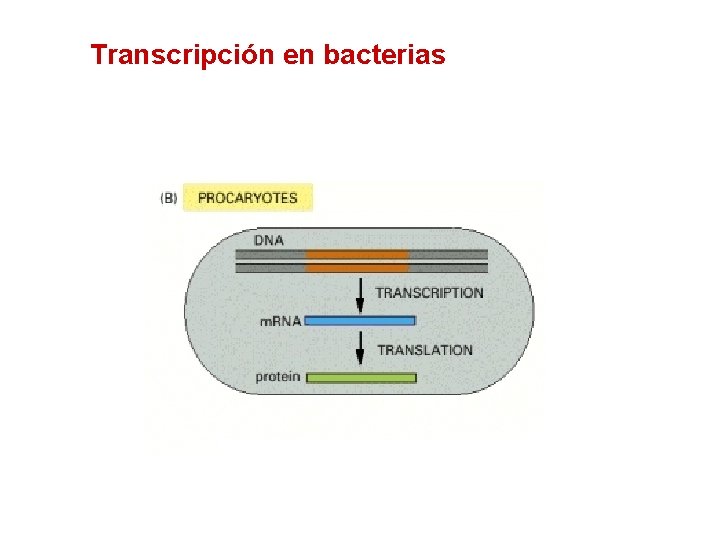
Transcripción en bacterias

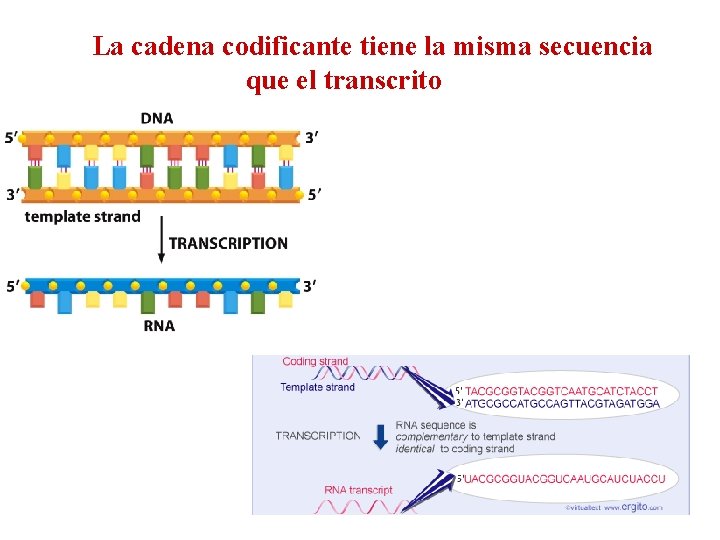
La cadena codificante tiene la misma secuencia que el transcrito

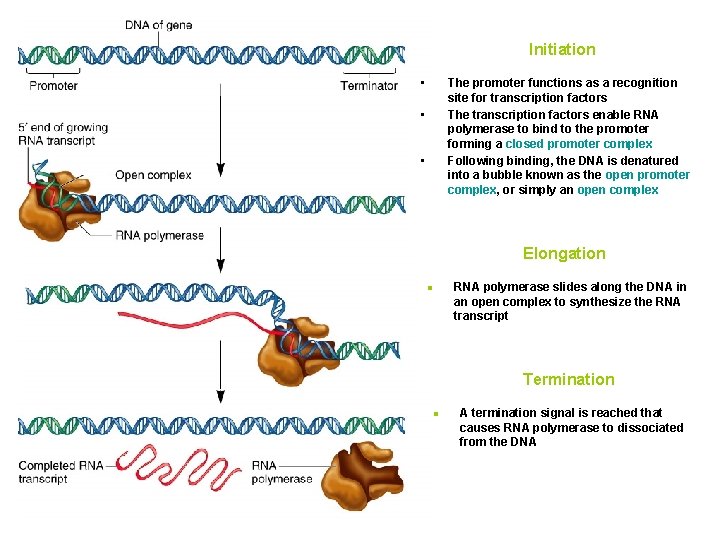
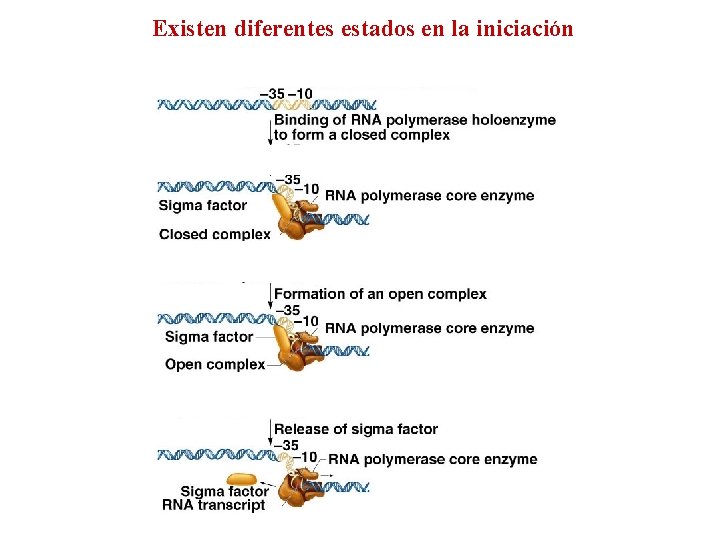
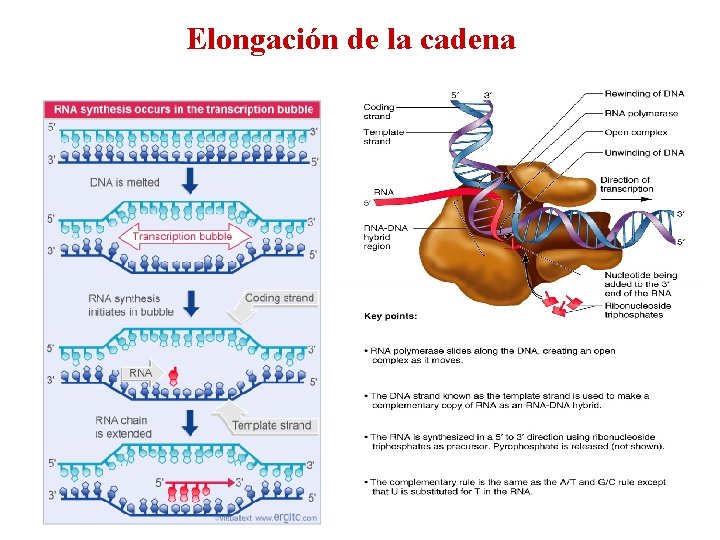
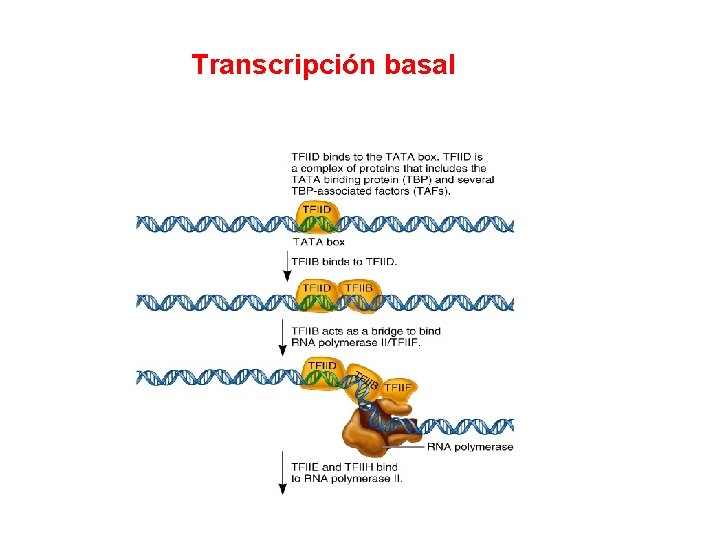
Initiation • The promoter functions as a recognition site for transcription factors The transcription factors enable RNA polymerase to bind to the promoter forming a closed promoter complex Following binding, the DNA is denatured into a bubble known as the open promoter complex, or simply an open complex • • Elongation RNA polymerase slides along the DNA in an open complex to synthesize the RNA transcript n Termination n A termination signal is reached that causes RNA polymerase to dissociated from the DNA

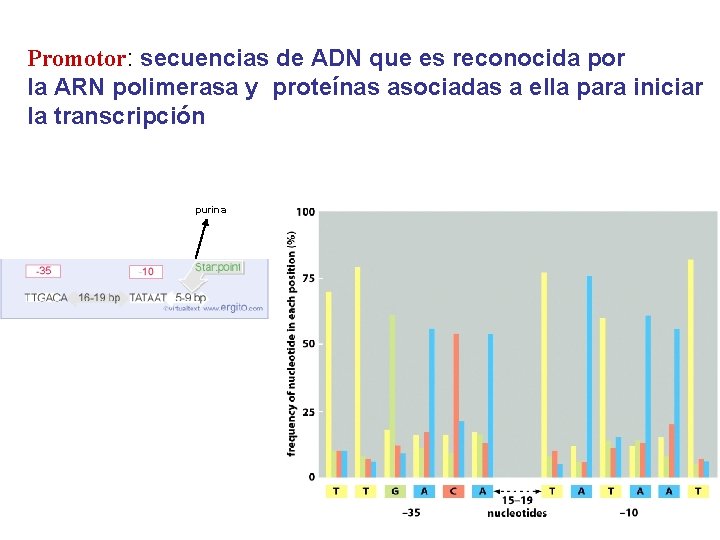
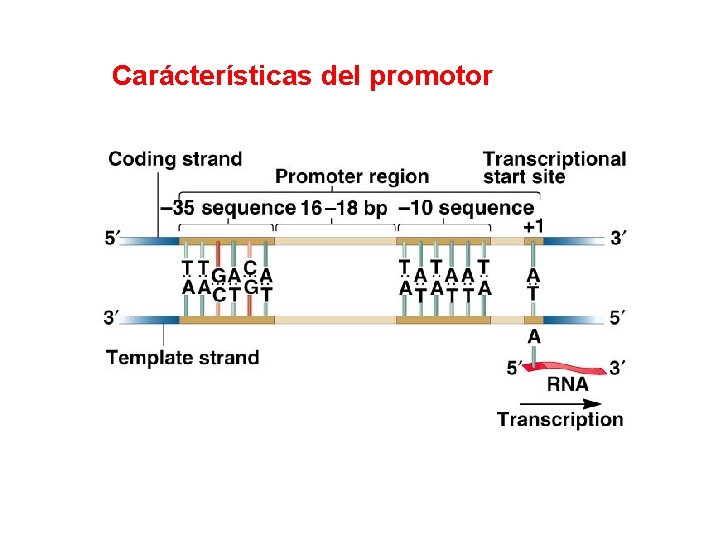
Promotor: secuencias de ADN que es reconocida por la ARN polimerasa y proteínas asociadas a ella para iniciar la transcripción purina

Carácterísticas del promotor

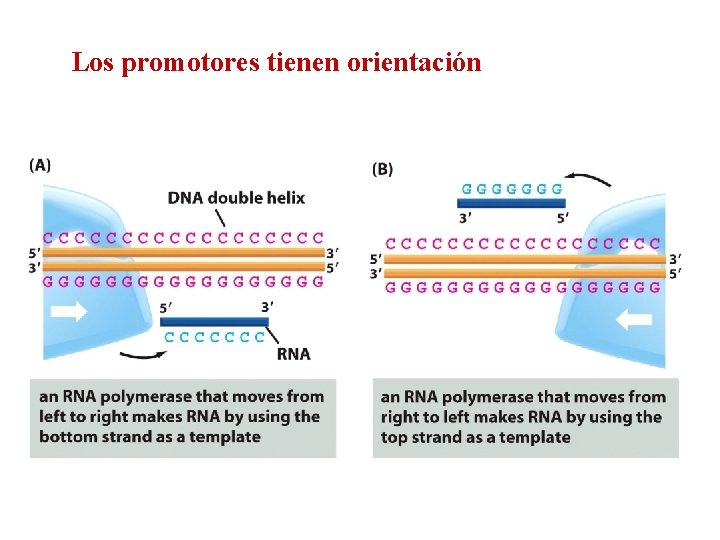
Los promotores tienen orientación

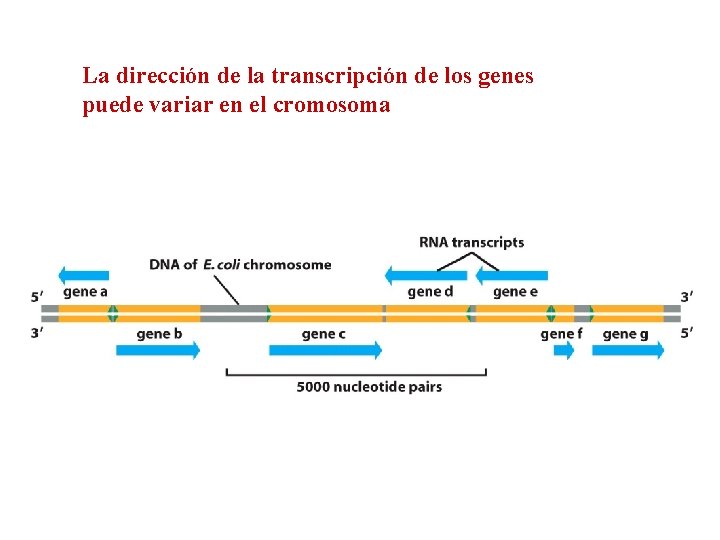
La dirección de la transcripción de los genes puede variar en el cromosoma

La ARN polimerasa es la encargada de copiar el ADN

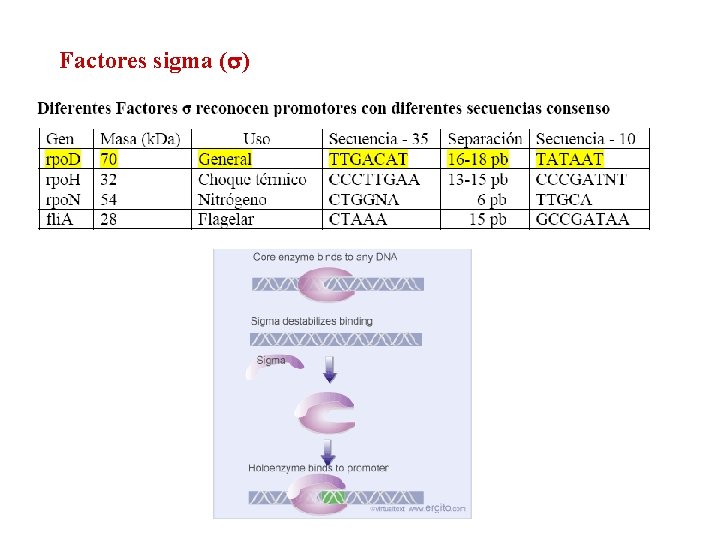
Factores sigma (s)

Existen diferentes estados en la iniciación

Elongación de la cadena

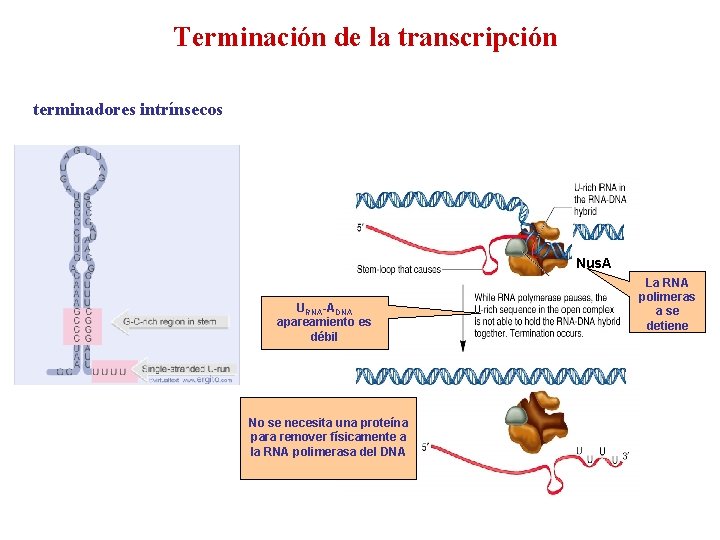
Terminación de la transcripción terminadores intrínsecos Nus. A URNA-ADNA apareamiento es débil No se necesita una proteína para remover físicamente a la RNA polimerasa del DNA La RNA polimeras a se detiene

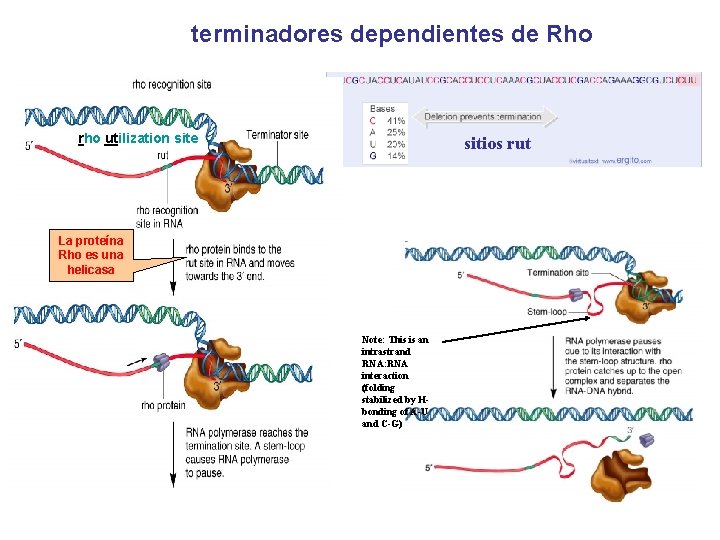
terminadores dependientes de Rho rho utilization site sitios rut La proteína Rho es una helicasa Note: This is an intrastrand RNA: RNA interaction (folding stabilized by Hbonding of A-U and C-G)

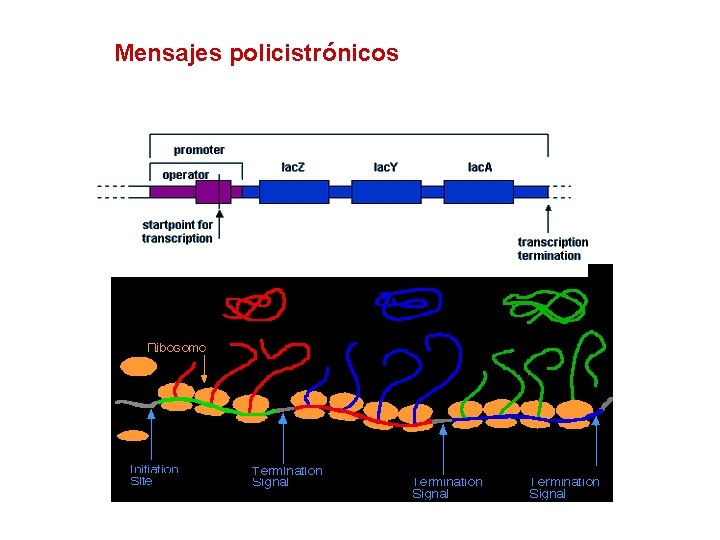
Mensajes policistrónicos

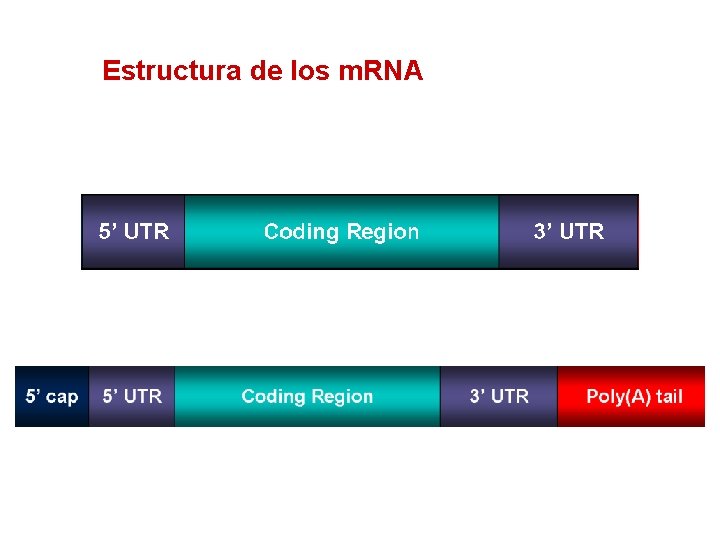
Estructura de los m. RNA

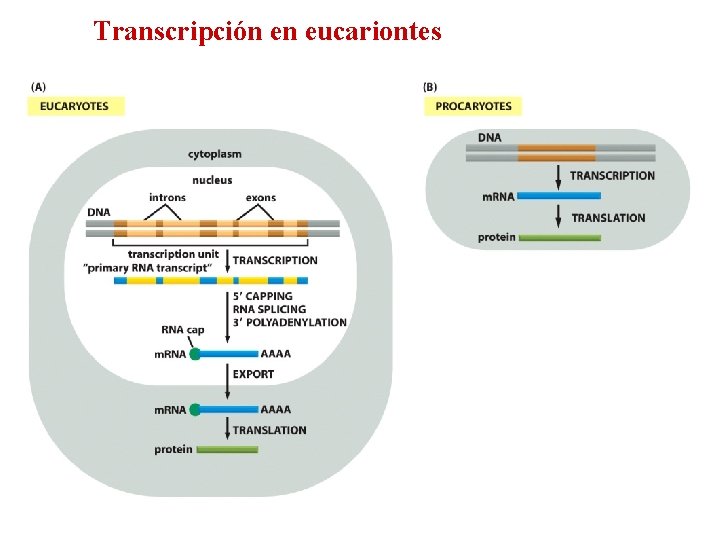
Transcripción en eucariontes

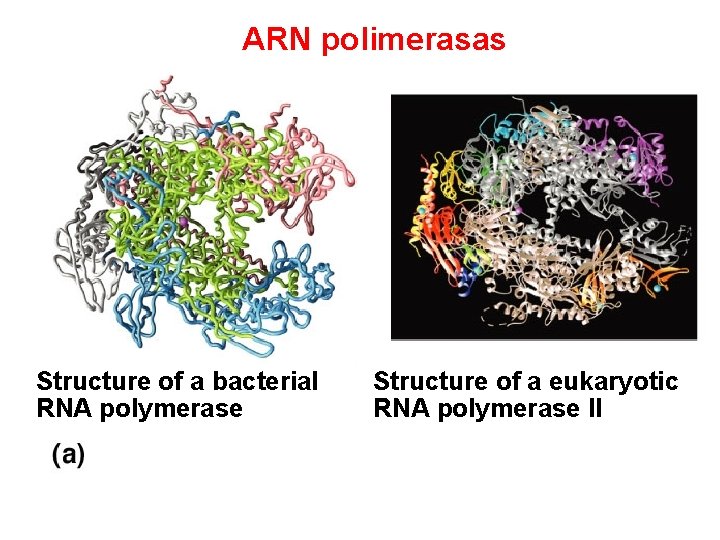
ARN polimerasas Structure of a bacterial RNA polymerase Structure of a eukaryotic RNA polymerase II

Diferentes tipos de ARN polimerasas

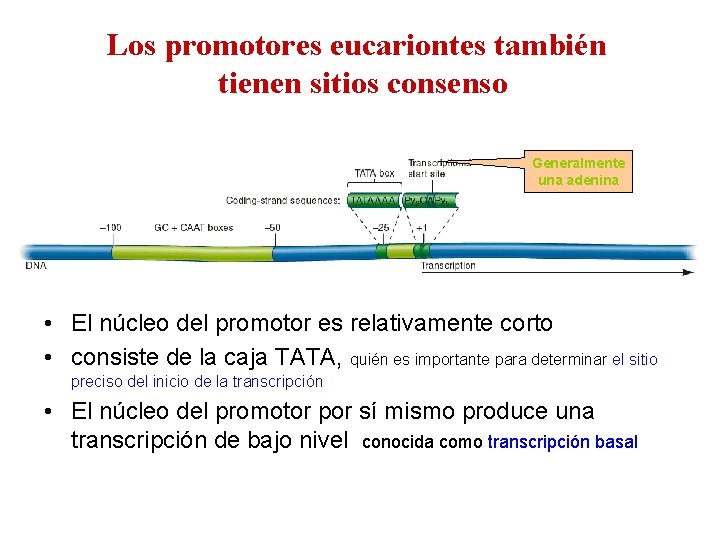
Los promotores eucariontes también tienen sitios consenso Generalmente una adenina • El núcleo del promotor es relativamente corto • consiste de la caja TATA, quién es importante para determinar el sitio preciso del inicio de la transcripción • El núcleo del promotor por sí mismo produce una transcripción de bajo nivel conocida como transcripción basal

Transcripción basal

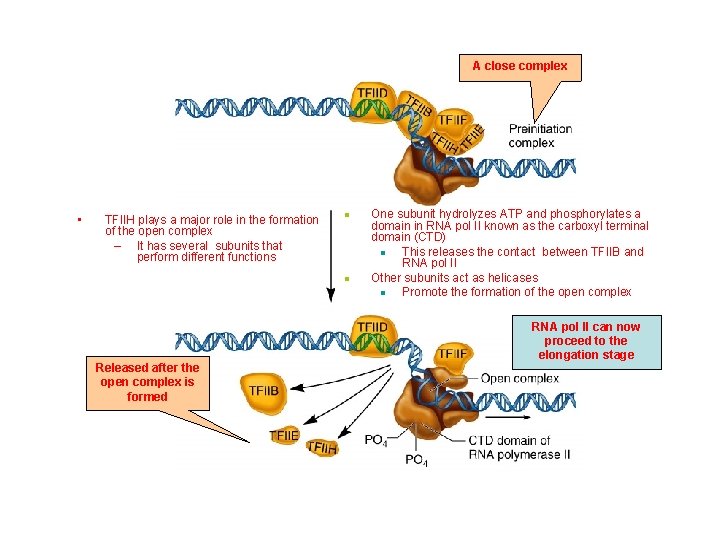
A close complex • TFIIH plays a major role in the formation of the open complex – It has several subunits that perform different functions n n Released after the open complex is formed One subunit hydrolyzes ATP and phosphorylates a domain in RNA pol II known as the carboxyl terminal domain (CTD) n This releases the contact between TFIIB and RNA pol II Other subunits act as helicases n Promote the formation of the open complex RNA pol II can now proceed to the elongation stage

Factores de transcripción

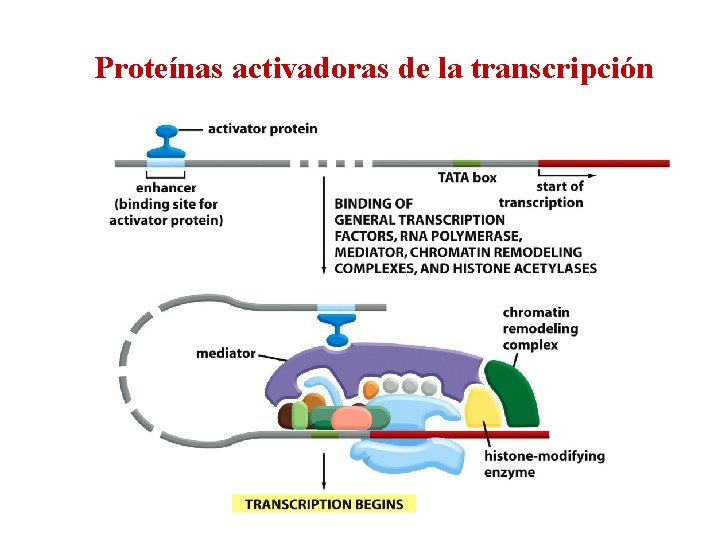
Proteínas activadoras de la transcripción

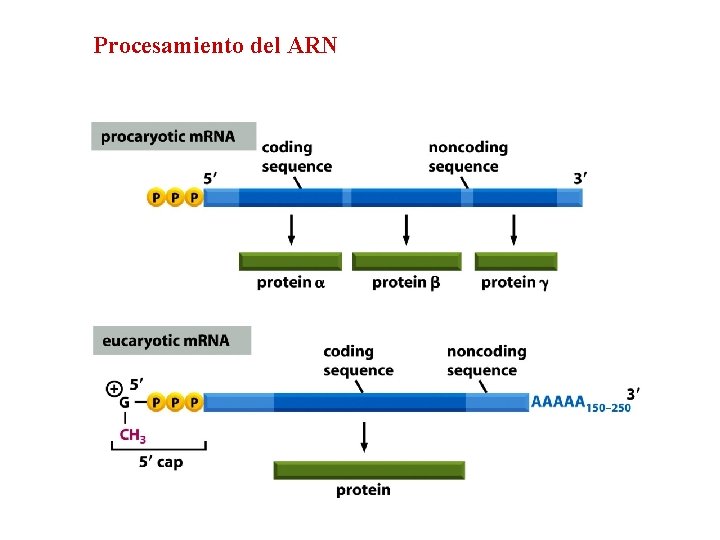
Procesamiento del ARN

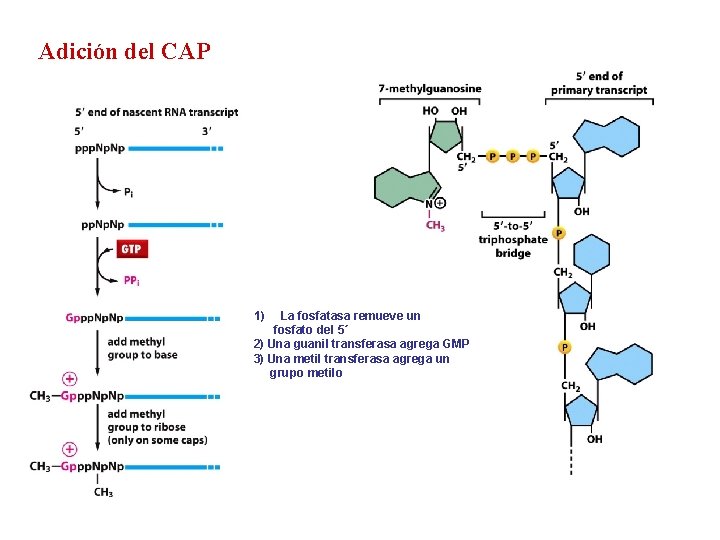
Adición del CAP 1) La fosfatasa remueve un fosfato del 5´ 2) Una guanil transferasa agrega GMP 3) Una metil transferasa agrega un grupo metilo

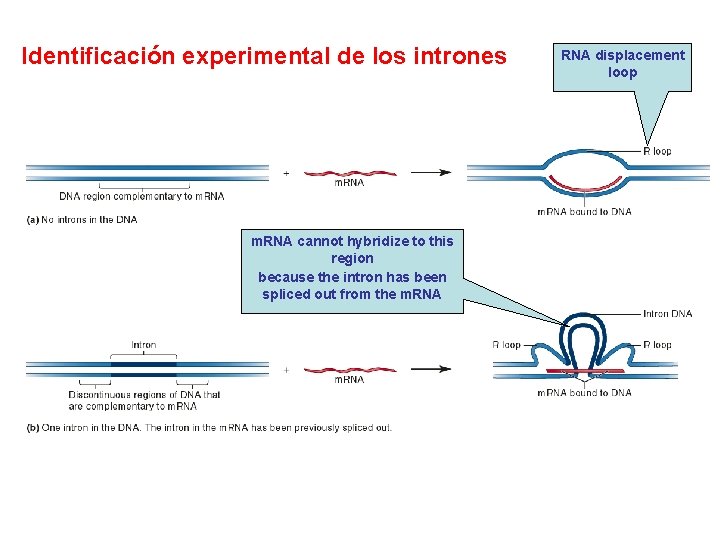
Identificación experimental de los intrones m. RNA cannot hybridize to this region because the intron has been spliced out from the m. RNA displacement loop

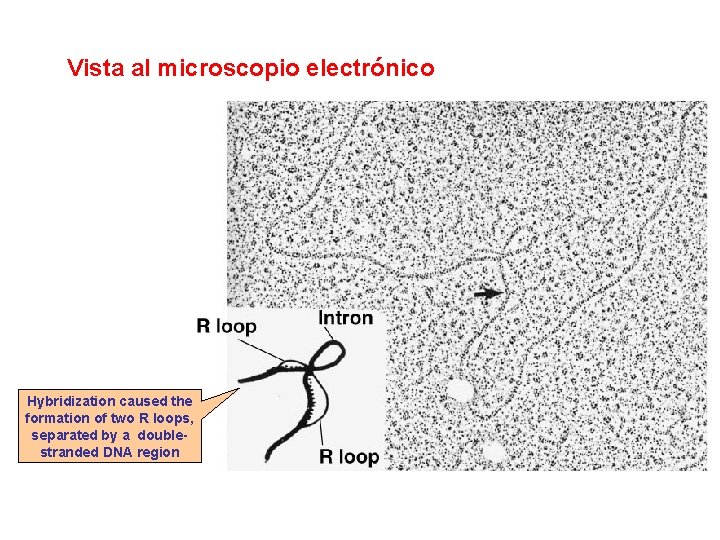
Vista al microscopio electrónico Hybridization caused the formation of two R loops, separated by a doublestranded DNA region

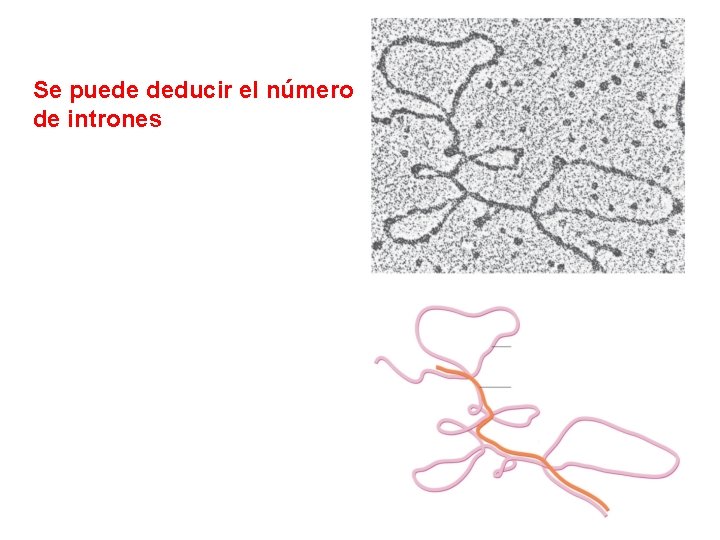
Se puede deducir el número de intrones

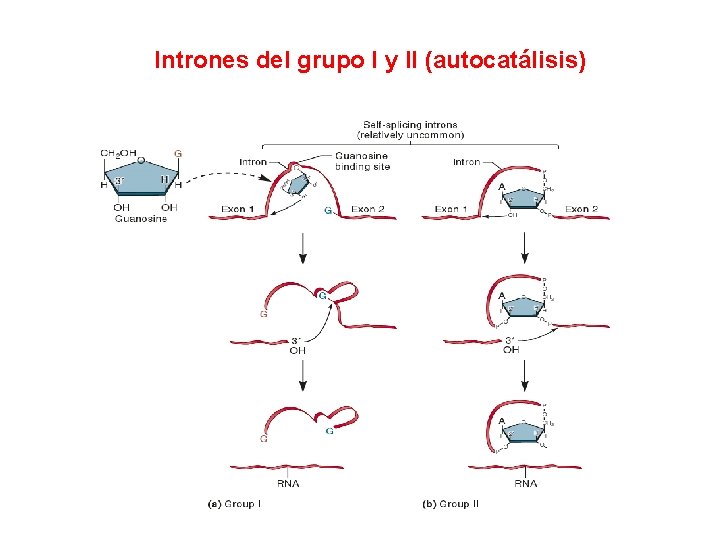
Intrones del grupo I y II (autocatálisis)

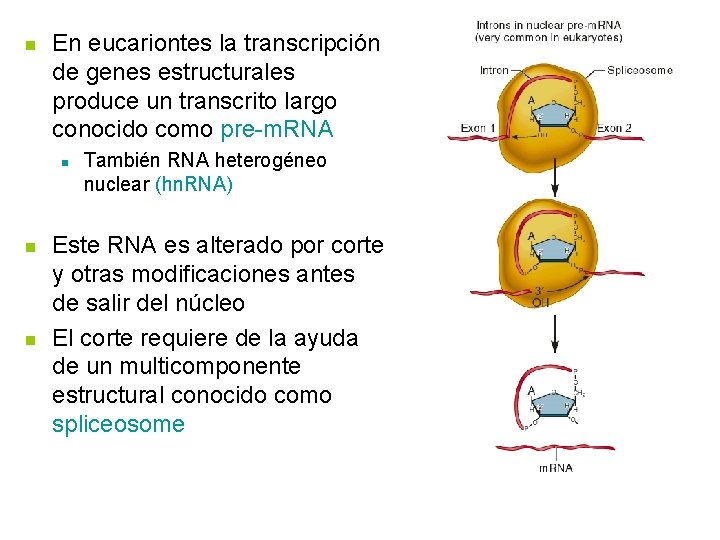
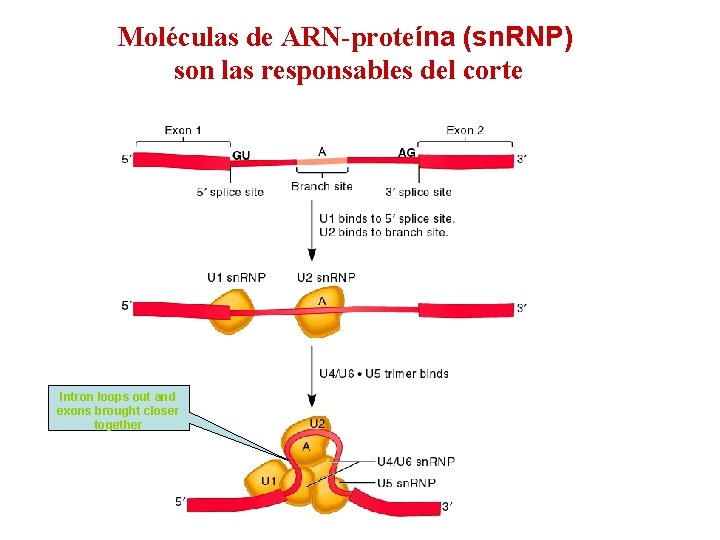
n En eucariontes la transcripción de genes estructurales produce un transcrito largo conocido como pre-m. RNA n n n También RNA heterogéneo nuclear (hn. RNA) Este RNA es alterado por corte y otras modificaciones antes de salir del núcleo El corte requiere de la ayuda de un multicomponente estructural conocido como spliceosome

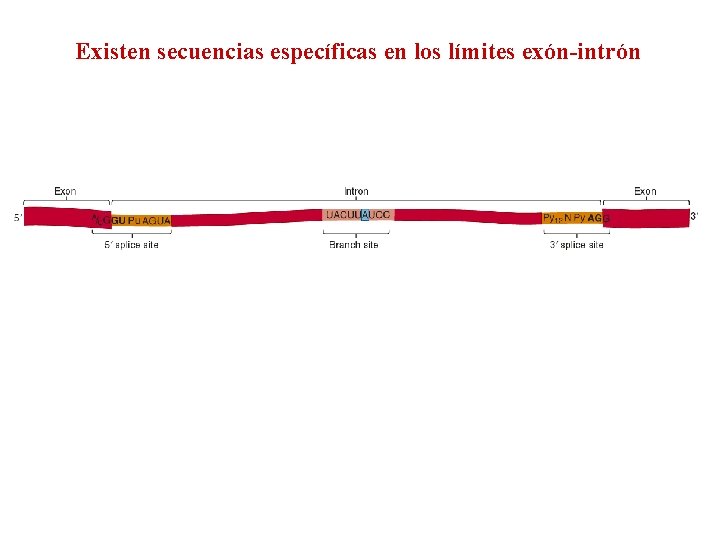
Existen secuencias específicas en los límites exón-intrón

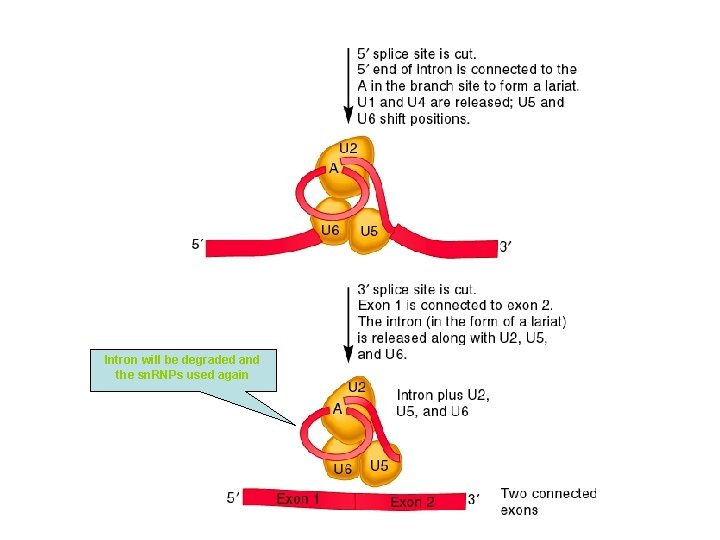
Moléculas de ARN-proteína (sn. RNP) son las responsables del corte Intron loops out and exons brought closer together

Intron will be degraded and the sn. RNPs used again



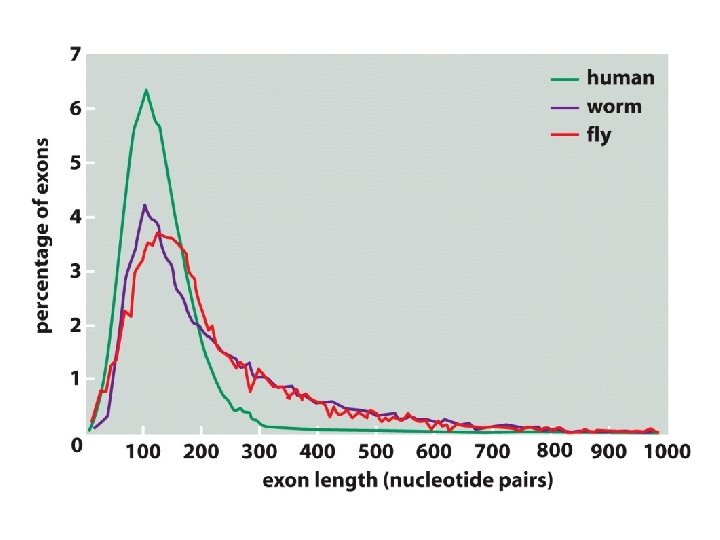
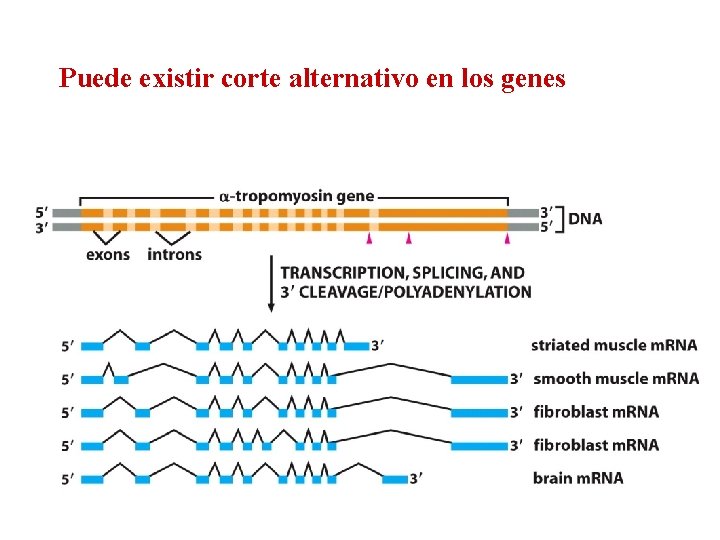
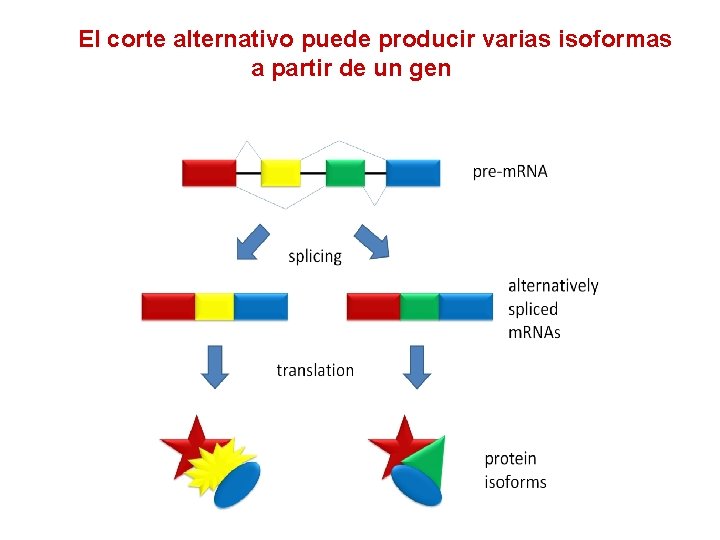
Puede existir corte alternativo en los genes

El corte alternativo puede producir varias isoformas a partir de un gen

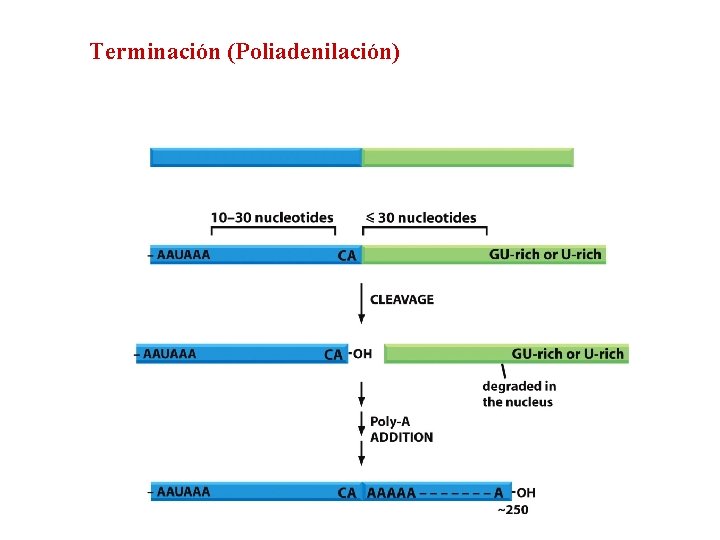
Terminación (Poliadenilación)

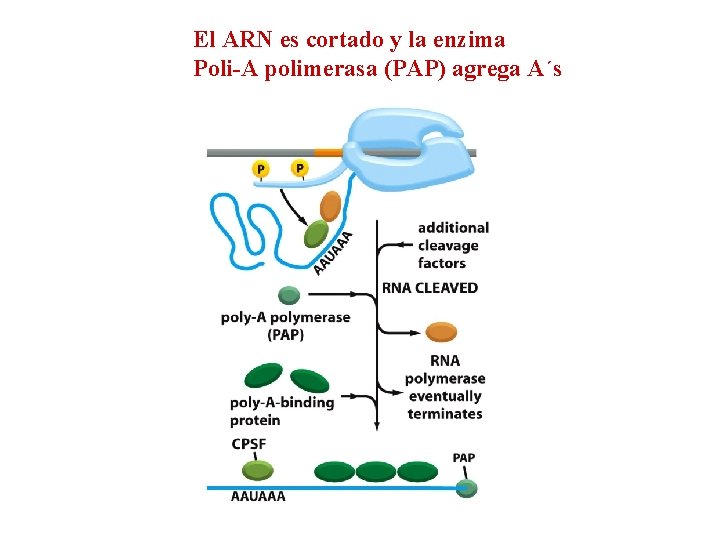
El ARN es cortado y la enzima Poli-A polimerasa (PAP) agrega A´s

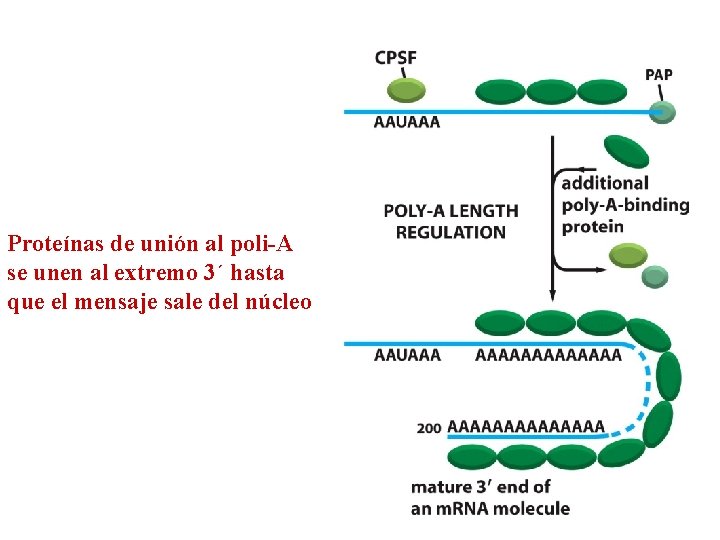
Proteínas de unión al poli-A se unen al extremo 3´ hasta que el mensaje sale del núcleo


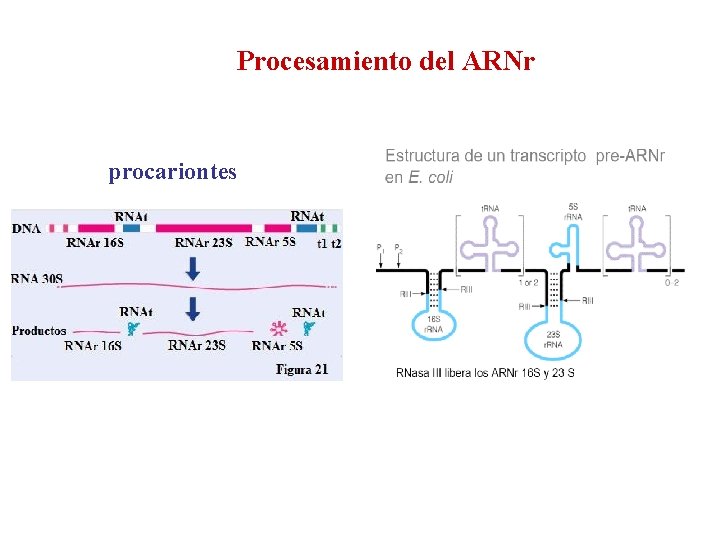
Procesamiento del ARNr procariontes

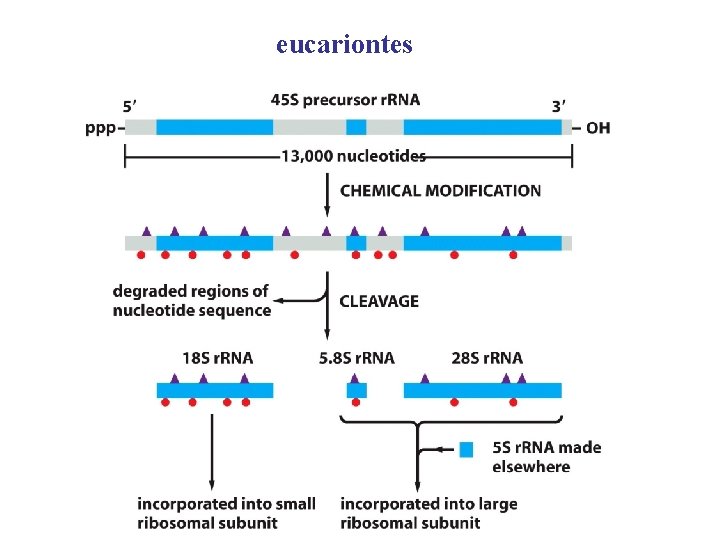
eucariontes

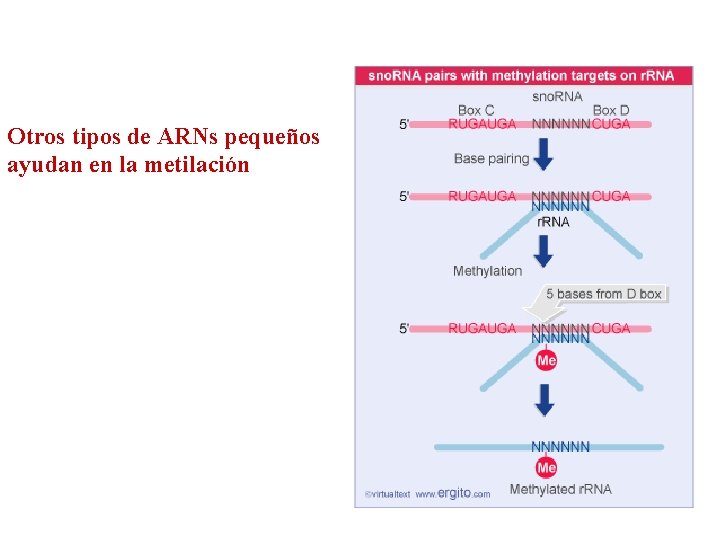
Otros tipos de ARNs pequeños ayudan en la metilación

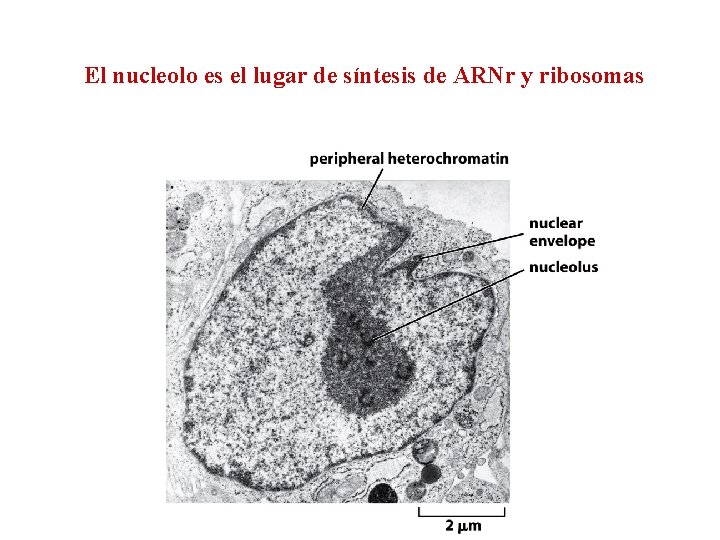
El nucleolo es el lugar de síntesis de ARNr y ribosomas


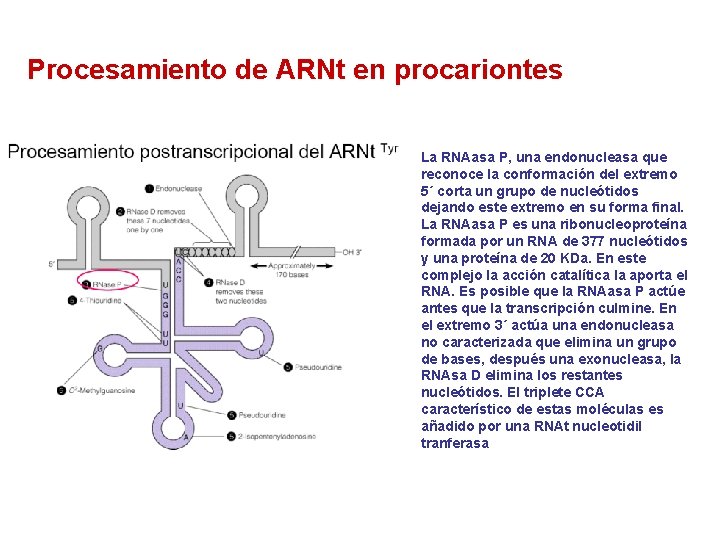
Procesamiento de ARNt en procariontes La RNAasa P, una endonucleasa que reconoce la conformación del extremo 5´ corta un grupo de nucleótidos dejando este extremo en su forma final. La RNAasa P es una ribonucleoproteína formada por un RNA de 377 nucleótidos y una proteína de 20 KDa. En este complejo la acción catalítica la aporta el RNA. Es posible que la RNAasa P actúe antes que la transcripción culmine. En el extremo 3´ actúa una endonucleasa no caracterizada que elimina un grupo de bases, después una exonucleasa, la RNAsa D elimina los restantes nucleótidos. El triplete CCA característico de estas moléculas es añadido por una RNAt nucleotidil tranferasa

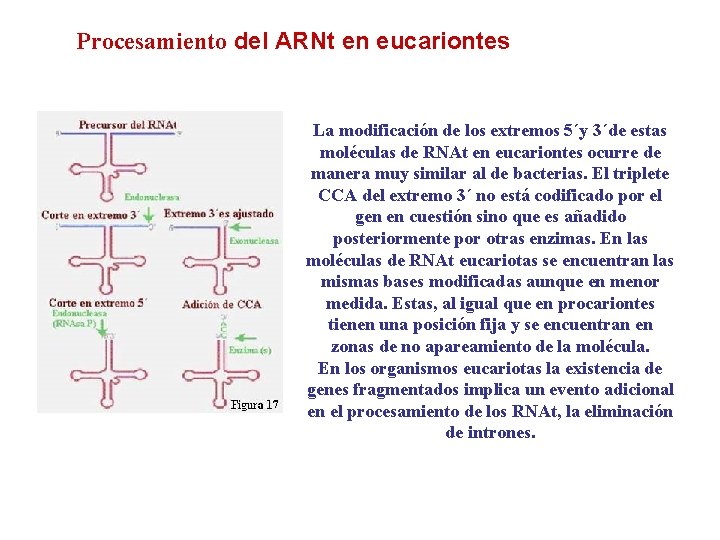
Procesamiento del ARNt en eucariontes La modificación de los extremos 5´y 3´de estas moléculas de RNAt en eucariontes ocurre de manera muy similar al de bacterias. El triplete CCA del extremo 3´ no está codificado por el gen en cuestión sino que es añadido posteriormente por otras enzimas. En las moléculas de RNAt eucariotas se encuentran las mismas bases modificadas aunque en menor medida. Estas, al igual que en procariontes tienen una posición fija y se encuentran en zonas de no apareamiento de la molécula. En los organismos eucariotas la existencia de genes fragmentados implica un evento adicional en el procesamiento de los RNAt, la eliminación de intrones.

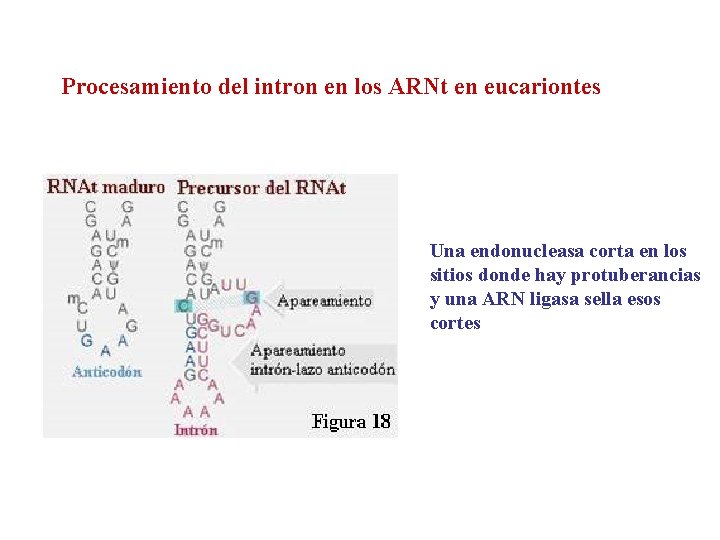
Procesamiento del intron en los ARNt en eucariontes Una endonucleasa corta en los sitios donde hay protuberancias y una ARN ligasa sella esos cortes

INHIBIDORES DE LA TRANSCRIPCIÓN ACTINOMICINA D y ACRIDINA: inhiben la elongación. Se intercalan en el DNA entre sucesivos pares GC, deformando la doble hélice, impidiendo el avance de la RNA polimerasa. RIFAMPICINA: antibiótico que se une a la subunidad de la RNA polimerasa bacteriana, impidiendo el inicio de la transcripción. -AMANITINA: inhibidor específico de la transcripción en eucariotas. Tóxico veneno producido por el hongo Amanita phalloides. Bloquea la síntesis de m. RNAs por la Pol II y a altas concentraciones a Pol III. NO AFECTA A LA RNA POLIMERASA BACTERIANA.

Las rifamicinas son antibióticos producidos por Streptomyces mediterranei, con buena actividad contra bacterias Gram-positivas y contra Mycobacterium tuberculosis. Su mecanismo de acción estriba en la inhibición del inicio de la transcripción, uniéndose de modo no covalente a la subunidad ß de la ARN polimerasa bacteriana.

-amanitina Es un octapéptido bicíclico que se obtiene del hongo Amanita phalloides. Inhibe la transcripción de la RNA polimerasa II eucarionte.