Analyses phylogntiques Infrer et tudier lhistoire volutive des


- Slides: 14

Analyses phylogénétiques Inférer et étudier l'histoire évolutive des gènes Jean-François Dufayard (CIRAD, équipe ID)

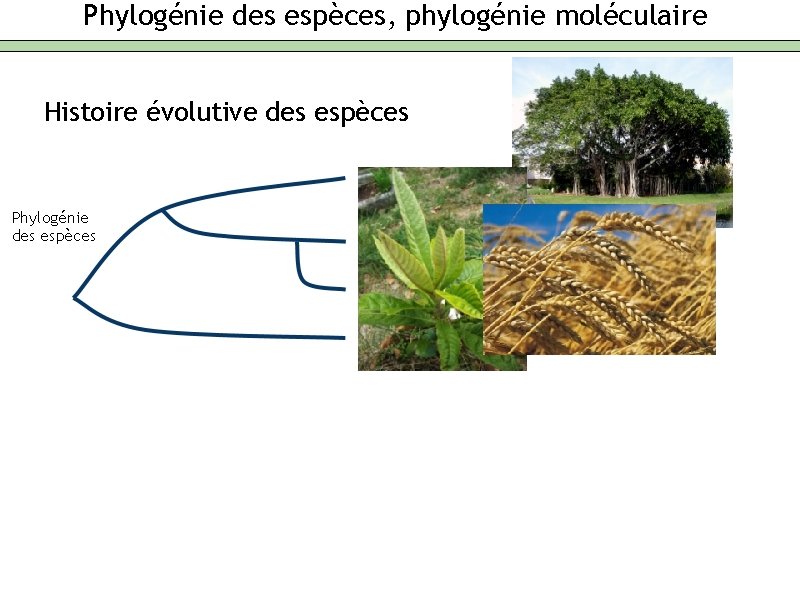
Phylogénie des espèces, phylogénie moléculaire Histoire évolutive des espèces Phylogénie des espèces

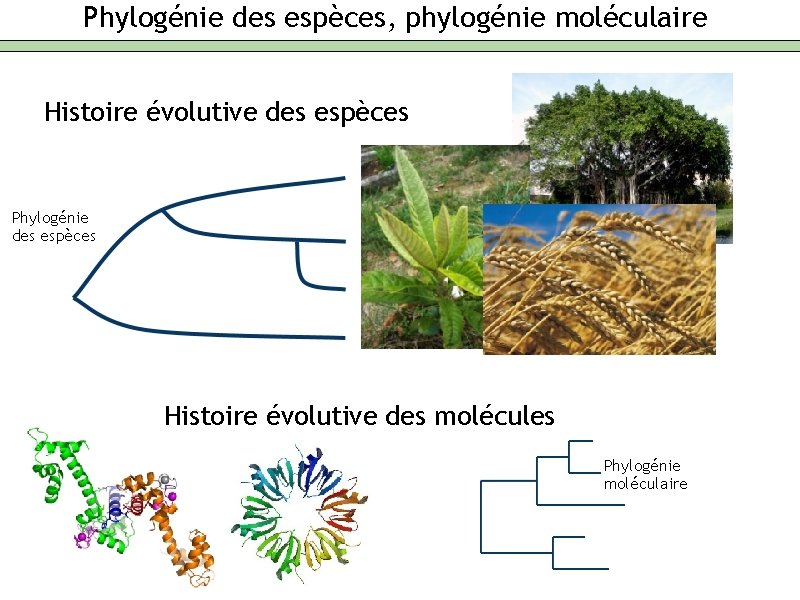
Phylogénie des espèces, phylogénie moléculaire Histoire évolutive des espèces Phylogénie des espèces Histoire évolutive des molécules Phylogénie moléculaire

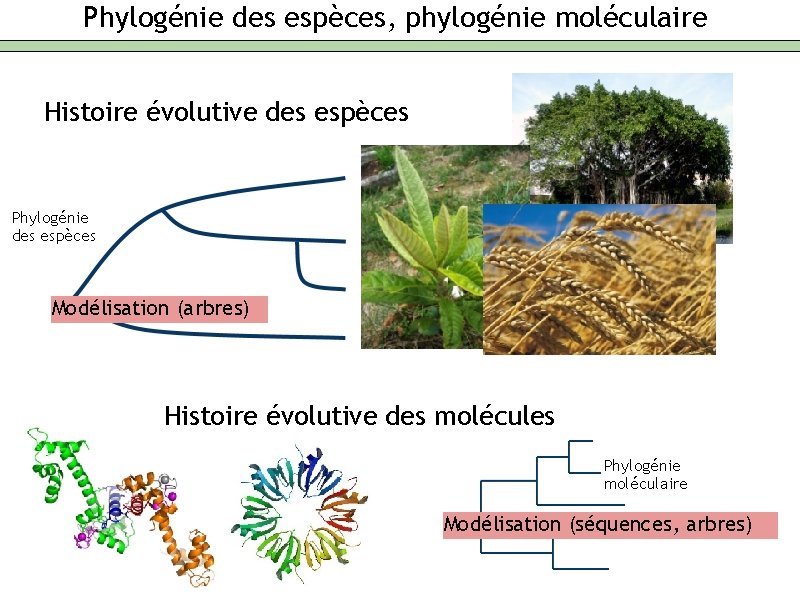
Phylogénie des espèces, phylogénie moléculaire Histoire évolutive des espèces Phylogénie des espèces Modélisation (arbres) Histoire évolutive des molécules Phylogénie moléculaire Modélisation (séquences, arbres)

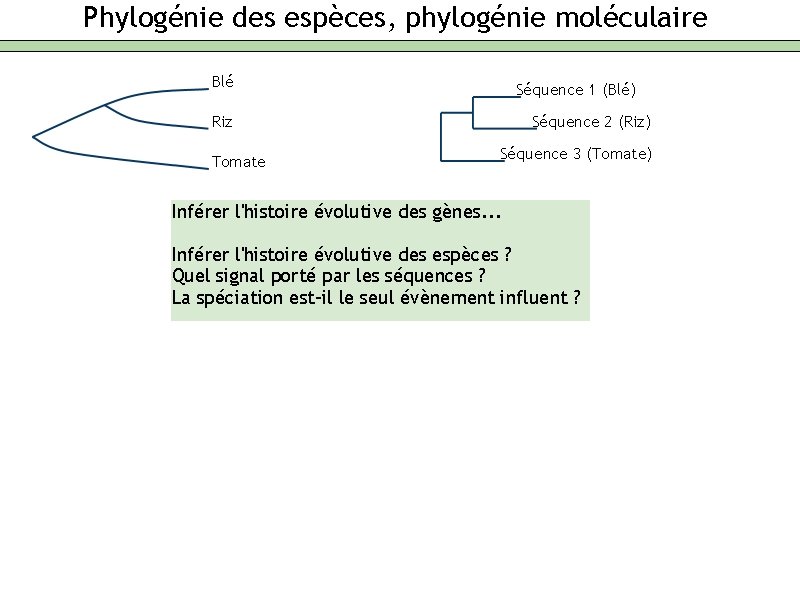
Phylogénie des espèces, phylogénie moléculaire Blé Séquence 1 (Blé) Riz Tomate Séquence 2 (Riz) Séquence 3 (Tomate) Inférer l'histoire évolutive des gènes. . . Inférer l'histoire évolutive des espèces ? Quel signal porté par les séquences ? La spéciation est-il le seul évènement influent ?

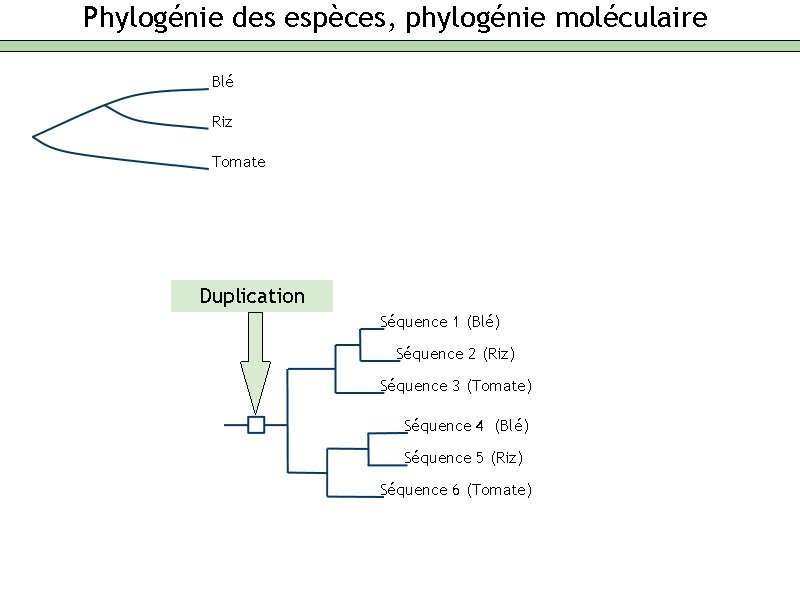
Phylogénie des espèces, phylogénie moléculaire Blé Riz Tomate Duplication Séquence 1 (Blé) Séquence 2 (Riz) Séquence 3 (Tomate) Séquence 4 (Blé) Séquence 5 (Riz) Séquence 6 (Tomate)

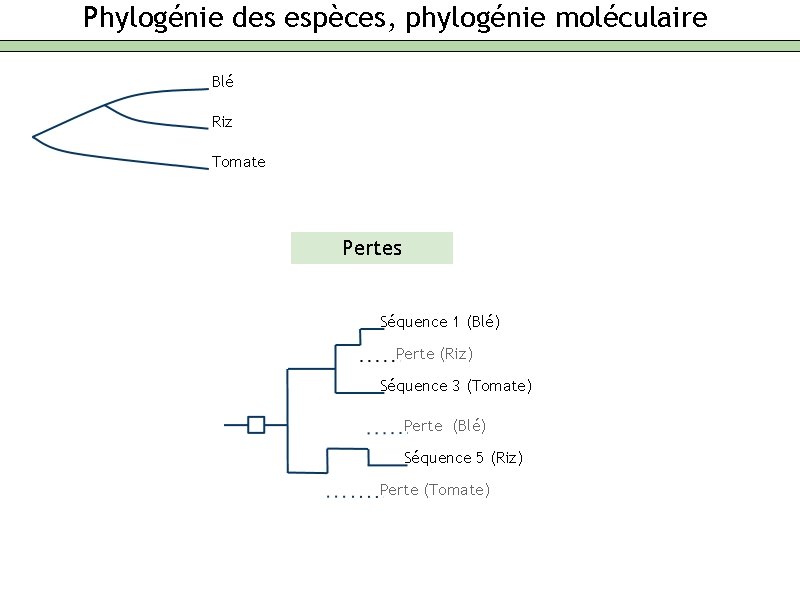
Phylogénie des espèces, phylogénie moléculaire Blé Riz Tomate Pertes Séquence 1 (Blé) Perte (Riz) Séquence 3 (Tomate) Perte (Blé) Séquence 5 (Riz) Perte (Tomate)

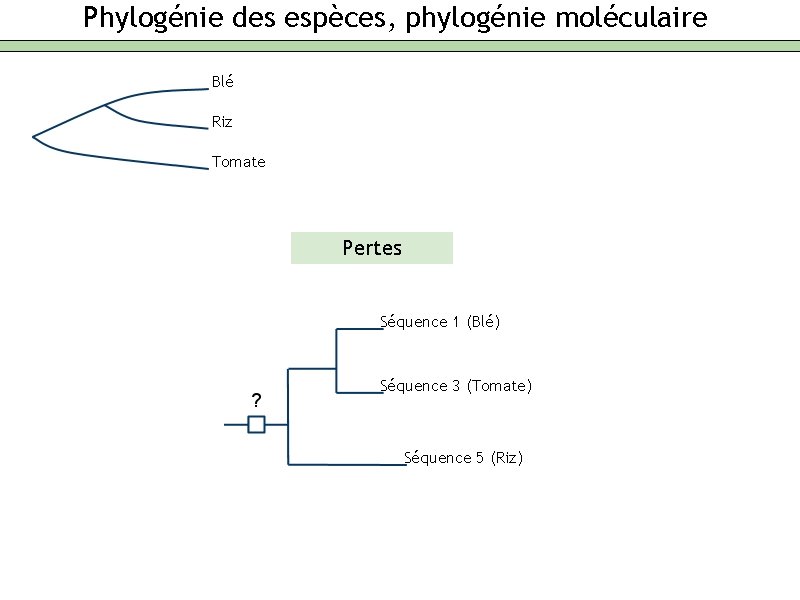
Phylogénie des espèces, phylogénie moléculaire Blé Riz Tomate Pertes Séquence 1 (Blé) Séquence 3 (Tomate) Séquence 5 (Riz)

Phylogénie des espèces, phylogénie moléculaire Blé Riz Tomate Réconciliation d'arbres Séquence 1 (Blé) Perte (Riz) Séquence 3 (Tomate) Perte (Blé) Séquence 5 (Riz) Perte (Tomate) Séquence 5 (Riz)

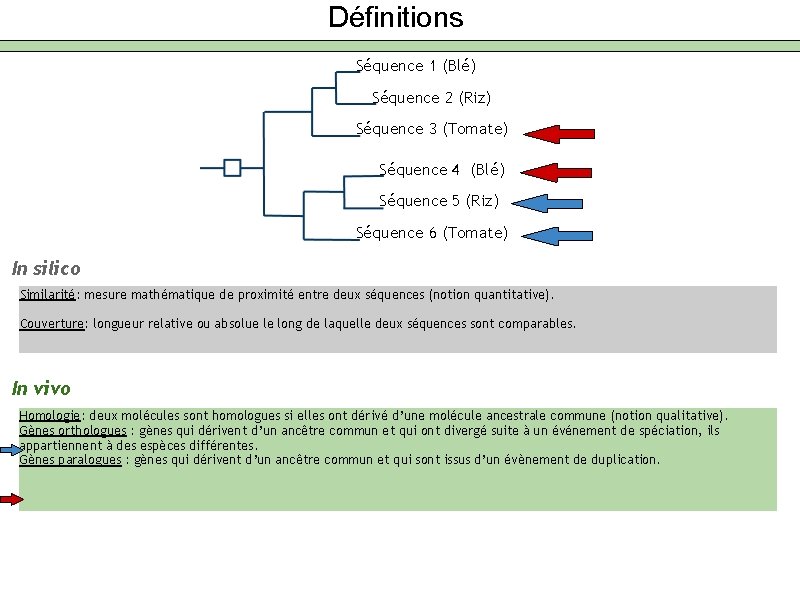
Définitions Séquence 1 (Blé) Séquence 2 (Riz) Séquence 3 (Tomate) Séquence 4 (Blé) Séquence 5 (Riz) Séquence 6 (Tomate) In silico Similarité: mesure mathématique de proximité entre deux séquences (notion quantitative). Couverture: longueur relative ou absolue le long de laquelle deux séquences sont comparables. In vivo Homologie: deux molécules sont homologues si elles ont dérivé d’une molécule ancestrale commune (notion qualitative). Gènes orthologues : gènes qui dérivent d’un ancêtre commun et qui ont divergé suite à un événement de spéciation, ils appartiennent à des espèces différentes. Gènes paralogues : gènes qui dérivent d’un ancêtre commun et qui sont issus d’un évènement de duplication.

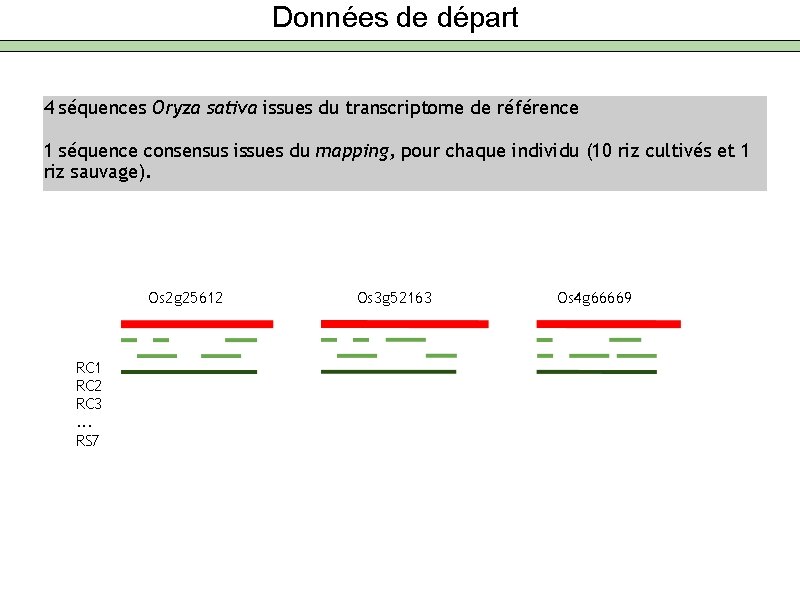
Données de départ 4 séquences Oryza sativa issues du transcriptome de référence 1 séquence consensus issues du mapping, pour chaque individu (10 riz cultivés et 1 riz sauvage). Os 2 g 25612 RC 1 RC 2 RC 3. . . RS 7 Os 3 g 52163 Os 4 g 66669

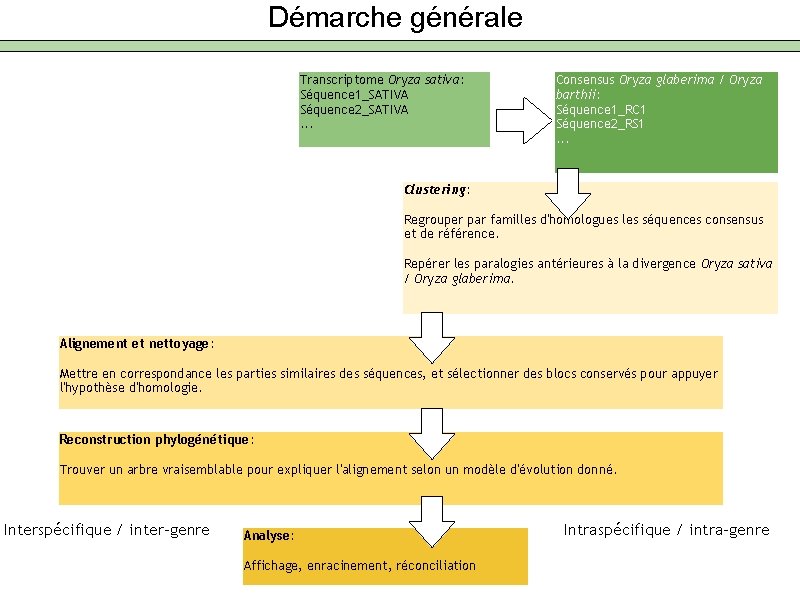
Démarche générale Transcriptome Oryza sativa: Séquence 1_SATIVA Séquence 2_SATIVA. . . Consensus Oryza glaberima / Oryza barthii: Séquence 1_RC 1 Séquence 2_RS 1. . . Clustering: Regrouper par familles d'homologues les séquences consensus et de référence. Repérer les paralogies antérieures à la divergence Oryza sativa / Oryza glaberima. Alignement et nettoyage: Mettre en correspondance les parties similaires des séquences, et sélectionner des blocs conservés pour appuyer l'hypothèse d'homologie. Reconstruction phylogénétique: Trouver un arbre vraisemblable pour expliquer l'alignement selon un modèle d'évolution donné. Interspécifique / inter-genre Analyse: Affichage, enracinement, réconciliation Intraspécifique / intra-genre

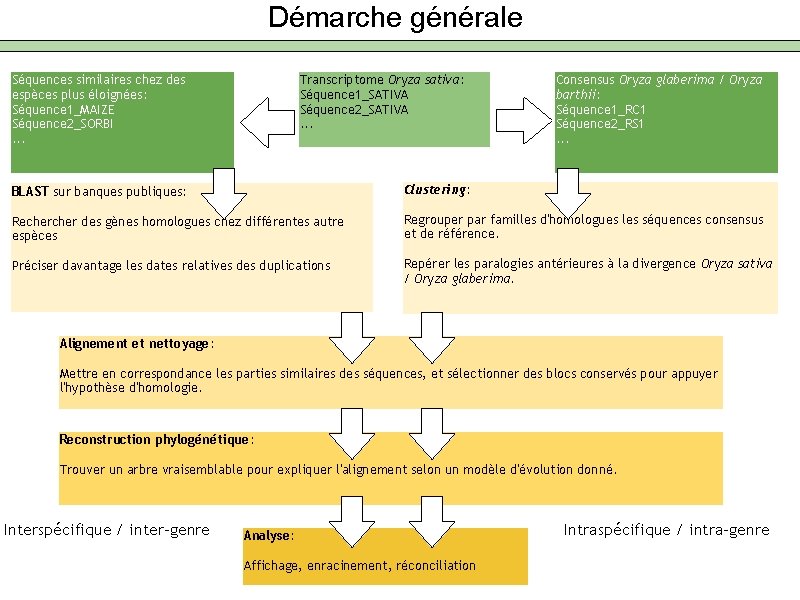
Démarche générale Séquences similaires chez des espèces plus éloignées: Séquence 1_MAIZE Séquence 2_SORBI. . . Transcriptome Oryza sativa: Séquence 1_SATIVA Séquence 2_SATIVA. . . Consensus Oryza glaberima / Oryza barthii: Séquence 1_RC 1 Séquence 2_RS 1. . . BLAST sur banques publiques: Clustering: Recher des gènes homologues chez différentes autre espèces Regrouper par familles d'homologues les séquences consensus et de référence. Préciser davantage les dates relatives duplications Repérer les paralogies antérieures à la divergence Oryza sativa / Oryza glaberima. Alignement et nettoyage: Mettre en correspondance les parties similaires des séquences, et sélectionner des blocs conservés pour appuyer l'hypothèse d'homologie. Reconstruction phylogénétique: Trouver un arbre vraisemblable pour expliquer l'alignement selon un modèle d'évolution donné. Interspécifique / inter-genre Analyse: Affichage, enracinement, réconciliation Intraspécifique / intra-genre

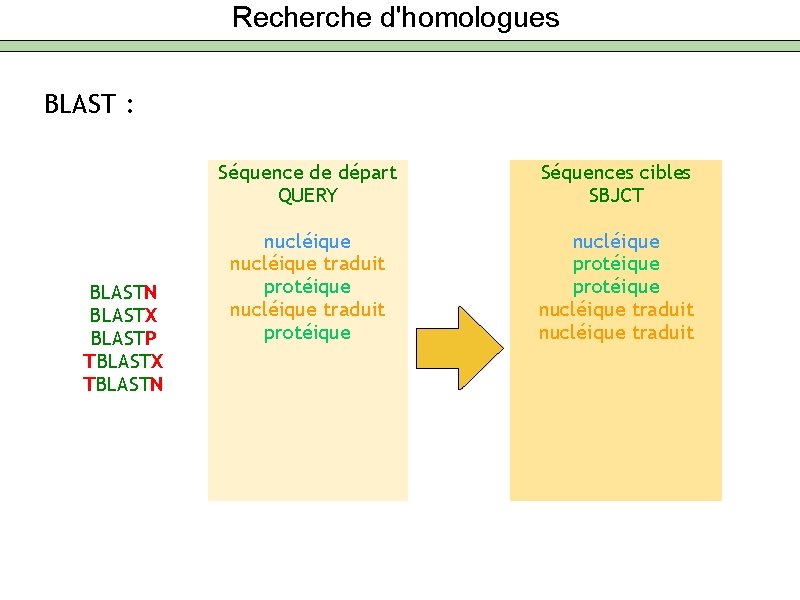
Recherche d'homologues BLAST : BLASTN BLASTX BLASTP TBLASTX TBLASTN Séquence de départ QUERY Séquences cibles SBJCT nucléique traduit protéique nucléique traduit
 Tudier
Tudier Conclusion sur lhistoire de linformatique
Conclusion sur lhistoire de linformatique Infrer
Infrer Des des des
Des des des Importance of critical thinking in nursing process
Importance of critical thinking in nursing process Rhetorical analysis
Rhetorical analysis Geicp
Geicp Concln
Concln La diffusion des idées des lumières
La diffusion des idées des lumières Diversification des espaces et des acteurs de la production
Diversification des espaces et des acteurs de la production Robin des bois des alpes
Robin des bois des alpes Il existe des personnes qui sont des lumières pour tous
Il existe des personnes qui sont des lumières pour tous Volume d un corps
Volume d un corps Triple des meet in the middle attack
Triple des meet in the middle attack Valeur de mode
Valeur de mode


























