Synthse de protines dans le cytosol On met




- Slides: 20

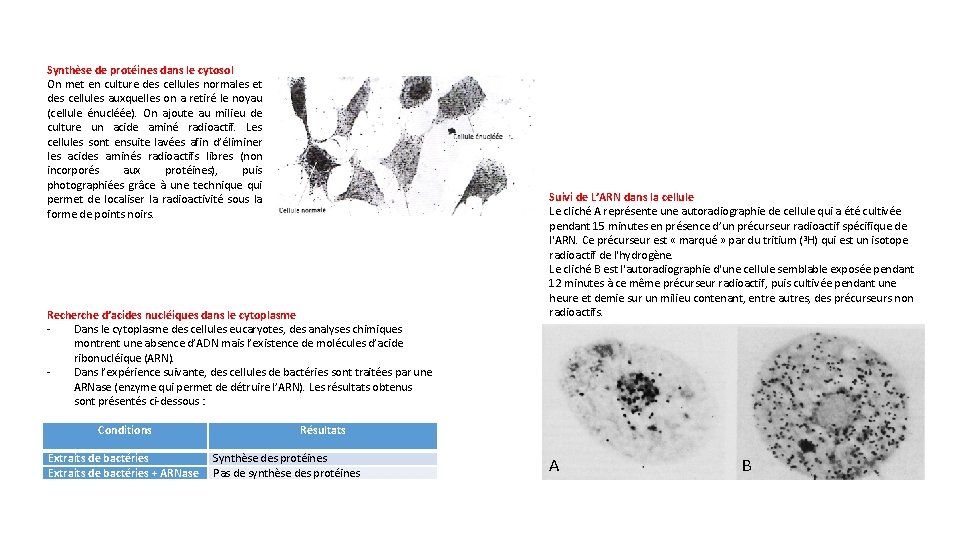
Synthèse de protéines dans le cytosol On met en culture des cellules normales et des cellules auxquelles on a retiré le noyau (cellule énucléée). On ajoute au milieu de culture un acide aminé radioactif. Les cellules sont ensuite lavées afin d’éliminer les acides aminés radioactifs libres (non incorporés aux protéines), puis photographiées grâce à une technique qui permet de localiser la radioactivité sous la forme de points noirs. Recherche d’acides nucléiques dans le cytoplasme Dans le cytoplasme des cellules eucaryotes, des analyses chimiques montrent une absence d’ADN mais l’existence de molécules d’acide ribonucléique (ARN). Dans l’expérience suivante, des cellules de bactéries sont traitées par une ARNase (enzyme qui permet de détruire l’ARN). Les résultats obtenus sont présentés ci-dessous : Conditions Extraits de bactéries + ARNase Suivi de L’ARN dans la cellule Le cliché A représente une autoradiographie de cellule qui a été cultivée pendant 15 minutes en présence d’un précurseur radioactif spécifique de l'ARN. Ce précurseur est « marqué » par du tritium (3 H) qui est un isotope radioactif de l'hydrogène. Le cliché B est l'autoradiographie d'une cellule semblable exposée pendant 12 minutes à ce même précurseur radioactif, puis cultivée pendant une heure et demie sur un milieu contenant, entre autres, des précurseurs non radioactifs. Résultats Synthèse des protéines Pas de synthèse des protéines A B

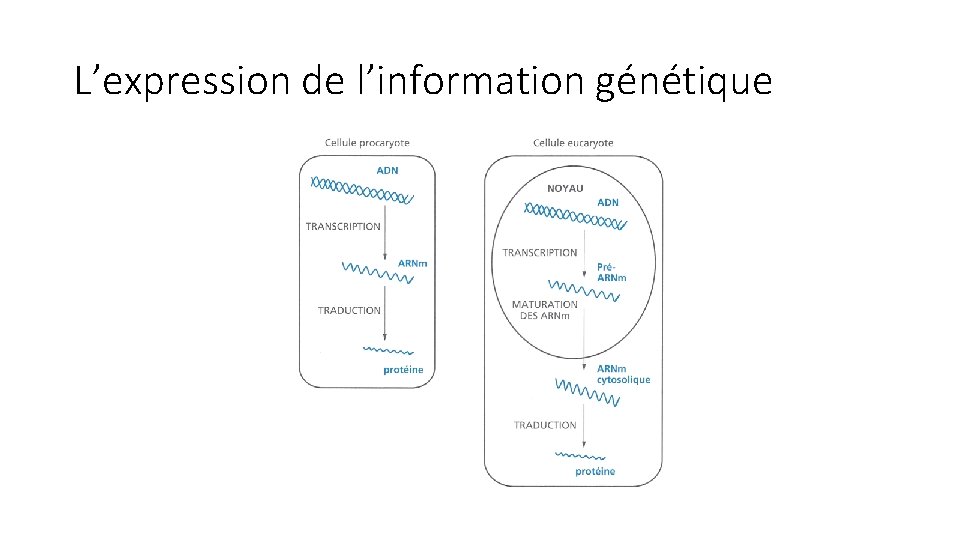
L’expression de l’information génétique

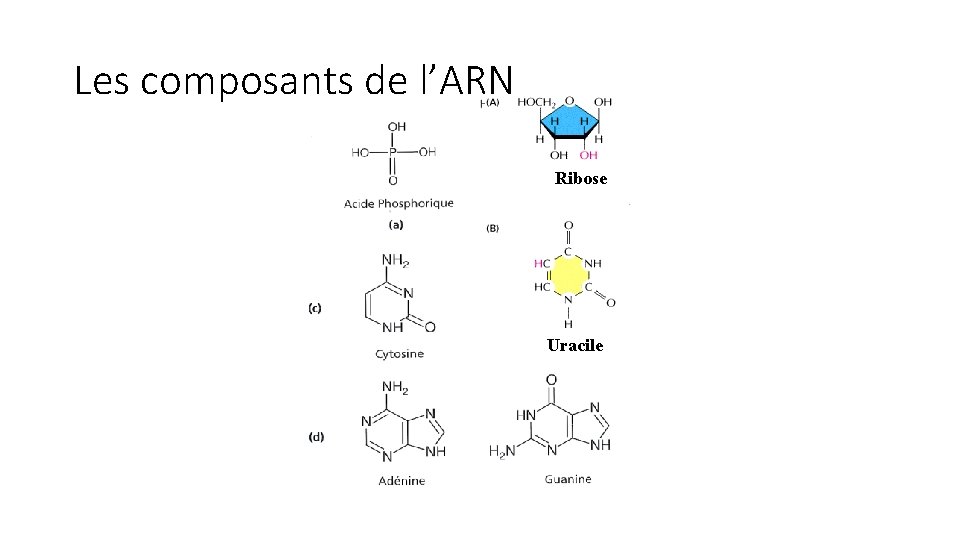
Les composants de l’ARN Ribose Uracile

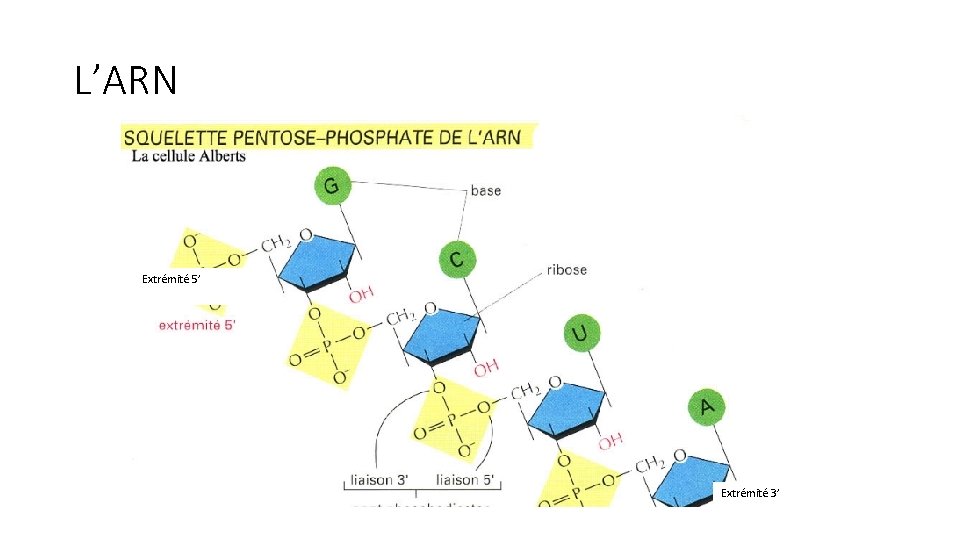
L’ARN Extrémité 5’ Extrémité 3’

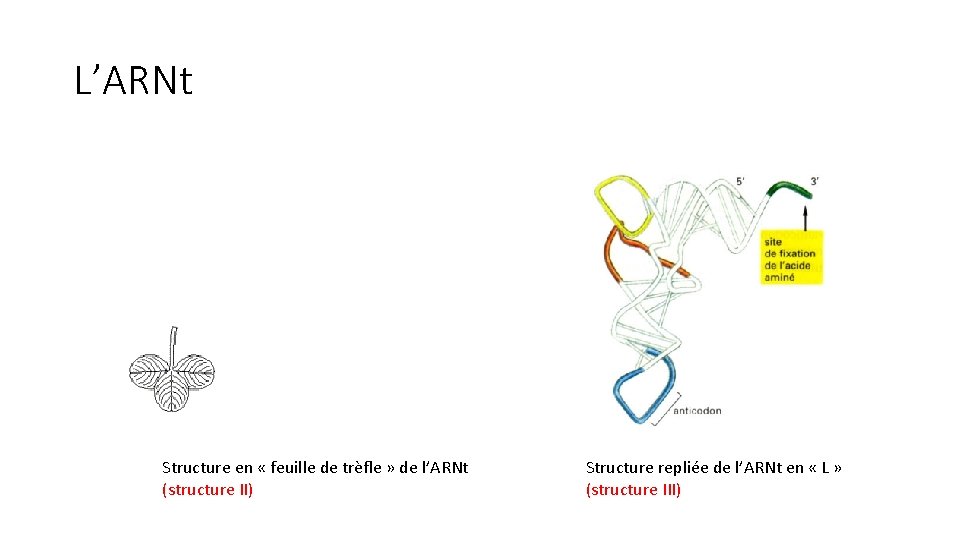
L’ARNt Structure en « feuille de trèfle » de l’ARNt (structure II) Structure repliée de l’ARNt en « L » (structure III)

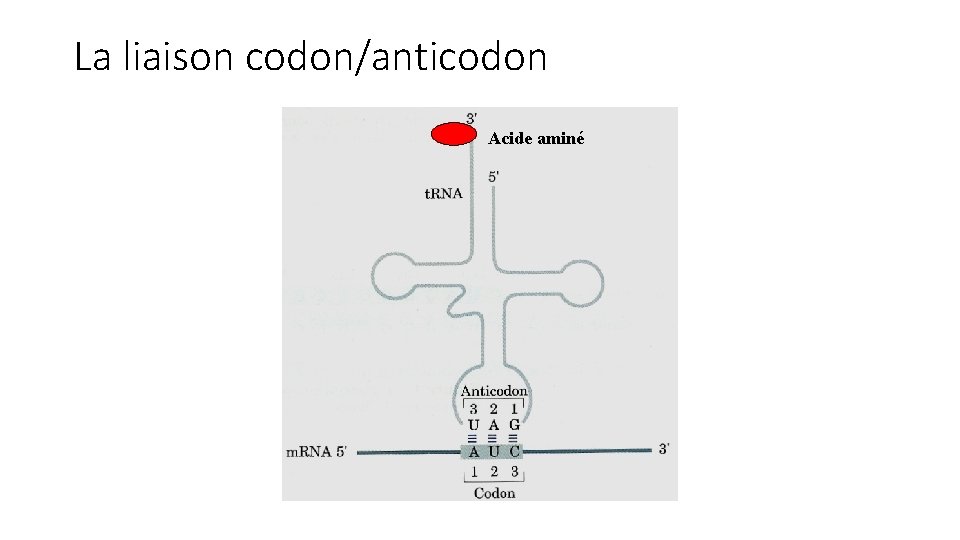
La liaison codon/anticodon Acide aminé

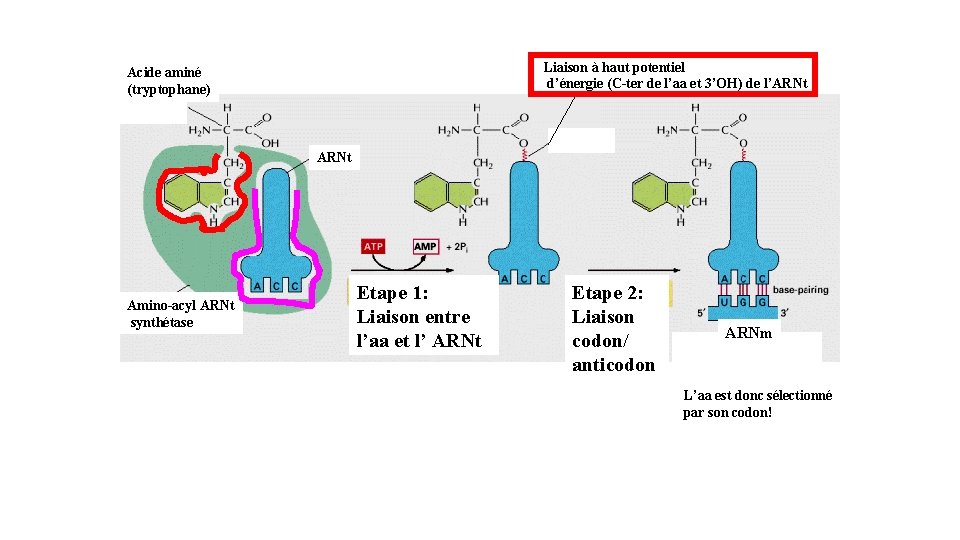
Liaison à haut potentiel d’énergie (C-ter de l’aa et 3’OH) de l’ARNt Acide aminé (tryptophane) ARNt Amino-acyl ARNt synthétase Etape 1: Liaison entre l’aa et l’ ARNt Etape 2: Liaison codon/ anticodon ARNm L’aa est donc sélectionné par son codon!

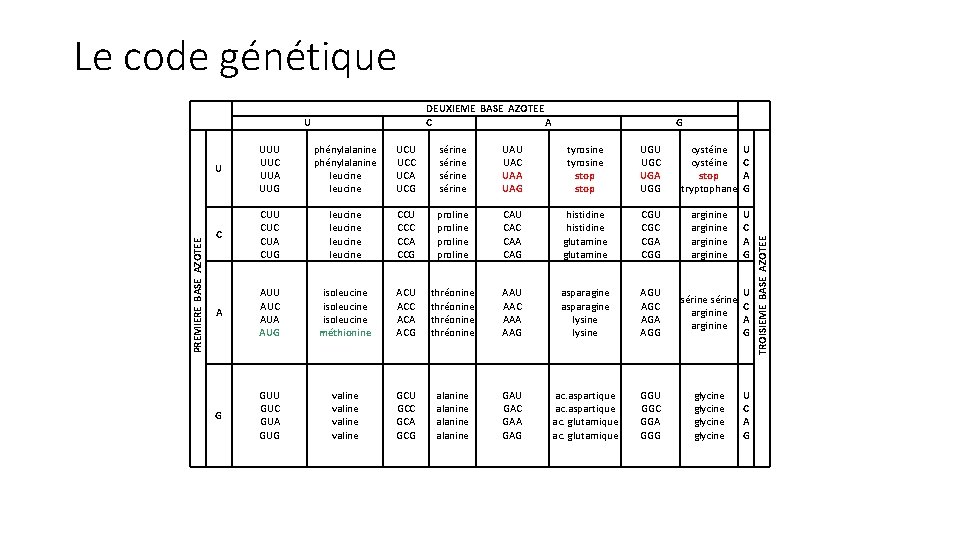
Le code génétique DEUXIEME BASE AZOTEE C A G U UUC UUA UUG phénylalanine leucine UCU UCC UCA UCG sérine UAU UAC UAA UAG tyrosine stop UGU UGC UGA UGG cystéine stop tryptophane U C A G C CUU CUC CUA CUG leucine CCU CCC CCA CCG proline CAU CAC CAA CAG histidine glutamine CGU CGC CGA CGG arginine U C A G A AUU AUC AUA AUG isoleucine méthionine ACU ACC ACA ACG thréonine AAU AAC AAA AAG asparagine lysine AGU AGC AGA AGG G GUU GUC GUA GUG valine GCU GCC GCA GCG alanine GAU GAC GAA GAG ac. aspartique ac. glutamique GGU GGC GGA GGG U sérine C arginine A arginine G glycine U C A G TROISIEME BASE AZOTEE PREMIERE BASE AZOTEE U

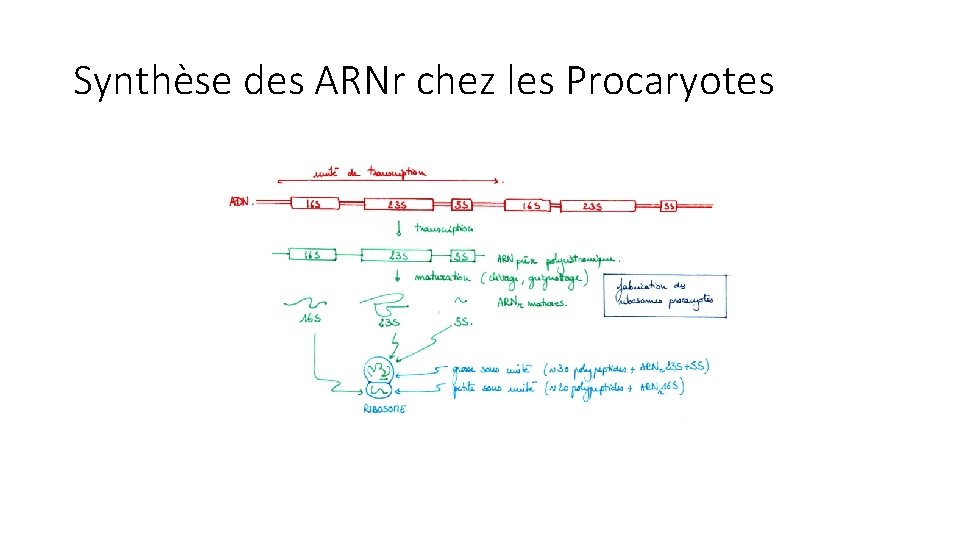
Comparaison des ribosomes procaryotes et eucaryotes Petite sous unité Grosse sous unité Procaryote ARN 16 S + 20 chaînes polypeptidiques ARN 23 S ARN 5 S + 30 chaînes polypeptidiques Ribosome 70 S eucaryote Sous unité 30 S ARN 18 S Ribosome 80 S Sous unité 40 S Sous unité 50 S ARN 28 S ARN 5, 8 S ARN 5 S Sous unité 60 S

Synthèse des ARNr chez les Procaryotes

Le nucléole

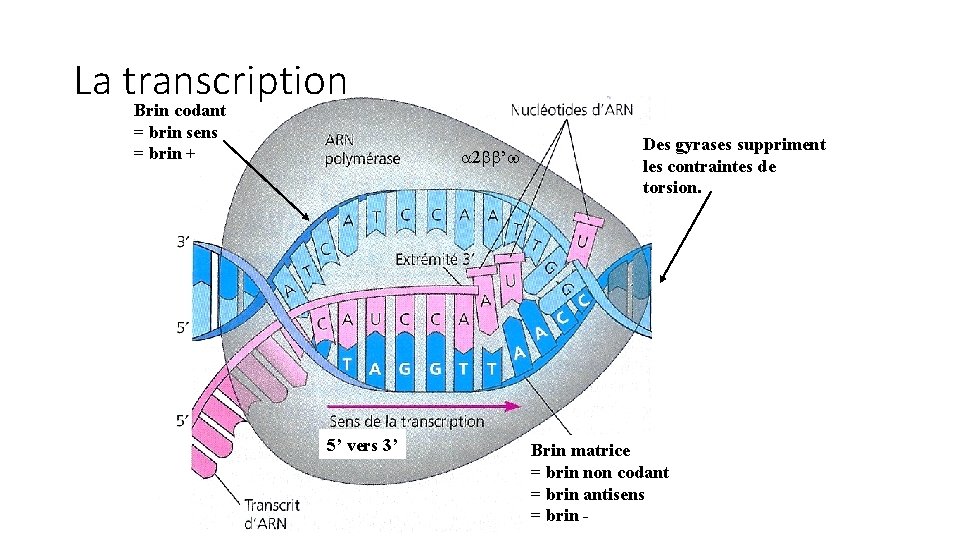
La transcription Brin codant = brin sens = brin + a 2 bb’w 5’ vers 3’ Des gyrases suppriment les contraintes de torsion. Brin matrice = brin non codant = brin antisens = brin -

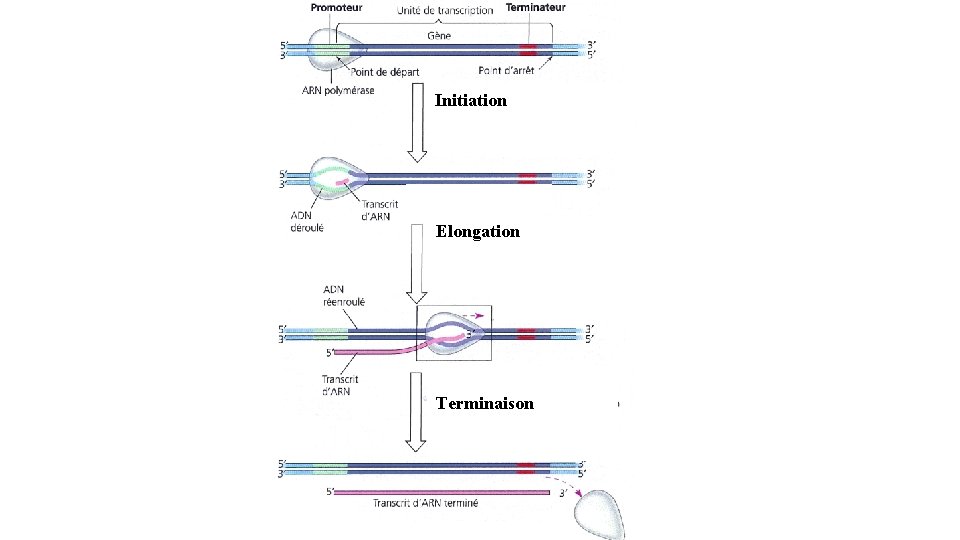
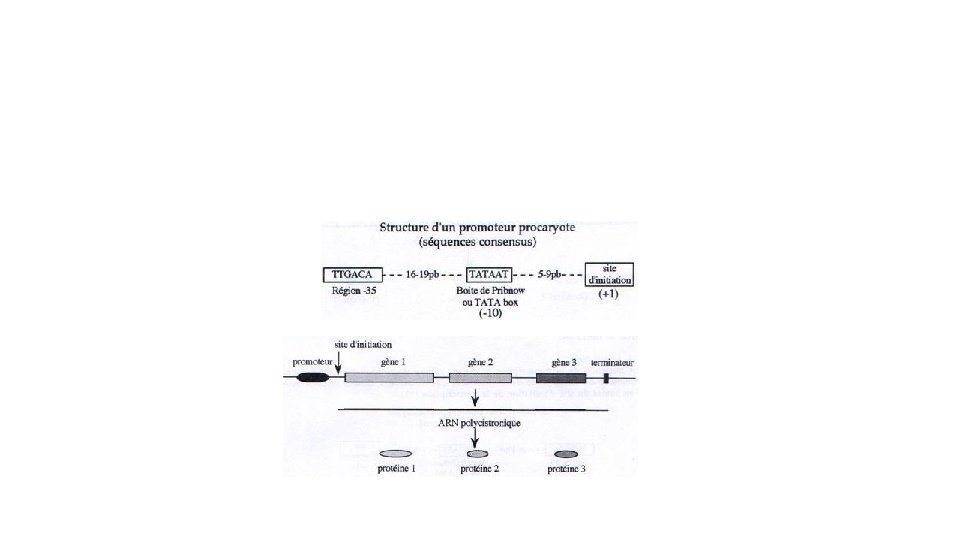
Initiation Elongation Terminaison

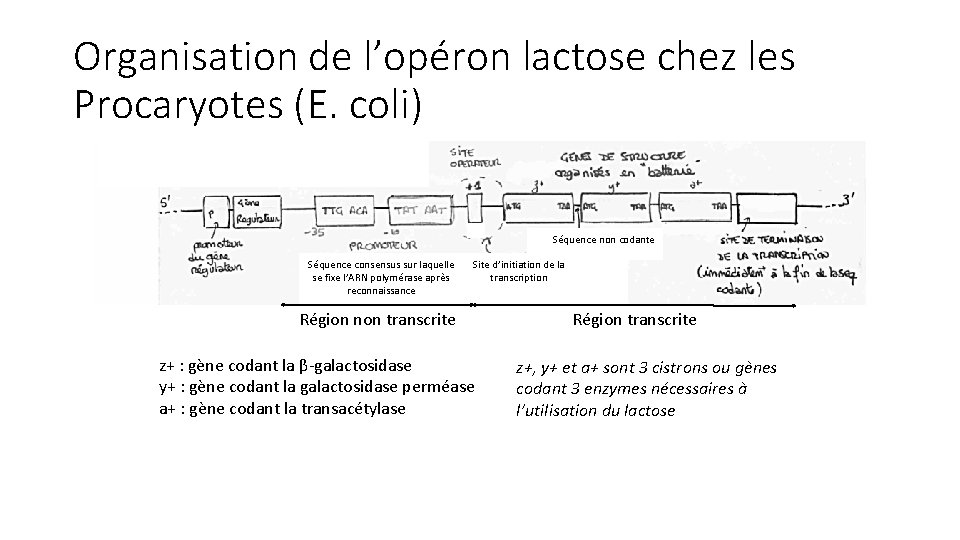
Organisation de l’opéron lactose chez les Procaryotes (E. coli) Séquence non codante Séquence consensus sur laquelle se fixe l’ARN polymérase après reconnaissance Site d’initiation de la transcription Région non transcrite z+ : gène codant la β-galactosidase y+ : gène codant la galactosidase perméase a+ : gène codant la transacétylase Région transcrite z+, y+ et a+ sont 3 cistrons ou gènes codant 3 enzymes nécessaires à l’utilisation du lactose

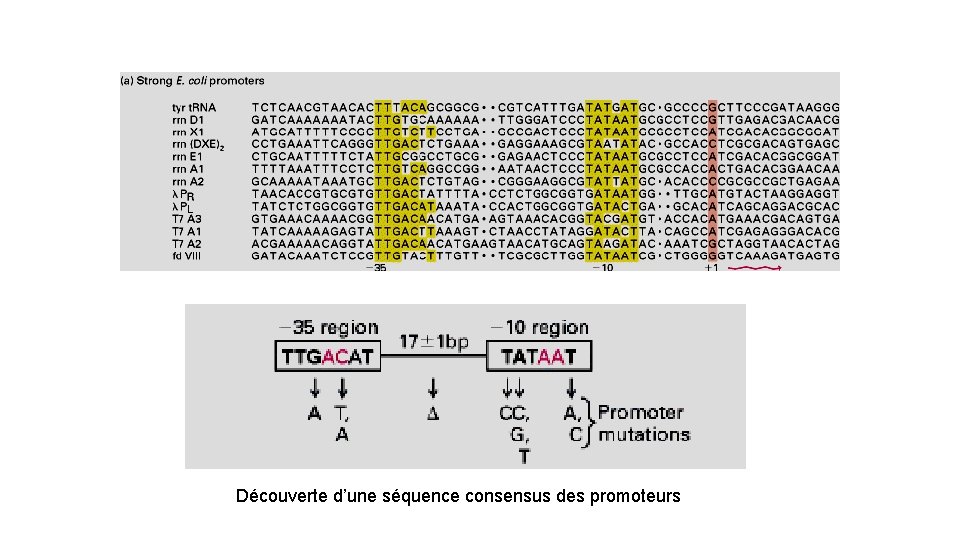
Séquence consensus: séquence idéalisée d’une région donnée d’un acide nucléique ou d’une protéine dans laquelle chaque position représente la base ou l’acide aminé rencontré le plus fréquemment. Comparaison des séquences promotrices de différents gènes (sens)

Découverte d’une séquence consensus des promoteurs

La transcription observée au MET figures en « arbre de Noël » : plusieurs molécules d’ARNm de longueurs différentes, en cours de formation. Plusieurs ARN polymérases se succèdent le long d’un même segment d’ADN et entament la fabrication « à la chaîne » d’ARN m identiques, qui seront autant de copies d’un même gène.



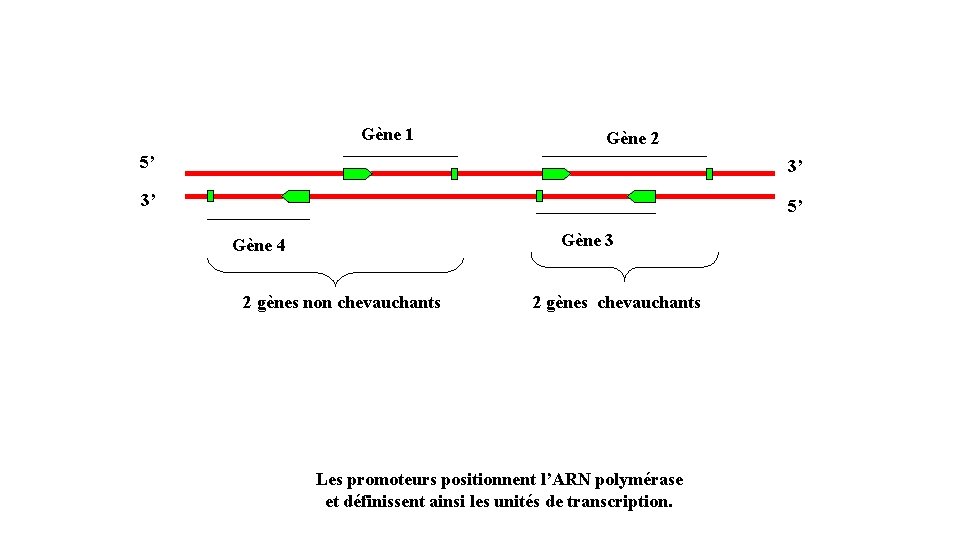
Gène 1 Gène 2 5’ 3’ 3’ 5’ Gène 3 Gène 4 2 gènes non chevauchants 2 gènes chevauchants Les promoteurs positionnent l’ARN polymérase et définissent ainsi les unités de transcription.