Citogenetica evoluzionistica La citogenetica evoluzionistica pu riferirsi a




- Slides: 13

Citogenetica evoluzionistica La citogenetica evoluzionistica può riferirsi a due aspetti del rapporto fra cromosomi ed evoluzione: 1) l’uso dei cromosomi come marcatori di processi evolutivi; 2) il ruolo dei cromosomi nei processi evolutivi. Sul primo punto la “citogenetica comparata” consente da un lato di “classificare i cariotipi” ovvero specie/popolazioni in base al cariotipo, dall’altro di definire le relazioni filogenetiche fra i gruppi tassonomici in base alla sequenza degli eventi mutazionali che hanno dato vita a successivi riordinamenti cromosomici, fino a costruire “alberi filogenetici”. Sul secondo punto si indaga il ruolo delle mutazioni cromosomiche (numeriche e strutturali) nell’anagenesi (cambiamento nel tempo di un pool genico) e nella cladogenesi (separazione tra pool genici). L’obiettivo del modulo è rivolto al ruolo delle mutazioni cromosomiche nei processi evolutivi, soprattutto nella cladogenesi.

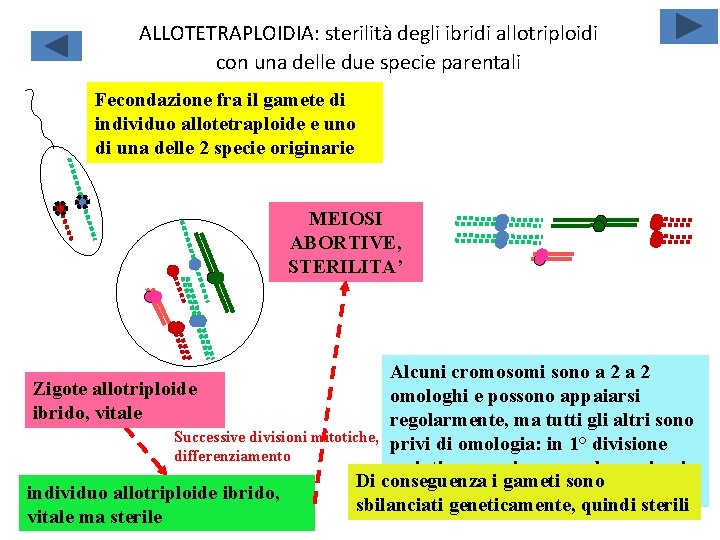
ALLOTETRAPLOIDIA: sterilità degli ibridi allotriploidi con una delle due specie parentali Fecondazione fra il gamete di individuo allotetraploide e uno di una delle 2 specie originarie MEIOSI ABORTIVE, STERILITA’ Alcuni cromosomi sono a 2 Zigote allotriploide omologhi e possono appaiarsi ibrido, vitale regolarmente, ma tutti gli altri sono Successive divisioni mitotiche, privi di omologia: in 1° divisione differenziamento meiotica non riescono ad appaiarsi e Di conseguenza i gameti sono segregano casualmente individuo allotriploide ibrido, sbilanciati geneticamente, quindi sterili vitale ma sterile

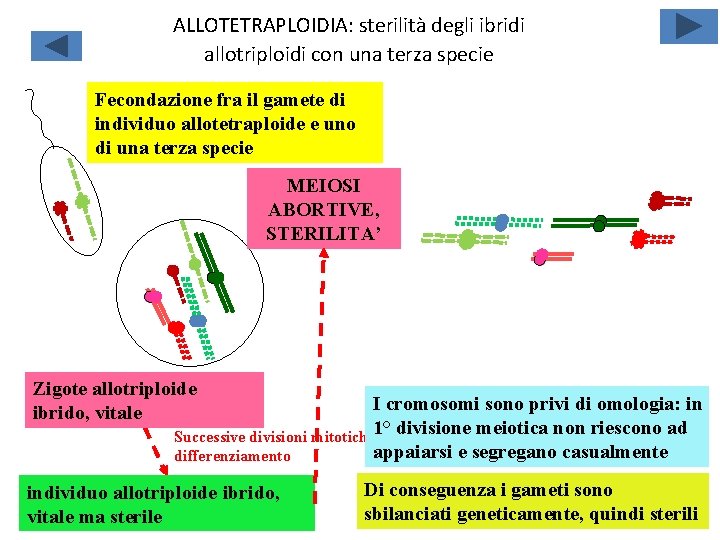
ALLOTETRAPLOIDIA: sterilità degli ibridi allotriploidi con una terza specie Fecondazione fra il gamete di individuo allotetraploide e uno di una terza specie MEIOSI ABORTIVE, STERILITA’ Zigote allotriploide ibrido, vitale I cromosomi sono privi di omologia: in Successive divisioni mitotiche, 1° divisione meiotica non riescono ad appaiarsi e segregano casualmente differenziamento individuo allotriploide ibrido, vitale ma sterile Di conseguenza i gameti sono sbilanciati geneticamente, quindi sterili

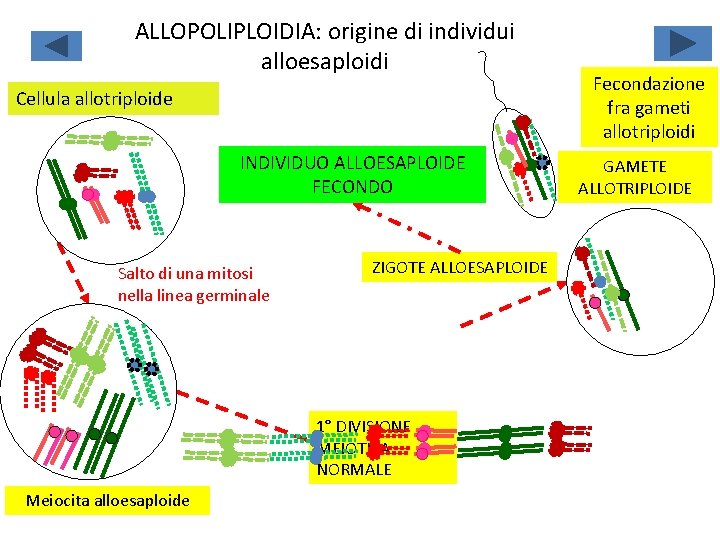
ALLOPOLIPLOIDIA: origine di individui alloesaploidi Cellula allotriploide INDIVIDUO ALLOESAPLOIDE FECONDO Salto di una mitosi nella linea germinale ZIGOTE ALLOESAPLOIDE 1° DIVISIONE MEIOTICA NORMALE Meiocita alloesaploide Fecondazione fra gameti allotriploidi GAMETE ALLOTRIPLOIDE

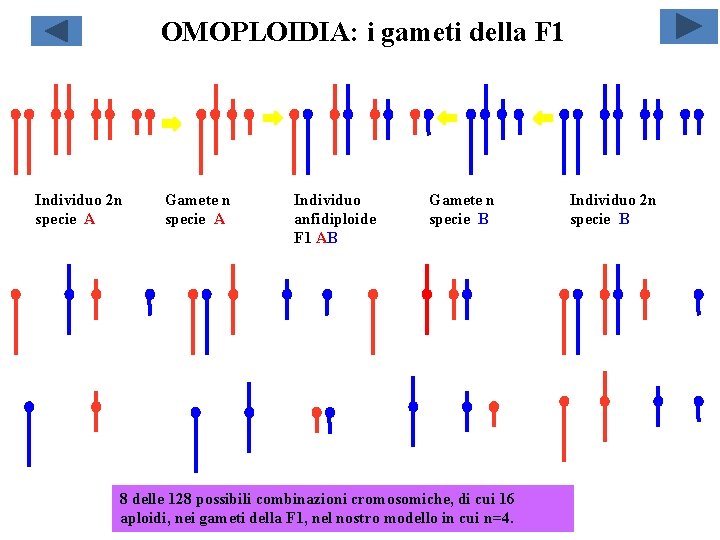
OMOPLOIDIA: i gameti della F 1 Individuo 2 n specie A Gamete n specie A Individuo anfidiploide F 1 AB Gamete n specie B 8 delle 128 possibili combinazioni cromosomiche, di cui 16 aploidi, nei gameti della F 1, nel nostro modello in cui n=4. Individuo 2 n specie B

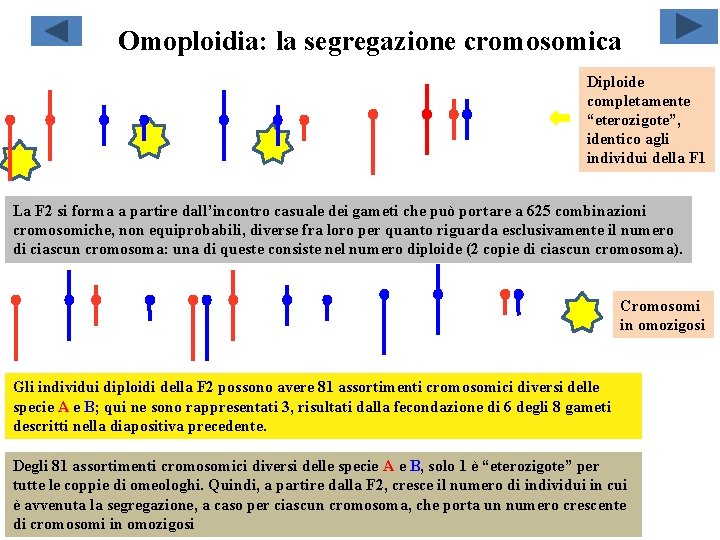
Omoploidia: la segregazione cromosomica Diploide completamente “eterozigote”, identico agli individui della F 1 La F 2 si forma a partire dall’incontro casuale dei gameti che può portare a 625 combinazioni cromosomiche, non equiprobabili, diverse fra loro per quanto riguarda esclusivamente il numero di ciascun cromosoma: una di queste consiste nel numero diploide (2 copie di ciascun cromosoma). Cromosomi in omozigosi Gli individui diploidi della F 2 possono avere 81 assortimenti cromosomici diversi delle specie A e B; qui ne sono rappresentati 3, risultati dalla fecondazione di 6 degli 8 gameti descritti nella diapositiva precedente. Degli 81 assortimenti cromosomici diversi delle specie A e B, solo 1 è “eterozigote” per tutte le coppie di omeologhi. Quindi, a partire dalla F 2, cresce il numero di individui in cui è avvenuta la segregazione, a caso per ciascun cromosoma, che porta un numero crescente di cromosomi in omozigosi

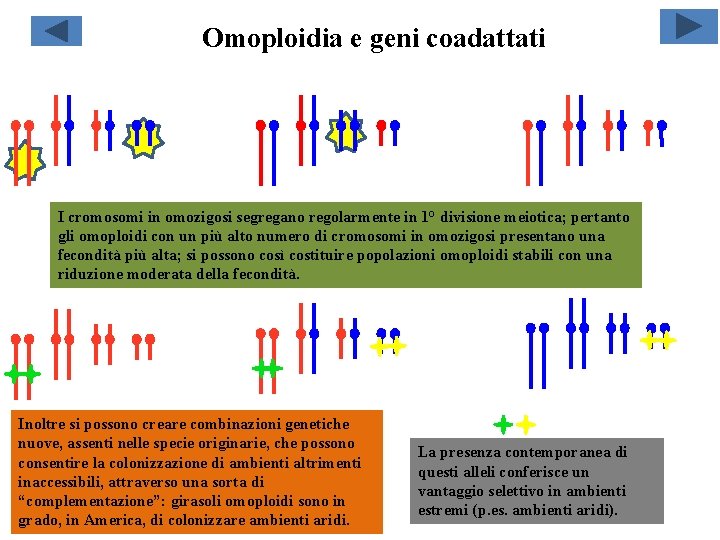
Omoploidia e geni coadattati I cromosomi in omozigosi segregano regolarmente in 1° divisione meiotica; pertanto gli omoploidi con un più alto numero di cromosomi in omozigosi presentano una fecondità più alta; si possono così costituire popolazioni omoploidi stabili con una riduzione moderata della fecondità. Inoltre si possono creare combinazioni genetiche nuove, assenti nelle specie originarie, che possono consentire la colonizzazione di ambienti altrimenti inaccessibili, attraverso una sorta di “complementazione”: girasoli omoploidi sono in grado, in America, di colonizzare ambienti aridi. La presenza contemporanea di questi alleli conferisce un vantaggio selettivo in ambienti estremi (p. es. ambienti aridi).

Mutazione, migrazione e selezione MUTAZIONE MIGRAZIONE Se A 2 muta in A 1 a un tasso costante m per generazione, allora: Se da una popolazione donatrice, in cui A 1 ha una frequenza P, immigra una frazione m della popolazione ricevente per generazione, allora: p=mq p=m(P- p) SELEZIONE L’idoneità riproduttiva (“fitness” – W) di un genotipo (per il genotipo A 1 A 2, WA 1 A 2) è la sua probabilità di sopravvivere e produrre progenie feconda. Si usa come misura della fitness “normalizzata” (W’) che si ottiene dividendo il valore della fitness di ciascun genotipo per il valore di fitness più alto tra i genotipi del gene interessato. La selezione determina Quindi 0<W’<1) un cambiamento delle frequenze alleliche a causa della differente La fitness media di una fitness degli alleli: popolazione è La fitness media 2 W’=p W’A 1 A 1+2 pq. W’A 1 A 2+ dell’allele A 1 è p=pq(W’A 1 -W’A 2)/W’ 2 q W’ W’ =p. W’ +q. W’ A 2 A 2 Il progressivo cambiamento delle frequenze alleliche può portare ai valori p=0 o p=1 A 1 A 1 Quando p=1, l’allele A 1 è fissato nella popolazione; quando p=0, l’allele A 1 è eliminato A 1 A 2 Quando un allele è fissato e gli altri sono eliminati, la popolazione da polimorfa diviene monomorfa e in essa non sono più possibili variazioni delle frequenze alleliche

Variazioni della frequenze alleliche dovute a diversi tipi di selezione A 1 A 1 A 1 A 2 A 2 A 2 Dp Direzionale positiva recessiva 1 1 -s sp 2 q/(1 -sq(2 p+q)) Direzionale positiva dominante 1 1 1 -s spq 2/(1 -sq 2) Stabilizzatrice 1 -s 1 1 -s spq(q-p)/(1 -s(p 2+q 2) Diversificatrice 1 1 -s 1 spq(p-q)/(1 -2 spq) s=1 -W’(per ogni genotipo)= coefficiente di selezione Bilanciamento fra mutazione e selezione Se un allele dannoso A 1 con coefficiente di selezione s viene introdotto in una popolazione a un tasso costante di mutazione per generazione m, sono raggiunte le seguenti frequenze all’equilibrio p^. Per un allele recessivo p^=(m/s)1/2 Per un allele dominante p^=m/s

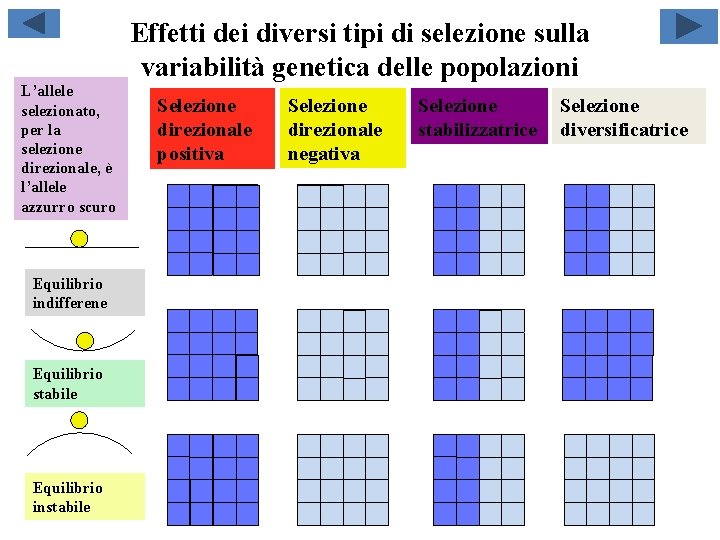
Effetti dei diversi tipi di selezione sulla variabilità genetica delle popolazioni L’allele selezionato, per la selezione direzionale, è l’allele azzurro scuro Equilibrio indifferene Equilibrio stabile Equilibrio instabile Selezione direzionale positiva Selezione direzionale negativa Selezione stabilizzatrice Selezione diversificatrice

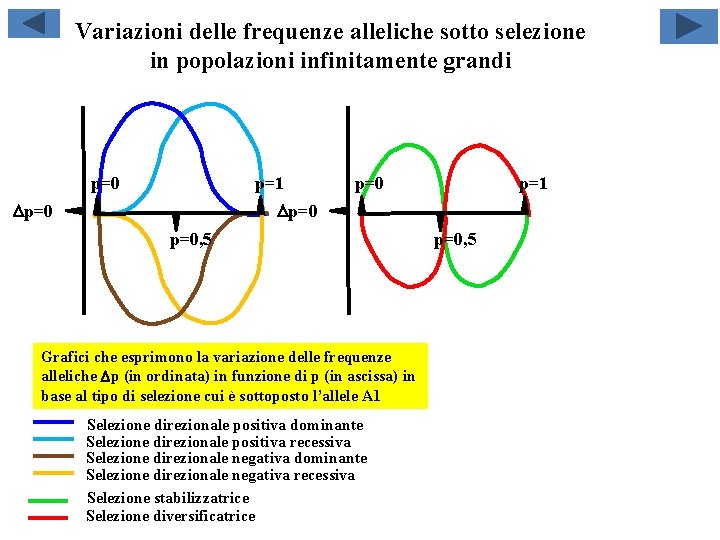
Variazioni delle frequenze alleliche sotto selezione in popolazioni infinitamente grandi p=0 p=1 Dp=0 p=0, 5 Grafici che esprimono la variazione delle frequenze alleliche Dp (in ordinata) in funzione di p (in ascissa) in base al tipo di selezione cui è sottoposto l’allele A 1 Selezione direzionale positiva dominante Selezione direzionale positiva recessiva Selezione direzionale negativa dominante Selezione direzionale negativa recessiva Selezione stabilizzatrice Selezione diversificatrice p=0, 5

Diversi tipi di selezione: conseguenze evolutive La selezione direzionale Avvantaggia uno dei 2 genotipi omozigoti (A 1 A 1) e svantaggia l’altro genotipo omozigote (A 2 A 2) e il genotipo eterozigote (A 1 A 2) (effetto vantaggioso recessivo per A 1, svantaggioso dominante per A 2: W’A 1 A 1>W’A 1 A 2=W’A 2 A 2) Avvantaggia uno dei 2 genotipi omozigoti (A 1 A 1) e il genotipo eterozigote (A 1 A 2) e svantaggia l’altro genotipo omozigote (A 2 A 2) (effetto vantaggioso dominante per A 1, svantaggioso recessivo per A 2: W’A 1 A 1=W’A 1 A 2>W’A 2 A 2) L’effetto della selezione direzionale è comunque la fissazione dell’allele avvantaggiato e l’eliminazione dell’allele svantaggiato La selezione stabilizzatrice Avvantaggia il genotipo eterozigote (A 1 A 2) e svantaggia i genotipi omozigoti (A 1 A 1, A 2 A 2) W’A 1 A 2>W’A 1 A 1; W’A 1 A 2>W’A 2 A 2) L’effetto della selezione stabilizzatrice è la persistenza di entrambi gli alleli con frequenze all’equilibrio diverse da 0 e 1; il valore di queste frequenze dipende dalle fitness dei genotipi La selezione diversificatrice Svantaggia il genotipo eterozigote (A 1 A 2) e avvantaggia i genotipi omozigoti (A 1 A 1, A 2 A 2) W’A 1 A 2<W’A 1 A 1; W’A 1 A 2<W’A 2 A 2) L’effetto della selezione diversificatrice è la fissazione di uno dei due alleli e l’eliminazione dell’altro; quale allele venga fissato e quale eliminato dipende dalle frequenze alleliche L’assenza di selezione Nessun genotipo è avvantaggiato (neutralità: W’A 1 A 1=W’A 1 A 2=WA’ 2 A 2=1)

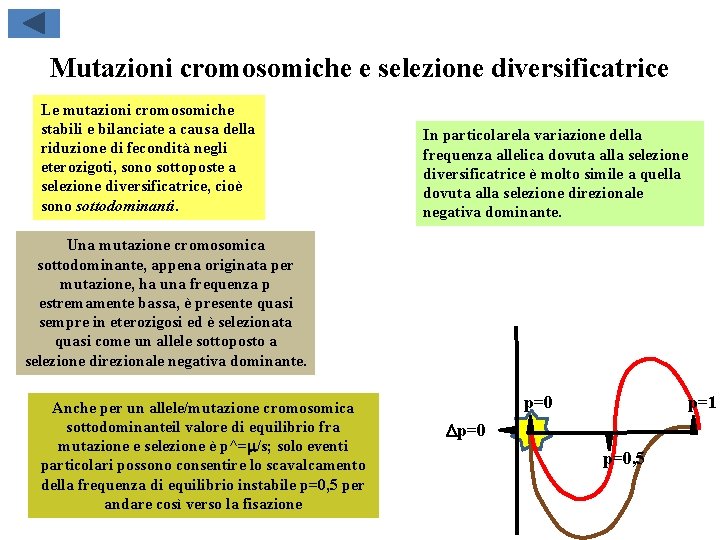
Mutazioni cromosomiche e selezione diversificatrice Le mutazioni cromosomiche stabili e bilanciate a causa della riduzione di fecondità negli eterozigoti, sono sottoposte a selezione diversificatrice, cioè sono sottodominanti. In particolarela variazione della frequenza allelica dovuta alla selezione diversificatrice è molto simile a quella dovuta alla selezione direzionale negativa dominante. Una mutazione cromosomica sottodominante, appena originata per mutazione, ha una frequenza p estremamente bassa, è presente quasi sempre in eterozigosi ed è selezionata quasi come un allele sottoposto a selezione direzionale negativa dominante. Anche per un allele/mutazione cromosomica sottodominanteil valore di equilibrio fra mutazione e selezione è p^=m/s; solo eventi particolari possono consentire lo scavalcamento della frequenza di equilibrio instabile p=0, 5 per andare così verso la fisazione p=0 p=1 Dp=0 p=0, 5