Aula 24 Goodies Goodies related to animals plants
















![#another example, now with data iris data(iris) my. PCAiris=rda(scale(iris[, 1: 4])) par(mfrow=c(1, 2), mar=c(4, #another example, now with data iris data(iris) my. PCAiris=rda(scale(iris[, 1: 4])) par(mfrow=c(1, 2), mar=c(4,](https://slidetodoc.com/presentation_image_h2/f0ac69c75d66a96fba59aa96b1f38a1a/image-26.jpg)
- Slides: 28

Aula 24 Goodies * Goodies related to animals, plants and numbers… *

Resultados detalhados na página da aula T 23

Ecologia Numérica: Avaliação: – 50% exame teórico + 50% exame teórico-prático com recurso a computador e software – Duração de cerca de 2 horas – Precisam de ter pelo menos 8 a ambas as partes para serem aprovados Datas: 1 a Época 2 a Época Especial Teórico 22 Jan 2019 – 16: 30 -19: 30 08 Fev 2019 – 16: 30 -19: 30 22 Jul 2019 – 14: 30 -16: 30 Teórico-Prático 22 Jan 2019 – 8: 00 -16: 00 08 Fev 2019 – 9: 00 -15: 00 22 Jul 2019 – 11: 30 -13: 30 Para ser possível a aprovação da disciplina, os alunos terão que garantir presença em pelo menos 10 das aulas teórico-práticas, não sendo exigida presença obrigatória nas aulas teóricas.

EXAMES Prático Teórico 1. Com recurso ao computador 2. Logo com consulta 3. Logo com perguntas que implicam ligar o cérebro 4. Feito a pares (por razões logísticas), mas quem quiser fazer sozinho pode 5. Perguntas parecidas com o que foi sendo implementado nas TPs 6. Principal atributo a ser avaliado é a capacidade de interpretação, vs. capacidade de decorar coisas 7. Será necessário inscreverem-se no exame e selecionar o horário desejado 1. Precisam de uma máquina de calcular simples, não aceito uso de telemóveis! 2. Não tem consulta – tentativas de consulta não autorizadas não serão admitidas e implicam anulação do exame 3. Foca-se nos aspetos mais teóricos/conceptuais 4. No entanto, privilegio o raciocínio critico e a criatividade em detrimento da “decoração”

Ecologia Numérica - Aula Teórica 24 – 04 -12 -2018 https: //www. azquotes. com/quote/727622

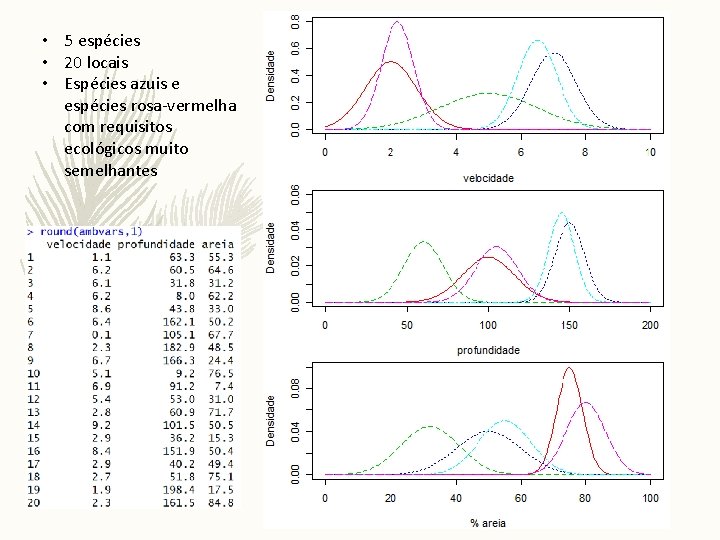
• 5 espécies • 20 locais • Espécies azuis e espécies rosa-vermelha com requisitos ecológicos muito semelhantes

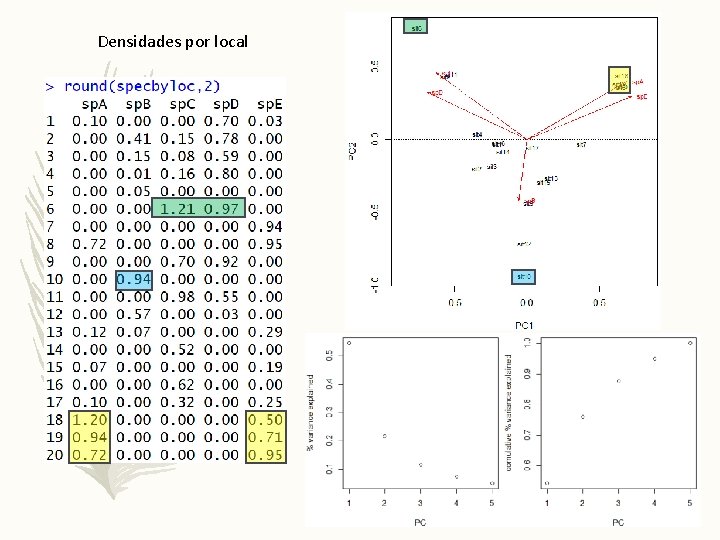
• • 3 variáveis ambientais (velocidade, profundidade, % areia) 5 espécies 20 locais Espécies azuis e espécies rosa-vermelha com requisitos ecológicos muito semelhantes • Cada local encontra-se num espaço multivariado (de dimensão 3) no que diz respeito às variáveis ambientais que o caracterizam • Cada local encontra-se num espaço multivariado (de dimensão 5 em relação às espécies que lá estão presentes) • O objectivo de uma PCA é representar os locais (e as variáveis) num espaço dimensional reduzido (tipicamente, 2 dimensões, ou seja, um biplot) Matrizes: 1. taxa por locais e 2. variáveis ambientais por locais

Densidades por local

• Relações de proximidade: 1. Entre locais, em função das espécies neles presentes 2. Entre locais, em função das variáveis ambientais que apresentam 3. Entre espécies, em função das variáveis ambientais que preferem 4. Entre espécies, em função dos locais em que se encontram simultaneamente presentes ou ausentes Objectivos: 1. compreender e descrever as relações de proximidade entre os locais para explicar quais os fatores determinantes na organização (na ordenação) das comunidades 2. perceber como é que as variáveis ambientais contribuem para estruturas os gradientes observados nas comunidades animais

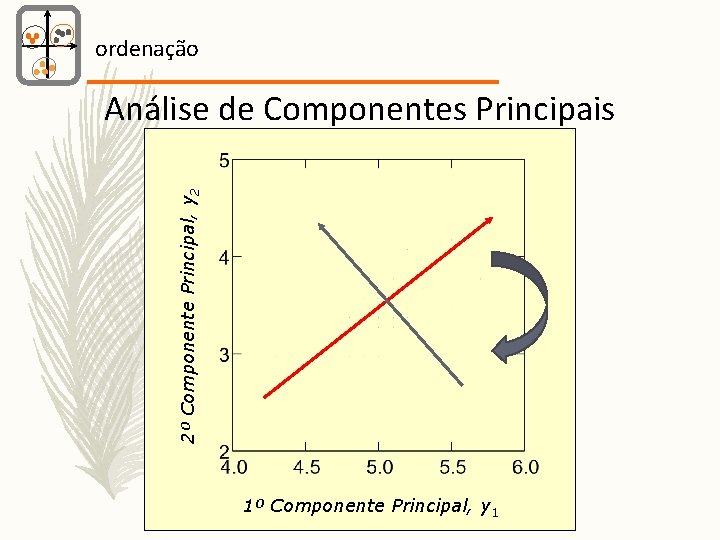
ordenação Análise de Componentes Principais Método A partir de k variáveis originais : x 1, x 2, . . . , xk: Produzem-se k novas variáveis: y 1, y 2, . . . , yk: y 1 = a 11 x 1 + a 12 x 2 +. . . + a 1 kxk y 2 = a 21 x 1 + a 22 x 2 +. . . + a 2 kxk. . . yk = ak 1 x 1 + ak 2 x 2 +. . . + akkxk Os y’s são combinações lineares dos x’s (i. e. das abundancies das espécies)

ordenação Análise de Componentes Principais Método Tal que: yk's são ortogonais, i. e. não correlacionados y 1 explica a maior quantidade possível da variância y 2 explica a maior quantidade possível da variância remanescente … etc.

ordenação 2º Componente Principal, y 2 Análise de Componentes Principais 1º Componente Principal, y 1

ordenação Análise de Componentes Principais Método A partir de k variáveis originais : x 1, x 2, . . . , xk: Produzem-se k novas variáveis: y 1, y 2, . . . , yk: y 1 = a 11 x 1 + a 12 x 2 +. . . + a 1 kxk y 2 = a 21 x 1 + a 22 x 2 +. . . + a 2 kxk. . . yk = ak 1 x 1 + ak 2 x 2 +. . . + akkxk Componentes Principais

ordenação Análise de Componentes Principais A extração dos CP’s pode ser baseada na: – Matriz de variância-covariância (argumento de princomp cor=FALSE, quando as variáveis são medidas nas mesmas unidades), ou – Matriz de correlação (argumento de princomp cor=TRUE, ou apenas scale(dados), mais adequada por exemplo se queremos usar sobre abundancias de espécies para descriminar locais)

ordenação Análise de Componentes Principais Então, sendo y 1 = a 11 x 1 + a 12 x 2 +. . . + a 1 kxk y 2 = a 21 x 1 + a 22 x 2 +. . . + a 2 kxk. . . yk = ak 1 x 1 + ak 2 x 2 +. . . + akkxk xj’s são padronizados (redução e centragem) se for utilizada a matriz de correlação (no R, scale(dados))

ordenação Análise de Componentes Principais {a 11, a 12, . . . , a 1 k} é o 1º vector próprio (eigenvector) da matriz de correlação/covariância, o qual inclui os coeficientes da 1ª componente principal; {a 21, a 22, . . . , a 2 k} é o 2º vector próprio (eigenvector) da matriz de correlação/covariância, o qual inclui os coeficientes da 2ª componente principal; … {ak 1, ak 2, . . . , akk} é o k-ésimo vector próprio (eigenvector) da matriz de correlação/covariância, o qual inclui os coeficientes da k-ésima componente principal.

ordenação Análise de Componentes Principais O score (coordenada) da i-ésima observação no jésimo componente principal é yi, j = aj 1 xi 1 + aj 2 xi 2 +. . . + ajkxik

ordenação Análise de Componentes Principais 2º Componente Principal, y 2 Scores (coordenadas) xi 2 yi, 1 yi, 2 xi 1 1º Componente Principal, y 1

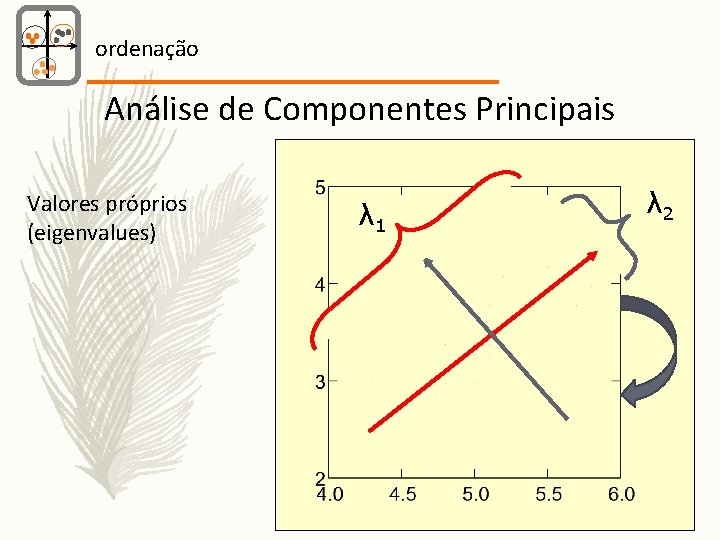
ordenação Análise de Componentes Principais A quantidade de variância explicada pelo: – 1º componente principal, λ 1, é o 1º valor próprio (eigenvalue) – 2º componente principal, λ 2, é o 2º valor próprio (eigenvalue) –. . . λ 1 > λ 2 > λ 3 > λ 4 >. . .

ordenação Análise de Componentes Principais Valores próprios (eigenvalues) λ 1 λ 2

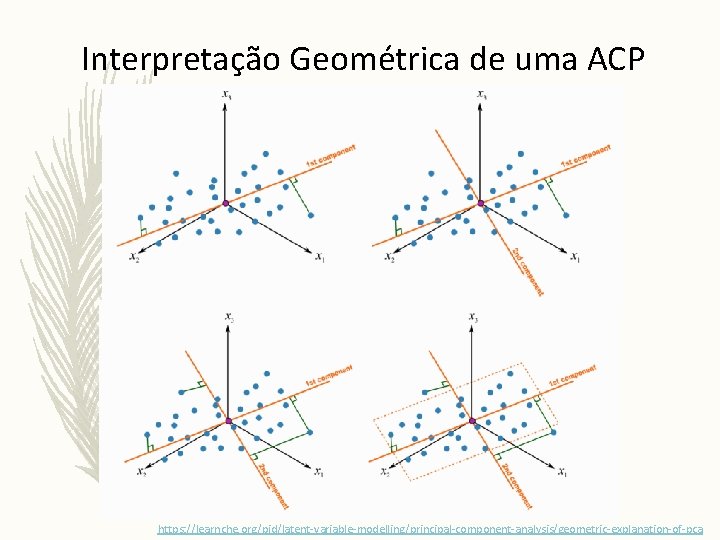
Interpretação Geométrica de uma ACP https: //learnche. org/pid/latent-variable-modelling/principal-component-analysis/geometric-explanation-of-pca

Interpretação Geométrica de uma ACP https: //learnche. org/pid/latent-variable-modelling/principal-component-analysis/geometric-explanation-of-pca

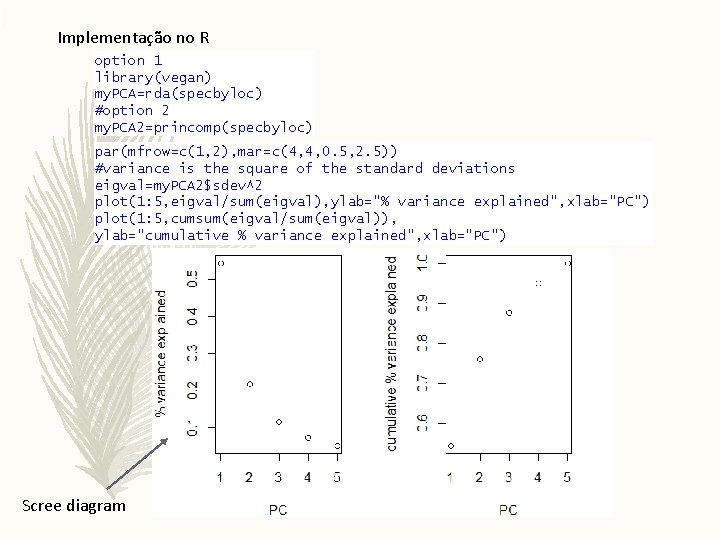
Implementação no R option 1 library(vegan) my. PCA=rda(specbyloc) #option 2 my. PCA 2=princomp(specbyloc) par(mfrow=c(1, 2), mar=c(4, 4, 0. 5, 2. 5)) #variance is the square of the standard deviations eigval=my. PCA 2$sdev^2 plot(1: 5, eigval/sum(eigval), ylab="% variance explained", xlab="PC") plot(1: 5, cumsum(eigval/sum(eigval)), ylab="cumulative % variance explained", xlab="PC") Scree diagram

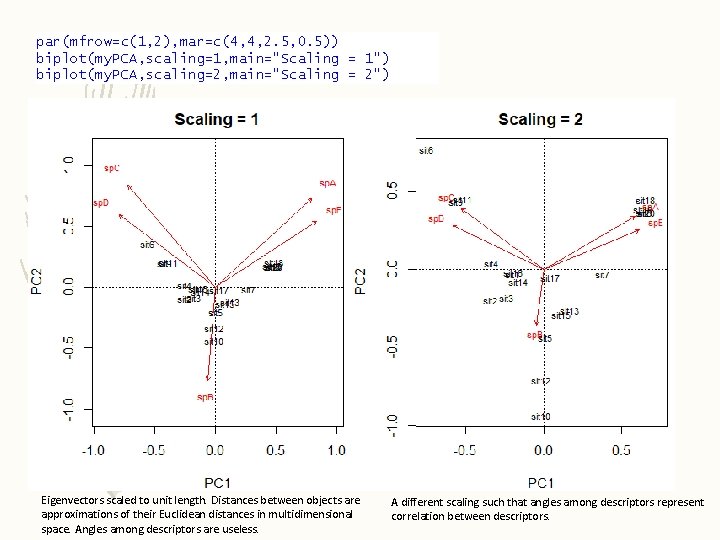
par(mfrow=c(1, 2), mar=c(4, 4, 2. 5, 0. 5)) biplot(my. PCA, scaling=1, main="Scaling = 1") biplot(my. PCA, scaling=2, main="Scaling = 2") Eigenvectors scaled to unit length. Distances between objects are approximations of their Euclidean distances in multidimensional space. Angles among descriptors are useless. A different scaling such that angles among descriptors represent correlation between descriptors.

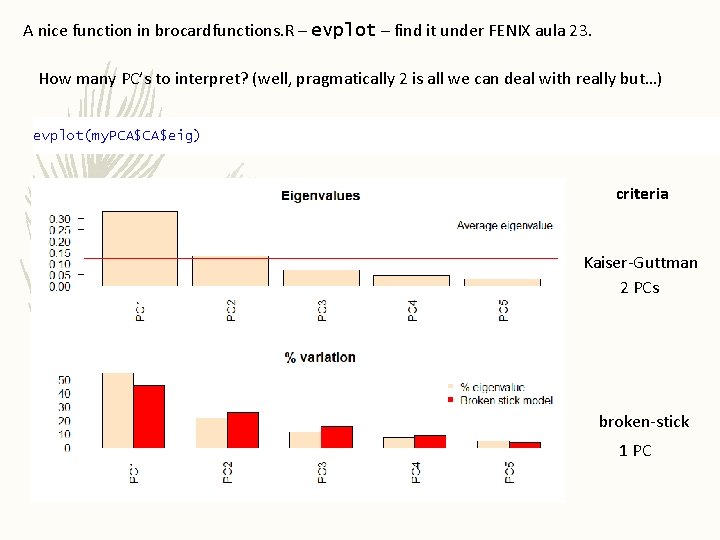
A nice function in brocardfunctions. R – evplot – find it under FENIX aula 23. How many PC’s to interpret? (well, pragmatically 2 is all we can deal with really but…) evplot(my. PCA$CA$eig) criteria Kaiser-Guttman 2 PCs broken-stick 1 PC
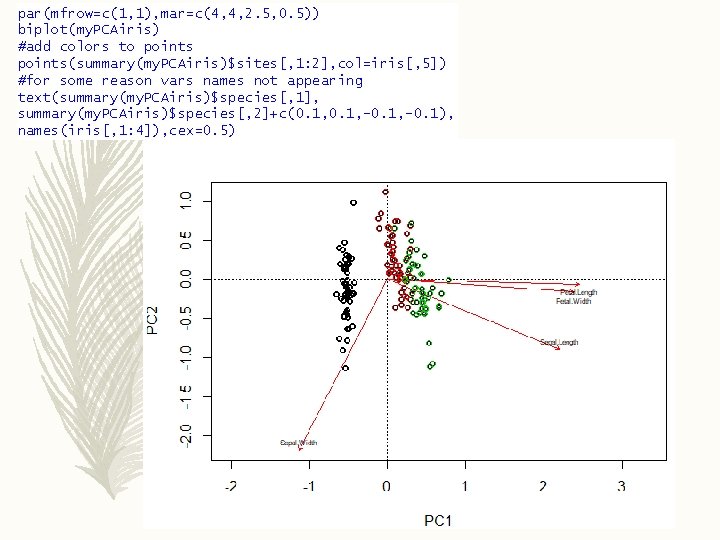
![another example now with data iris datairis my PCAirisrdascaleiris 1 4 parmfrowc1 2 marc4 #another example, now with data iris data(iris) my. PCAiris=rda(scale(iris[, 1: 4])) par(mfrow=c(1, 2), mar=c(4,](https://slidetodoc.com/presentation_image_h2/f0ac69c75d66a96fba59aa96b1f38a1a/image-26.jpg)
#another example, now with data iris data(iris) my. PCAiris=rda(scale(iris[, 1: 4])) par(mfrow=c(1, 2), mar=c(4, 4, 0. 5, 2. 5)) eigvali=my. PCAiris$CA$eig plot(1: 4, eigvali/sum(eigvali), ylab="% variance explained", xlab="PC") plot(1: 4, cumsum(eigvali/sum(eigvali)), ylab="cumulative % variance explained", xlab="PC")

par(mfrow=c(1, 1), mar=c(4, 4, 2. 5, 0. 5)) biplot(my. PCAiris) #add colors to points(summary(my. PCAiris)$sites[, 1: 2], col=iris[, 5]) #for some reason vars names not appearing text(summary(my. PCAiris)$species[, 1], summary(my. PCAiris)$species[, 2]+c(0. 1, -0. 1), names(iris[, 1: 4]), cex=0. 5)

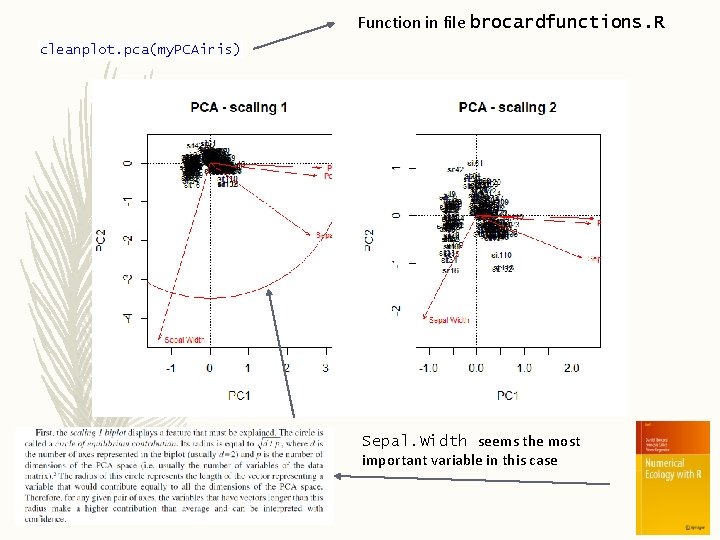
Function in file brocardfunctions. R cleanplot. pca(my. PCAiris) Sepal. Width seems the most important variable in this case
 Carnivore
Carnivore Is a horse a producer consumer or decomposer
Is a horse a producer consumer or decomposer G h patel college of engineering and technology
G h patel college of engineering and technology Goodies frozen egg product expiration date
Goodies frozen egg product expiration date Python goodies
Python goodies Goodies superman
Goodies superman 2 types of fitness
2 types of fitness Health-related skill
Health-related skill Https://a-z-animals.com/animals/
Https://a-z-animals.com/animals/ Venn diagram of plant and animal cells
Venn diagram of plant and animal cells Piney woods ecoregion of texas
Piney woods ecoregion of texas Grade 5 natural science term 1
Grade 5 natural science term 1 Site of meiosis in plants and animals
Site of meiosis in plants and animals Science class five
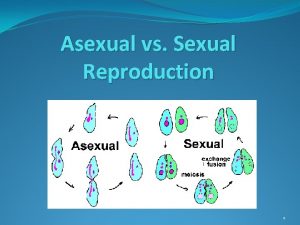
Science class five Plants and animals reproduction venn diagram
Plants and animals reproduction venn diagram Differences between mitosis in plants and animals
Differences between mitosis in plants and animals Central plains industry
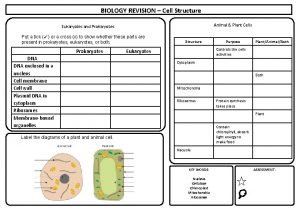
Central plains industry Structure of cell
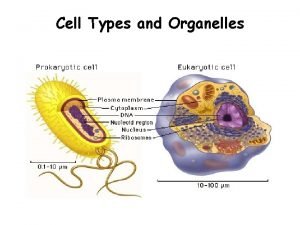
Structure of cell When does respiration take place in animals and plants?
When does respiration take place in animals and plants? Every living plants and animals must have
Every living plants and animals must have Animals in temperate forest biome
Animals in temperate forest biome Number of chromosomes in animals and plants
Number of chromosomes in animals and plants Grassland biome animals and plants
Grassland biome animals and plants Plant keystone species
Plant keystone species Chromosome number
Chromosome number Nitrogen important
Nitrogen important Gas exchange in plants and animals venn diagram
Gas exchange in plants and animals venn diagram North central plains plants and animals
North central plains plants and animals C3 plants vs c4 plants
C3 plants vs c4 plants