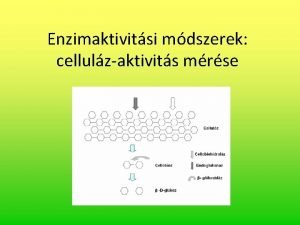
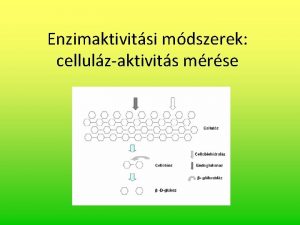
A NUKLEINSAVAK MANIPULCIJA SORN HASZNLATOS ENZIMEK RESTRIKCIS ENDONUKLEZOK





























- Slides: 72

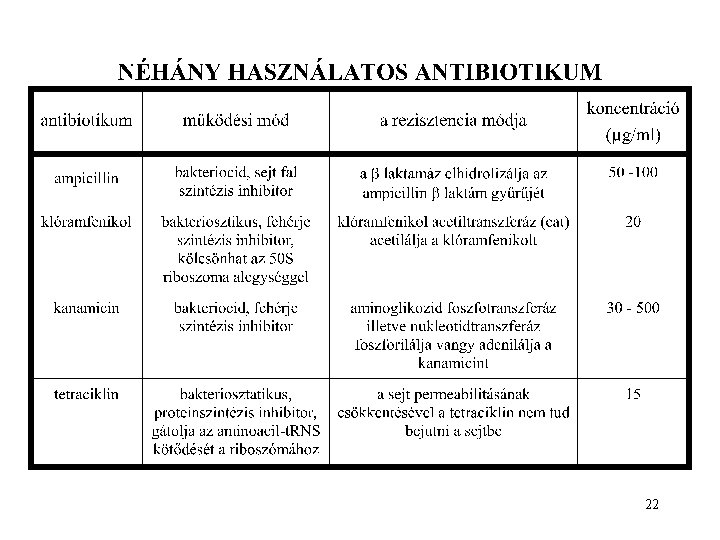
A NUKLEINSAVAK MANIPULÁCIÓJA SORÁN HASZNÁLATOS ENZIMEK RESTRIKCIÓS ENDONUKLEÁZOK 1970 – A molekuláris biológia forradalmának kezdete A Haemophilus influenzae restrikciós endunuklázának felfedezése Három fő csoport I típus Specifikus szekvenicákat ismer fel, de elvándorol a DNS-en (~1000 -5000 bázis) mielőtt az egyik szálat elhasítja, nukleotideokat szabadít fel (~75) a hasítás helyén. Egy másik endonuckleáz kell a második szál hasításához Pl. Eco. K. Mg 2+, ATP és SAM (S-adenosyl methionine) SAM kell hozzá 1

A NUKLEINSAVAK MANIPULÁCIÓJA SORÁN HASZNÁLATOS ENZIMEK II típus RESTRIKCIÓS ENDONUKLEÁZOK Speciális célszekvenciát ismer fel és hasítja a DNS-t mindkét szálon a felismerési szekvenciában vagy annak környezetében pl. Eco. RI Mg 2+ kell Ez a típus az elterjedt III. típus I és II. Kombinációja. Mindkét DNS szálat hasítja egy meghatározott távolságra a felismerési szekvenciától pl. Hga. I Mg 2+-t és. ATP-t igényel 2

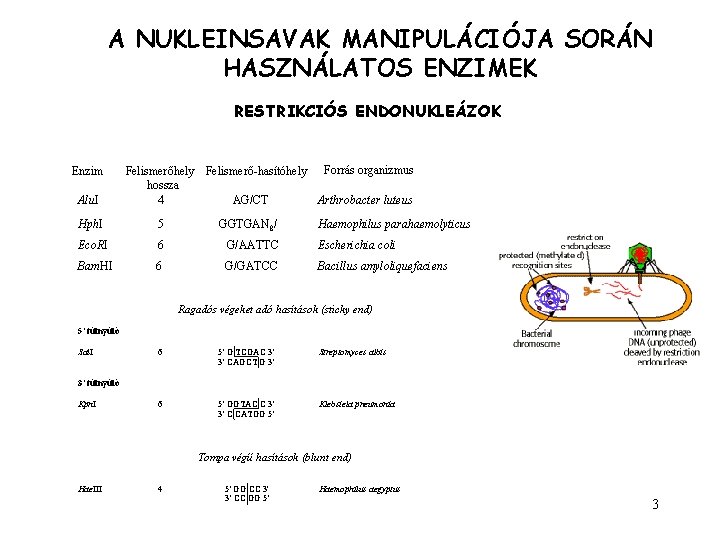
A NUKLEINSAVAK MANIPULÁCIÓJA SORÁN HASZNÁLATOS ENZIMEK RESTRIKCIÓS ENDONUKLEÁZOK Enzim Alu. I Felismerőhely Felismerő-hasítóhely Forrás organizmus hossza 4 AG/CT Arthrobacter luteus Hph. I 5 GGTGAN 8/ Haemophilus parahaemolyticus Eco. RI 6 G/AATTC Escherichia coli Bam. HI 6 G/GATCC Bacillus amyloliquefaciens Ragadós végeket adó hasítások (sticky end) 5' túlnyúló Sal. I 6 5' G TCGAC 3' 3' CAGCT G 3' Streptomyces albis 6 5' GGTAC C 3' 3' C CATGG 5' Klebsiela pneumonia 3' túlnyúló Kpn. I Tompa végű hasítások (blunt end) Hae. III 4 5' GG CC 3' 3' CC GG 5' Haemophilus aegyptus 3

Palindrom példák KIS EREK MENTÉN LÁP SÍK ÖLÉN ODA VAN A BÁNYA RABJA JAJ BARANYÁBAN A VADON ÉLŐ KIS PÁLNÉT NEM KERESIK 4

Isoschisomers Atc. I Kpn. I Xma. I Sma. I 6 5' GGTAC/C 3' 6 5' C/CCGGG 3' 5' CCC/GGG 3' Kompatibilis véget adó enzimek Sal. I Xho. I 6 5' G/TCGAC 3' 5' C/TCGAG 3' FEHÉRJE TÚLTERMELTETÉS SZEMPONTJÁBÓL KITÜNTETT ENZIMEK Afl. III Bsp. HI Nco. I Nde. I 6 5' A/CPu. Py. GT 3' 6 5' T/CATGA 3' 6 5' C/CATGG 3' 6 5' CA/TATG 3' 5

DNS METILÁZOK - dam metiláz (dezoxiadenin metiláz) 5’ GATC 3’ felismerő hely N 6 pozícióban metilez adenin sok dam metiláz érzékeny enzim van pl. Mbo. I, Xho. I, egyenként ellenőrizni kell, érzékenység esetén - dam- törzs használata - nem érzékeny izoskizomer használata (ha van) (Mbo. I - Sau 3 AI) 6

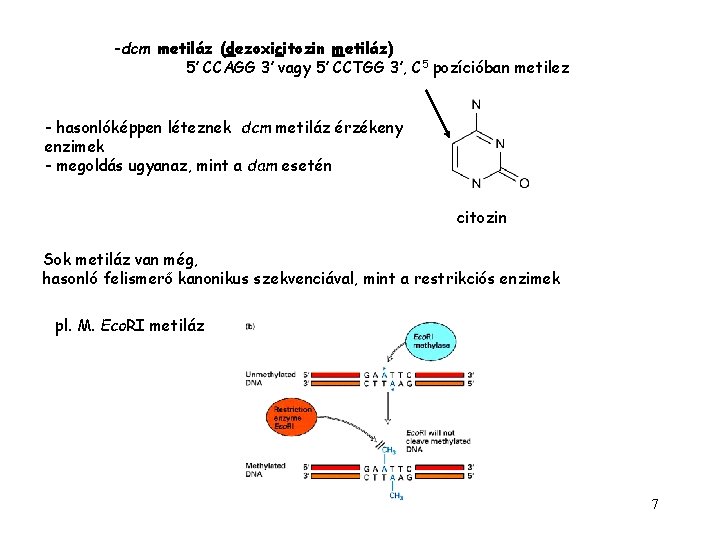
-dcm metiláz (dezoxicitozin metiláz) 5’ CCAGG 3’ vagy 5’ CCTGG 3’, C 5 pozícióban metilez - hasonlóképpen léteznek dcm metiláz érzékeny enzimek - megoldás ugyanaz, mint a dam esetén citozin Sok metiláz van még, hasonló felismerő kanonikus szekvenciával, mint a restrikciós enzimek pl. M. Eco. RI metiláz 7

Polimerázok - DNS függő DNS polimerázok - RNS függő DNS polimerázok - Templátfüggetlen DNS polimeráz - DNS függő RNS polimeráz 5’ 3’ polimeráz aktivitás 8

DNS függő DNS polimerázok 9

10

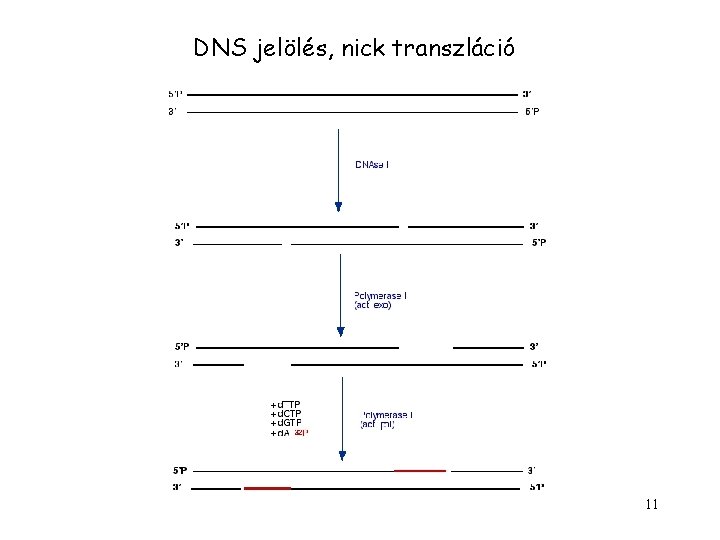
DNS jelölés, nick transzláció 11

12

Templát független DNS polimeráz 13

RNS függő DNS polimeráz 14

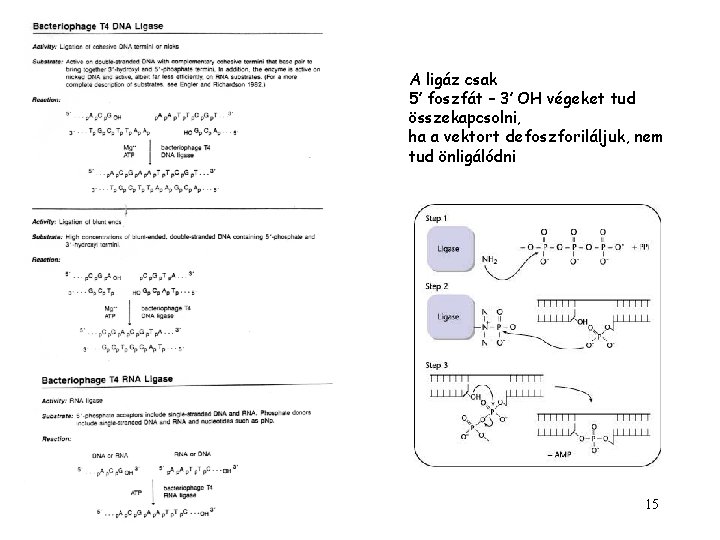
A ligáz csak 5’ foszfát – 3’ OH végeket tud összekapcsolni, ha a vektort defoszforiláljuk, nem tud önligálódni 15

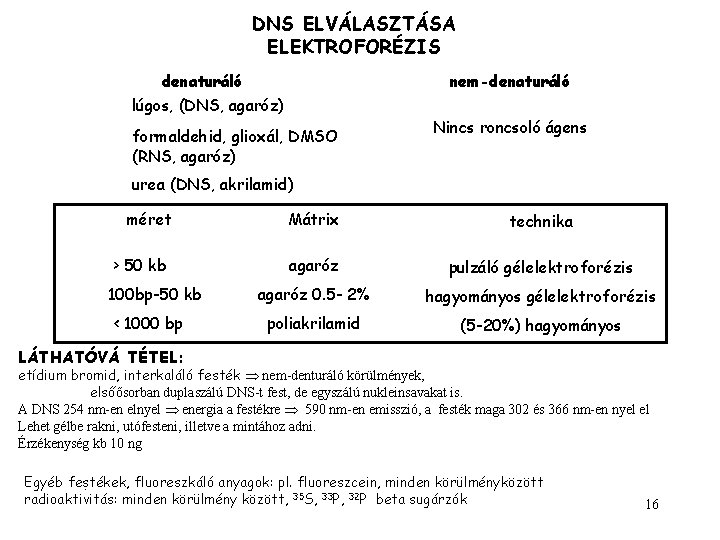
DNS ELVÁLASZTÁSA ELEKTROFORÉZIS denaturáló lúgos, (DNS, agaróz) nem-denaturáló formaldehid, glioxál, DMSO (RNS, agaróz) Nincs roncsoló ágens urea (DNS, akrilamid) méret > 50 kb 100 bp-50 kb < 1000 bp Mátrix technika agaróz pulzáló gélelektroforézis agaróz 0. 5 - 2% hagyományos gélelektroforézis poliakrilamid (5 -20%) hagyományos LÁTHATÓVÁ TÉTEL: etídium bromid, interkaláló festék nem-denturáló körülmények, elsőősorban duplaszálú DNS-t fest, de egyszálú nukleinsavakat is. A DNS 254 nm-en elnyel energia a festékre 590 nm-en emisszió, a festék maga 302 és 366 nm-en nyel el Lehet gélbe rakni, utófesteni, illetve a mintához adni. Érzékenység kb 10 ng Egyéb festékek, fluoreszkáló anyagok: pl. fluoreszcein, minden körülményközött. radioaktivitás: minden körülmény között, 35 S, 33 P, 32 P beta sugárzók 16

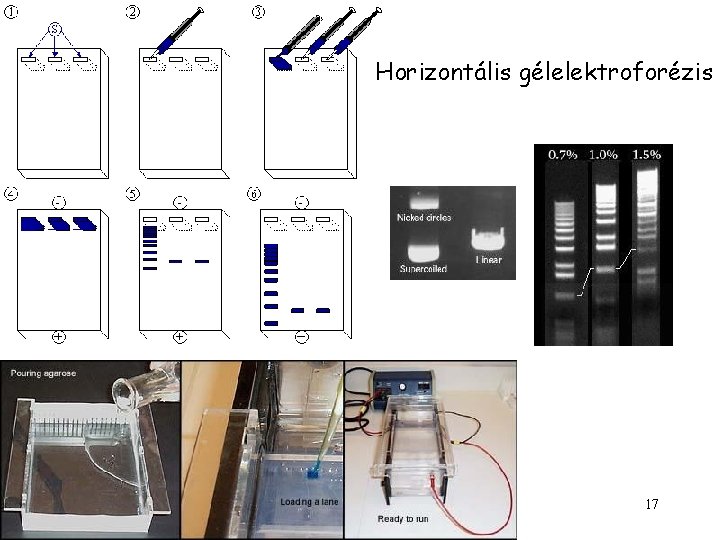
Horizontális gélelektroforézis 17

DNS IZOLÁLÁSA GÉLBŐL - közös pont: először elektroforetikus úton elválasztjuk a DNS-t - a megfelelő sávot kivágjuk steril pengével AGARÓZ - dialízis, dializáló hártyában, elektrodialízis a kapott DNS további tisztítása fenolos extrakcióval, alkoholos kicsapással, univerzális, széles mérettartomány - fagyasztásos módszer : a kivágott – DNS-t tartalmazó - agarózdarabot – 80 o. C-on megfagyasztjuk az agarós szerkezete roncsolódik, ebből a DNS egy szűrőn keresztül centrifugálással kinyerhető további tisztítás szükséges, univerzális - kromatográfiás módszerek 6 M Na. I mellett a DNS 55 o. C-on (az agaróz megolvad) szilikagél felületére kötődik, a mátrix mosása után innen o 55 C-on alacsony só(víz, TE) eluálható, majdnem univerzális, elég széles mérettartomány DEAE membránba futtatjuk a DNS-t, innen magassókoncentrációval (1. 5 M) magas hőmérsékleten eluálható nagy tisztaság, szűk, alacsony mérettartomány < 1. 5 – 2 kb esetén jó a kitermelés POLIAKRILAMID (PAGE) a kivágott darabot passzív módon vagy elektromos térben eluáljuk majd töményítjük, ioncserésen tisztítjuk 18

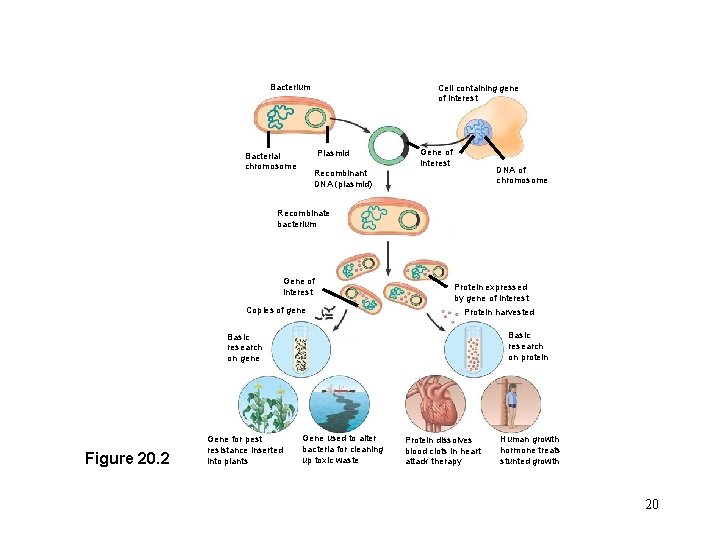
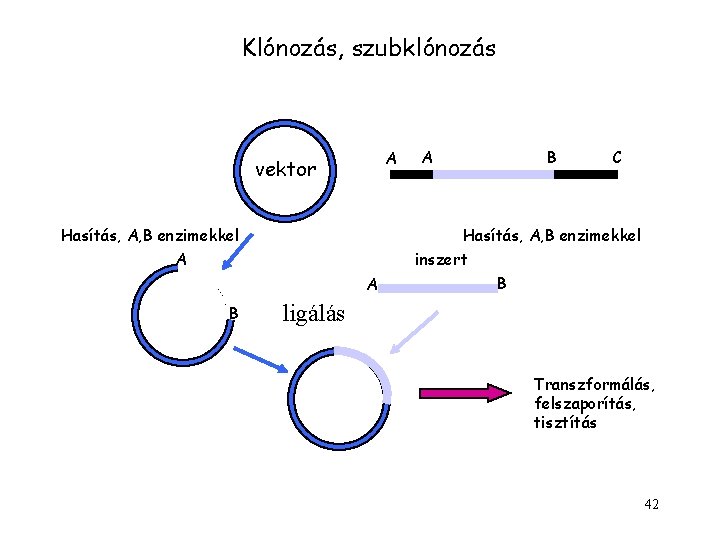
Klónozás fogalma Egy általunk kiválasztott DNS darabot vektor segítségével gazdasejtbe juttatunk és ott felszaporítunk Szubklónozás: további kisebb darabok hasonló felszaporítása vektor Hasítás, A, B enzimekkel A inszert A B B ligálás Transzformálás, felszaporítás, tisztítás Vektor: olyan nukleinsav hordozó, amellyel nukleinsavakat sejtbe lehet juttatni, 19 Felhasználás: klónozás, fehérje termeltetés, genetikai manipulációk stb.

Bacterium Cell containing gene of interest Plasmid Bacterial chromosome Gene of interest DNA of chromosome Recombinant DNA (plasmid) Recombinate bacterium Gene of interest Copies of gene 3 Protein expressed by gene of interest Protein harvested Basic research on protein Basic research on gene Figure 20. 2 Gene for pest resistance inserted into plants Gene used to alter bacteria for cleaning up toxic waste Protein dissolves blood clots in heart attack therapy Human growth hormone treats stunted growth 20

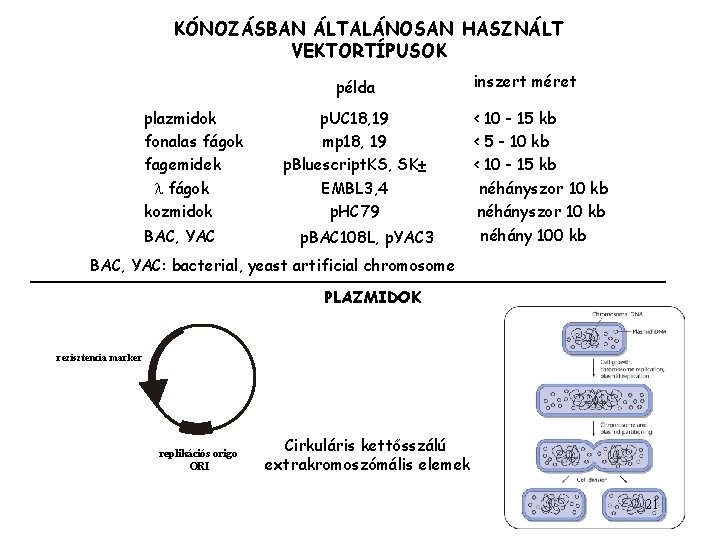
KÓNOZÁSBAN ÁLTALÁNOSAN HASZNÁLT VEKTORTÍPUSOK példa plazmidok fonalas fágok fagemidek l fágok kozmidok BAC, YAC p. UC 18, 19 mp 18, 19 p. Bluescript. KS, SK± EMBL 3, 4 p. HC 79 p. BAC 108 L, p. YAC 3 inszert méret < 10 - 15 kb < 5 - 10 kb < 10 - 15 kb néhányszor 10 kb néhány 100 kb BAC, YAC: bacterial, yeast artificial chromosome PLAZMIDOK rezisztencia marker replikációs origo ORI Cirkuláris kettősszálú extrakromoszómális elemek 21

22

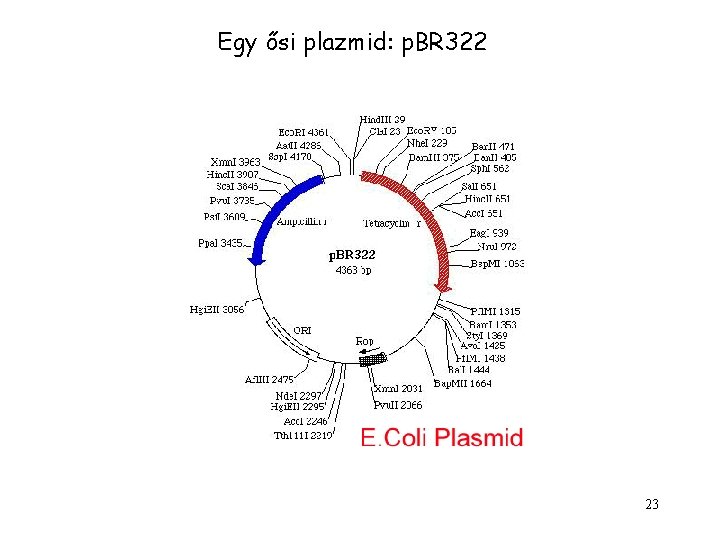
Egy ősi plazmid: p. BR 322 23

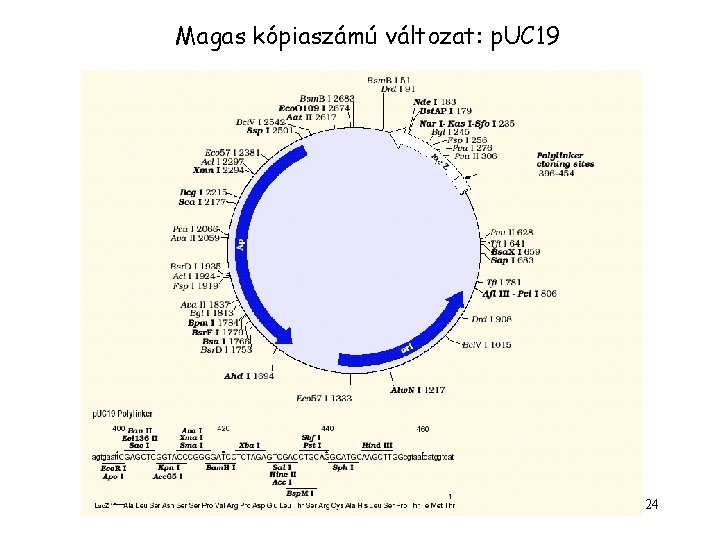
Magas kópiaszámú változat: p. UC 19 24

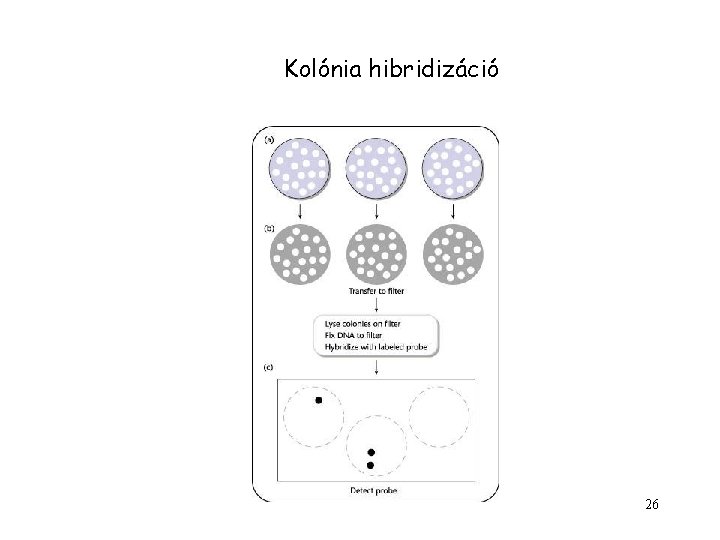
Inszertet tartalmazó klónok kiválasztása -antibiotikum rezisztencia, ld. p. BR 3222, két antibiotikum, az egyik elromlik, ha inszert épül be, fáradtságos szurkálások, két antibiotikum rezisztencia gén szükséges - kolónia hibridizáció, univerzális mindig használható - plazmid tisztítás, térképezés restrikciós emésztéssel hosszú fáradtságos - polimeráz láncreakció sejteken, kombinatorikus gyors ha nincs más szelekció - kék fehér színszelekció, - pozitív szelekciós vektorok, kondicionálisan letális gén a vektoron, az inszert beépül, elrontja a gént megszűnik a letalitás - auxotrofiát komplementáló génbe történő klónozás ugyanaz a probléma, mint az antibiotikumok esetén 25

Kolónia hibridizáció 26

Lac. Z a komplementáció F' plazmidon: defektív - galaktozidáz gén, hiányzik 11 - 41. aminosav közötti régió bevitt vektor: tartalmazza a lac. Z szabályozó régiót és az 1 -146 aminosavat a kettő együtt: aktív – galaktozidáz X-gal szubsztráttal kék telep a bevitt N-terminális fragmentben : polilinker régió (leolvasási keret marad) ebbe lehet klónozni fragmentumot, ha kis fragmentum és leolvasási keret nem romlik el X-gal szubsztráttal kék telep, ha elromlik vagy nagy fragment X-gal szubsztráttal fehér telep 27

PLAZMIDOK SEJTBE JUTTATÁSÁNAK MÓDJAI 1. Kémiai transzformálás Kompetens sejt: a DNS felvételére alkalmassá tett sejt A sejteket felnövesztés után centrifugáljuk speciális kétértékű kationokat (Ca 2+, Mn 2+) tartalmazó oldattal kezeljük, sejtfal permeabilitást növelő ágenst (DMSO) adunk hozzá : transzformációs hatékonyság: transzformáns /µg DNS elvi szám a transzformációt 1 ng mennyiségű DNS-sel hajtjuk végre normál érték: 106 – 108 nagyon jó: 109 a transzformáció hatékonyságát meghatározó tényezők: -oldatok edények tisztasága, - sejtek növekedési sebessége, a növesztés fázisa, hőmérséklete - hősokk hőmérséklete hossza - permeabilizáló faktor a lineáris DNS transzformációs gyakorisága kb 2 nagyságrenddel alacsonyabb, mint a cirkulárisé egyéb fogások: -spheroplast készítés ozmotikum jelenlétében és ezt transzformáljuk - a DNS-t liposzómába csomagoljuk transzfomálás előtt 28

Elektroporáció A sejteket felnövesztés után kis vezetőképességű, glicerines (nagy ellenállású 600 ) pufferben szuszpendáljuk nagy feszültségű impulzust adunk rá kb 5 ms-ig transzformációs hatékonység 20 - 50 x jobb (1010/µg DNS) sejttípusonként optimalizálni kell maghatározó faktorok: - az oldat ellenállása - az impulzus nagysága, hossza - permeabilizáló, redox potenciált befolyásoló faktorok adagolása 29

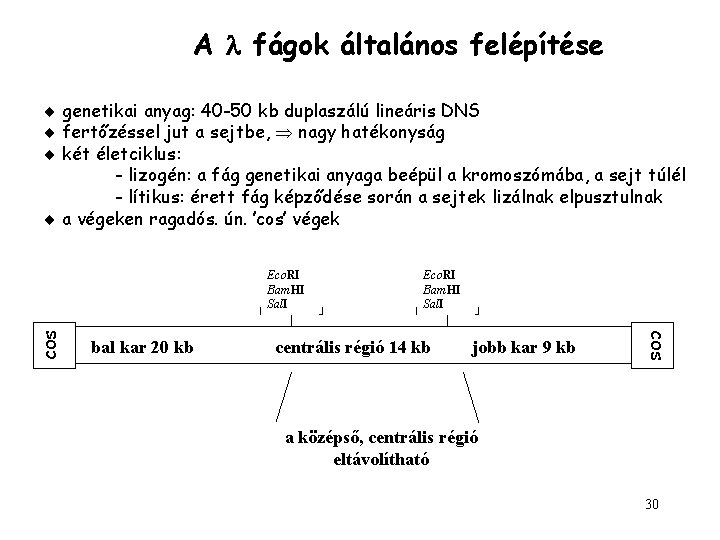
A fágok általános felépítése genetikai anyag: 40 -50 kb duplaszálú lineáris DNS fertőzéssel jut a sejtbe, nagy hatékonyság két életciklus: - lizogén: a fág genetikai anyaga beépül a kromoszómába, a sejt túlél - lítikus: érett fág képződése során a sejtek lizálnak elpusztulnak a végeken ragadós. ún. ’cos’ végek bal kar 20 kb Eco. RI Bam. HI Sal. I centrális régió 14 kb jobb kar 9 kb cos Eco. RI Bam. HI Sal. I a középső, centrális régió eltávolítható 30

31

Speciális λ fág fajták 32

Mesterséges kromoszómák: BAC (bacterial artificial chromosome) vektorok 33

Mesterséges kromoszómák: YAC (yeast artificial chromosome) vektorok 34

KÖNYVTÁRAK TÍPUSAI - genomiális a teljes genomból készül, elvileg minden információt tartalmaz (pl. nem transzlálódó régió, szabályozó régiók, intronok) mind prokariótákból, mind eukariótákból - c. DNS az aktívan termelődő m. RNS-ekből készül csak az érett m. RNS szekvenciáját tartalmazza (nincs intron szabályozó régió stb) csak eukariótákból expressziós a c. DNS-eket ún. expressziós kazettába klónozzuk, ezáltal a kódolt fehérje (ha a m. RNS kódol ilyet) aktívan termelődhet Követelmények a könyvtárakkal szemben - fedje le a teljes genomot, illetve m. RNS populációt 35 - ne legyen redundáns

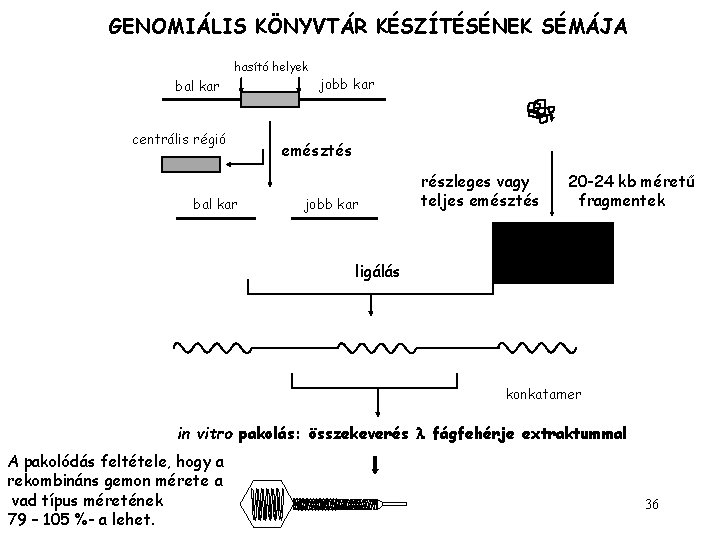
GENOMIÁLIS KÖNYVTÁR KÉSZÍTÉSÉNEK SÉMÁJA hasító helyek bal kar centrális régió bal kar jobb kar emésztés jobb kar részleges vagy teljes emésztés 20 -24 kb méretű fragmentek ligálás konkatamer in vitro pakolás: összekeverés fágfehérje extraktummal A pakolódás feltétele, hogy a rekombináns gemon mérete a vad típus méretének 79 – 105 %- a lehet. 36

c. DNS könyvtár A m. RNS-ekkel szemben támasztott követelmények Integritás A m. RNS mérete, degradált-e Megoldás: denaturáló gélelektroforézis (várt méret 0. 5 - 8 kb, a többség 1. 5 -2 kb) Lehet-e teljes hosszúságú c. DNS-t szintetizáltatni Megoldás: elõzetes reverz transzkripció jelölt nukloetidokkal elektroforézis Lehet-e nagy molekulasúlyú fehérjéket in vitro szintetizálni Megoldás: in vitro transzláció jelölt aminosavakkal A kérdéseses fehérjét tudjuk-e szintetizáltatni Megoldás: in vitro transzláció + immunoprecipitáció 37

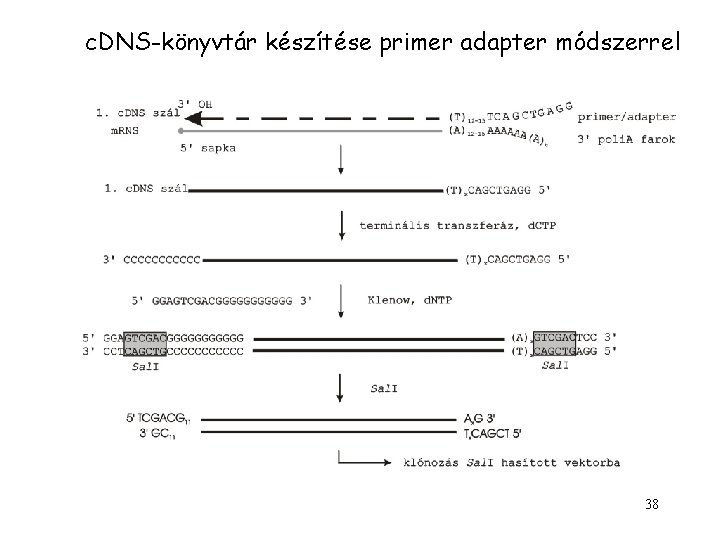
c. DNS-könyvtár készítése primer adapter módszerrel 38

RENDELKEZÉSRE ÁLLÓ VAGY SZÜKSÉGES INFORMÁCIÓK A KERESETT FEHÉRJÉRŐL, ILLETVE GÉNJÉRŐL - nem tudunk semmit - ismeretek a fehérje funkciójáról csak fenotípus alapján jellemezhető van tisztított fehérje mutagenezis (transzpozon) kromoszomális séta fehérje funkció tesztelhető expressziós könyvtárak már van ilyen fehérje illetve gén más típusú sejtekből fehérje szekvenálás heterológ próba használata könyvtárak átvizsgálásához ellenanyag termeltetés DNS próba tervezés expressziós könyvtárak EGYÉB pl. ha a fehérje szabályozása ismert szubsztraktív hibridizáció - számítógép, adatbankok genom szekvenálások 39

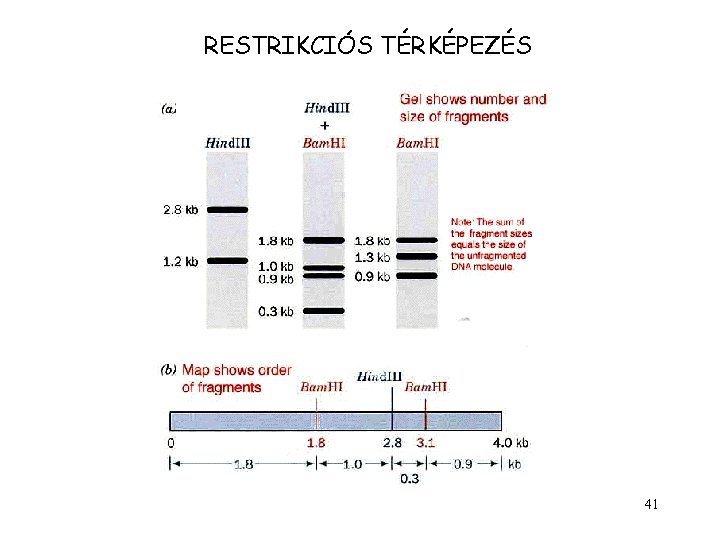
A POZITÍV KLÓNOK TOVÁBBI FELDOLGOZÁSA A könyvtárakból kapott klónok általában túl nagyok közvetlen felhasználáshoz, ezért további munkálatok szükségesek: 1. AZ INSZERT RESTRIKCIÓS TÉRKÉPEZÉSE 2. A TÉRKÉP ALAPJÁN AZ INSZERT KISEBB DARABOKBAN TÖRTÉNŐ SZUBKLÓNOZÁSA 3. AZ SZUBKLÓNOK SZEKVENCIÁJÁNAK MEGÁLLAPÍTÁSA 4. AZ INSZERT SZEKVENCIÁJÁNAK MEGÁLLAPÍTÁSA 5. SZÁMÍTÓGÉPES ADATFELDOLGOZÁS A SZEKVENCIÁN BELÜLI GÉNEK, ELEMEK AZONOSÍTÁSA 40

RESTRIKCIÓS TÉRKÉPEZÉS 41

Klónozás, szubklónozás A vektor Hasítás, A, B enzimekkel A A B C Hasítás, A, B enzimekkel inszert B ligálás Transzformálás, felszaporítás, tisztítás 42

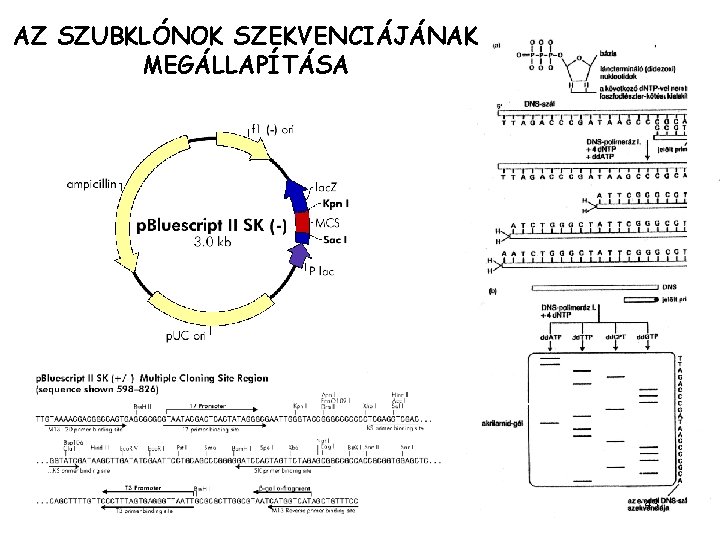
AZ SZUBKLÓNOK SZEKVENCIÁJÁNAK MEGÁLLAPÍTÁSA 43

FEHÉRJE TERMELTETŐ RENDSZEREK PROKARIÓTÁK E. coli ismert, Bármilyen fehérje, feltéve, ha - nem túl nagy - nem túl kicsi - nem túl hidrofób - nincs túl sok cisztein benne szekréciós rendszere minimális Bacillus subtilis jól ismert, ha nem is annyira, mint az E. coli van szekréciós rendszere EUKARIÓTÁK élesztő ügyorsan szaporodó eukarióta sejtvonal, üsok ismeretanyag üviszonylag könnyű kezelhetőség üposzttranszlációs modifikációk lehetősége emlős sejtvonalak v minden fajta fehérje termeltethető bennük v tranziens expressziós rendszerek viszonylag gyors, de korlátozott lehetôségek, v stabil expressziós rendszerek v hosszú, fáradságos optimalizálást 44 igényel

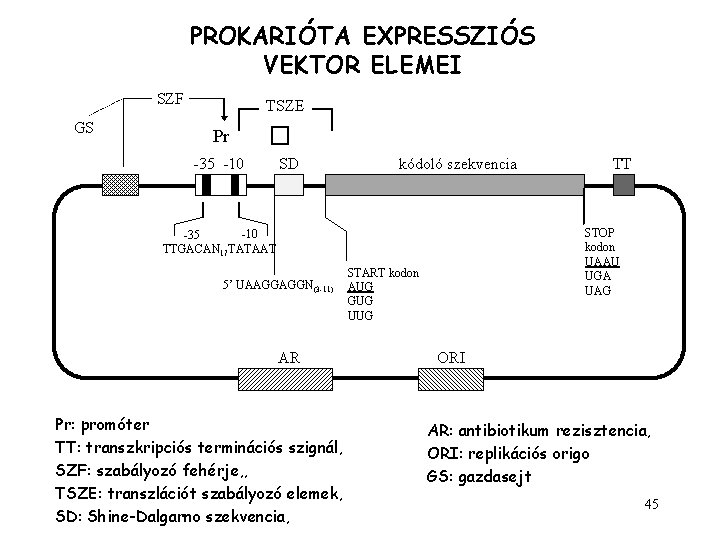
PROKARIÓTA EXPRESSZIÓS VEKTOR ELEMEI SZF GS TSZE Pr � -35 -10 SD kódoló szekvencia STOP kodon UAAU UGA UAG -10 -35 TTGACAN 17 TATAAT 5’ UAAGGAGGN(3 -11) AR Pr: promóter TT: transzkripciós terminációs szignál, SZF: szabályozó fehérje, , TSZE: transzlációt szabályozó elemek, SD: Shine-Dalgarno szekvencia, TT START kodon AUG GUG UUG ORI AR: antibiotikum rezisztencia, ORI: replikációs origo GS: gazdasejt 45

c. DNS-ből készült expressziós könyvtárból - Immunológiai detektálás poli- vagy monoklonális ellenanyaggal - Aktivitás mérés (csak bizonyos szerencsés esetekben) - Valamilyen módon jelölt liganddal (ha van a keresett fehérjének) 46

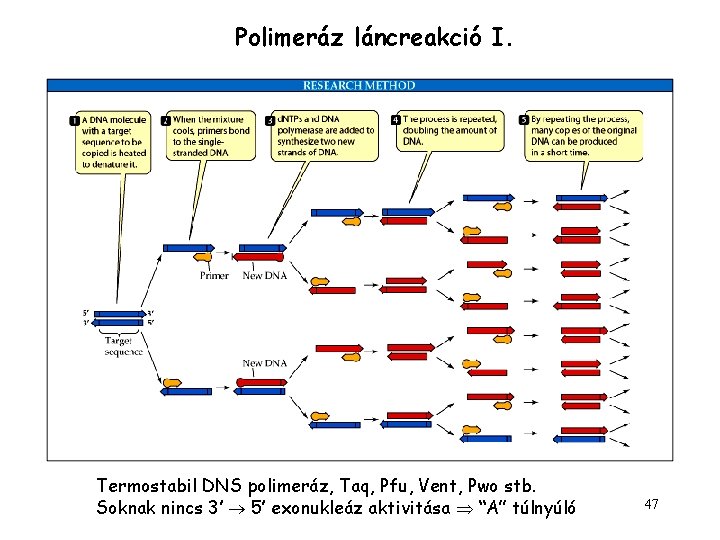
Polimeráz láncreakció I. Termostabil DNS polimeráz, Taq, Pfu, Vent, Pwo stb. Soknak nincs 3’ 5’ exonukleáz aktivitása “A” túlnyúló 47

Polimeráz láncreakció II. 48

A PCR DIAGNOSZTIKAI ALKALMAZÁSAI I. - FERTŐZÉSEK KIMUTATÁSA minden élőlény genetikai anyaga nukleinsav, ez tartalmaz specifikus szekvenciaelemeket speciálisan tervezett primerek segítségével PCR a termék megjelenése fertőzésre utal 49

Pontmutációk kimutatása RFLP-vel. Rerstrikciós fragment length polimorfizmus Normal -globin allele 201 bp 175 bp Dde. I Large fragment Dde. I Sickle-cell mutant -globin allele Large fragment 376 bp Dde. I (a) Dde. I restriction sites in normal and sickle-cell alleles of -globin gene. Normal allele Sickle-cell allele Large fragment 376 bp 201 bp 175 bp Figure 20. 9 a, b (b) Electrophoresis of restriction fragments from normal and sickle-cell alleles. 50

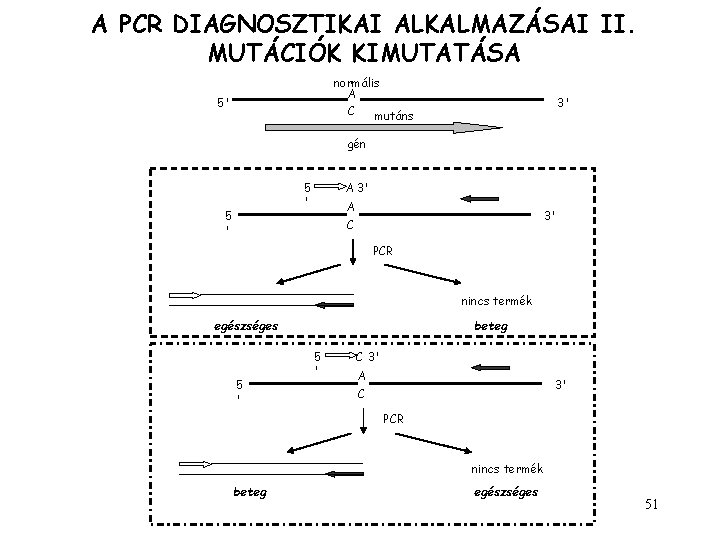
A PCR DIAGNOSZTIKAI ALKALMAZÁSAI II. MUTÁCIÓK KIMUTATÁSA normális A C mutáns 5' 3' gén 5 ' A 3' C PCR nincs termék egészséges 5 ' beteg 5 ' C 3' A 3' C PCR nincs termék beteg egészséges 51

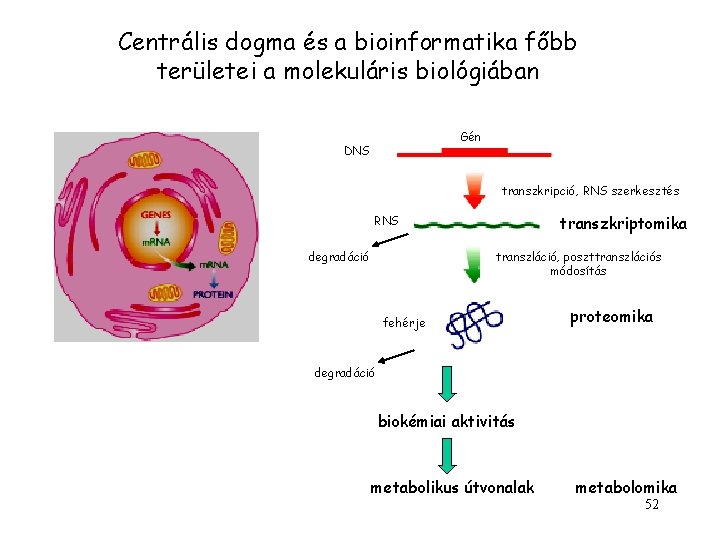
Centrális dogma és a bioinformatika főbb területei a molekuláris biológiában Gén DNS transzkripció, RNS szerkesztés transzkriptomika RNS degradáció transzláció, poszttranszlációs módosítás fehérje proteomika degradáció biokémiai aktivitás metabolikus útvonalak metabolomika 52

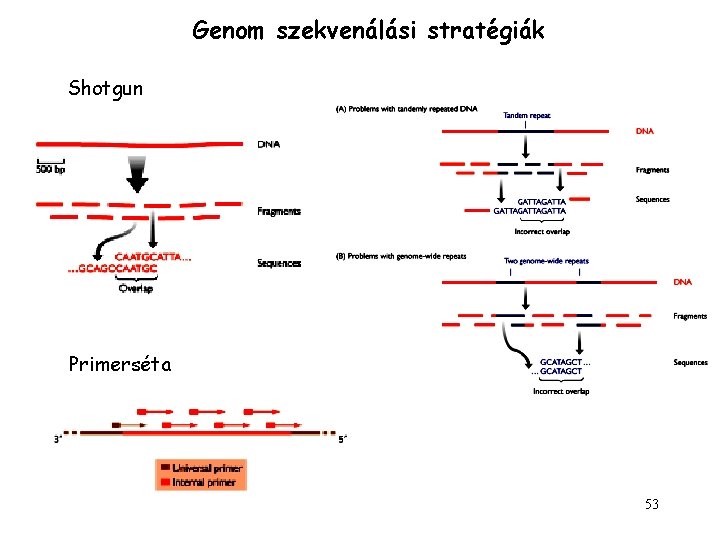
Genom szekvenálási stratégiák Shotgun Primerséta 53

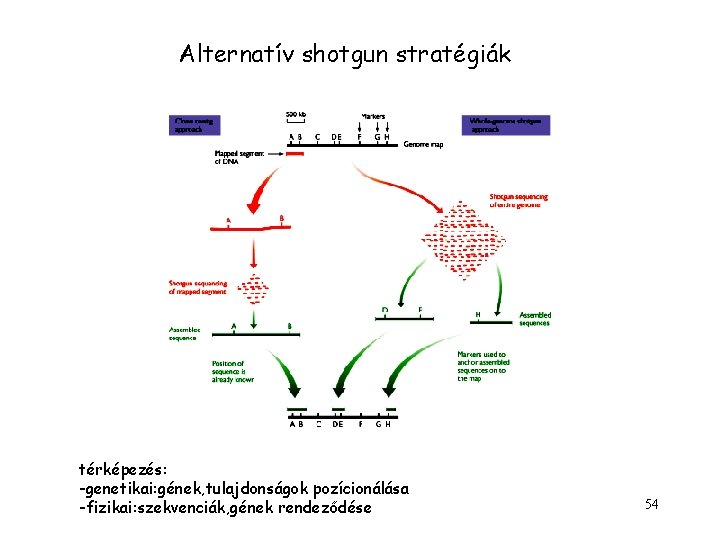
Alternatív shotgun stratégiák térképezés: -genetikai: gének, tulajdonságok pozícionálása -fizikai: szekvenciák, gének rendeződése 54

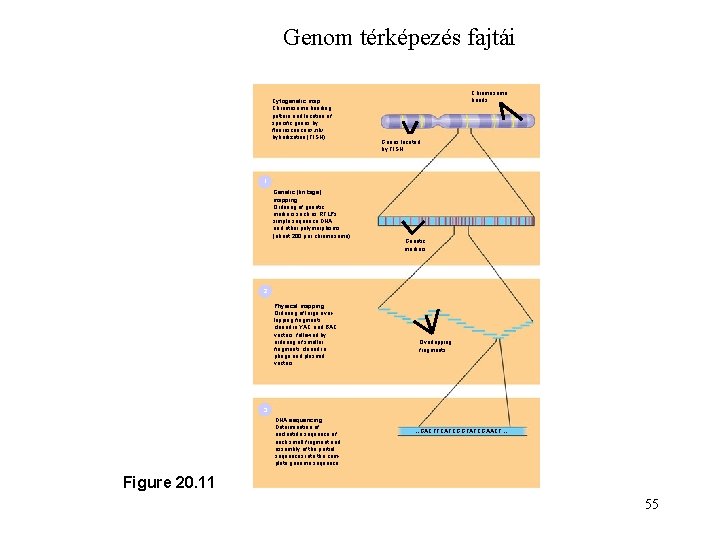
Genom térképezés fajtái Cytogenetic map Chromosome banding pattern and location of specific genes by fluorescence in situ hybridization (FISH) Chromosome bands Genes located by FISH 1 Genetic (linkage) mapping Ordering of genetic markers such as RFLPs, simple sequence DNA, and other polymorphisms (about 200 per chromosome) Genetic markers 2 Physical mapping Ordering of large overlapping fragments cloned in YAC and BAC vectors, followed by ordering of smaller fragments cloned in phage and plasmid vectors Overlapping fragments 3 DNA sequencing Determination of nucleotide sequence of each small fragment and assembly of the partial sequences into the complete genome sequence …GACTTCATCGGTATCGAACT… Figure 20. 11 55

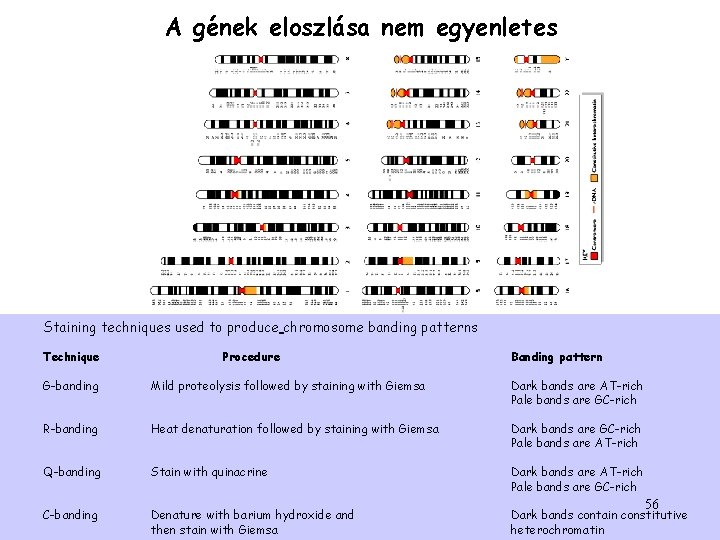
A gének eloszlása nem egyenletes Staining techniques used to produce chromosome banding patterns Technique G-banding Mild proteolysis followed by staining with Giemsa R-banding Dark bands are AT-rich Pale bands are GC-rich Heat denaturation followed by staining with Giemsa Q-banding Dark bands are GC-rich Pale bands are AT-rich Stain with quinacrine Dark bands are AT-rich Pale bands are GC-rich Denature with barium hydroxide and then stain with Giemsa Dark bands contain constitutive heterochromatin C-banding Procedure Banding pattern 56

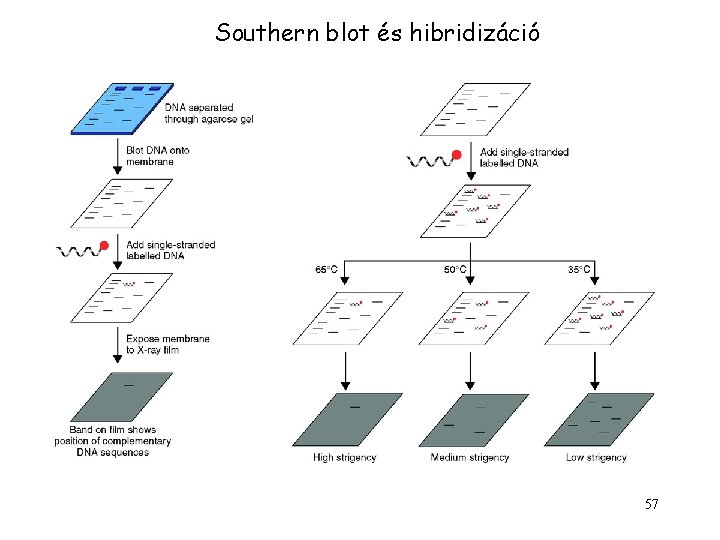
Southern blot és hibridizáció 57

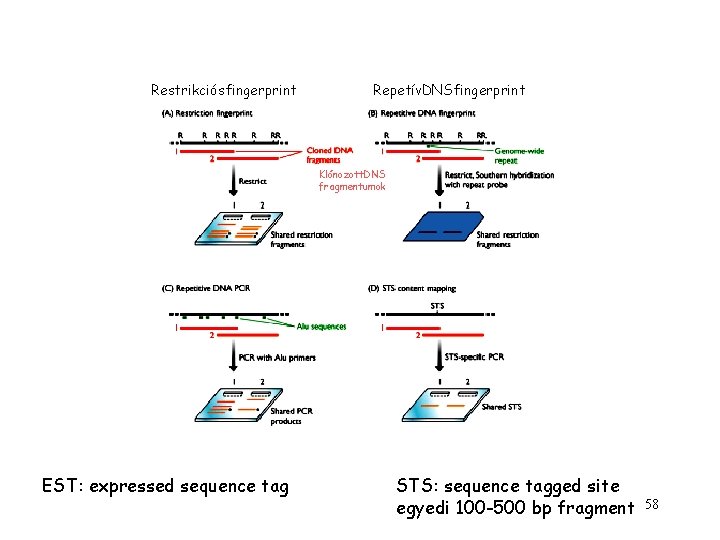
Restrikciósfingerprint Repetív. DNSfingerprint Klónozott. DNS fragmentumok EST: expressed sequence tag STS: sequence tagged site egyedi 100 -500 bp fragment 58

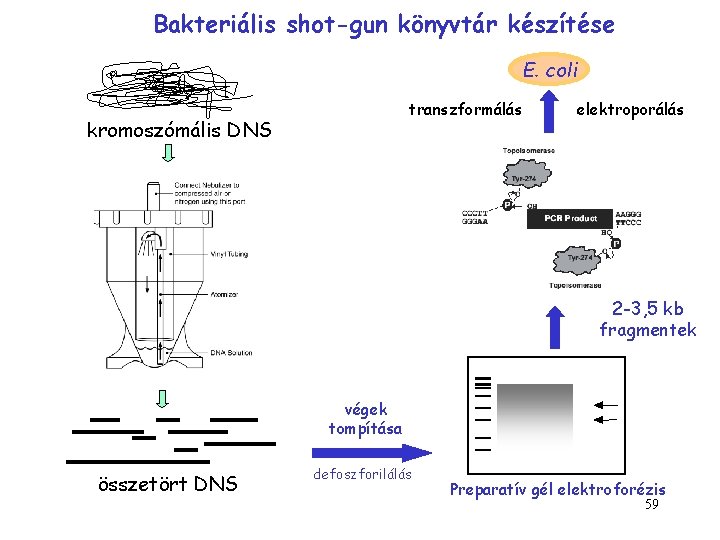
Bakteriális shot-gun könyvtár készítése E. coli transzformálás kromoszómális DNS elektroporálás nebulizátor 2 -3, 5 kb fragmentek végek tompítása összetört DNS defoszforilálás Preparatív gél elektroforézis 59

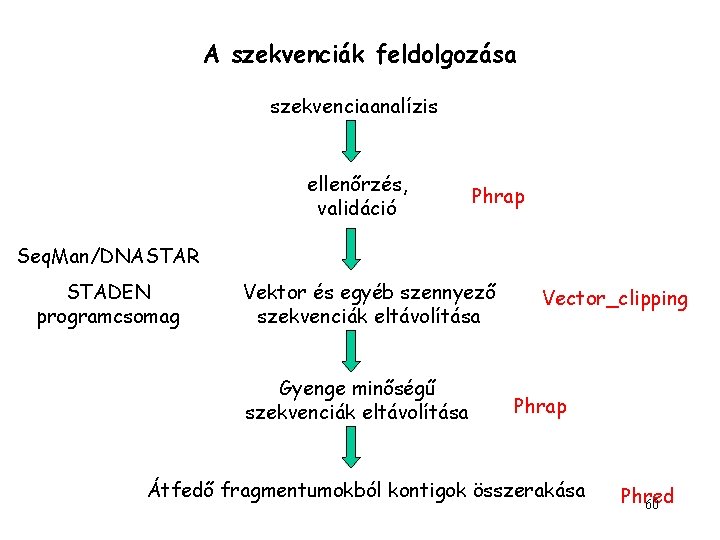
A szekvenciák feldolgozása szekvenciaanalízis ellenőrzés, validáció Phrap Seq. Man/DNASTAR STADEN programcsomag Vektor és egyéb szennyező szekvenciák eltávolítása Gyenge minőségű szekvenciák eltávolítása Vector_clipping Phrap Átfedő fragmentumokból kontigok összerakása Phred 60

De ha sikerül, és van szekvenciánk Mi van rajta, van-e gén? Honnan tudjuk, hogy Valamit találtunk, találtunk-e gént? CTCGAGACGCTGTTTCTGGGGTCATTCTTGGCGGGCTGCAACTGCTGGTGTGACCGACGCGACCTGGCAGGCCGCGGTGCGCAACTGGCCGGGCGGACTAATGGTGGAGCAAAAGA TCGGCATGTCCAGCGCACCTGAAGCTTGGGTGGTTGCTGCAATAGCAGCCTTATTGGCATGGCGAAGGGCGGTTTGGCCAATGTGGGGGTTATCGCCGTTCCCTTGATGTCCCTGG TCAAGCCGCCGCTTACCGCTGCCGGATTGCTGCTCCCGATCTATGTCGTTTCTGATGCATTCGGCGTCTGGCTTTATCGGCACCGGTATTCTGCCTCCAATCTGCGCATCCTGATTCCTT CGGGATTTTTTGGGGTCCTGATTGGCTGGTTATTGGCCGGGCAGATCTCCGACGCGATTGCCAGTGTCATTGTTGGTTTCACCGGCTGCGGCTTCGTGGCTGTGCTGCTGGCACGACGAG GGGTGCCATCGGTGCCGCGTCAAGCCAACGTGCCCAAAGGATGGTTTCTGGGGGTGGCCACCGGCTTTACCAGCTTTTTGACTCATTCCGGTGCGGCGACCTTCCAGATGTTCGTGCTGC CGCAACGGCTGGACAAGACCATGTTCGCGGGCACATCAACGCTTACCTTTGCTGCCATAAACCTATTCAAGATTCCGTCCTACTGGGCATTGGGACAGCTTTCGACTTCCTCGGTCATGT CCGCGCTAGTGTTGATTCCGGTGGCCGGGACGTTCGCAGGTGTTTTTGCGACGCGCAGGCTATCGACATCCTGGTTCTTCATTCTGGTCCAGGCGATGTTGCTGGTGGTCTCCA TTCAGCTTCTGTGGAGGGGAATGTCGGATATCCTGAACTAGCTGGAGATCGCAATGTCAGAACGCTCAATCAGAATGTAATCTTGACATAGAATACCGTTCCGATTTATTGCTTCG AGTGAAGCTGCCCGTCCGCTGAGATGTCATGACATTTTCCCCGCTTGATTCCGCCCTGCTTGGACCGTTGTTCGCGACCGATGAAATGCGCACGGTCTTCTCCGAACGGCGTTTTTTGGC GGGAATGCTTCGTGTTGAAGTGGCCCTGGCGCGCAGGCGGCAGAGGGCCTTGTCAGTTCGGAATTGGCCGACGCGATCGAGGTTGTTGGTACTGCCGGGTTGGACCCCGAGGCGAT GGCGGCGACTACTCGCATGACAGGAGTGCCCGCAATATCGTCCGTGCGGTGCAATCGGCCCTGCCGCCCTCACTGGCGGGTGGATTTCGGCGCCACCAGTCAAGACATCGT GGATACGGCCCACGCGCTCCAGCTGGCCGAGGCACTCGATATTATAGAAGTCGATTTACACGCCACTGTCAGCGCAATGATGAATCTGGCCGCTGCTCACTGCAATACACCCTGTATCGG GCGCACGGCCTTGCAGCACGCAGCGCCAGTTACGTTCGGCTACAAGGCGTCCGGCTGGTGCGTTGCCCTGGCGGAGCATCTGGTGCAGCTTCCCGCGCTGCGAAAGCGGGTTCTGGTGGC GTCGCTAGGGGGGCCGGTTGGTACCCTTGCCGCGATGGAGGAGCGGGCCGACGCTGTACTGGAGGGTTTCGCTGCGGACCTGGGGTTGGCCATTCCCGCCCTGGCACACGCAGCG GGCCCGGATCGTCGAGGTGGCCAGTTGGCCATATTGCTGGGAATTCTGGCAAAAATGGCCACCGATGTCGTTCACTTGTCCTCCACGGAAGTGCGCGAGCTTTCCGAACCTGTAGC GCCGGGCAGGGGGGGCTCCTCGGCGATGCCTCACAAGCGGAACCCGATTTCCTCGATTACCATCCTGTCCCAGCATGCTGCGGCAGGGGCCCAGCTCTCCATTCTCGTGAACGGCATGGC CAGTCTGCACGAACGTCCGGTGGGGGCGTGGCATTCGGAATGGTTGGCTCTGCCGACGCTGTTCGGCCTTGCCGGCGGTGCCGTGCGCGAGGGCAGGTTTCTGGCCGAGGGGCTGCTGGT CGATGCCGACCAGATGGGTCGCAATCTACAATTGACCAATGGCCTGATTTTCAGCGACGCGGTAGCCGGCCAGTTGGCAAAGCACTTGGGTCGGGCCGAGGCTTATGCCGCTGTCGAGGA TGCCGCCGCCGAGGTGTTGCGTTCAGGCGGCAGCTTTCAGGGTCAGCTGAACCAGCGCCTGCCCGATCACCGCGACGCTATTGCTTTTGATACGACGCCGGCGATCCAGGCCGG GGCCGCTGCCGTAGTGCGCTGGATCATGTGGCTCGTATTCTTGGACCCGCCTCTACCATCGGATTTCAAGGAGGCTAATGACGACACTGTTTGAGGCGACGACCATCCCGA TTTGCGAGGGCCCGCGCGACCAGACCGCCGAGATCCTTTTCGAGATGCCGCCGGGTGCGTGGGATACCCATTTTCATGTTTTTGGCCCAGTTTCATCGTTTCCATACGCAGAACACAGGC TCTATTCCCCACCGGAGTCGCCACTTGAGGATTATCTGGTGTTGATGGAGGCTTTGGGGATCGAGCGCGGCGTTTGTGTCCATCCGAATGTTCATGGTGCCGACAATTCGGTGACGCTCG 61 ACGCAGTTGCGCGGTCCGATGGTCGTCTGCTGGCGGTGATCAAGCCACATCACGAGATGACTTTTGTTCAGCTGCGGGACATGAAGGCGCAGGGGGTCTGCGGGGTACGTTTTGCCTTCA ATCCGCAGCATGGCTCGGGCGAGTTGGATACTCGTTTGTTCGAGCGTATGTTGGACTGGTGCCGCGACCTAGGCTGGTGCGTAAAATTGCATTTCGCGCCCGCTGGACGGTCTGG CTGAACGTTTGGCGCGCGTCGATATTCCGATCATCATCGATCATTTCGGGTGGACACCGCGCAAGGTGTGGATCAGCCGCACTTCCTGCGTTTGCTCGATCTGGCCAAACTGGACC

Fehérjeszekvenciák összehasonlítása 62

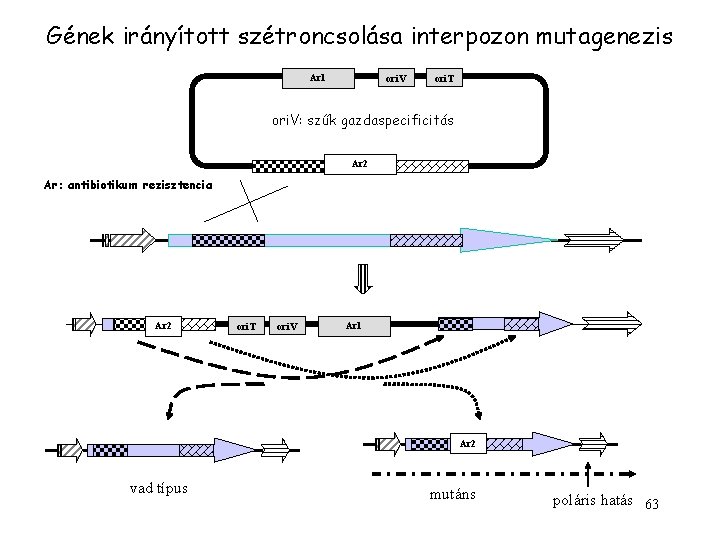
Gének irányított szétroncsolása interpozon mutagenezis Ar 1 ori. V ori. T ori. V: szűk gazdaspecificitás Ar 2 Ar: antibiotikum rezisztencia Ar 2 ori. T ori. V Ar 1 Ar 2 vad típus mutáns poláris hatás 63

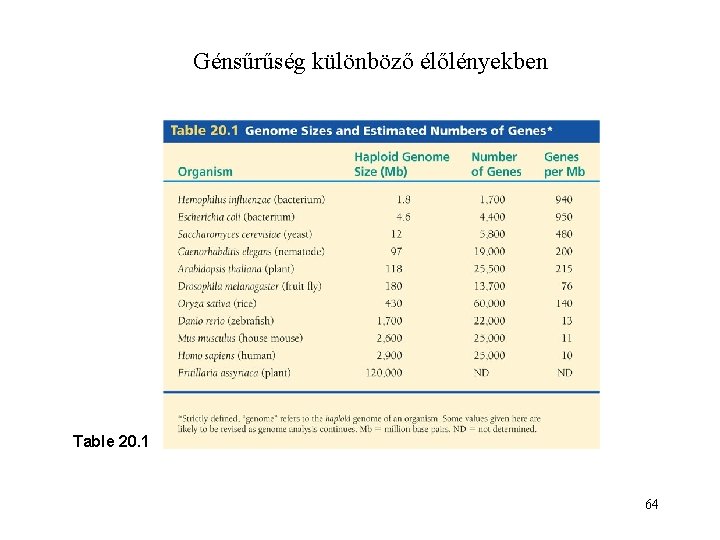
Génsűrűség különböző élőlényekben Table 20. 1 64

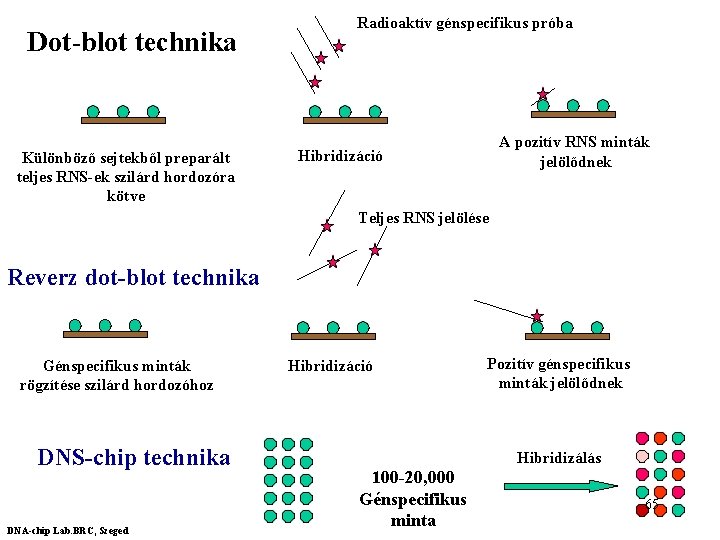
Dot-blot technika Különböző sejtekből preparált teljes RNS-ek szilárd hordozóra kötve Radioaktív génspecifikus próba A pozitív RNS minták jelölődnek Hibridizáció Teljes RNS jelölése Reverz dot-blot technika Génspecifikus minták rögzítése szilárd hordozóhoz DNS-chip technika DNA-chip Lab. BRC, Szeged Hibridizáció Pozitív génspecifikus minták jelölődnek Hibridizálás 100 -20, 000 Génspecifikus minta 65

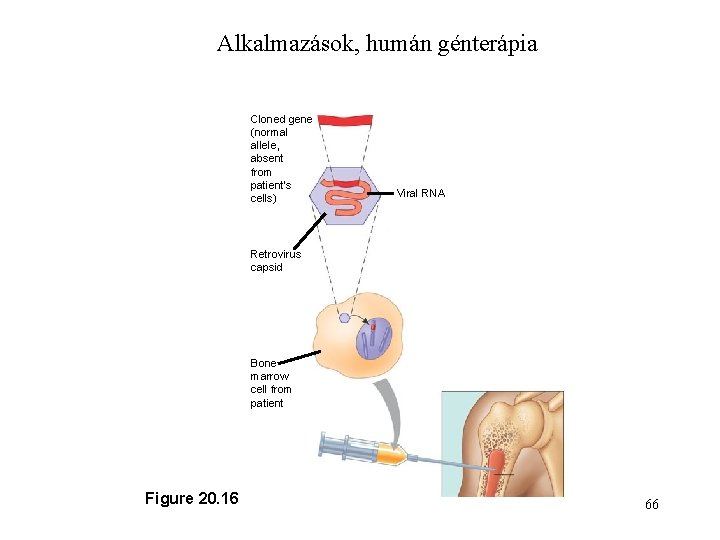
Alkalmazások, humán génterápia Cloned gene (normal allele, absent from patient’s cells) Viral RNA 2 Retrovirus capsid Bone marrow cell from patient Figure 20. 16 66

Alkalmazások, transzgenikus állatok Figure 20. 18 67

Alkalmazások, transzgenikus növények APPLICATION Genes conferring useful traits, such as pest resistance, herbicide resistance, delayed ripening, and increased nutritional value, can be transferred from one plant variety or species to another using the Ti plasmid as a vector. TECHNIQUE 1 Figure 20. 19 The Ti plasmid is isolated from the bacterium Agrobacterium tumefaciens. The segment of the plasmid that integrates into the genome of host cells is called T DNA. 2 Isolated plasmids and foreign DNA containing a gene of interest are incubated with a restriction enzyme that cuts in the middle of T DNA. After base pairing occurs between the sticky ends of the plasmids and foreign DNA fragments, DNA ligase is added. Some of the resulting stable recombinant plasmids contain the gene of interest. 3 Recombinant plasmids can be introduced into cultured plant cells by electroporation. Or plasmids can be returned to Agrobacterium, which is then applied as a liquid suspension to the leaves of susceptible plants, infecting them. Once a plasmid is taken into a plant cell, its T DNA integrates into the cell‘s chromosomal DNA. RESULTS Transformed cells carrying the transgene of interest can regenerate complete plants that exhibit the new trait conferred by the transgene. Agrobacterium tumefaciens Ti plasmid Site where restriction enzyme cuts DNA with the gene of interest T DNA Recombinant Ti plasmid Plant with new trait 68

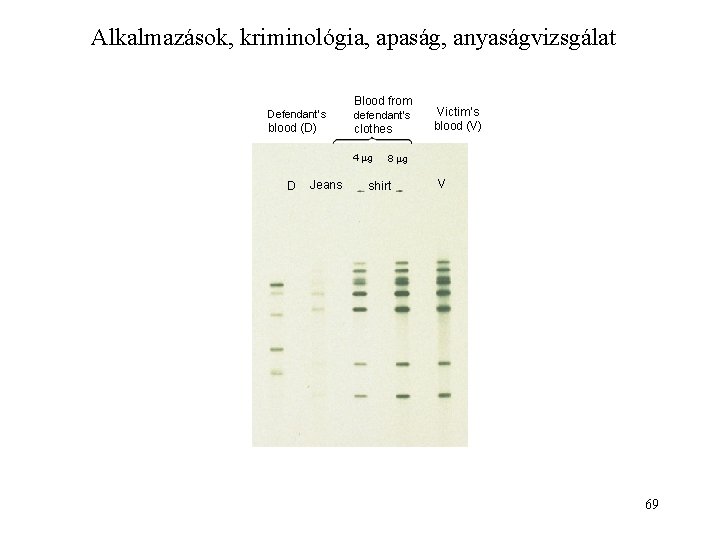
Alkalmazások, kriminológia, apaság, anyaságvizsgálat Defendant’s blood (D) Blood from defendant’s clothes 4 g D Jeans Victim’s blood (V) 8 g shirt V 69

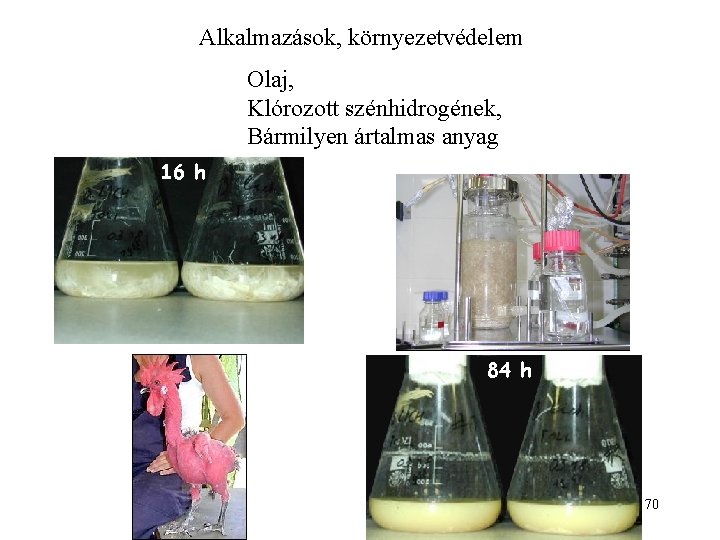
Alkalmazások, környezetvédelem Olaj, Klórozott szénhidrogének, Bármilyen ártalmas anyag 16 h 84 h 70

Szulfonált aromás vegyületek lebontása Nitrokémia Rt. elfolyó szennyvízben nagy mennyiségű szulfanilsav azofestékek, növényvédőszerek, antibiotikumok alapanyaga Igény biológiai eltávolításra • Izoláltunk egy baktériumot, mely képes a szulfanilsavat, mint egyedüli szén-, nitrogénés kénforrást hasznosítani • ezzel a tulajdonságával egyedülálló a világon 71

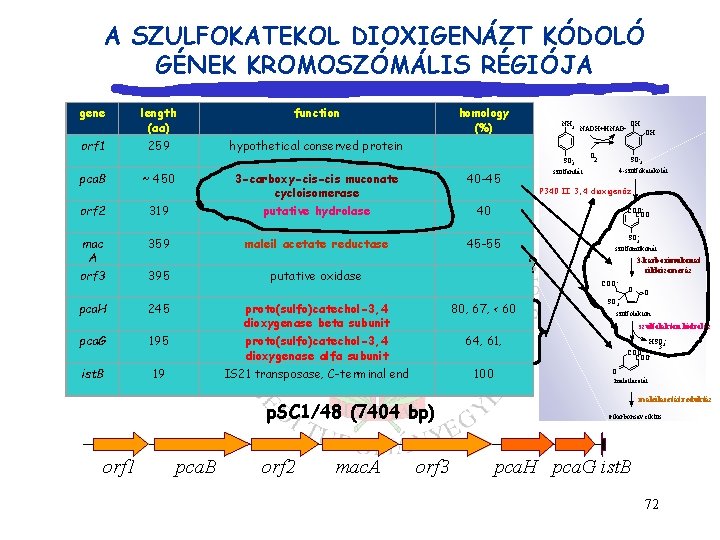
A SZULFOKATEKOL DIOXIGENÁZT KÓDOLÓ GÉNEK KROMOSZÓMÁLIS RÉGIÓJA gene orf 1 length (aa) function 259 hypothetical conserved protein homology (%) NH 2 NADH+HNAD + + SO -3 pca. B ~ 450 3 -carboxy-cis muconate cycloisomerase 40 -45 orf 2 319 putative hydrolase 40 mac A 359 maleil acetate reductase 45 -55 orf 3 395 pca. H putative oxidase 245 proto(sulfo)catechol-3, 4 dioxygenase beta subunit 80, 67, < 60 64, 61, ist. B 19 IS 21 transposase, C-terminal end 100 p. SC 1/48 (7404 bp) mac. A SO 3 szulfomukonát 3 -karboximukonat cikloizomeráz COO - O SO 3 - O szulfolakton hidroláz proto(sulfo)catechol-3, 4 dioxygenase alfa subunit orf 2 SO-3 4 -szulfokatekolát P 340 II 3, 4 dioxigenáz ? 195 pca. B OH COOCOO pca. G orf 1 szulfanilát O 2 OH orf 3 HSO 33 COO- COO O maleilacetát reduktáz trikarbonsav ciklus pca. H pca. G ist. B 72