ESCUELA SUPERIOR DEL LITORAL FACULTAD DE INGENIERA MARTIMA











- Slides: 22

ESCUELA SUPERIOR DEL LITORAL FACULTAD DE INGENIERÍA MARÍTIMA Y CIENCIAS DEL MAR “SILENCIAMIENTO DE POSIBLES GENES ANTIVIRALES EN Litopenaeus vannamei Y SU EFECTO EN LA SUSCEPTIBILIDAD AL VIRUS DEL SÍNDROME DE LA MANCHA BLANCA (WSSV)” TESIS DE GRADO Elaborado por: �Mirabá Guerrero Mariuxi �Morán Castillo Aida

La charla de hoy I. Introducción: • Virus del Síndrome de la Mancha Blanca (WSSV) • ARN de interferencia (ARNi) • Efectos de inyectar ds. RNA en camarón L. vannamei II. Métodos III. Resultados: • Bioinformática • Fenotipos encontrados IV. Conclusiones

Objetivos • Identificar, de entre 54 genes candidatos, genes potencialmente involucrados en la respuesta antiviral de L. vannamei usando la técnica de silenciamiento genético (RNAi, siglas en inglés) mediante la inyección de ARN bicatenario (ds. RNA, siglas en inglés). • Determinar las posibles proteínas codificadas por cada uno de los genes evaluados mediante análisis bioinformático de la base de datos genéticos (NCBI, National Center for Biotechnology Information).

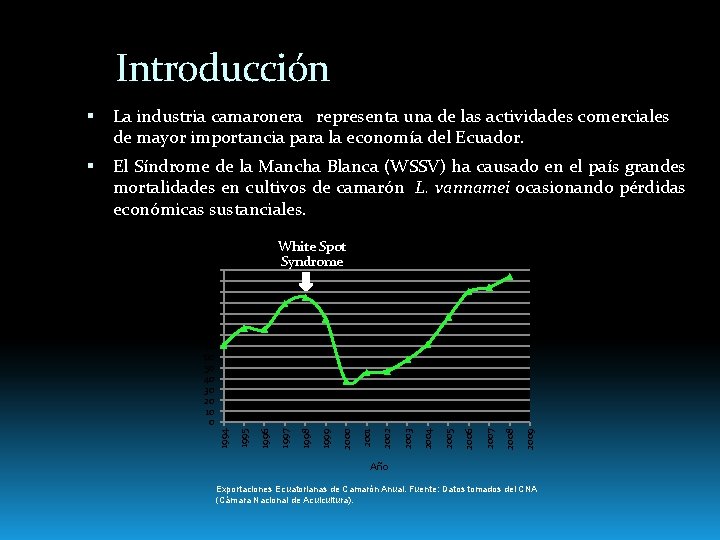
Introducción La industria camaronera representa una de las actividades comerciales de mayor importancia para la economía del Ecuador. El Síndrome de la Mancha Blanca (WSSV) ha causado en el país grandes mortalidades en cultivos de camarón L. vannamei ocasionando pérdidas económicas sustanciales. 2009 2008 2007 2006 2005 2004 2003 2002 2001 2000 1999 1998 1996 1995 140 130 120 110 100 90 80 70 60 50 40 30 20 10 0 1997 White Spot Syndrome 1994 Millions Año Exportaciones Ecuatorianas de Camarón Anual. Fuente: Datos tomados del CNA (Cámara Nacional de Acuicultura).

Virus del Síndrome de la Mancha Blanca (WSSV) Morfología de WSSV Virión Núcleo -capside Fuente: Virus Taxonomy: Eighth Report of the International Committee on the Taxonomy Viruses Familia, Nimaviridae Género Whispovirus ADN circular de doble cadena y envoltura lipídica exterior a la nucleocápside 275 nm de longitud y 85 nm de diámetro

Virus del Síndrome de la Mancha Blanca (WSSV) Descripción de la Enfermedad Manchas Blancas Aparición de manchas blancas en la cara interna de la cutícula de los crustáceos Manifestación de signos de estrés Reacción + anticuerpo Tejido sano Tejido infectado (WSSV) Tejido hematopoyetico teñido con Anticuerpos contra WSSV Letargia Coloración rojiza Anorexia Natación en superficie Muerte en horas o en unos pocos días Mortalidades altas en cultivo Enfermedad es atenuada a temperaturas altas Transmisión Horizontal

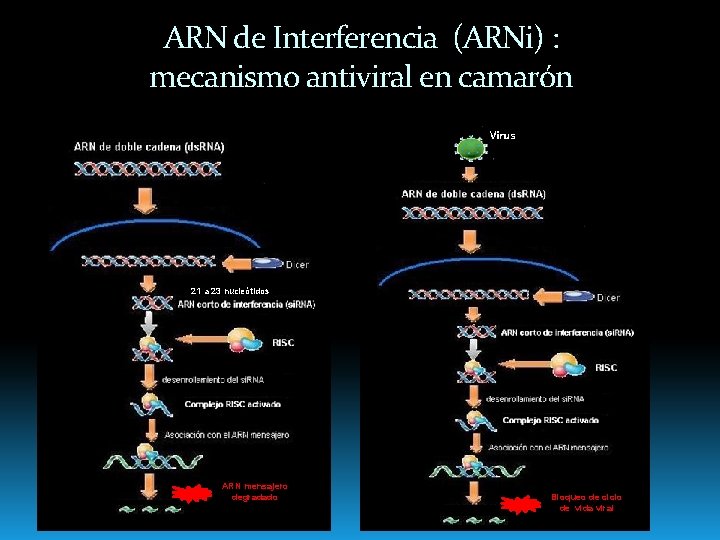
ARN de Interferencia (ARNi) : mecanismo antiviral en camarón 21 a 23 nucleótidos ARN mensajero degradado Bloqueo de ciclo de vida viral

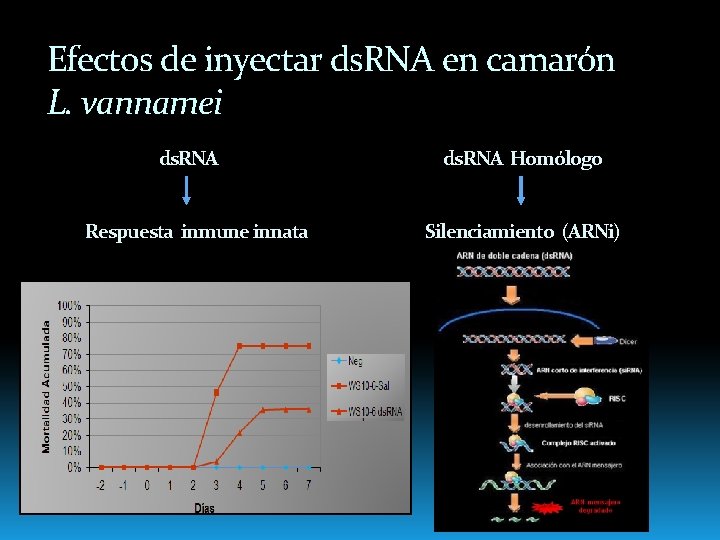
Efectos de inyectar ds. RNA en camarón L. vannamei ds. RNA Respuesta inmune innata ds. RNA Homólogo Silenciamiento (ARNi)

HIPOTESIS: Para genes importantes en la respuesta antiviral de L. vannamei, la inyección de un ds. RNA homólogo a los genes candidatos resulta en una respuesta inmune deficiente en comparación con ds. RNAs usados como controles en este estudio. Criterios para la selección de genes candidatos 1. Expresión aumenta en respuesta a una inyección de ds. RNA. 2. Expresión aumenta en respuesta a infección con WSSV. 3. Genes importantes para el sistema inmune del camarón

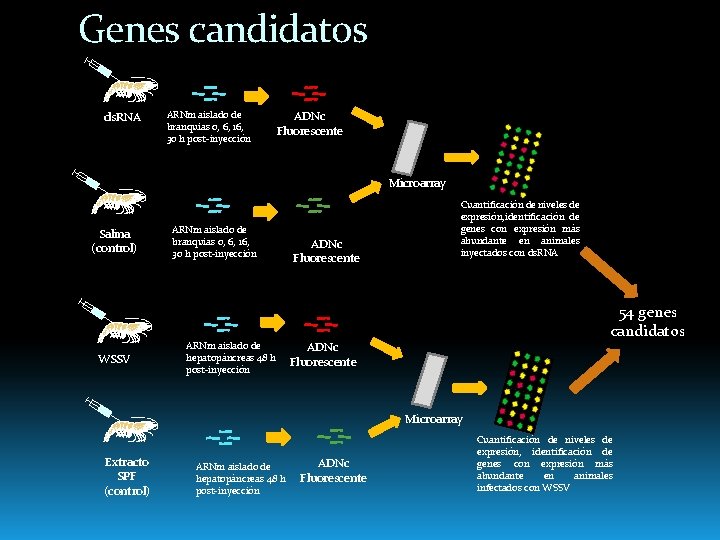
Genes candidatos ds. RNA ARNm aislado de branquias 0, 6, 16, 30 h post-inyección ADNc Fluorescente Microarray Salina (control) WSSV ARNm aislado de branquias 0, 6, 16, 30 h post-inyección ARNm aislado de hepatopáncreas 48 h post-inyección ADNc Fluorescente Cuantificación de niveles de expresión, identificación de genes con expresión más abundante en animales inyectados con ds. RNA 54 genes candidatos ADNc Fluorescente Microarray Extracto SPF (control) ARNm aislado de hepatopáncreas 48 h post-inyección ADNc Fluorescente Cuantificación de niveles de expresión, identificación de genes con expresión más abundante en animales infectados con WSSV

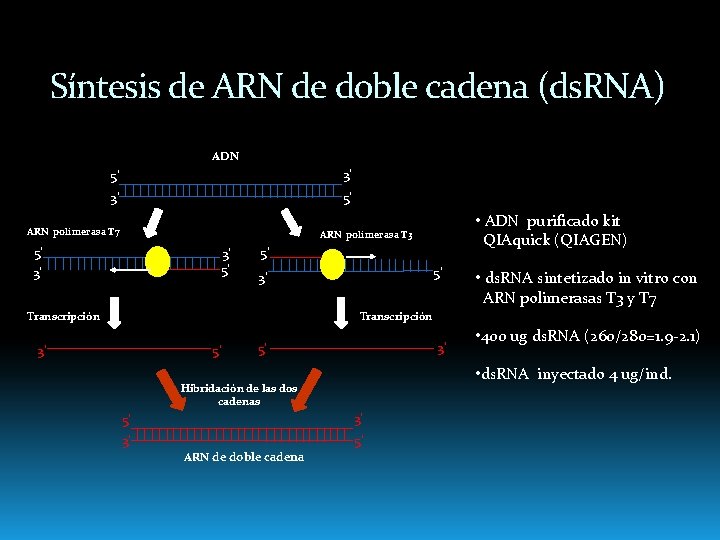
Síntesis de ARN de doble cadena (ds. RNA) ADN 5’ 3’ 3’ 5’ ARN polimerasa T 7 • ADN purificado kit QIAquick (QIAGEN) ARN polimerasa T 3 5’ 3’ 3’ 5’ 5’ 5’ 3’ Transcripción • ds. RNA sintetizado in vitro con ARN polimerasas T 3 y T 7 Transcripción 3’ 5’ • ds. RNA inyectado 4 ug/ind. Hibridación de las dos cadenas 5’ 3’ 3’ 5’ ARN de doble cadena • 400 ug ds. RNA (260/280=1. 9 -2. 1)

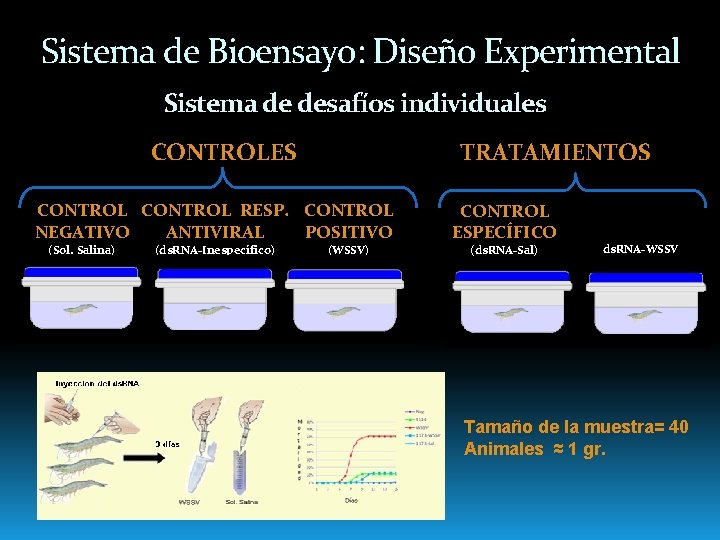
Sistema de Bioensayo: Diseño Experimental Sistema de desafíos individuales CONTROLES TRATAMIENTOS CONTROL RESP. CONTROL NEGATIVO ANTIVIRAL POSITIVO (Sol. Salina) (ds. RNA-Inespecífico) (WSSV) CONTROL ESPECÍFICO (ds. RNA-Sal) ds. RNA-WSSV Tamaño de la muestra= 40 Animales ≈ 1 gr.

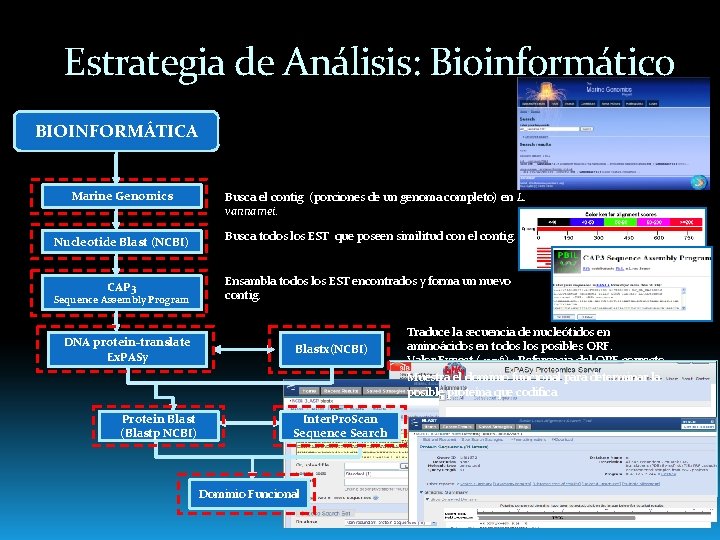
Estrategia de Análisis: Bioinformático BIOINFORMÁTICA Marine Genomics Busca el contig (porciones de un genoma completo) en L. vannamei. Nucleotide Blast (NCBI) Busca todos los EST que poseen similitud con el contig. CAP 3 Ensambla todos los EST encontrados y forma un nuevo contig. Sequence Assembly Program DNA protein-translate Ex. PASy Protein Blast (Blastp NCBI) Blastx(NCBI) Inter. Pro. Scan Sequence Search Dominio Funcional Traduce la secuencia de nucleótidos en aminoácidos en todos los posibles ORF. Valor Expect (<10 -6) ; Referencia del ORF correcto. Muestra el dominio funcional para determinar la posible proteína que codifica

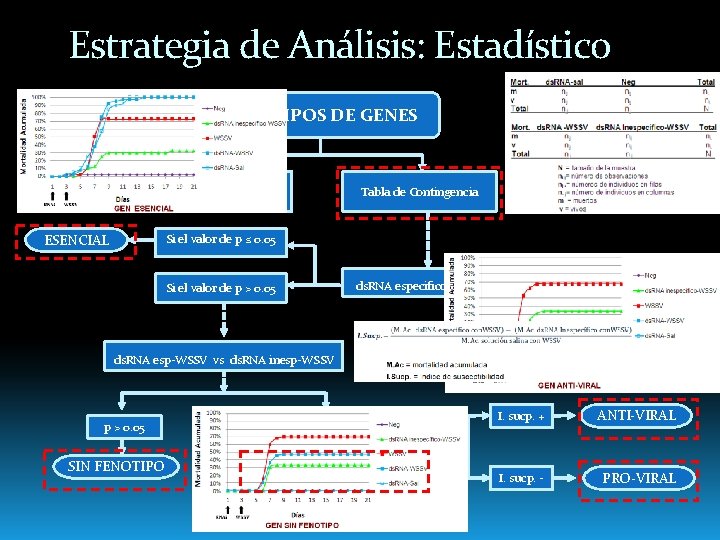
Estrategia de Análisis: Estadístico FENOTIPOS DE GENES Mortalidad de cada tratamiento ESENCIAL Tabla de Contingencia Si el valor de p ≤ 0. 05 ds. RNA especifico-sal vs Neg. Si el valor de p > 0. 05 ds. RNA esp-WSSV vs ds. RNA inesp-WSSV p > 0. 05 SIN FENOTIPO p ≤ 0. 05 Índice de susceptibilidad I. sucp. + ANTI-VIRAL I. sucp. - PRO-VIRAL

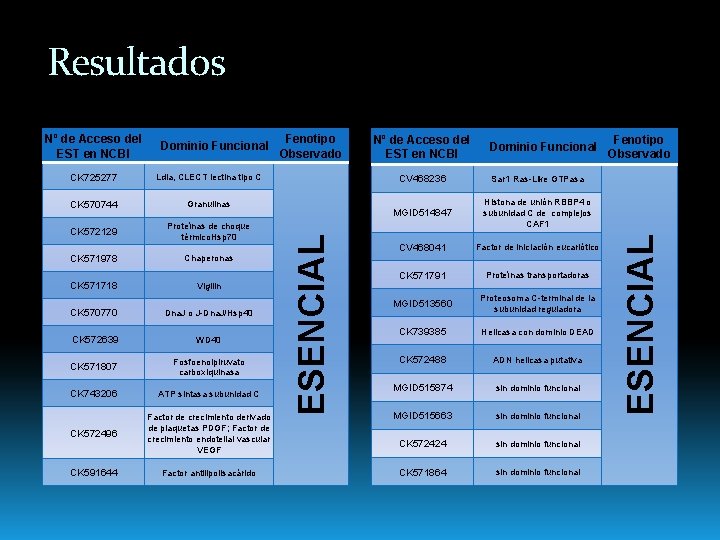
Resultados CK 725277 Ldla, CLECT lectina tipo C CK 570744 Granulinas CK 572129 Proteínas de choque térmico. Hsp 70 CK 571978 Chaperonas CK 571718 Vigilin CK 570770 Dna. J o J-Dna. J/Hsp 40 CK 572639 WD 40 CK 571807 Fosfoenolpiruvato carboxiquinasa CK 743206 ATP sintasa subunidad C CK 572496 Factor de crecimiento derivado de plaquetas PDGF; Factor de crecimiento endotelial vascular VEGF CK 591644 Factor antilipolisacárido Fenotipo Observado Nº de Acceso del EST en NCBI Dominio Funcional CV 468236 Sar 1 Ras-Like GTPasa MGID 514847 Histona de unión RBBP 4 o subunidad C de complejos CAF 1 CV 468041 Factor de iniciación eucariótico CK 571791 Proteínas transportadoras MGID 513560 Proteosoma C-terminal de la subunidad reguladora CK 739385 Helicasa con dominio DEAD CK 572488 ADN helicasa putativa MGID 515874 sin dominio funcional MGID 515663 sin dominio funcional CK 572424 sin dominio funcional CK 571864 sin dominio funcional Fenotipo Observado ESENCIAL Dominio Funcional ESENCIAL Nº de Acceso del EST en NCBI

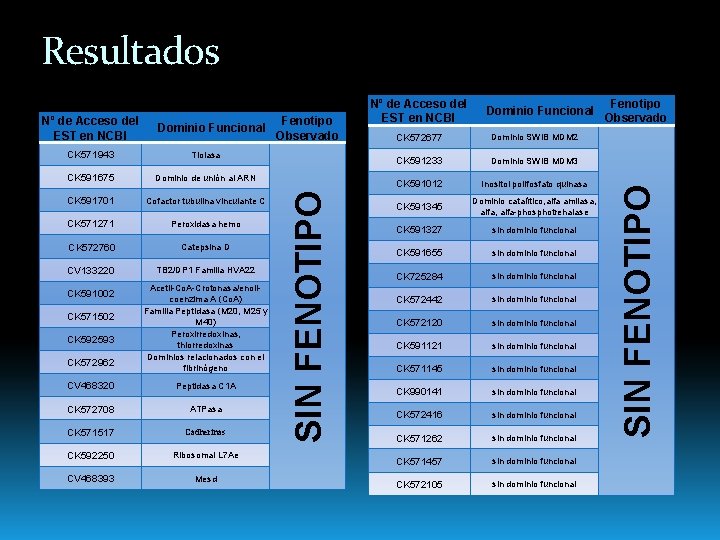
Resultados CK 571943 Tiolasa CK 591675 Dominio de unión al ARN CK 591701 Cofactor tubulina vinculante C CK 571271 Peroxidasa hemo CK 572760 Catepsina D CV 133220 TB 2/DP 1 Familia HVA 22 CK 591002 CK 571502 CK 592593 CK 572962 Acetil-Co. A-Crotonasa/enoilcoenzima A (Co. A) Familia Peptidasa (M 20, M 25 y M 40) Peroxirredoxinas, thiorredoxinas Dominios relacionados con el fibrinógeno CV 468320 Peptidasa C 1 A CK 572708 ATPasa CK 571517 Cadherinas CK 592250 Ribosomal L 7 Ae CV 468393 Mesd Fenotipo Observado Dominio Funcional CK 572677 Dominio SWIB MDM 2 CK 591233 Dominio SWIB MDM 3 CK 591012 Inositol polifosfato quinasa CK 591345 Dominio catalítico, alfa amilasa, alfa-phosphotrehalase CK 591327 sin dominio funcional CK 591655 sin dominio funcional CK 725284 sin dominio funcional CK 572442 sin dominio funcional CK 572120 sin dominio funcional CK 591121 sin dominio funcional CK 571145 sin dominio funcional CK 990141 sin dominio funcional CK 572416 sin dominio funcional CK 571262 sin dominio funcional CK 571457 sin dominio funcional CK 572105 sin dominio funcional Fenotipo Observado SIN FENOTIPO Dominio Funcional SIN FENOTIPO Nº de Acceso del EST en NCBI

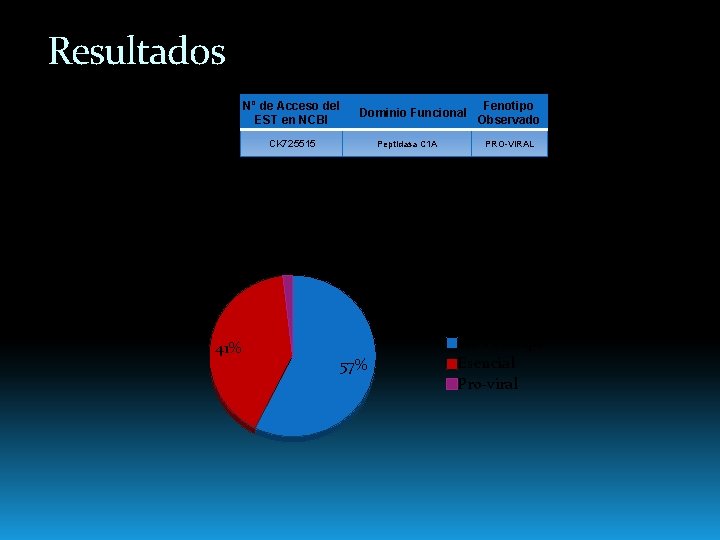
Resultados Nº de Acceso del EST en NCBI Dominio Funcional CK 725515 Peptidasa C 1 A Fenotipo Observado PRO-VIRAL Fenotipos Identificados 2% 41% 57% Sin Fenotipo Esencial Pro-viral

Discusión-Conclusiones Uno de los 54 genes estudiados presentó un fenotipo de potencial interés (posible gen pro-viral). El 41% de los genes evaluados fueron esenciales para la supervivencia del camarón, aún en ausencia de infección experimental Encontrar genes antivirales tiene aplicación en la selección de camarones más resistentes a enfermedades. Identificar genes antivirales tiene relevancia para comprender a nivel molecular los mecanismos antivirales en el camarón.

Discusión-Conclusiones El silenciamiento con ds. RNA (ARNi) representa un método promisorio para la evaluación funcional de genes candidatos.

Recomendaciones En base a los resultados, se recomienda realizar más bioensayos en busca de genes antivirales en L. vannamei realizando réplicas para cada tratamiento. Confirmar los fenotipos identificados en este y en futuros trabajos mediante la cuantificación de los niveles de ARN de los genes bloqueados Realizar análisis a nivel histológico de animales que presentaron alta mortalidad en ausencia de WSSV (fenotipo esencial).

Agradecimientos • FINANCIAMIENTO: MUSC-USA BIOGEMAR-EC • ESPOL: Ph. D. Javier Robalino (Director de Tesis) Ph. D. Marcelo Muñoz Ph. D. Washington Cardenas • BIOGEMAR: Ing. Walter Intriago Msc. Ricardo Cedeño • CENAIM: Lcda. Irma Bentancourt Ph. D. José Melena

Gracias =)
 Massa martima
Massa martima Escuela tradicional linea del tiempo
Escuela tradicional linea del tiempo 1.ingenieramédicaprogramadoraperiodistahijastra
1.ingenieramédicaprogramadoraperiodistahijastra Universidad alonso de ojeda
Universidad alonso de ojeda Competencias abet
Competencias abet Análisis horizontal del estado de resultados
Análisis horizontal del estado de resultados Litoral mapa
Litoral mapa Pedagogia existencialista
Pedagogia existencialista Escuela tradicional y escuela nueva
Escuela tradicional y escuela nueva Hacer de una escuela una buena escuela
Hacer de una escuela una buena escuela Femenine
Femenine Isfd escuela normal superior n 1 aula virtual
Isfd escuela normal superior n 1 aula virtual Escuela de enfermeria cecilia grierson
Escuela de enfermeria cecilia grierson Bandera de la escuela normal superior sady tobon calle
Bandera de la escuela normal superior sady tobon calle Escuela justo josé de urquiza
Escuela justo josé de urquiza Escuela normal superior dr alejandro carbó
Escuela normal superior dr alejandro carbó Escuela normal superior de la laguna cursos intensivos
Escuela normal superior de la laguna cursos intensivos Escuela normal superior convencion
Escuela normal superior convencion Escuela superior de comercio manuel belgrano
Escuela superior de comercio manuel belgrano Escuela superior militar cosme renella barbato
Escuela superior militar cosme renella barbato Escuela superior de pnl regalos
Escuela superior de pnl regalos Escuela preparatoria instituto superior benjamín franklin
Escuela preparatoria instituto superior benjamín franklin Escuela de educacion superior tecnico profesional pnp
Escuela de educacion superior tecnico profesional pnp