Chromatyna a epigenetyka Chromatyna wypenia jdro komrkowe 23

















- Slides: 76

Chromatyna a epigenetyka

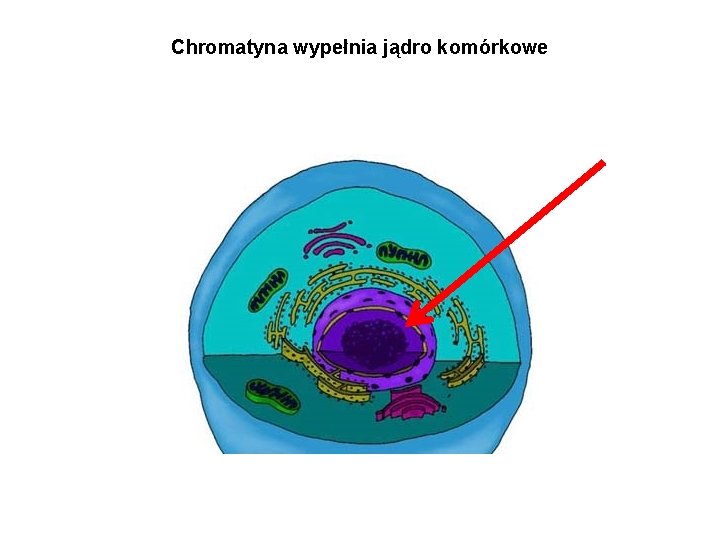
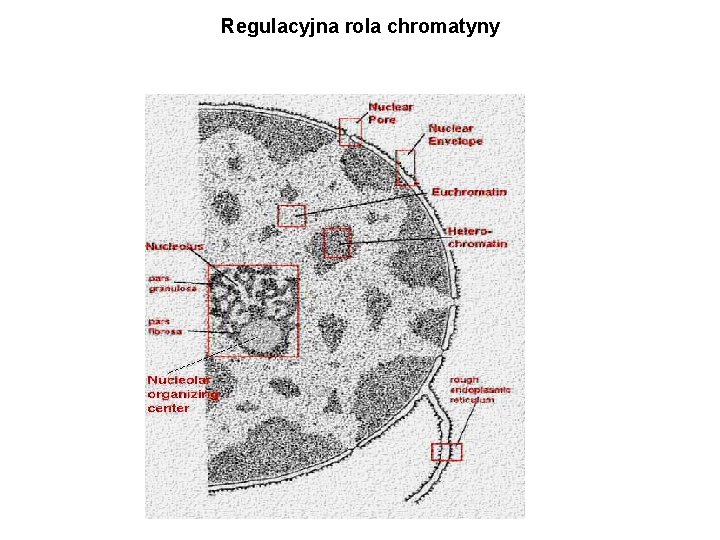
Chromatyna wypełnia jądro komórkowe

23 ludzkie chromosomy w jądrach fibroblastów we wczesnej profazie

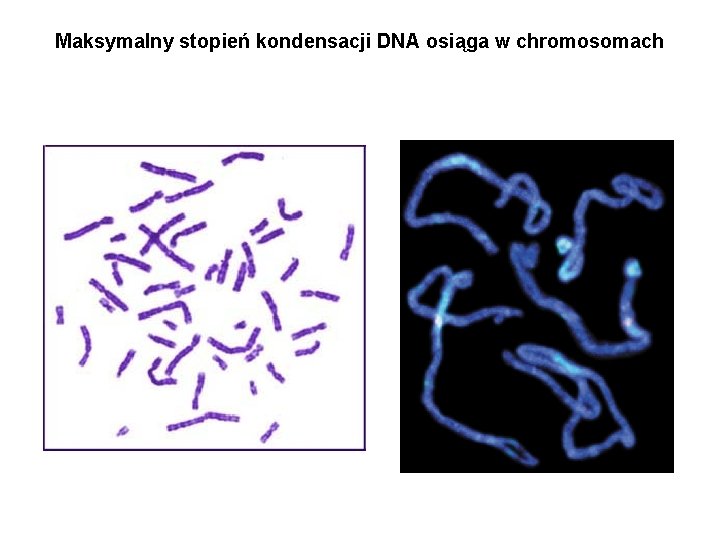
Maksymalny stopień kondensacji DNA osiąga w chromosomach

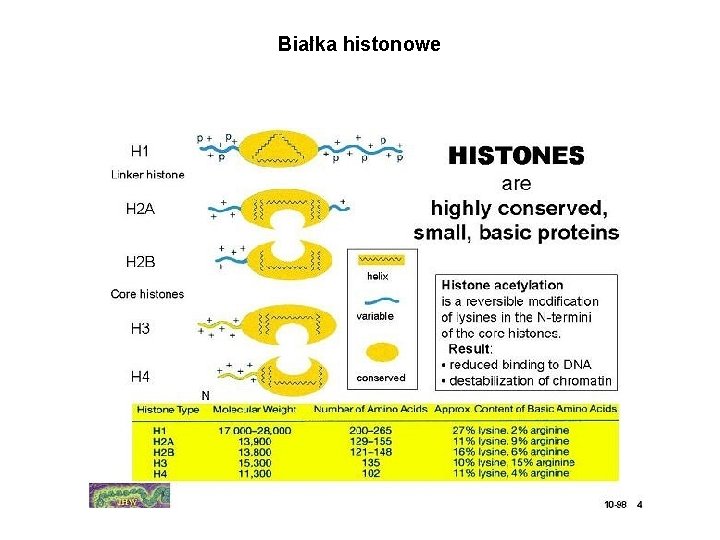
Białka histonowe

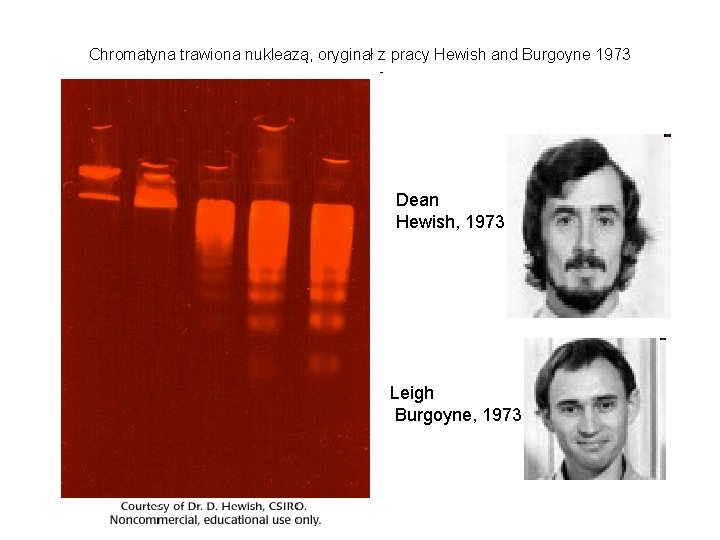
Chromatyna trawiona nukleazą, oryginał z pracy Hewish and Burgoyne 1973 Dean Hewish, 1973 Leigh Burgoyne, 1973

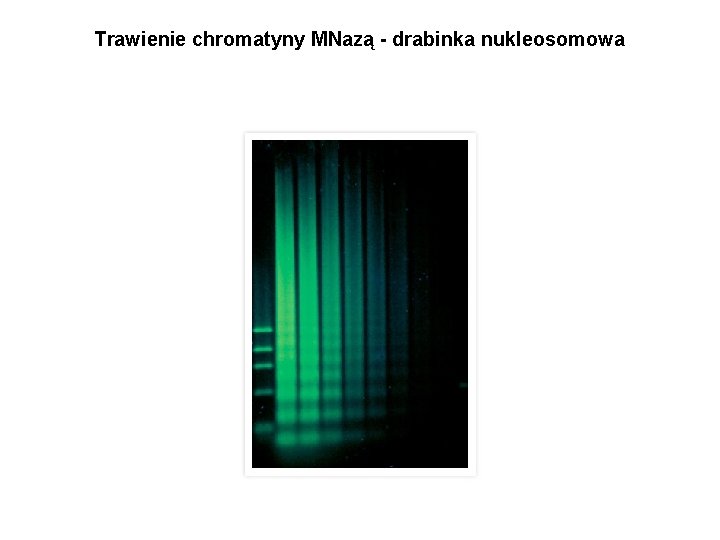
Trawienie chromatyny MNazą - drabinka nukleosomowa

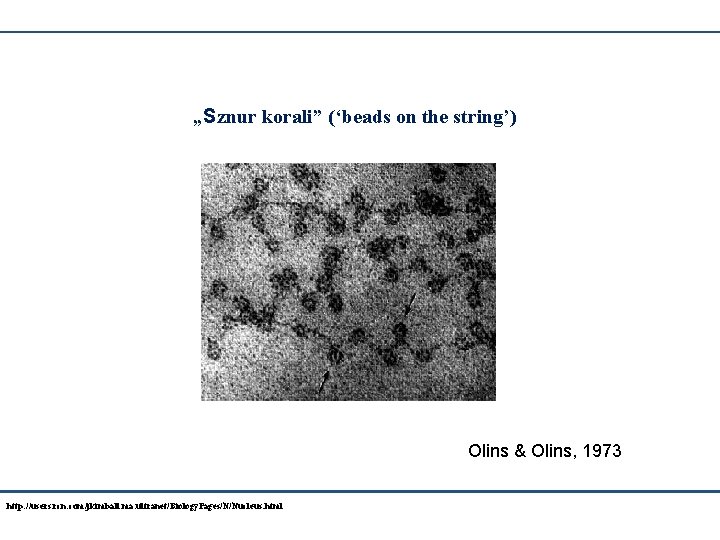
„Sznur korali” (‘beads on the string’) Olins & Olins, 1973 http: //users. rcn. com/jkimball. ma. ultranet/Biology. Pages/N/Nucleus. html

Roger Kornberg w 1974 r. zaproponował model, w którym DNA owinięty jest wokół rdzenia histonowego tworząc nukleosom NUKLEOSOM JEST PODSTAWOWĄ JEDNOSTKĄ STRUKTURALNĄ CHROMATYNY

Fałd histonowy

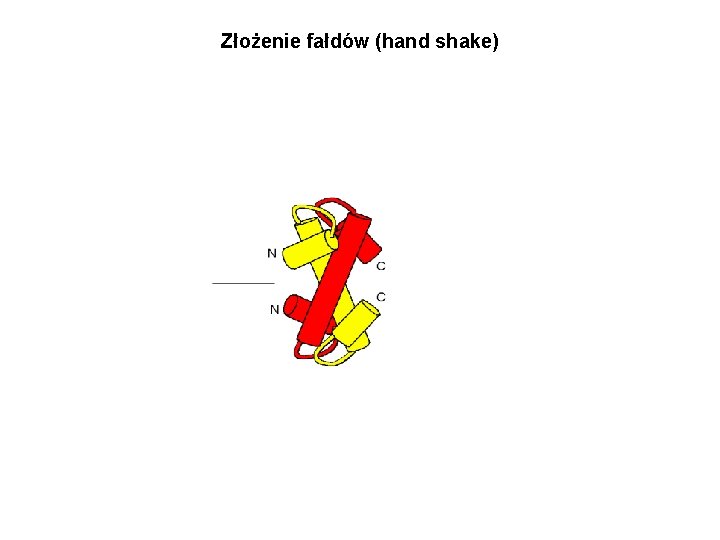
Złożenie fałdów (hand shake)

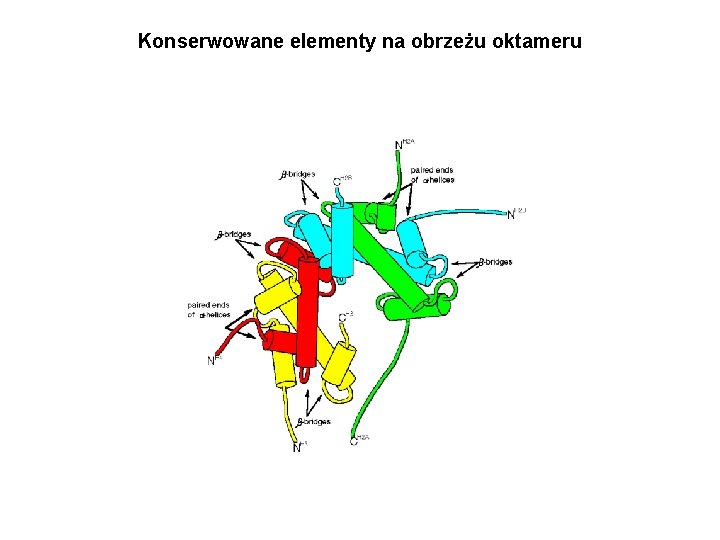
Konserwowane elementy na obrzeżu oktameru

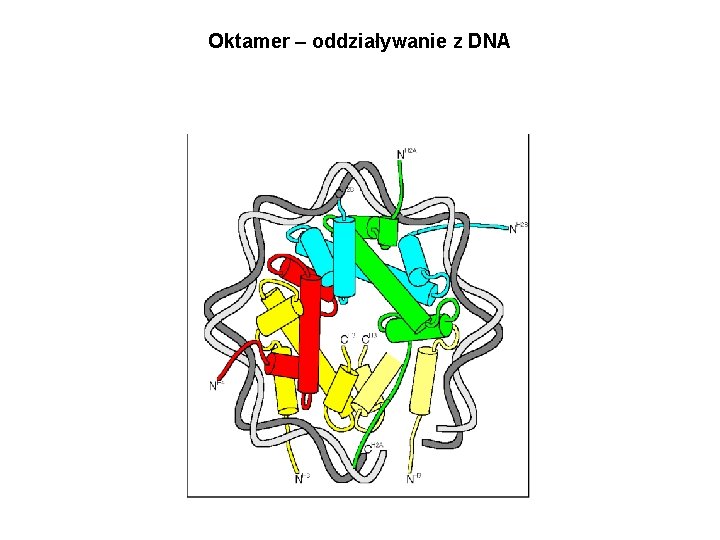
Oktamer – oddziaływanie z DNA

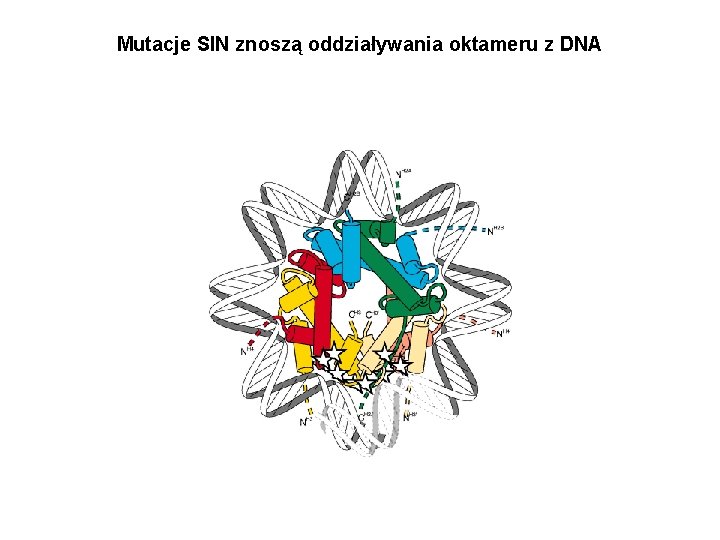
Mutacje SIN znoszą oddziaływania oktameru z DNA

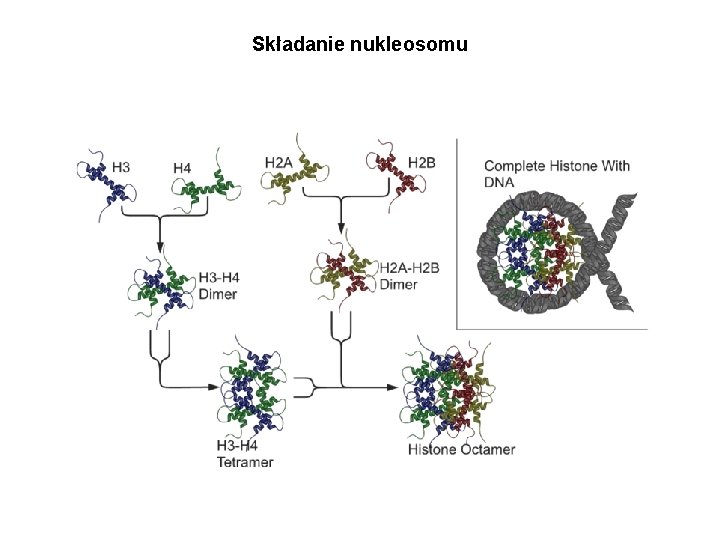
Składanie nukleosomu

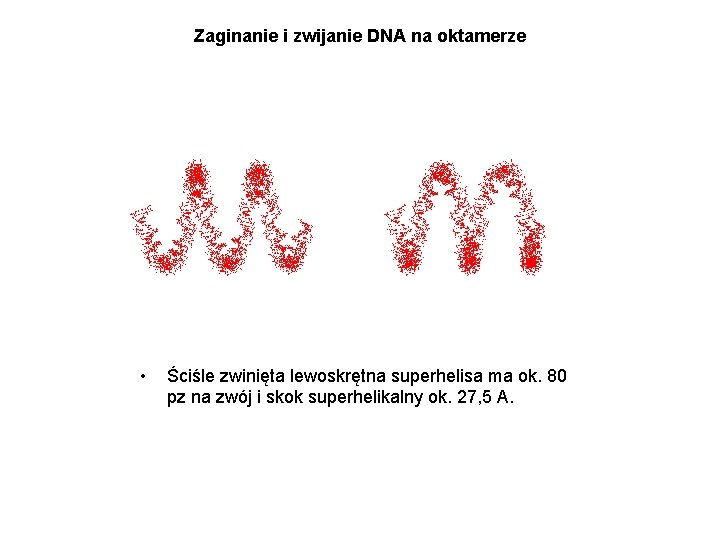
Zaginanie i zwijanie DNA na oktamerze • Ściśle zwinięta lewoskrętna superhelisa ma ok. 80 pz na zwój i skok superhelikalny ok. 27, 5 A.

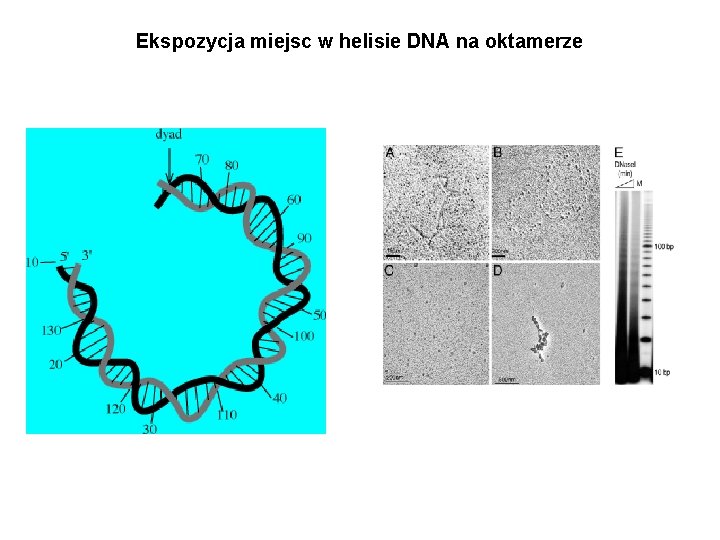
Ekspozycja miejsc w helisie DNA na oktamerze

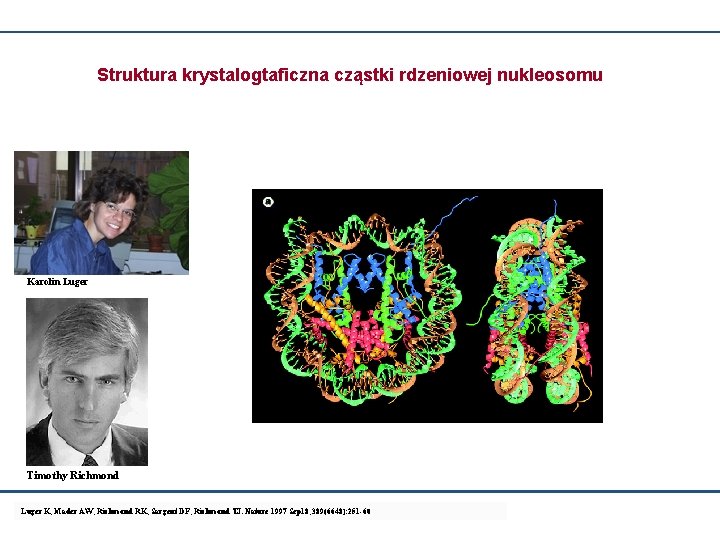
Struktura krystalogtaficzna cząstki rdzeniowej nukleosomu Karolin Luger Timothy Richmond Luger K, Mader AW, Richmond RK, Sargent DF, Richmond TJ. Nature 1997 Sep 18; 389(6648): 251 -60


Schemat nukleosomu

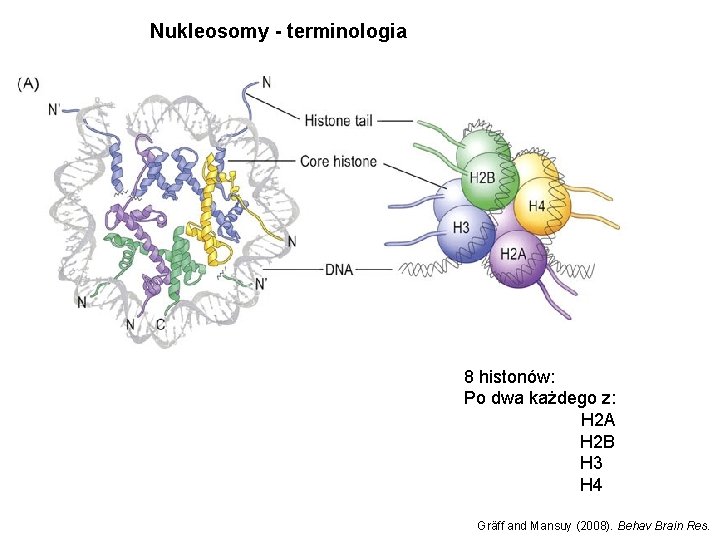
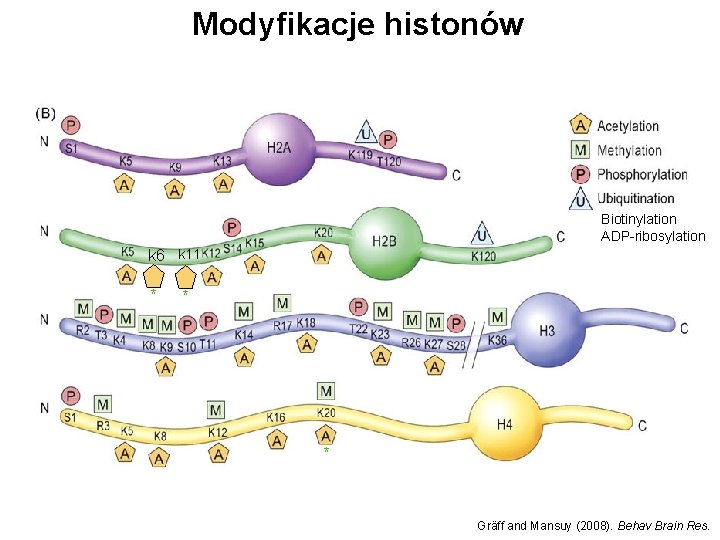
Nukleosomy - terminologia 8 histonów: Po dwa każdego z: H 2 A H 2 B H 3 H 4 Gräff and Mansuy (2008). Behav Brain Res.

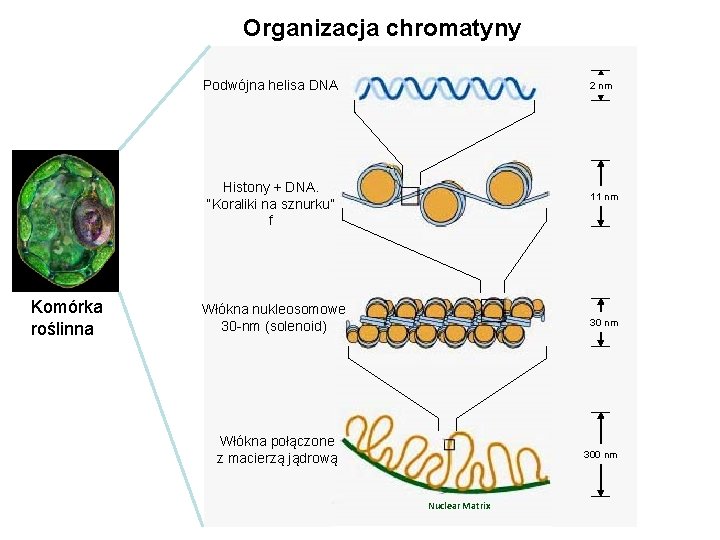
Organizacja chromatyny Podwójna helisa DNA 2 nm Histony + DNA. “Koraliki na sznurku” f Komórka roślinna 11 nm Włókna nukleosomowe 30 -nm (solenoid) 30 nm Włókna połączone z macierzą jądrową 300 nm Nuclear Matrix

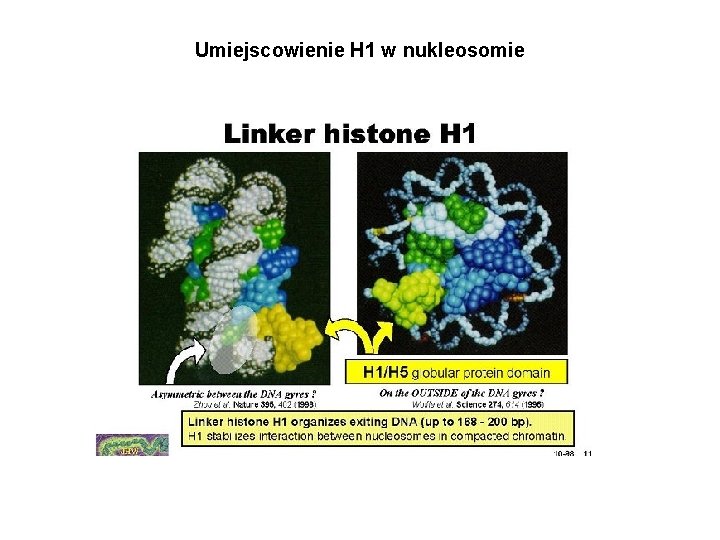
Umiejscowienie H 1 w nukleosomie

Regulacyjna rola chromatyny

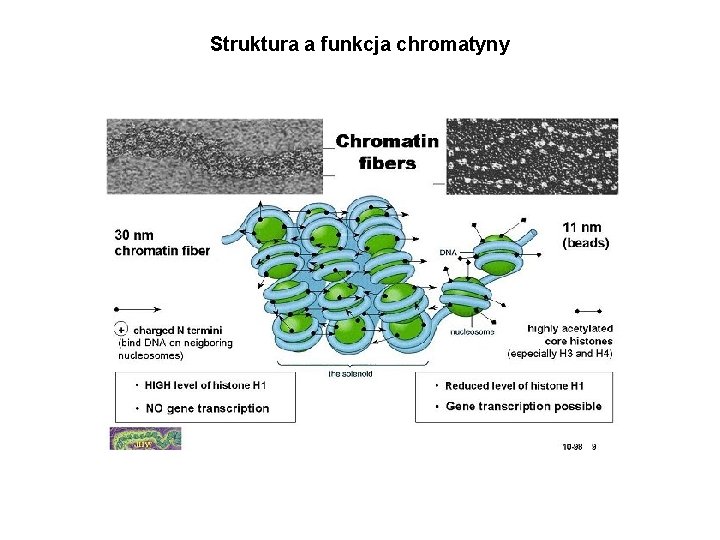
Struktura a funkcja chromatyny

Zmiany struktury chromatyny • modyfikacje DNA • modyfikacje potranslacyjne histonów • wyspecjalizowane warianty histonów • ATP-zależna przebudowa (remodeling) chromatyny

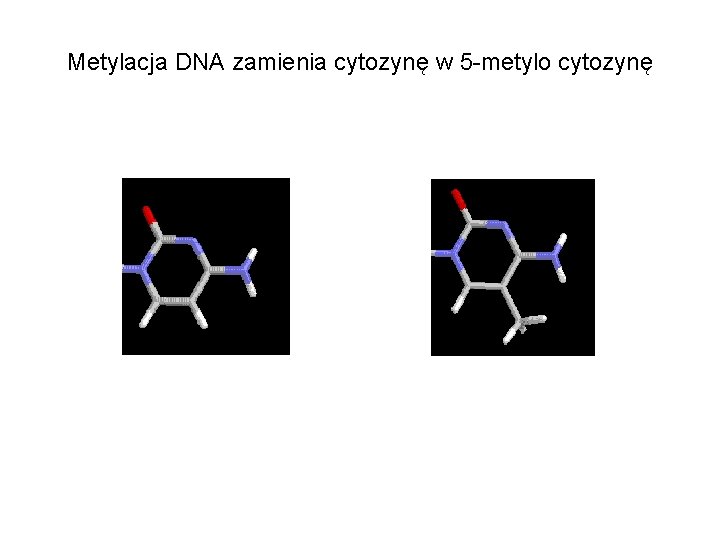
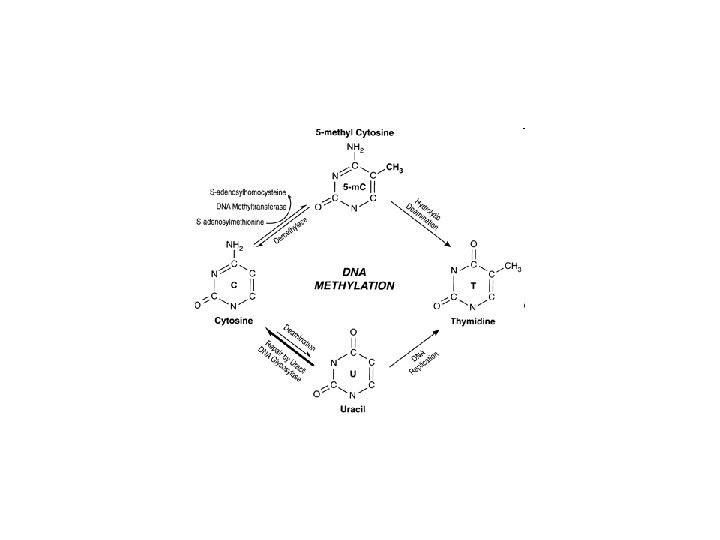
Metylacja DNA, Metylomy

Metylacja DNA zamienia cytozynę w 5 -metylo cytozynę


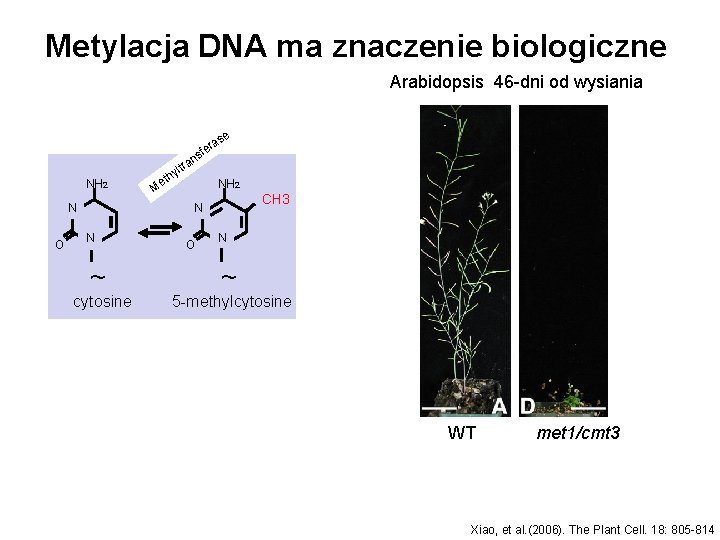
Metylacja DNA ma znaczenie biologiczne Arabidopsis 46 -dni od wysiania e as r sfe n a r NH 2 N O ylt h t e NH 2 M CH 3 N N ~ cytosine O N ~ 5 -methylcytosine WT met 1/cmt 3 Xiao, et al. (2006). The Plant Cell. 18: 805 -814

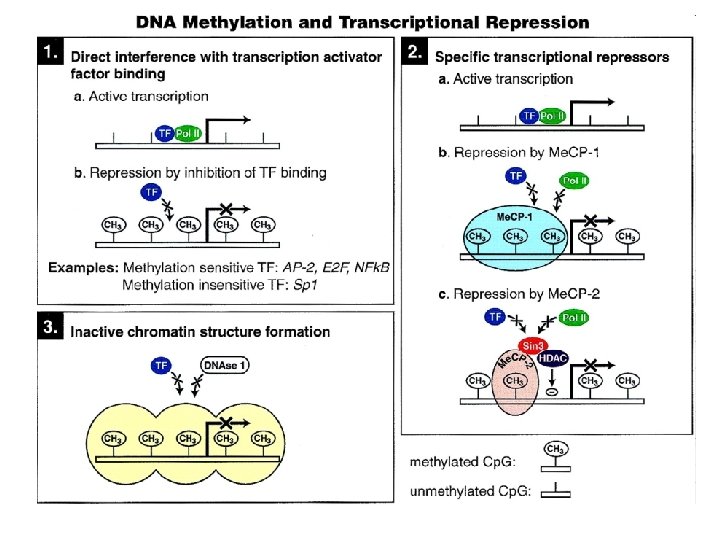
Metylacja DNA u eukariontów • Nie jest uniwersalna, występuje u ssaków i roślin kwiatowych • Jest zmienna gatunkowo, tkankowo i chromosomowo • Rozpoznawana przez rodzinę białek zawierających domenę (MBD) wiążącą metylowany DNA • Ściąga kompleksy białkowe indukujące zmiany w lokalnej strukturze chromosomów • Wyłącza ekspresję genów • Zakłócenia w normalnym wzorze metylacji DNA są niemal powszechnie wykrywane w nowotworach


Transpozony • Fragmenty DNA, które mogą się wstawiać w nowe miejsce w chromosomie • Niektóre są zdolne do autokopiowania, co prowadzi do wzrostu ich ilości w genomie • Odpowiadają za wielkoskalowe zmiany w chromosomach, jak również pojedyncze wydarzenia mutacyjne

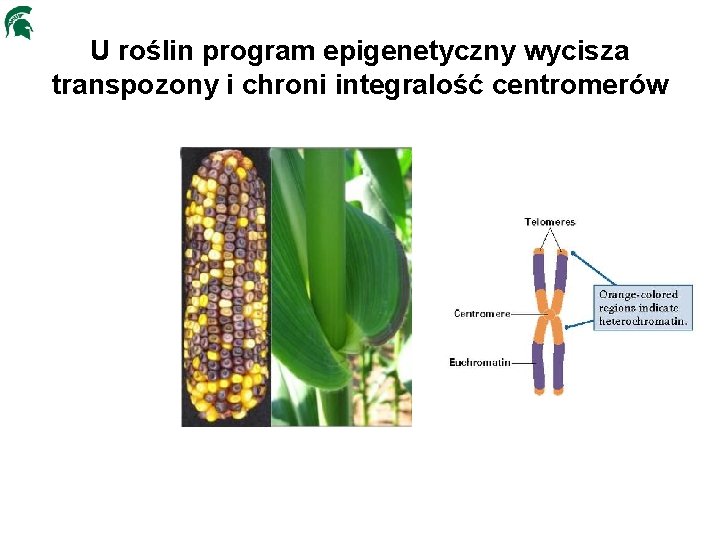
U roślin program epigenetyczny wycisza transpozony i chroni integralość centromerów

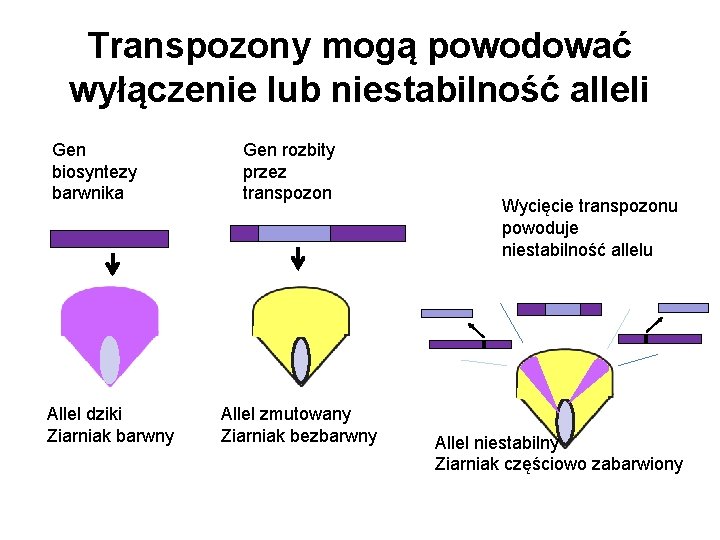
Transpozony mogą powodować wyłączenie lub niestabilność alleli Gen biosyntezy barwnika Allel dziki Ziarniak barwny Gen rozbity przez transpozon Allel zmutowany Ziarniak bezbarwny Wycięcie transpozonu powoduje niestabilność allelu Allel niestabilny Ziarniak częściowo zabarwiony

Metylacja DNA jest niezbędna do wyciszania transpozonów Niebieski = gęstość genów Czerwony = Gęstość elementów powtarzalnych Utrata funkcji met 1 lub ddm 1 (decrease in DNA methylation 1) mutanty mają niedometylowany DNA Zielony = Metylowany DNA Brązowy = Metylowany DNA w mutancie met 1 Zhang, et al. (2006). Cell 126: 1189 -1201

Metylotransferazy DNA w Arabidopsis MET 1 (METHYLTRANSFERASE 1) • Miejsca 5'-CG-3' • Wyciszanie transpozonów, elementów powtórzonych, piętnowanie genów (imprinting) CMT 3 (CHROMOMETHYLASE 3) • Miejsca 5'-CHG-3' • (H= A, C or T) • Oddziaływanie z modyfikakcjami histonów DRM 1, DRM 2 (DOMAINS REARRANGED 1 and 2) • Miejsca 5'-CHH-3' • Metyluje głównie elementy powtarzalne • Wymaga aktywnej indukcji przez si. RNA © 2013 American Society of Plant Biologists

Aktywacja transpozonów w mutancie ddm 1 indukuje mutacje Dziki Po 1 pokoleniu Po 3 pokoleniach Po 5 pokoleniach Po wyłączeniu DDM 1, fenotyp rośliny staje się coraz bardziej nienormalny, w miarę akumulowania indukowanych przez transpozony. Kakutani, et al. (1996). PNAS. 93: 12406 -12411

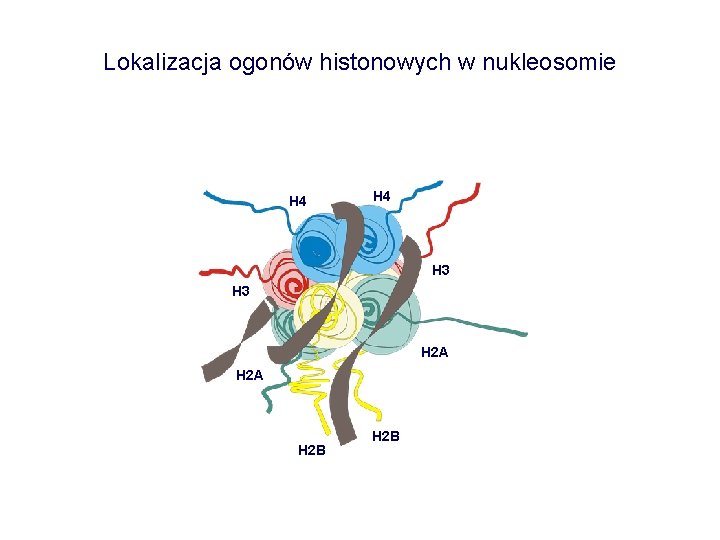
Lokalizacja ogonów histonowych w nukleosomie H 4 H 3 H 2 A H 2 B

Enzymy modyfikujące histony i DNA • HAT – acetylotransferazy histonów (bromodomena) • HDAC – deacteylazy histonów • HMT – metylotransferazy histonów (chromodomena). CMT 3 – DNA metylotransferaza (chromodomena)

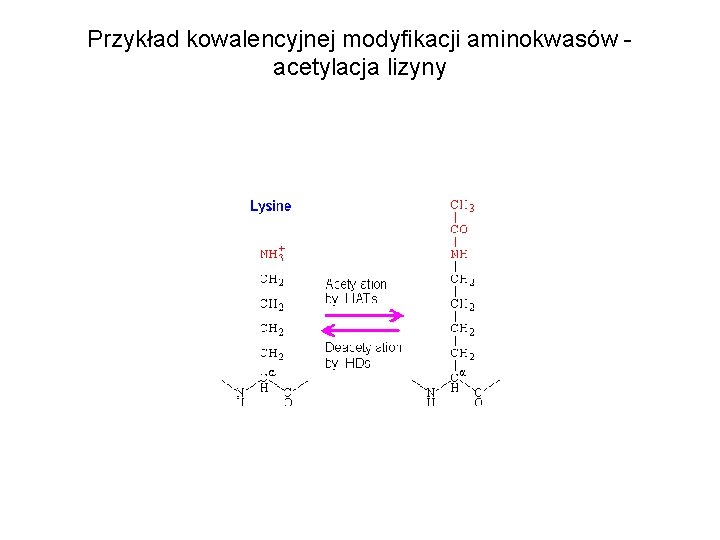
Przykład kowalencyjnej modyfikacji aminokwasów acetylacja lizyny

Elektroforetyczny dowód acetylacji histonów

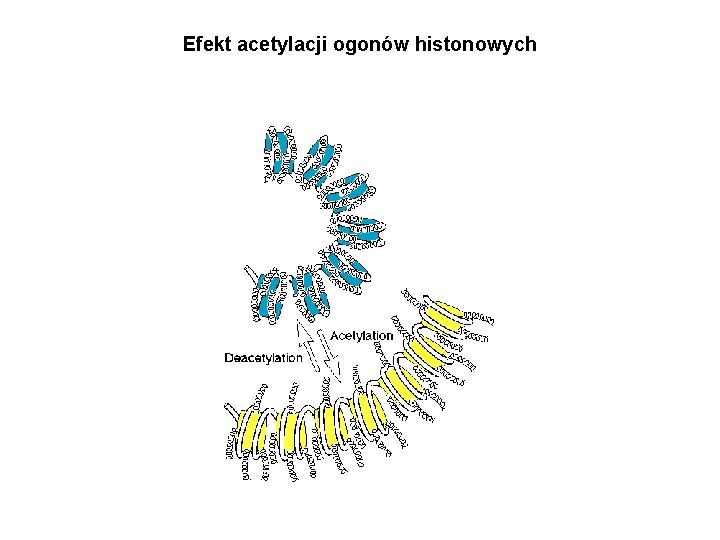
Efekt acetylacji ogonów histonowych

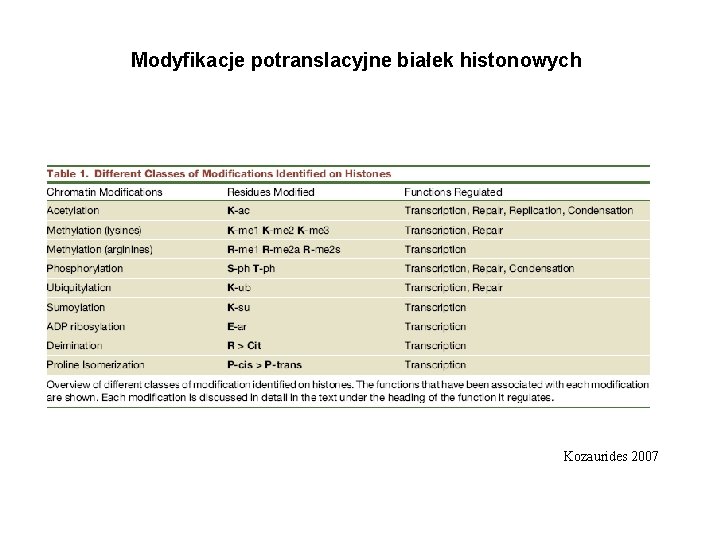
Modyfikacje potranslacyjne białek histonowych Kozaurides 2007

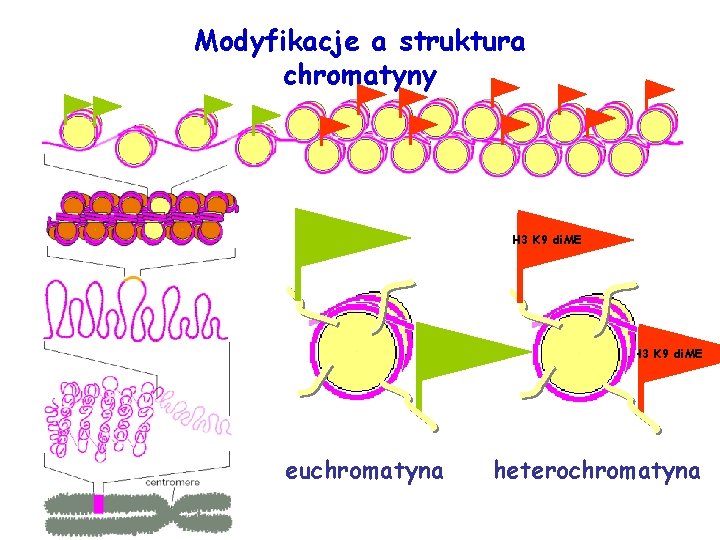
Modyfikacje a struktura chromatyny H 3 K 9 di. ME euchromatyna heterochromatyna

Modyfikacje histonów Biotinylation ADP-ribosylation k 6 k 11 * * * Gräff and Mansuy (2008). Behav Brain Res.

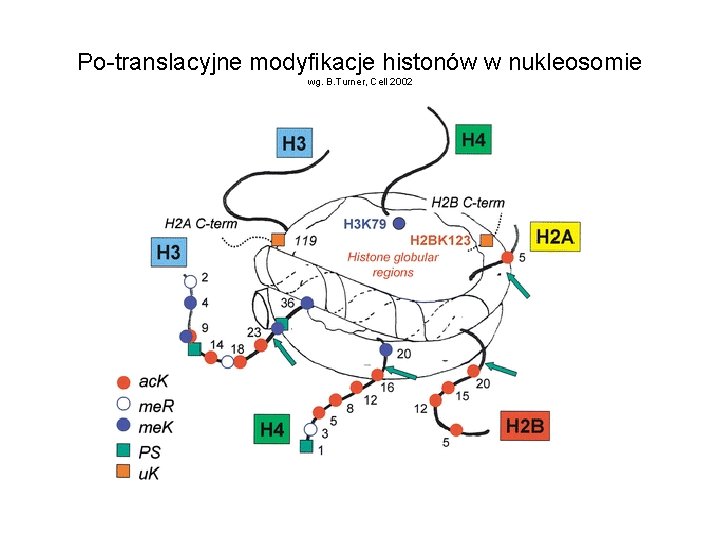
Po-translacyjne modyfikacje histonów w nukleosomie wg. B. Turner, Cell 2002

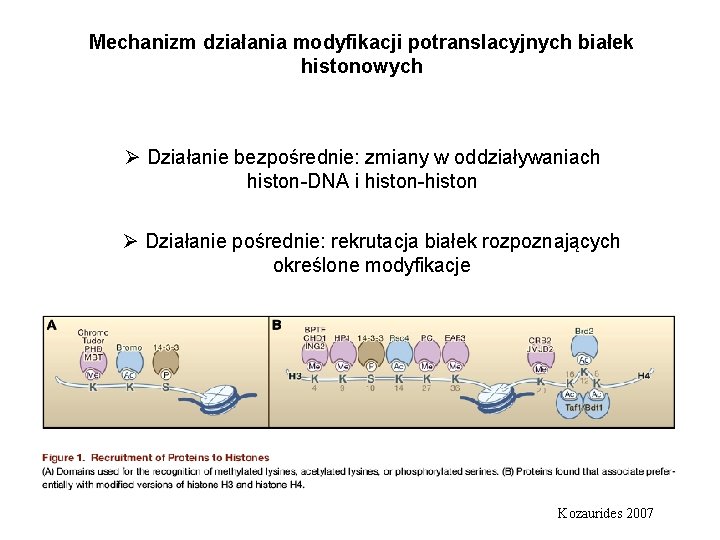
Mechanizm działania modyfikacji potranslacyjnych białek histonowych Ø Działanie bezpośrednie: zmiany w oddziaływaniach histon-DNA i histon-histon Ø Działanie pośrednie: rekrutacja białek rozpoznających określone modyfikacje Kozaurides 2007

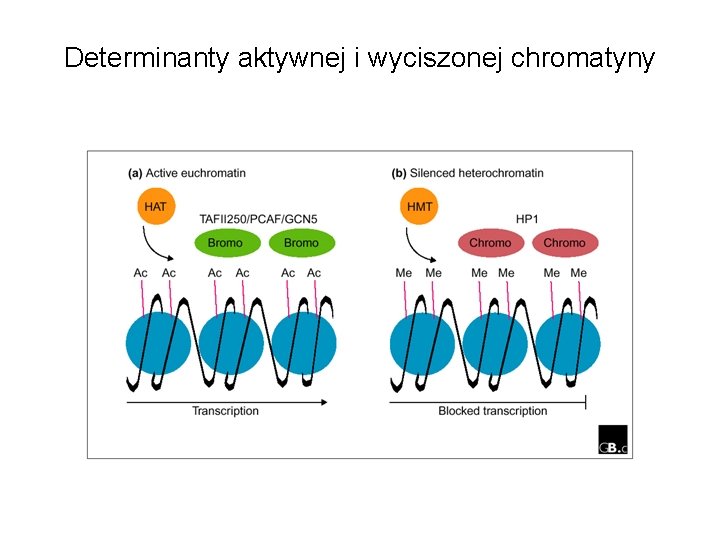
Przykład przeciwstawnych funkcji modyfikacji histonów • H 3/H 4 Ac. Lys/H 3 met. Lys 4(H 3 K 4) związane z rejonami aktywnej transkrypcji H 3/H 4 Lys+/H 3 met. Lys 9 (H 3 K 9) związane z rejonami wygaszonej transkrypcji.

Determinanty aktywnej i wyciszonej chromatyny

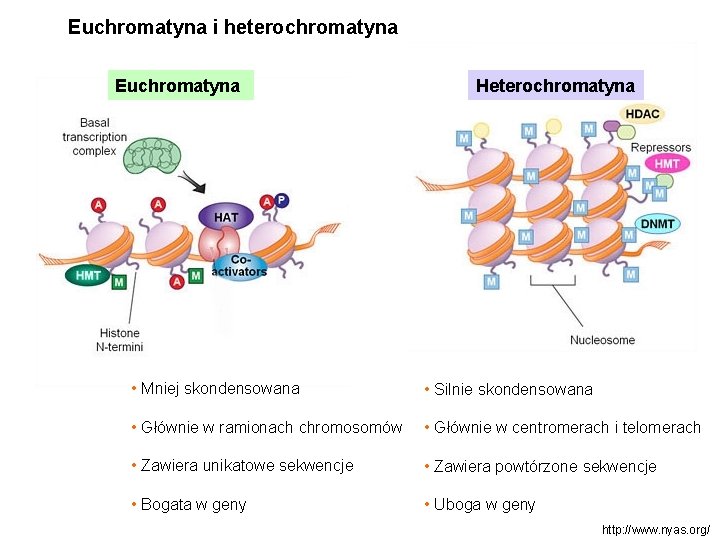
Euchromatyna i heterochromatyna Euchromatyna Heterochromatyna • Mniej skondensowana • Silnie skondensowana • Głównie w ramionach chromosomów • Głównie w centromerach i telomerach • Zawiera unikatowe sekwencje • Zawiera powtórzone sekwencje • Bogata w geny • Uboga w geny http: //www. nyas. org/

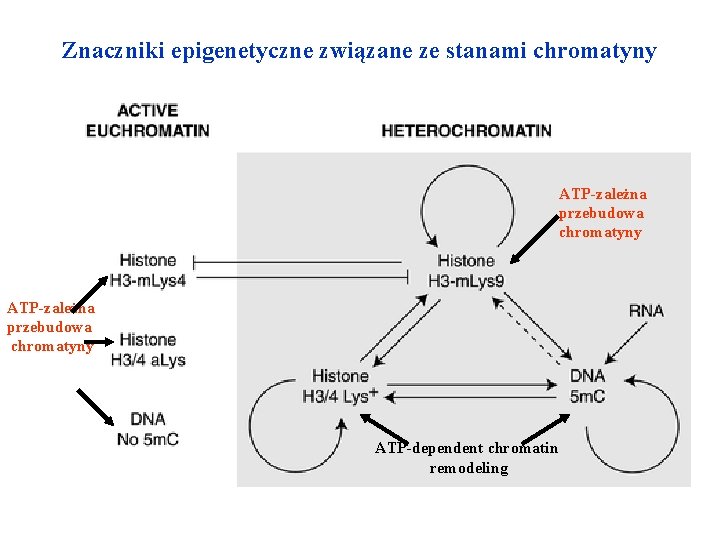
Znaczniki epigenetyczne związane ze stanami chromatyny ATP-zależna przebudowa chromatyny ATP-dependent chromatin remodeling

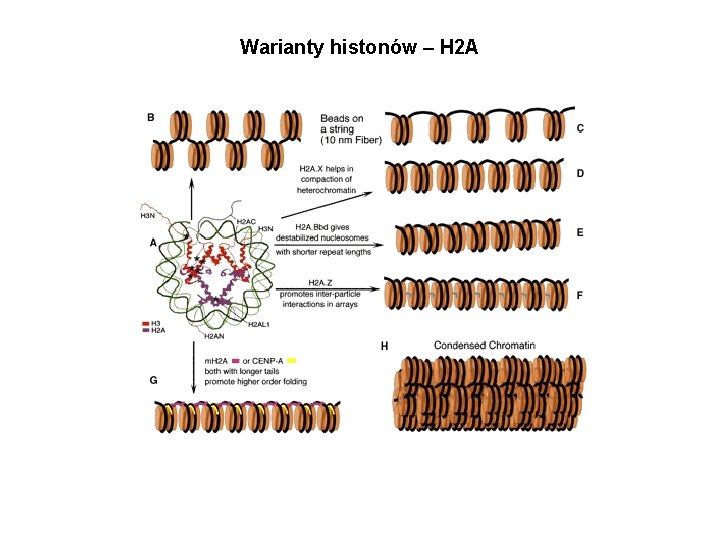
Warianty histonów – H 2 A

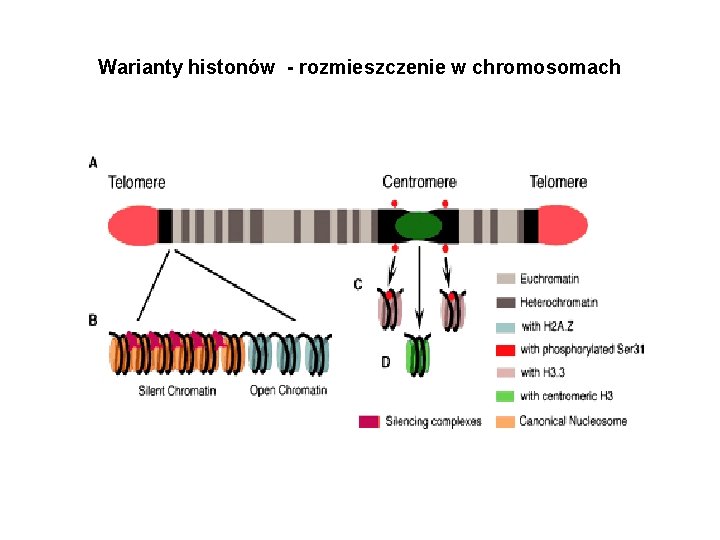
Warianty histonów - rozmieszczenie w chromosomach

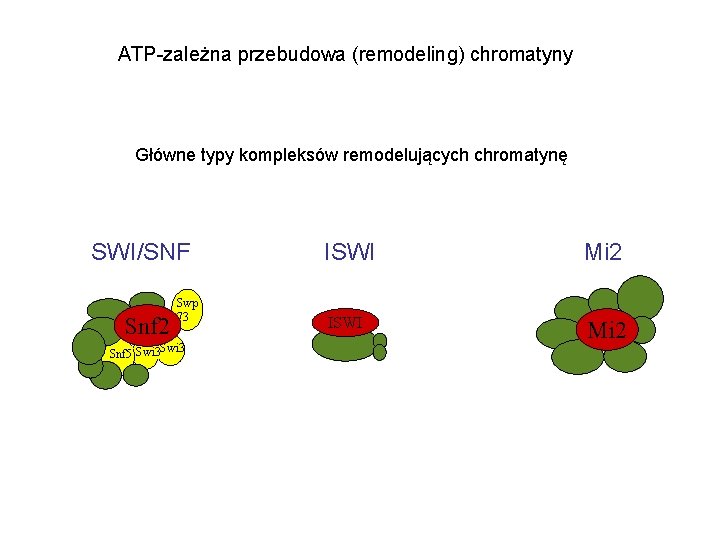
ATP-zależna przebudowa (remodeling) chromatyny Główne typy kompleksów remodelujących chromatynę SWI/SNF Snf 2 Swp 73 Snf 5 Swi 3 ISWI Mi 2

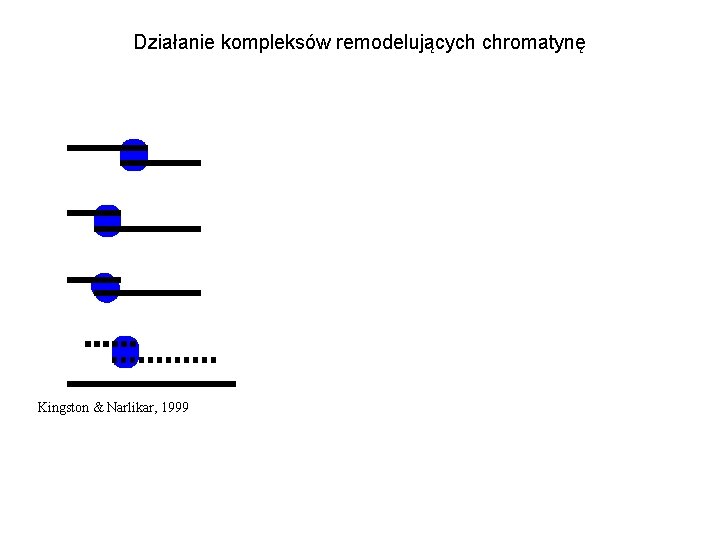
Działanie kompleksów remodelujących chromatynę Kingston & Narlikar, 1999

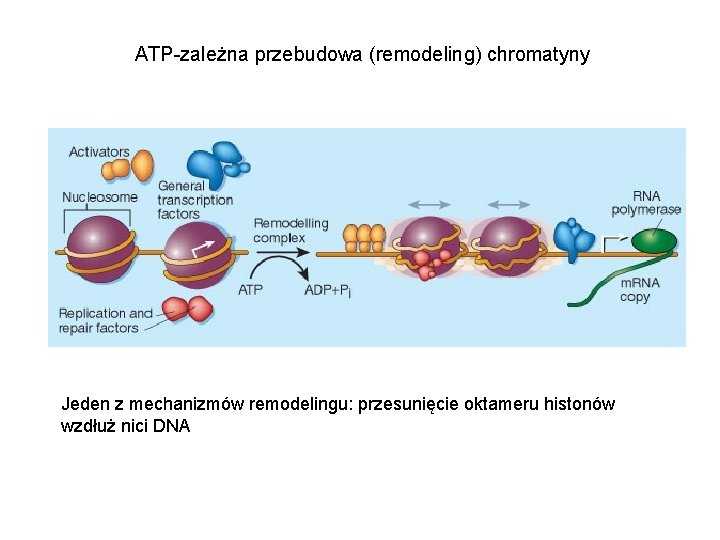
ATP-zależna przebudowa (remodeling) chromatyny Jeden z mechanizmów remodelingu: przesunięcie oktameru histonów wzdłuż nici DNA

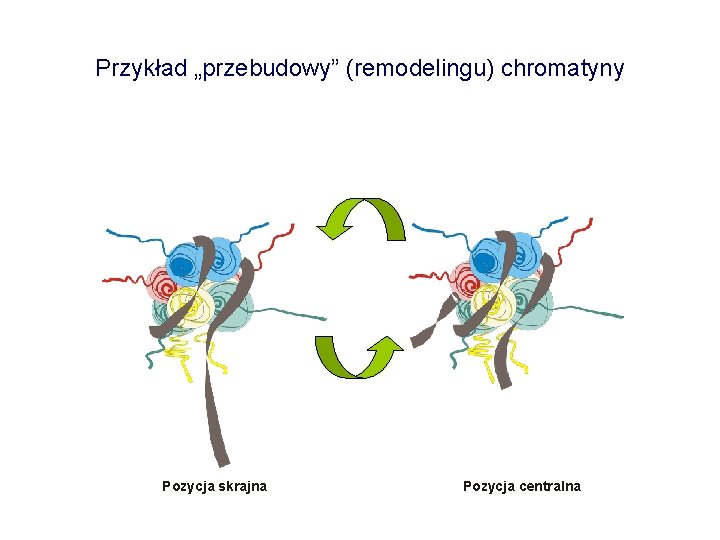
Przykład „przebudowy” (remodelingu) chromatyny Pozycja skrajna Pozycja centralna

Pozycjonowanie nukleosomów

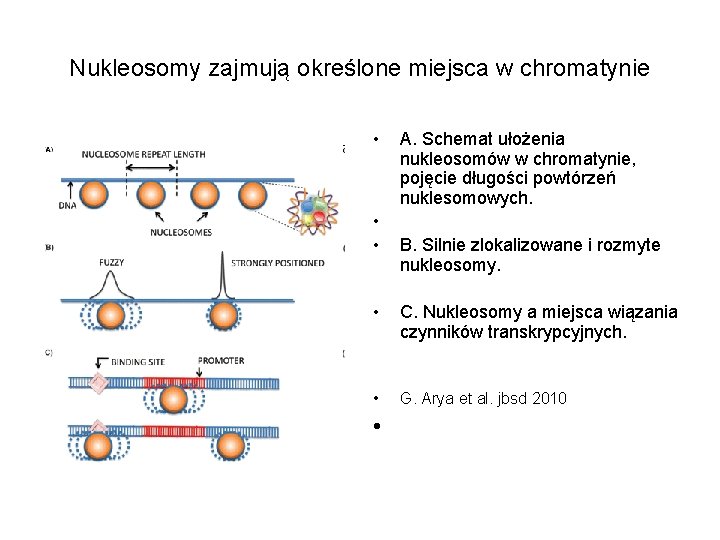
Nukleosomy zajmują określone miejsca w chromatynie • • • A. Schemat ułożenia nukleosomów w chromatynie, pojęcie długości powtórzeń nuklesomowych. B. Silnie zlokalizowane i rozmyte nukleosomy. • C. Nukleosomy a miejsca wiązania czynników transkrypcyjnych. • G. Arya et al. jbsd 2010 •

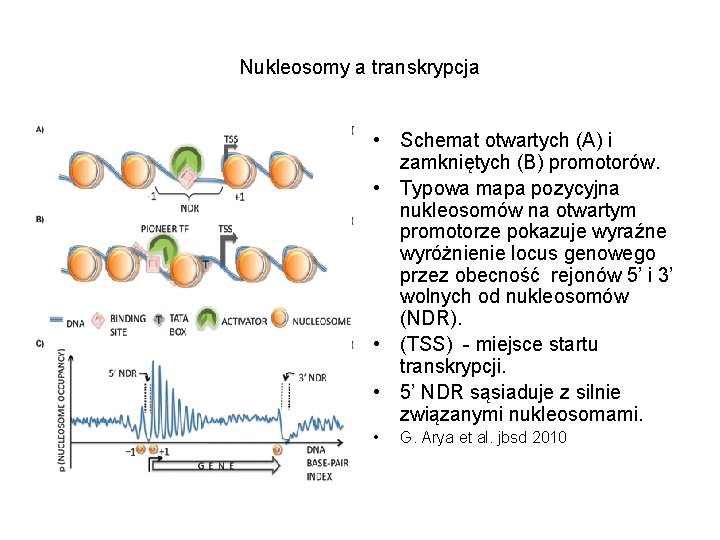
Nukleosomy a transkrypcja • Schemat otwartych (A) i zamkniętych (B) promotorów. • Typowa mapa pozycyjna nukleosomów na otwartym promotorze pokazuje wyraźne wyróżnienie locus genowego przez obecność rejonów 5’ i 3’ wolnych od nukleosomów (NDR). • (TSS) - miejsce startu transkrypcji. • 5’ NDR sąsiaduje z silnie związanymi nukleosomami. • G. Arya et al. jbsd 2010

Dziedziczenie epigenetyczne • Dziedziczne modyfikacje funkcji genów nie związane ze zmianami w sekwencji DNA • Mechanizmy epigenetyczne są związane z modulacją chromatyny i obejmują: modyfikacje histonów, metylację DNA, przebudowę nukleosomów (remodeling), system RNAi.

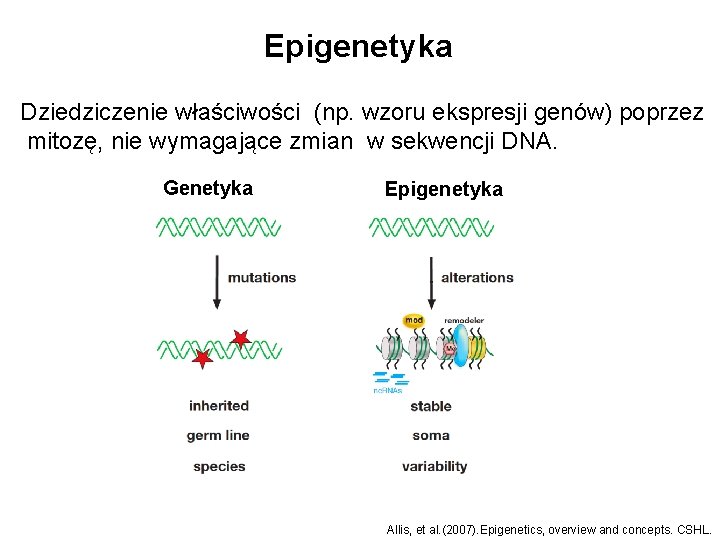
Epigenetyka Dziedziczenie właściwości (np. wzoru ekspresji genów) poprzez mitozę, nie wymagające zmian w sekwencji DNA. Genetyka Epigenetyka Allis, et al. (2007). Epigenetics, overview and concepts. CSHL.

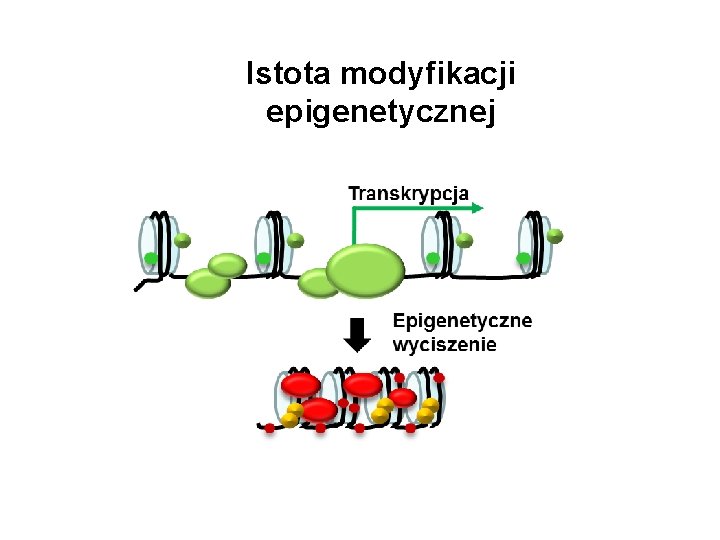
Istota modyfikacji epigenetycznej

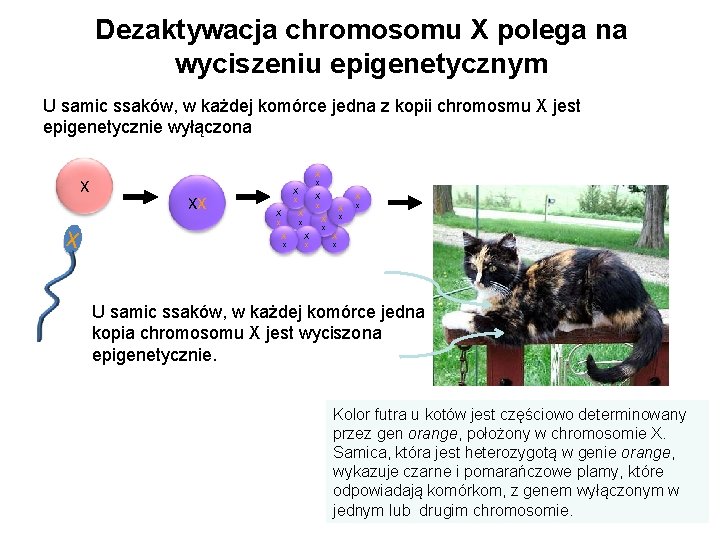
Dezaktywacja chromosomu X polega na wyciszeniu epigenetycznym U samic ssaków, w każdej komórce jedna z kopii chromosmu X jest epigenetycznie wyłączona X XX X X X X X U samic ssaków, w każdej komórce jedna kopia chromosomu X jest wyciszona epigenetycznie. Kolor futra u kotów jest częściowo determinowany przez gen orange, położony w chromosomie X. Samica, która jest heterozygotą w genie orange, wykazuje czarne i pomarańczowe plamy, które odpowiadają komórkom, z genem wyłączonym w jednym lub drugim chromosomie.

Dlaczego sklonowany kot nie jest dokładnie taki, jak oryginał? Rainbow Klon Rainbow

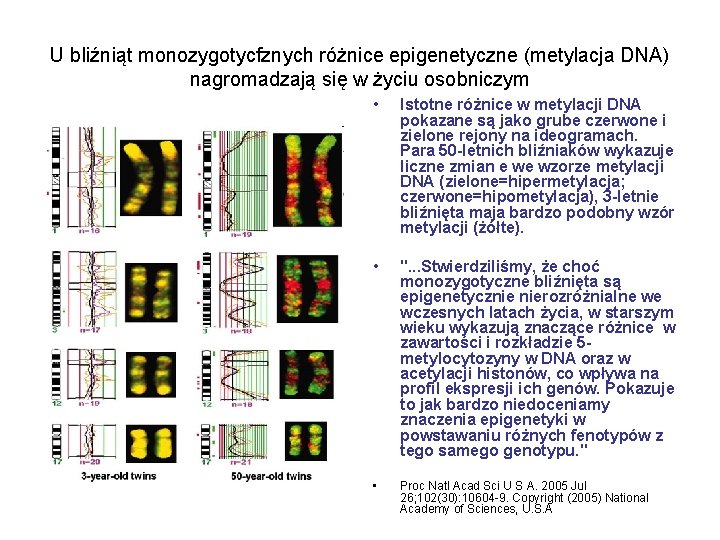
U bliźniąt monozygotycfznych różnice epigenetyczne (metylacja DNA) nagromadzają się w życiu osobniczym • Istotne różnice w metylacji DNA pokazane są jako grube czerwone i zielone rejony na ideogramach. Para 50 -letnich bliźniaków wykazuje liczne zmian e we wzorze metylacji DNA (zielone=hipermetylacja; czerwone=hipometylacja), 3 -letnie bliźnięta maja bardzo podobny wzór metylacji (żółte). • ". . . Stwierdziliśmy, że choć monozygotyczne bliźnięta są epigenetycznie nierozróżnialne we wczesnych latach życia, w starszym wieku wykazują znaczące różnice w zawartości i rozkładzie 5 metylocytozyny w DNA oraz w acetylacji histonów, co wpływa na profil ekspresji ich genów. Pokazuje to jak bardzo niedoceniamy znaczenia epigenetyki w powstawaniu różnych fenotypów z tego samego genotypu. " • Proc Natl Acad Sci U S A. 2005 Jul 26; 102(30): 10604 -9. Copyright (2005) National Academy of Sciences, U. S. A

Fenotyp mutantów w metylotransferazie histonów Mutanty są pozbawione metylacji w H 3 K 36. Dong, et al. (2008). Biochemical and Biophysical Research Communications. 373: 659 -664

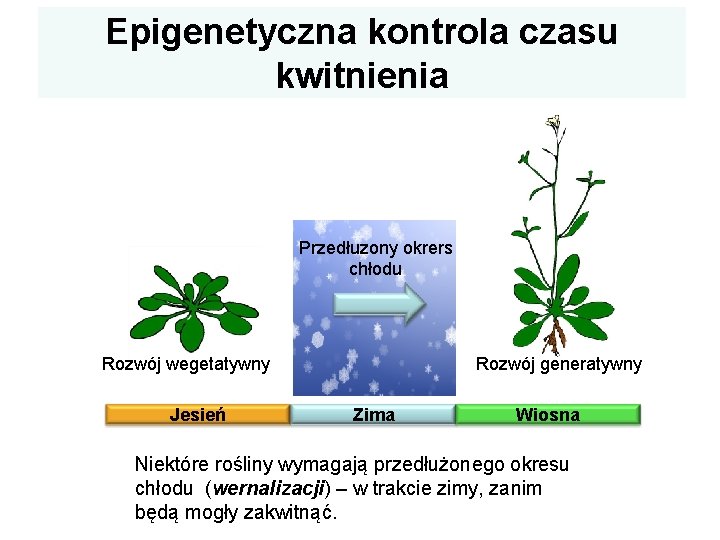
Epigenetyczna kontrola czasu kwitnienia Przedłuzony okrers chłodu Rozwój generatywny Rozwój wegetatywny Jesień Zima Wiosna Niektóre rośliny wymagają przedłużonego okresu chłodu (wernalizacji) – w trakcie zimy, zanim będą mogły zakwitnąć.

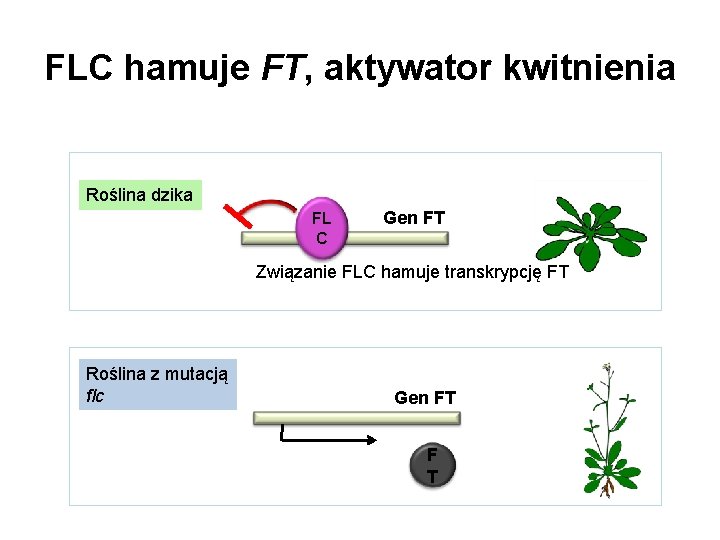
FLC hamuje FT, aktywator kwitnienia Roślina dzika FL C Gen FT Związanie FLC hamuje transkrypcję FT Roślina z mutacją flc Gen FT F T

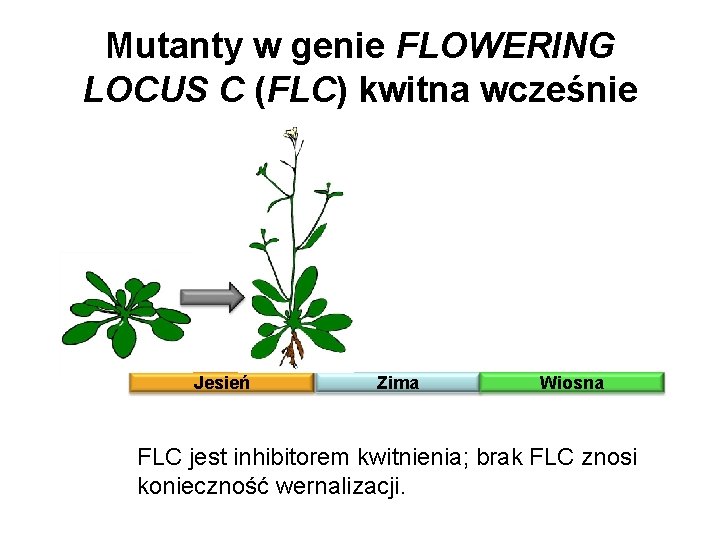
Mutanty w genie FLOWERING LOCUS C (FLC) kwitna wcześnie Jesień Zima Wiosna FLC jest inhibitorem kwitnienia; brak FLC znosi konieczność wernalizacji.

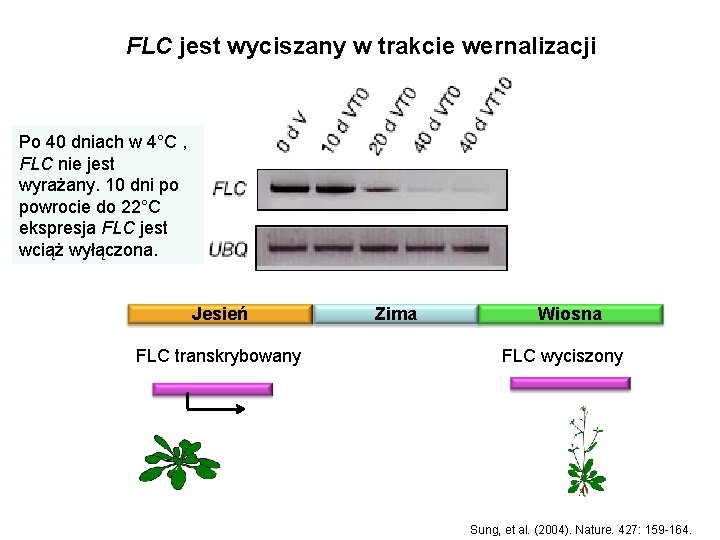
FLC jest wyciszany w trakcie wernalizacji Po 40 dniach w 4°C , FLC nie jest wyrażany. 10 dni po powrocie do 22°C ekspresja FLC jest wciąż wyłączona. Jesień FLC transkrybowany Zima Wiosna FLC wyciszony Sung, et al. (2004). Nature. 427: 159 -164.

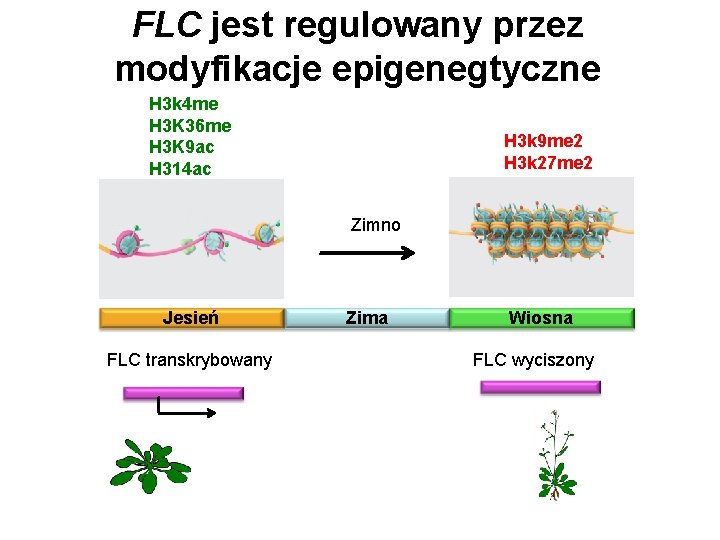
FLC jest regulowany przez modyfikacje epigenegtyczne H 3 k 4 me H 3 K 36 me H 3 K 9 ac H 314 ac H 3 k 9 me 2 H 3 k 27 me 2 Zimno Jesień FLC transkrybowany Zima Wiosna FLC wyciszony

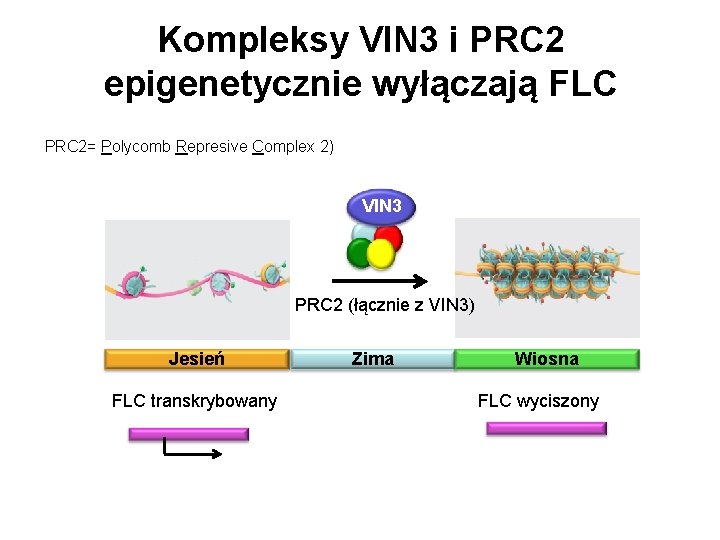
Kompleksy VIN 3 i PRC 2 epigenetycznie wyłączają FLC PRC 2= Polycomb Represive Complex 2) VIN 3 PRC 2 (łącznie z VIN 3) Jesień FLC transkrybowany Zima Wiosna FLC wyciszony

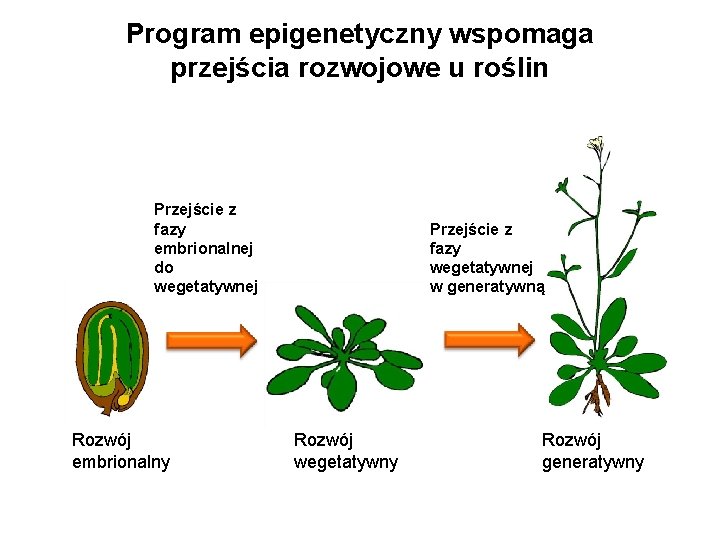
Program epigenetyczny wspomaga przejścia rozwojowe u roślin Przejście z fazy embrionalnej do wegetatywnej Rozwój embrionalny Przejście z fazy wegetatywnej w generatywną Rozwój wegetatywny Rozwój generatywny

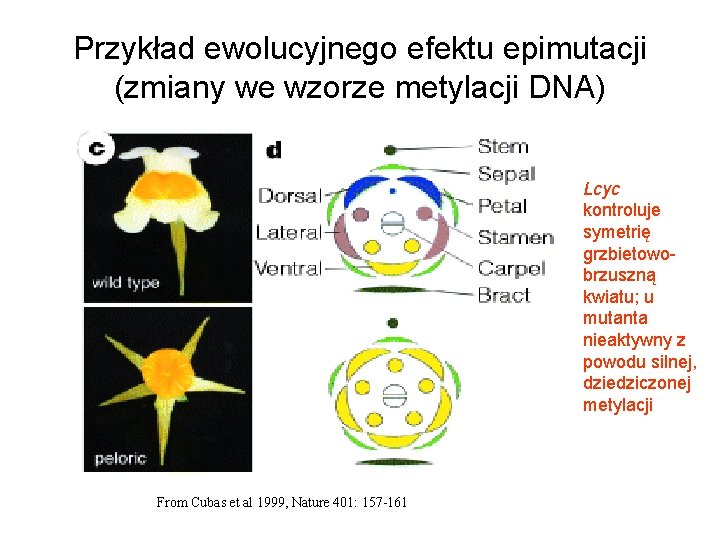
Przykład ewolucyjnego efektu epimutacji (zmiany we wzorze metylacji DNA) Lcyc kontroluje symetrię grzbietowobrzuszną kwiatu; u mutanta nieaktywny z powodu silnej, dziedziczonej metylacji From Cubas et al 1999, Nature 401: 157 -161