Virus Cos un Virus E una particella formata











- Slides: 38

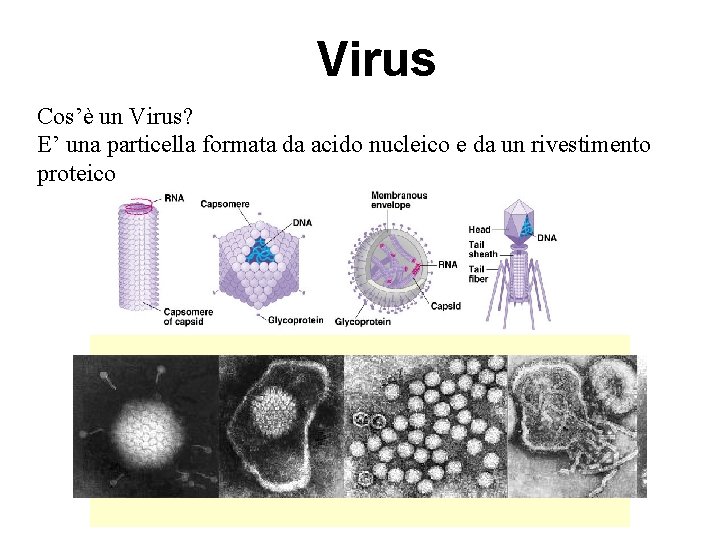
Virus Cos’è un Virus? E’ una particella formata da acido nucleico e da un rivestimento proteico

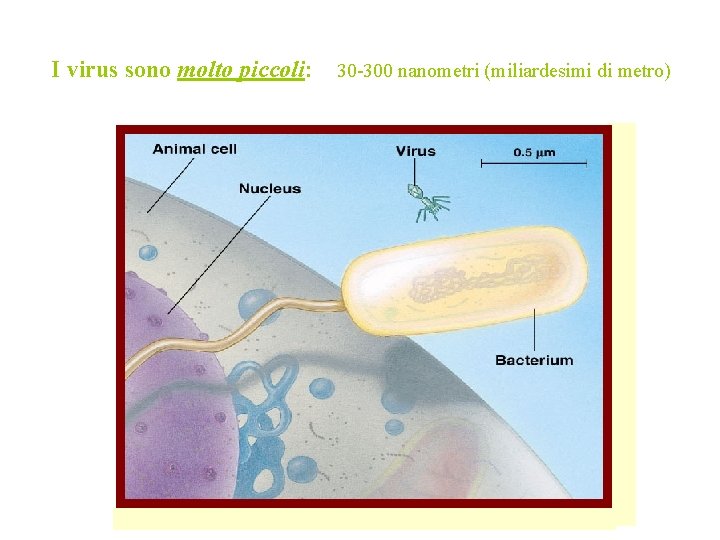
I virus sono molto piccoli: 30 -300 nanometri (miliardesimi di metro)

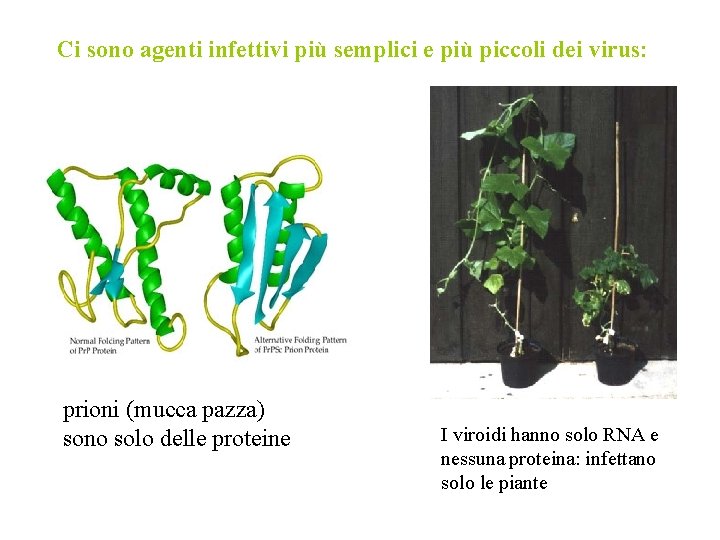
Ci sono agenti infettivi più semplici e più piccoli dei virus: prioni (mucca pazza) sono solo delle proteine I viroidi hanno solo RNA e nessuna proteina: infettano solo le piante

AGENTI INFETTANTI A STRUTTURA SUBCELLULARE VIRUS parassiti endocellulari obbligati formati esclusivamente da acido nucleico (genoma) + involucri proteici. Dimensioni di 20 -300 nm di diametro. Esistono anche strutture infettanti diverse: VIROIDI genoma senza involucri patogeni delle piante formati da piccole molecole di RNA PRIONI proteine infettanti

Struttura dei virus Definizioni • • • Virione: particella virale completa Genoma: DNA o RNA Capside: rivestimento proteico del genoma Nucleocapside: capside + genoma Matrice o tegumento: zona fra il capside e pericapside composta da proteine virali • Pericapside: rivestimento lipidico e glicoproteico esterno in parte di derivazione cellulare. Le glicoproteine sono di origine virale.

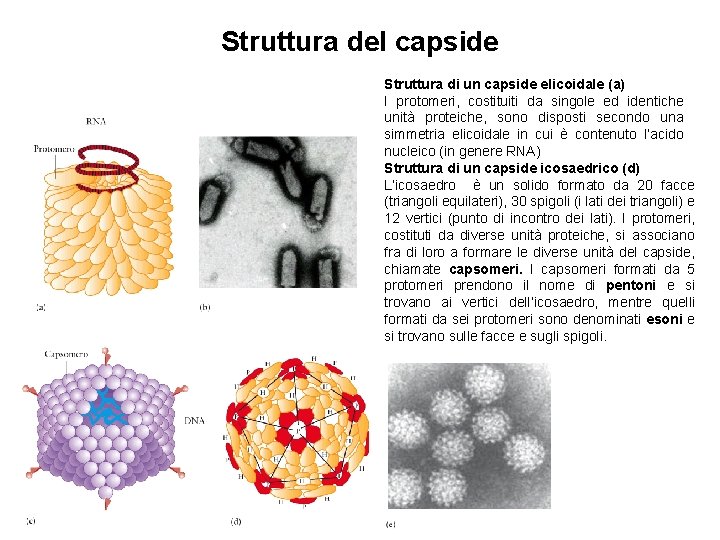
Struttura del capside Struttura di un capside elicoidale (a) I protomeri, costituiti da singole ed identiche unità proteiche, sono disposti secondo una simmetria elicoidale in cui è contenuto l’acido nucleico (in genere RNA) Struttura di un capside icosaedrico (d) L’icosaedro è un solido formato da 20 facce (triangoli equilateri), 30 spigoli (i lati dei triangoli) e 12 vertici (punto di incontro dei lati). I protomeri, costituti da diverse unità proteiche, si associano fra di loro a formare le diverse unità del capside, chiamate capsomeri. I capsomeri formati da 5 protomeri prendono il nome di pentoni e si trovano ai vertici dell’icosaedro, mentre quelli formati da sei protomeri sono denominati esoni e si trovano sulle facce e sugli spigoli.

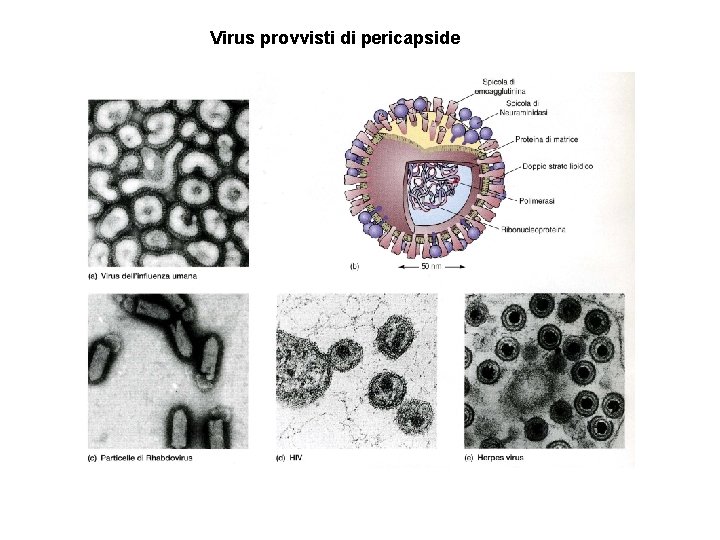
Virus provvisti di pericapside

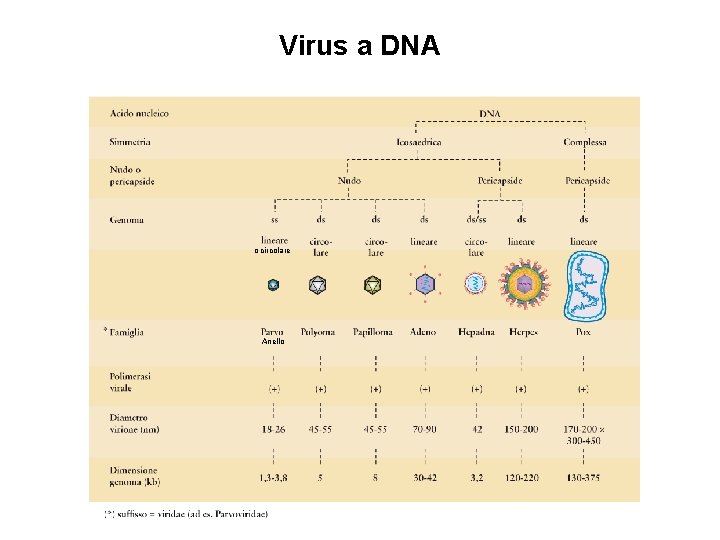
ACIDO NUCLEICO VIRALE Deossiribovirus esistono virus a DNA lineare a doppia elica (es. herpesvirus), a DNA circolare a doppia elica (es. papillomavirus), a DNA circolare con tratti a singola e a doppia elica (es. hepadnavirus), a DNA a singola elica (es. parvovirus) Ribovirus esistono virus a RNA monocatenario (es. paramyxovirus), a RNA segmentato (es. orthomyxovirus), a RNA bicatenario (es. reovirus). Virus a polarità positiva (RNA+) il genoma virale funziona direttamente da messaggero (m. RNA). Virus a polarità negativa (RNA-) il genoma virale serve da stampo per lo m. RNA.

CLASSIFICAZIONE Le principali famiglie di virus di interesse medico si possono classificare secondo i seguenti parametri principali: • • tipo di acido nucleico (DNA o RNA) nei virus a RNA la presenza di genoma a polarità positiva o negativa struttura del genoma (singola o doppia elica, lineare o circolare, segmentata o non segmentata) presenza o assenza di involucro (virus con o senza pericapside ) Inoltre si possono considerare caratteristiche morfologiche (es: struttura e dimensioni) • caratteristiche biologiche (es. ospite infettato, modo di trasmissione, tessuti bersaglio e sede anatomica) caratteristiche genomiche (es. strategia replicativa, sequenza nucleotidica)

Classificazione dei virus • • • • Famiglia (suffisso –viridae) sottofamiglia (suffisso-virinae) genere (suffisso-virus) specie Es: Famiglia Herpesviridae Sottofamiglia Alphaherpesvirinae (alphaerpesvirus) Genere Herpes Simplex virus Specie: herpesvirus umano 1 (o virus herpes simplex 1) Famiglia Retroviridae Genere Lentivirus Specie: virus dell’immunodeficienza umana Nella pratica medica si continuano ad indicare i virus con il loro nome comune (virus dell’influenza, del morbillo, virus Ebola, ecc) All’interno della stessa specie si possono identificare, sulla base della sequenza nucleotidica, ulteriori sottogruppi: genotipi, sottotipi, cladi, varianti

Virus a RNA

Virus a DNA o circolare Anello

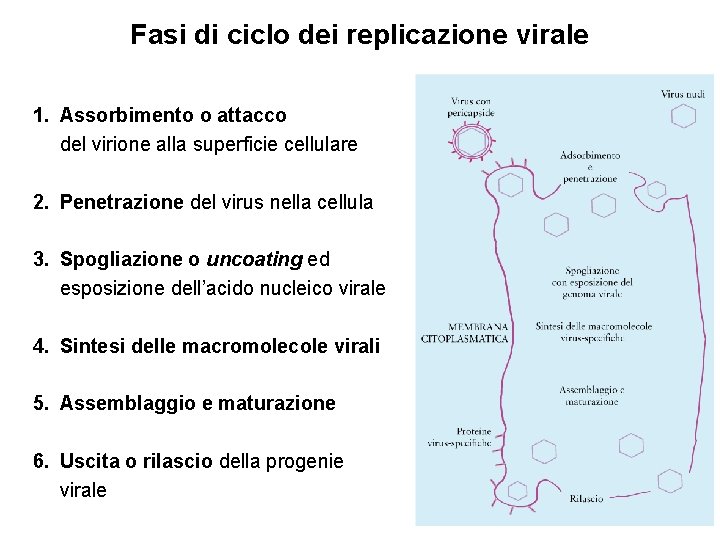
Fasi di ciclo dei replicazione virale 1. Assorbimento o attacco del virione alla superficie cellulare 2. Penetrazione del virus nella cellula 3. Spogliazione o uncoating ed esposizione dell’acido nucleico virale 4. Sintesi delle macromolecole virali 5. Assemblaggio e maturazione 6. Uscita o rilascio della progenie virale

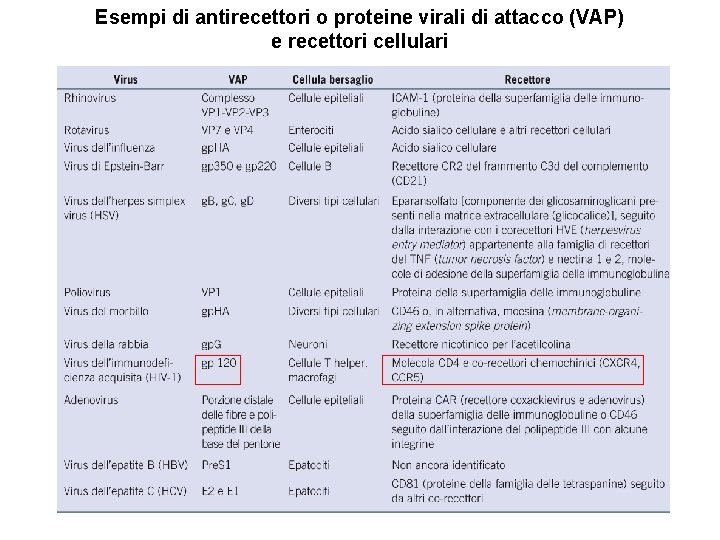
ASSORBIMENTO o ATTACCO • • Richiede l’interazione specifica fra strutture virali (antirecettori o proteine virali di attacco-VAP) e cellulari (recettori) posti sulla membrana plasmatica. Si conoscono diversi antirecettori e recettori Le caratteristiche del recettore determinano spettro d’ospite, tropismo cellulare e patogenesi. Ad esempio, (1) un recettore presente solo sui linfociti determina una patologia specifica emato-immunologica e una trasmissione ematica, (2) un recettore comune su cellule di diversi tessuti determina patologie variabili (polmoniti, encefaliti, enteriti, ecc) ed elevata trasmissibilità. I virus complessi possono avere più di un antirecettore (es. Herpesvirus) L’adsorbimento può essere facilitato dalla presenza di

Esempi di antirecettori o proteine virali di attacco (VAP) e recettori cellulari

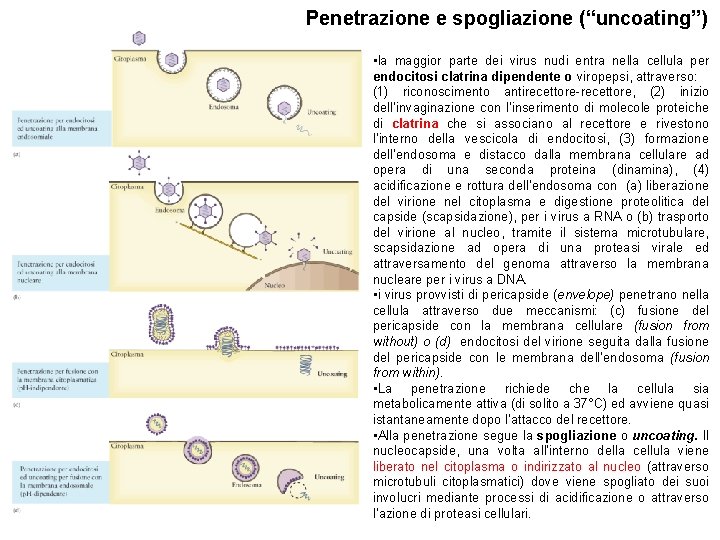
Penetrazione e spogliazione (“uncoating”) • la maggior parte dei virus nudi entra nella cellula per endocitosi clatrina dipendente o viropepsi, attraverso: (1) riconoscimento antirecettore-recettore, (2) inizio dell’invaginazione con l’inserimento di molecole proteiche di clatrina che si associano al recettore e rivestono l’interno della vescicola di endocitosi, (3) formazione dell’endosoma e distacco dalla membrana cellulare ad opera di una seconda proteina (dinamina), (4) acidificazione e rottura dell’endosoma con (a) liberazione del virione nel citoplasma e digestione proteolitica del capside (scapsidazione), per i virus a RNA o (b) trasporto del virione al nucleo, tramite il sistema microtubulare, scapsidazione ad opera di una proteasi virale ed attraversamento del genoma attraverso la membrana nucleare per i virus a DNA. • i virus provvisti di pericapside (envelope) penetrano nella cellula attraverso due meccanismi: (c) fusione del pericapside con la membrana cellulare (fusion from without) o (d) endocitosi del virione seguita dalla fusione del pericapside con le membrana dell’endosoma (fusion from within). • La penetrazione richiede che la cellula sia metabolicamente attiva (di solito a 37°C) ed avviene quasi istantaneamente dopo l’attacco del recettore. • Alla penetrazione segue la spogliazione o uncoating. Il nucleocapside, una volta all’interno della cellula viene liberato nel citoplasma o indirizzato al nucleo (attraverso microtubuli citoplasmatici) dove viene spogliato dei suoi involucri mediante processi di acidificazione o attraverso l’azione di proteasi cellulari.

SINTESI DELLE COMPONENTI VIRALI Il virus deve: • • • sintetizzare i propri RNA messaggeri utilizzare il metabolismo cellulare per sintetizzare le proteine virali replicare il proprio acido nucleico Generalizzando, le proteine virali si possono dividere in: • • precoci sintetizzate dopo la spogliazione, sono soprattutto di natura enzimatica e regolatoria, e intervengono nella replicazione dell’acido nucleico (polimerasi, ecc. ). Alcuni virus possiedono un’ulteriore suddivisione in proteine precocissime e precoci (es. herpesvirus). tardive sintetizzate dopo la replicazione dell’acido nucleico, sono soprattutto strutturali

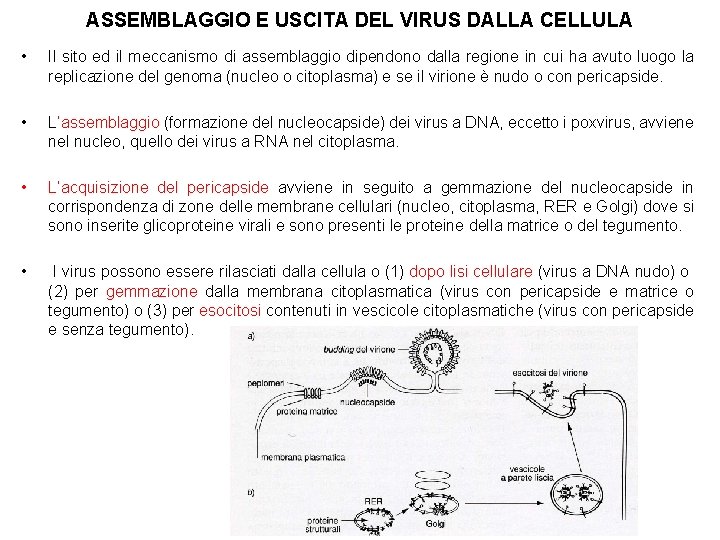
ASSEMBLAGGIO E USCITA DEL VIRUS DALLA CELLULA • Il sito ed il meccanismo di assemblaggio dipendono dalla regione in cui ha avuto luogo la replicazione del genoma (nucleo o citoplasma) e se il virione è nudo o con pericapside. • L’assemblaggio (formazione del nucleocapside) dei virus a DNA, eccetto i poxvirus, avviene nel nucleo, quello dei virus a RNA nel citoplasma. • L’acquisizione del pericapside avviene in seguito a gemmazione del nucleocapside in corrispondenza di zone delle membrane cellulari (nucleo, citoplasma, RER e Golgi) dove si sono inserite glicoproteine virali e sono presenti le proteine della matrice o del tegumento. • I virus possono essere rilasciati dalla cellula o (1) dopo lisi cellulare (virus a DNA nudo) o (2) per gemmazione dalla membrana citoplasmatica (virus con pericapside e matrice o tegumento) o (3) per esocitosi contenuti in vescicole citoplasmatiche (virus con pericapside e senza tegumento).

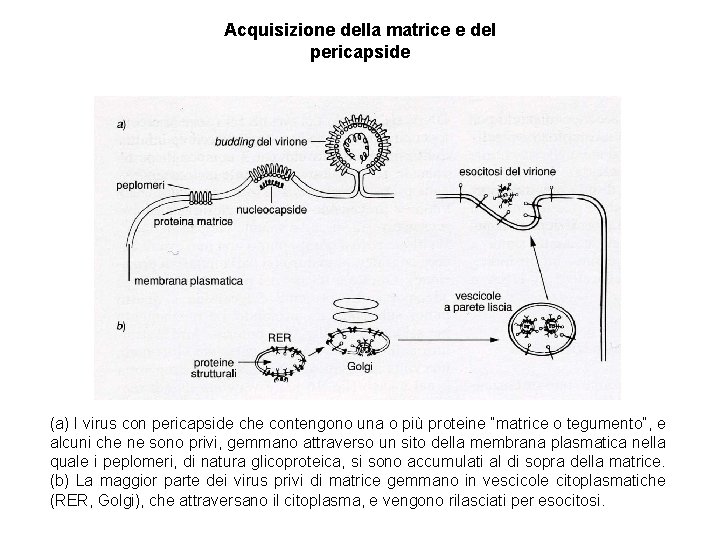
Acquisizione della matrice e del pericapside (a) I virus con pericapside che contengono una o più proteine “matrice o tegumento”, e alcuni che ne sono privi, gemmano attraverso un sito della membrana plasmatica nella quale i peplomeri, di natura glicoproteica, si sono accumulati al di sopra della matrice. (b) La maggior parte dei virus privi di matrice gemmano in vescicole citoplasmatiche (RER, Golgi), che attraversano il citoplasma, e vengono rilasciati per esocitosi.

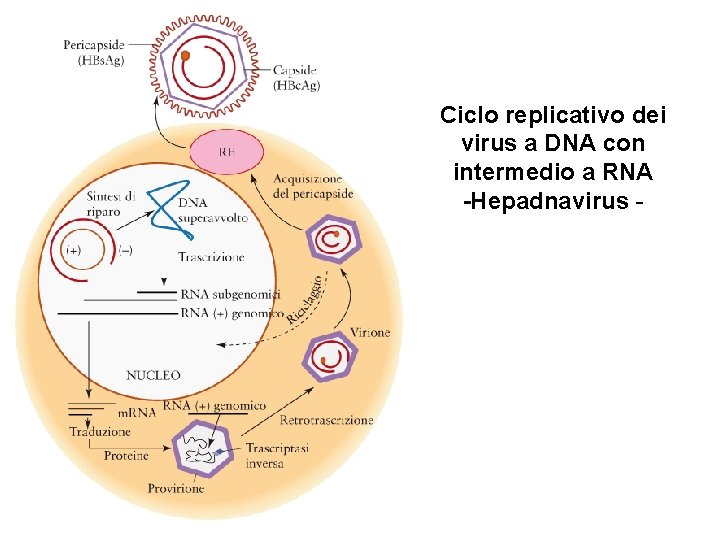
CLASSI REPLICATIVE Ogni specie virale ha strategie replicative diverse, in funzione della natura del materiale genetico e della sede di replicazione. • Si possono distinguere 7 classi replicative con sottoclassi: • • Replicazione Genoma Esempio Classe I DNA a doppia elica Herpesvirus Classe II DNA a singola elica Parvovirus Classe III RNA a doppia elica Reovirus Classe IV RNA+ a singola elica Picornavirus Classe V RNA- a singola elica Orthomyxovirus Classe VI RNA+ con intermedio a DNA Retrovirus Classe VII DNA con intermedio a RNA Epatite B

Classe I DNA a doppia elica. Questi virus hanno bisogno della RNA polimerasi cellulare e, per replicarsi, devono dirigersi al nucleo. Possono essere suddivisi in due sottoclassi che comprendono (a) i virus a replicazione esclusivamente nucleare e (b) i virus che si replicano principalmente nel citoplasma. La figu è un schema esemplificativo di replicazione dei virus della sottoclasse (a), che è costituita da tutti i virus a doppia elica di DNA (Papillomaviridae, Polyomaviridae, Adenoviridae, Herpesviridae, il cui DNA all’interno del nucleo viene trascritto dalla RNA polimerasi cellulare in una serie di m. RNA. I geni possono essere trascritti da ambedue i filamenti di DNA ed in opposte direzioni, possono contenere introni e richiedere processi di splicing da parte dell’apparato nucleare della cellula. La replicazione inizia da una sequenza del DNA genomico detta origine (ori) e il ciclo replicativo può avvenire in due tempi (es. Papillomaviridae, Poliomaviridae) con formazione di due gruppi di m. RNA (precoci e tardivi) che portano, rispettivamente, alla produzione di proteine ad attività enzimatica/regolatoria e a componenti strutturali del virione. Alternativamente il ciclo replicativo può richiedere tre fasi come ad esempio per la famiglia degli herpesvirus in cui sono dimostrabili tre coordinate sequenze di produzione di RNA messaggeri e di proteine, denominati α, β e γ o precocissimi, precoci e tardivi Le proteine α promuovono la trascrizione dei geni precoci. Le proteine β comprendono principalmente enzimi, fra cui la DNA polimerasi-DNA dipendente. La replicazione del genoma inizia non appena viene sintetizzata la DNA polimerasi e porta alla trascrizione dei geni tardivi che codificano proteine strutturali. Queste ultime, dopo essere state trasportate nel nucleo, sono assemblate in pro-capsidi vuoti all’interno dei quali è successivamente inserito il DNA virale neosintetizzato. All’interno del gruppo della classe Ia esistono alcune differenze, nonostante la strategia replicativa tra i virus sia abbastanza simile. Ad esempio, gli Herpesvirus, oltre alla DNA polimerasi codificano anche altri fattori virali, come la ribonucleotide sintetasi e la timidino chinasi, che favoriscono la replicazione del loro genoma, mentre gli Adenovirus codificano una DNA polimerasi la cui attività dipende da una serie di fattori cellulari, e i Polyomavirus e i Papillomavirus utilizzano la DNA polimerasi cellulare. N. B. Gli herpesvirus hanno due cicli biologici, uno di replicazione e uno di latenza (vedi parte speciale Herpesviridae). I papillomavirus hanno un ciclo di replicazione che dipende dalla differenziazione cellulare nei diversi strati dell’epidermide (vedi parte speciale) La sottoclasse b (Ib) è rappresentata dai Poxvirus che sono fra i virus animali di maggiori dimensioni. La loro replicazione avviene principalmente nel citoplasma in quanto nel loro virione è presente una RNA-polimerasi-DNAdipendente che avvia la trascrizione iniziale con la produzione di proteine precoci, tra cui una che consente la spogliazione del genoma e l’avvio delle tappe successive della replicazione, svolte da enzimi codificati dal virus. Si ipotizza che, comunque, vi sia un coinvolgimento del nucleo, dato che il virus non completa il processo replicativo in cellule enucleate; sembra, infatti, che alcuni messaggeri tardivi siano trascritti a livello nucleare.

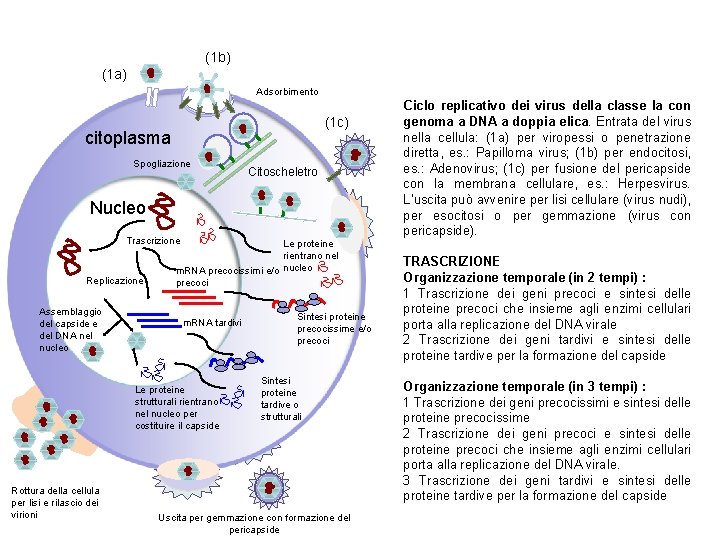
(1 b) (1 a) Adsorbimento (1 c) citoplasma Spogliazione Citoscheletro Nucleo Trascrizione Replicazione Assemblaggio del capside e del DNA nel nucleo Le proteine rientrano nel m. RNA precocissimi e/o nucleo precoci v m. RNA tardivi Le proteine strutturali rientrano nel nucleo per costituire il capside Rottura della cellula per lisi e rilascio dei virioni Sintesi proteine precocissime e/o precoci Sintesi proteine tardive o strutturali Uscita per gemmazione con formazione del pericapside Ciclo replicativo dei virus della classe Ia con genoma a DNA a doppia elica. Entrata del virus nella cellula: (1 a) per viropessi o penetrazione diretta, es. : Papilloma virus; (1 b) per endocitosi, es. : Adenovirus; (1 c) per fusione del pericapside con la membrana cellulare, es. : Herpesvirus. L’uscita può avvenire per lisi cellulare (virus nudi), per esocitosi o per gemmazione (virus con pericapside). TRASCRIZIONE Organizzazione temporale (in 2 tempi) : 1 Trascrizione dei geni precoci e sintesi delle proteine precoci che insieme agli enzimi cellulari porta alla replicazione del DNA virale 2 Trascrizione dei geni tardivi e sintesi delle proteine tardive per la formazione del capside Organizzazione temporale (in 3 tempi) : 1 Trascrizione dei geni precocissimi e sintesi delle proteine precocissime 2 Trascrizione dei geni precoci e sintesi delle proteine precoci che insieme agli enzimi cellulari porta alla replicazione del DNA virale. 3 Trascrizione dei geni tardivi e sintesi delle proteine tardive per la formazione del capside

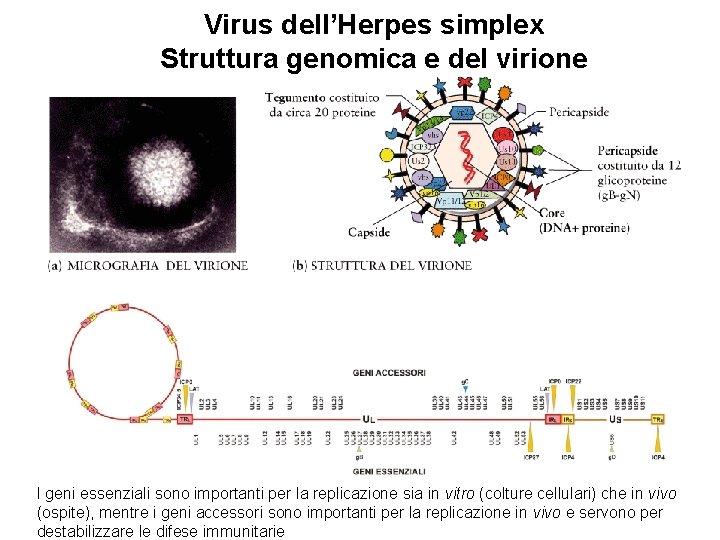
Virus dell’Herpes simplex Struttura genomica e del virione I geni essenziali sono importanti per la replicazione sia in vitro (colture cellulari) che in vivo (ospite), mentre i geni accessori sono importanti per la replicazione in vivo e servono per destabilizzare le difese immunitarie

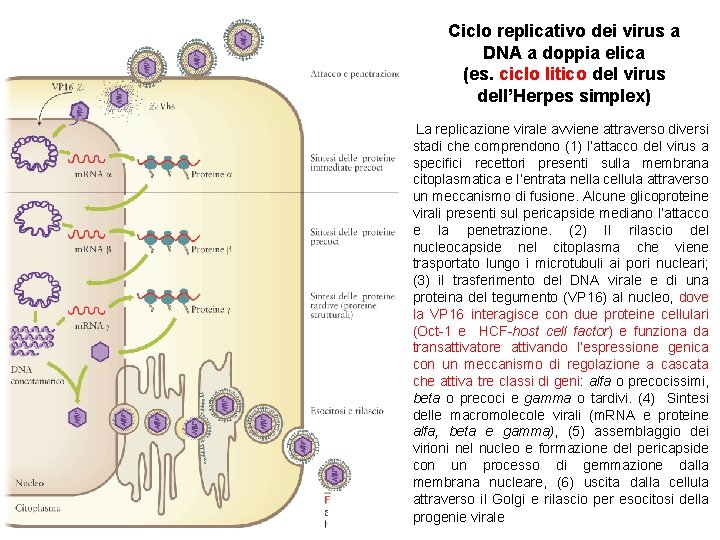
Ciclo replicativo dei virus a DNA a doppia elica (es. ciclo litico del virus dell’Herpes simplex) La replicazione virale avviene attraverso diversi stadi che comprendono (1) l’attacco del virus a specifici recettori presenti sulla membrana citoplasmatica e l’entrata nella cellula attraverso un meccanismo di fusione. Alcune glicoproteine virali presenti sul pericapside mediano l’attacco e la penetrazione. (2) Il rilascio del nucleocapside nel citoplasma che viene trasportato lungo i microtubuli ai pori nucleari; (3) il trasferimento del DNA virale e di una proteina del tegumento (VP 16) al nucleo, dove la VP 16 interagisce con due proteine cellulari (Oct-1 e HCF-host cell factor) e funziona da transattivatore attivando l’espressione genica con un meccanismo di regolazione a cascata che attiva tre classi di geni: alfa o precocissimi, beta o precoci e gamma o tardivi. (4) Sintesi delle macromolecole virali (m. RNA e proteine alfa, beta e gamma), (5) assemblaggio dei virioni nel nucleo e formazione del pericapside con un processo di gemmazione dalla membrana nucleare, (6) uscita dalla cellula attraverso il Golgi e rilascio per esocitosi della progenie virale

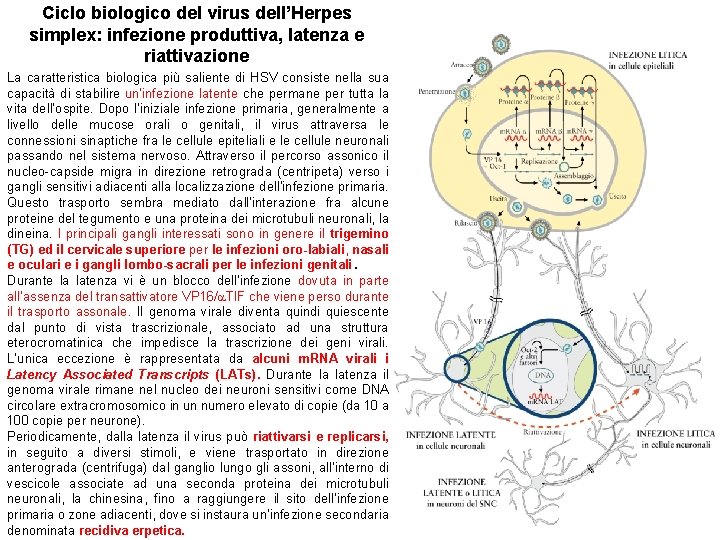
Ciclo biologico del virus dell’Herpes simplex: infezione produttiva, latenza e riattivazione La caratteristica biologica più saliente di HSV consiste nella sua capacità di stabilire un’infezione latente che permane per tutta la vita dell’ospite. Dopo l’iniziale infezione primaria, generalmente a livello delle mucose orali o genitali, il virus attraversa le connessioni sinaptiche fra le cellule epiteliali e le cellule neuronali passando nel sistema nervoso. Attraverso il percorso assonico il nucleo-capside migra in direzione retrograda (centripeta) verso i gangli sensitivi adiacenti alla localizzazione dell’infezione primaria. Questo trasporto sembra mediato dall’interazione fra alcune proteine del tegumento e una proteina dei microtubuli neuronali, la dineina. I principali gangli interessati sono in genere il trigemino (TG) ed il cervicale superiore per le infezioni oro-labiali, nasali e oculari e i gangli lombo-sacrali per le infezioni genitali. Durante la latenza vi è un blocco dell’infezione dovuta in parte all’assenza del transattivatore VP 16/ TIF che viene perso durante il trasporto assonale. Il genoma virale diventa quindi quiescente dal punto di vista trascrizionale, associato ad una struttura eterocromatinica che impedisce la trascrizione dei geni virali. L’unica eccezione è rappresentata da alcuni m. RNA virali i Latency Associated Transcripts (LATs). Durante la latenza il genoma virale rimane nel nucleo dei neuroni sensitivi come DNA circolare extracromosomico in un numero elevato di copie (da 100 copie per neurone). Periodicamente, dalla latenza il virus può riattivarsi e replicarsi, in seguito a diversi stimoli, e viene trasportato in direzione anterograda (centrifuga) dal ganglio lungo gli assoni, all’interno di vescicole associate ad una seconda proteina dei microtubuli neuronali, la chinesina, fino a raggiungere il sito dell’infezione primaria o zone adiacenti, dove si instaura un’infezione secondaria denominata recidiva erpetica.

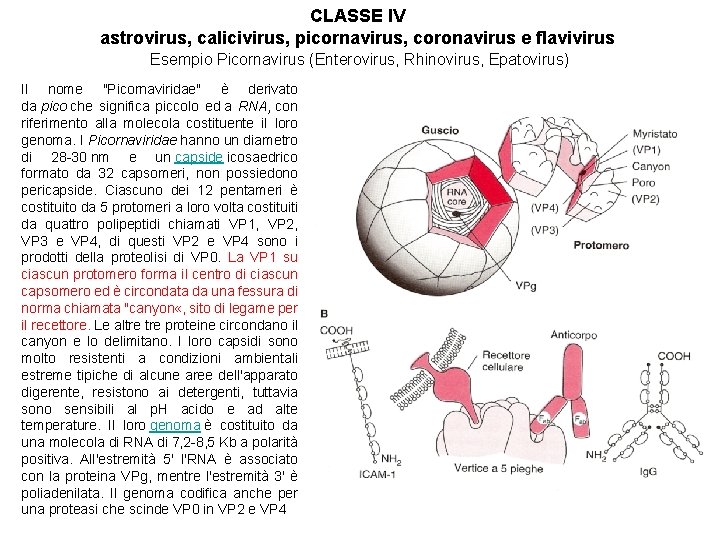
CLASSE IV astrovirus, calicivirus, picornavirus, coronavirus e flavivirus Esempio Picornavirus (Enterovirus, Rhinovirus, Epatovirus) Il nome "Picornaviridae" è derivato da pico che significa piccolo ed a RNA, con riferimento alla molecola costituente il loro genoma. I Picornaviridae hanno un diametro di 28 -30 nm e un capside icosaedrico formato da 32 capsomeri, non possiedono pericapside. Ciascuno dei 12 pentameri è costituito da 5 protomeri a loro volta costituiti da quattro polipeptidi chiamati VP 1, VP 2, VP 3 e VP 4, di questi VP 2 e VP 4 sono i prodotti della proteolisi di VP 0. La VP 1 su ciascun protomero forma il centro di ciascun capsomero ed è circondata da una fessura di norma chiamata "canyon «, sito di legame per il recettore. Le altre proteine circondano il canyon e lo delimitano. I loro capsidi sono molto resistenti a condizioni ambientali estreme tipiche di alcune aree dell'apparato digerente, resistono ai detergenti, tuttavia sono sensibili al p. H acido e ad alte temperature. Il loro genoma è costituito da una molecola di RNA di 7, 2 -8, 5 Kb a polarità positiva. All'estremità 5' l'RNA è associato con la proteina VPg, mentre l'estremità 3' è poliadenilata. Il genoma codifica anche per una proteasi che scinde VP 0 in VP 2 e VP 4

Ciclo replicativo dei Picornavirus Il ciclo replicativo ha inizio con l’interazione antirecettore-recettore e dura circa 3 -4 ore. Molti picornavirus entrano nella cellula per endocitosi mediata da recettore e la scapsidazione è favorita dall’acidità dell’endosoma, in altri avviene per traslocazione diretta dell’RNA genomico attraverso la membrana Porzione peptidica con attività proteasica

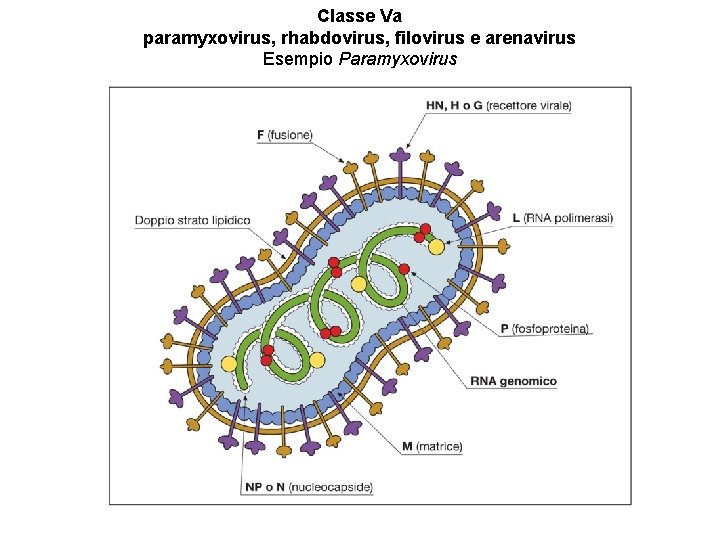
Classe Va paramyxovirus, rhabdovirus, filovirus e arenavirus Esempio Paramyxovirus

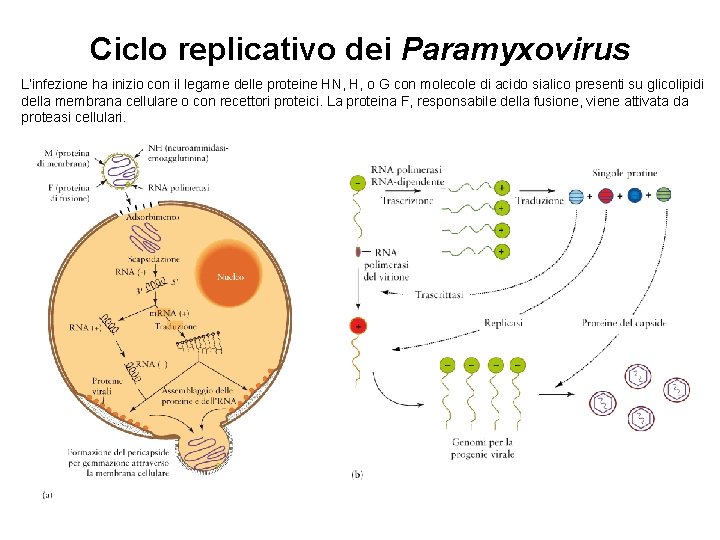
Ciclo replicativo dei Paramyxovirus L’infezione ha inizio con il legame delle proteine HN, H, o G con molecole di acido sialico presenti su glicolipidi della membrana cellulare o con recettori proteici. La proteina F, responsabile della fusione, viene attivata da proteasi cellulari.

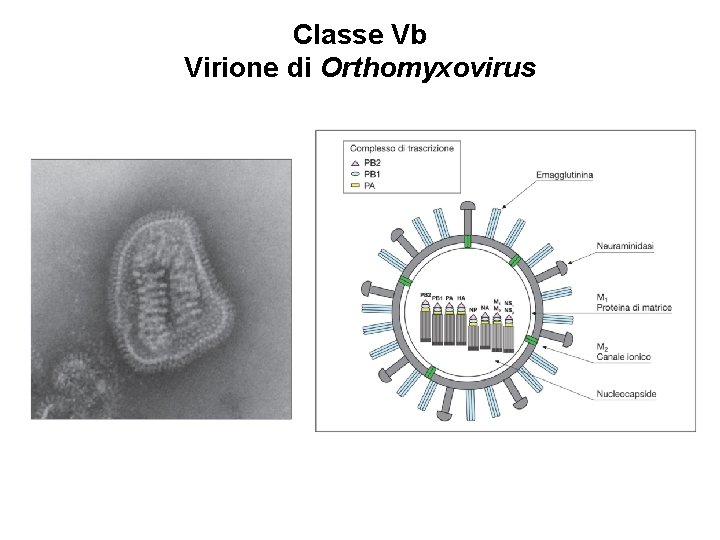
Classe Vb Virione di Orthomyxovirus

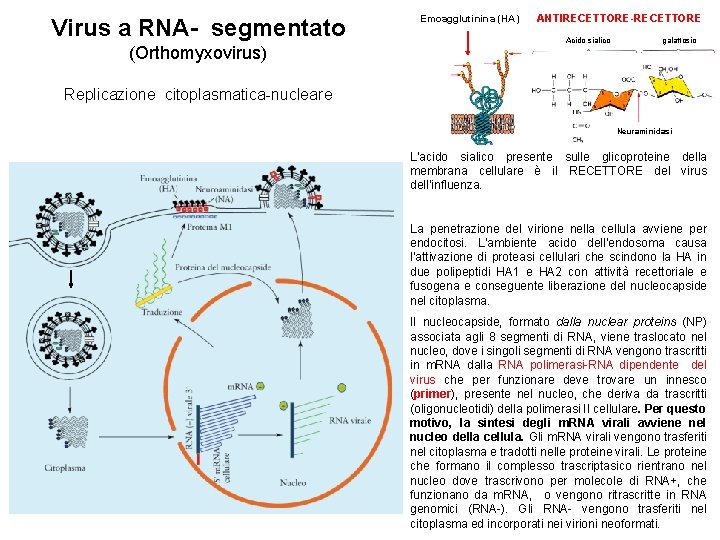
Virus a RNA- segmentato (Orthomyxovirus) Emoagglutinina (HA) ANTIRECETTORE-RECETTORE Acido sialico galattosio Replicazione citoplasmatica-nucleare Neuraminidasi L’acido sialico presente sulle glicoproteine della membrana cellulare è il RECETTORE del virus dell’influenza. La penetrazione del virione nella cellula avviene per endocitosi. L’ambiente acido dell’endosoma causa l’attivazione di proteasi cellulari che scindono la HA in due polipeptidi HA 1 e HA 2 con attività recettoriale e fusogena e conseguente liberazione del nucleocapside nel citoplasma. Il nucleocapside, formato dalla nuclear proteins (NP) associata agli 8 segmenti di RNA, viene traslocato nel nucleo, dove i singoli segmenti di RNA vengono trascritti in m. RNA dalla RNA polimerasi-RNA dipendente del virus che per funzionare deve trovare un innesco (primer), presente nel nucleo, che deriva da trascritti (oligonucleotidi) della polimerasi II cellulare. Per questo motivo, la sintesi degli m. RNA virali avviene nel nucleo della cellula. Gli m. RNA virali vengono trasferiti nel citoplasma e tradotti nelle proteine virali. Le proteine che formano il complesso trascriptasico rientrano nel nucleo dove trascrivono per molecole di RNA+, che funzionano da m. RNA, o vengono ritrascritte in RNA genomici (RNA-). Gli RNA- vengono trasferiti nel citoplasma ed incorporati nei virioni neoformati.

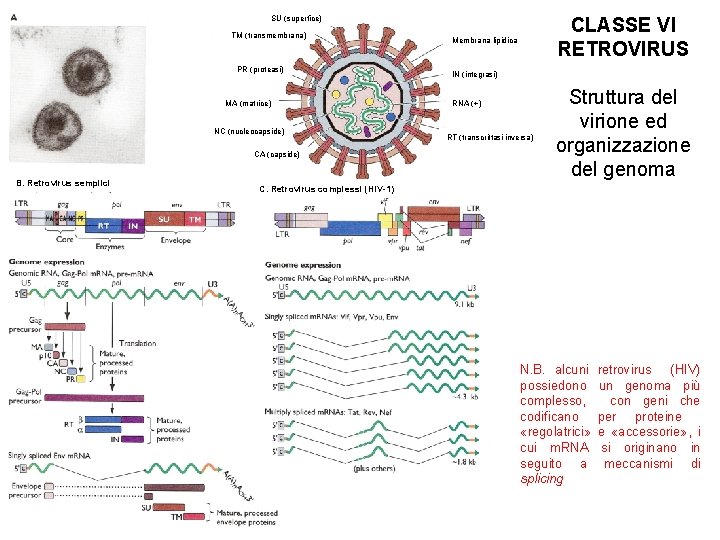
SU (superfice) TM (transmembrana) PR (proteasi) MA (matrice) NC (nucleocapside) CA (capside) B. Retrovirus semplici CLASSE VI RETROVIRUS Membrana lipidica IN (integrasi) RNA (+) RT (transcrittasi inversa) Struttura del virione ed organizzazione del genoma C. Retrovirus complessi (HIV-1) N. B. alcuni retrovirus (HIV) possiedono un genoma più complesso, con geni che codificano per proteine «regolatrici» e «accessorie» , i cui m. RNA si originano in seguito a meccanismi di splicing

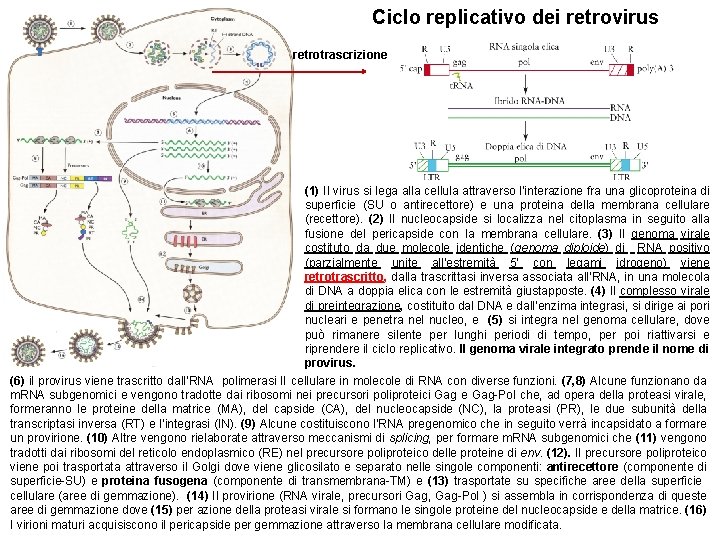
Ciclo replicativo dei retrovirus retrotrascrizione (1) Il virus si lega alla cellula attraverso l’interazione fra una glicoproteina di superficie (SU o antirecettore) e una proteina della membrana cellulare (recettore). (2) Il nucleocapside si localizza nel citoplasma in seguito alla fusione del pericapside con la membrana cellulare. (3) Il genoma virale costituto da due molecole identiche (genoma diploide) di RNA positivo (parzialmente unite all’estremità 5’ con legami idrogeno) viene retrotrascritto, dalla trascrittasi inversa associata all’RNA, in una molecola di DNA a doppia elica con le estremità giustapposte. (4) Il complesso virale di preintegrazione, costituito dal DNA e dall’enzima integrasi, si dirige ai pori nucleari e penetra nel nucleo, e (5) si integra nel genoma cellulare, dove può rimanere silente per lunghi periodi di tempo, per poi riattivarsi e riprendere il ciclo replicativo. Il genoma virale integrato prende il nome di provirus. (6) il provirus viene trascritto dall’RNA polimerasi II cellulare in molecole di RNA con diverse funzioni. (7, 8) Alcune funzionano da m. RNA subgenomici e vengono tradotte dai ribosomi nei precursori poliproteici Gag e Gag-Pol che, ad opera della proteasi virale, formeranno le proteine della matrice (MA), del capside (CA), del nucleocapside (NC), la proteasi (PR), le due subunità della transcriptasi inversa (RT) e l’integrasi (IN). (9) Alcune costituiscono l’RNA pregenomico che in seguito verrà incapsidato a formare un provirione. (10) Altre vengono rielaborate attraverso meccanismi di splicing, per formare m. RNA subgenomici che (11) vengono tradotti dai ribosomi del reticolo endoplasmico (RE) nel precursore poliproteico delle proteine di env. (12). Il precursore poliproteico viene poi trasportata attraverso il Golgi dove viene glicosilato e separato nelle singole componenti: antirecettore (componente di superficie-SU) e proteina fusogena (componente di transmembrana-TM) e (13) trasportate su specifiche aree della superficie cellulare (aree di gemmazione). (14) Il provirione (RNA virale, precursori Gag, Gag-Pol ) si assembla in corrispondenza di queste aree di gemmazione dove (15) per azione della proteasi virale si formano le singole proteine del nucleocapside e della matrice. (16) I virioni maturi acquisiscono il pericapside per gemmazione attraverso la membrana cellulare modificata.

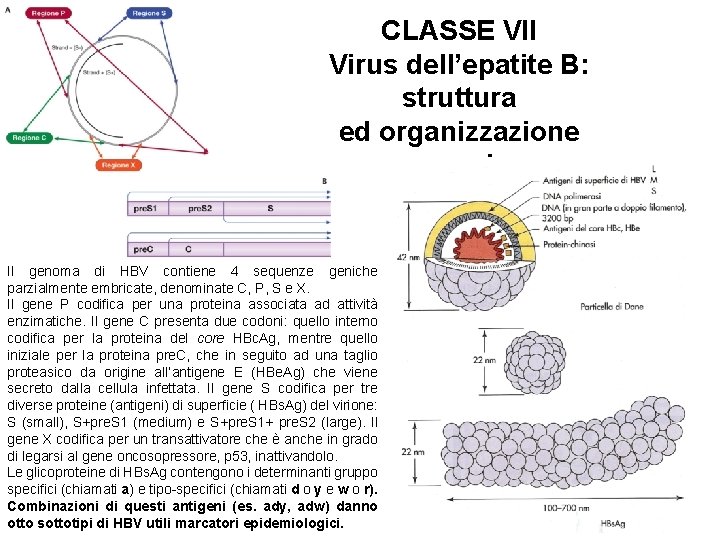
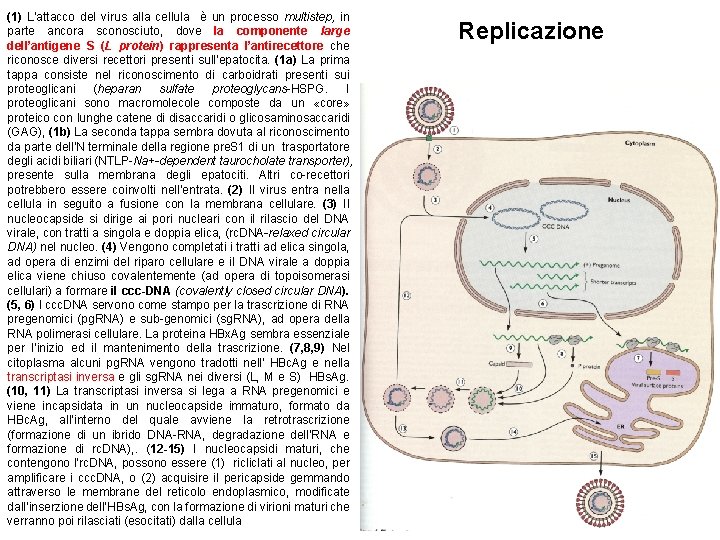
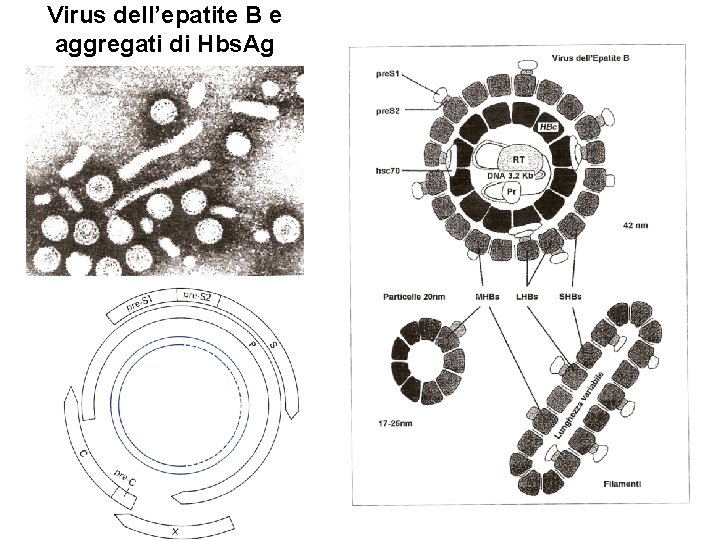
CLASSE VII Virus dell’epatite B: struttura ed organizzazione genomica Il genoma di HBV contiene 4 sequenze geniche parzialmente embricate, denominate C, P, S e X. Il gene P codifica per una proteina associata ad attività enzimatiche. Il gene C presenta due codoni: quello interno codifica per la proteina del core HBc. Ag, mentre quello iniziale per la proteina pre. C, che in seguito ad una taglio proteasico da origine all’antigene E (HBe. Ag) che viene secreto dalla cellula infettata. Il gene S codifica per tre diverse proteine (antigeni) di superficie ( HBs. Ag) del virione: S (small), S+pre. S 1 (medium) e S+pre. S 1+ pre. S 2 (large). Il gene X codifica per un transattivatore che è anche in grado di legarsi al gene oncosopressore, p 53, inattivandolo. Le glicoproteine di HBs. Ag contengono i determinanti gruppo specifici (chiamati a) e tipo-specifici (chiamati d o y e w o r). Combinazioni di questi antigeni (es. ady, adw) danno otto sottotipi di HBV utili marcatori epidemiologici.

(1) L’attacco del virus alla cellula è un processo multistep, in parte ancora sconosciuto, dove la componente large dell’antigene S (L protein) rappresenta l’antirecettore che riconosce diversi recettori presenti sull’epatocita. (1 a) La prima tappa consiste nel riconoscimento di carboidrati presenti sui proteoglicani (heparan sulfate proteoglycans-HSPG. I proteoglicani sono macromolecole composte da un «core» proteico con lunghe catene di disaccaridi o glicosaminosaccaridi (GAG), (1 b) La seconda tappa sembra dovuta al riconoscimento da parte dell’N terminale della regione pre. S 1 di un trasportatore degli acidi biliari (NTLP-Na+-dependent taurocholate transporter), presente sulla membrana degli epatociti. Altri co-recettori potrebbero essere coinvolti nell’entrata. (2) Il virus entra nella cellula in seguito a fusione con la membrana cellulare. (3) Il nucleocapside si dirige ai pori nucleari con il rilascio del DNA virale, con tratti a singola e doppia elica, (rc. DNA-relaxed circular DNA) nel nucleo. (4) Vengono completati i tratti ad elica singola, ad opera di enzimi del riparo cellulare e il DNA virale a doppia elica viene chiuso covalentemente (ad opera di topoisomerasi cellulari) a formare il ccc-DNA (covalently closed circular DNA). (5, 6) l ccc. DNA servono come stampo per la trascrizione di RNA pregenomici (pg. RNA) e sub-genomici (sg. RNA), ad opera della RNA polimerasi cellulare. La proteina HBx. Ag sembra essenziale per l’inizio ed il mantenimento della trascrizione. (7, 8, 9) Nel citoplasma alcuni pg. RNA vengono tradotti nell’ HBc. Ag e nella transcriptasi inversa e gli sg. RNA nei diversi (L, M e S) HBs. Ag. (10, 11) La transcriptasi inversa si lega a RNA pregenomici e viene incapsidata in un nucleocapside immaturo, formato da HBc. Ag, all’interno del quale avviene la retrotrascrizione (formazione di un ibrido DNA-RNA, degradazione dell’RNA e formazione di rc. DNA), . (12 -15) I nucleocapsidi maturi, che contengono l’rc. DNA, possono essere (1) ricliclati al nucleo, per amplificare i ccc. DNA, o (2) acquisire il pericapside gemmando attraverso le membrane del reticolo endoplasmico, modificate dall’inserzione dell’HBs. Ag, con la formazione di virioni maturi che verranno poi rilasciati (esocitati) dalla cellula Replicazione

FINE

Virus dell’epatite B e aggregati di Hbs. Ag

Ciclo replicativo dei virus a DNA con intermedio a RNA -Hepadnavirus -