Variantvurdering rapportering og RNA analyser Kristian Tveten Telemark






















- Slides: 30

Variantvurdering, rapportering og RNA analyser Kristian Tveten (Telemark) Bjørn Ivar Haukanes (Haukeland) Hilde Monica Stensland (UNN) Liss Anne Solberg Lavik (St Olav) Christa Schmidt (St Olav) Helge Rootvelt (OUS) Mari Ann Kulseth (OUS)

Klassifisering av varianter Klasse 5: Sykdomsgivende variant Klasse 4: Sannsynlig sykdomsgivende variant Klasse 3: Variant med usikker betydning(VUS) Klasse 2: Sannsynlig normalvariant Klasse 1: Normalvariant

Samler fakta om en variant for deretter å vekte hver fakta etter styrke for pathogenisitet eller for normalitet. Summen av vekting avgjør klasse. Richards et al. 2015 Genetics in Medicine 17; 405

Evidens for pathogenisitet Veldig sterk evidens PVS 1 Nullvariant (nonsens, frameshift, ± 1 el 2 spleisesete, startkodon, eksondelesjon) i gener hvor LOF er kjent sykdomsmekanisme Sterk evidens PS 1 PS 2 PS 3 PS 4 Samme aminosyreendring er tidligere etablert som pathogen variant De novo (foreldreskap er bekreftet) i en pasient med en sykdom uten familiehistorie In vitro/in vivo funksjonelle studier viser en ødeleggende effekt på m. RNA eller protein Prevalens av variant i affekterte individer er signifikant høyere enn i kontroller Moderat evidens PM 1 PM 2 PM 3 PM 4 PM 5 PM 6 Lokalisert i mutasjons hotspot og/eller i et kritisk og dokumentert funksjonelt domene i proteinet (f. eks. , aktivt sete i et enzym) uten påviste normalvarianter Ikke påvist i kontrollpopulasjon (eller med svært lav frekvens ved recessiv arvegang) For recessive tilstander, detektert i trans med en pathogen variant. Proteinlengden endres, in-frame del/ins i et ikke-repetert område eller ved tap av stoppkodon En missens som gir en ny aminosyreendring i et kodon hvor en annen aminosyreendring er tidligere rapportert pathogen variant Antatt de novo (foreldreskap ikke bekreftet) Støttende evidens PP 1 PP 2 PP 3 PP 4 PP 5 Ko-segregering med sykdom i flere affiserte familiemedlemmer Missens-variant i et gen med få normale missens-varianter og hvor missens-varianter er vanlig årsak til sykdom In silico evidens for pathogenisitet Pasientens fenotype eller familiehistorie er spesifikk for en sykdom knyttet til et gitt gen Anerkjent kilde har nylig rapportert varianten som pathogen uten tilgjengelig bevis

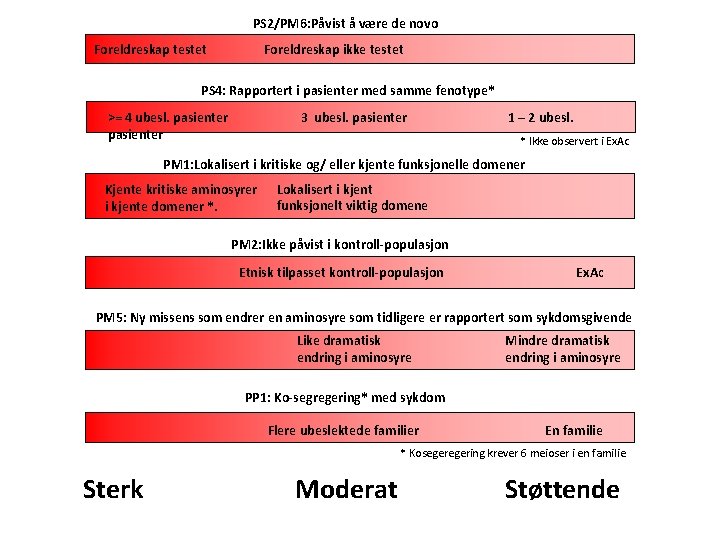
PS 2/PM 6: Påvist å være de novo Foreldreskap testet Foreldreskap ikke testet PS 4: Rapportert i pasienter med samme fenotype* >= 4 ubesl. pasienter 3 ubesl. pasienter 1 – 2 ubesl. * Ikke observert i Ex. Ac PM 1: Lokalisert i kritiske og/ eller kjente funksjonelle domener Kjente kritiske aminosyrer i kjente domener *. Lokalisert i kjent funksjonelt viktig domene PM 2: Ikke påvist i kontroll-populasjon Etnisk tilpasset kontroll-populasjon Ex. Ac PM 5: Ny missens som endrer en aminosyre som tidligere er rapportert som sykdomsgivende Like dramatisk endring i aminosyre Mindre dramatisk endring i aminosyre PP 1: Ko-segregering* med sykdom Flere ubeslektede familier En familie * Kosegering krever 6 meioser i en familie Sterk Moderat Støttende

Evidens for normalitet Sterk nok alene BA 1 Allelfrekvensen er større enn 5% i ESP, 1000 Genome, eller Ex. Ac Vi benytter >0, 5% ved dominant arvegang, >1% ved recessiv arvegang (noen kjente unntak) Sterk evidens BS 1 BS 2 BS 3 BS 4 Allelfrekvensen er større enn forventet for sykdommen Pasienter med alvorlig infantil sykdom skal ikke være inkludert i Ex. AC. Påvist i friske voksne individer; for recessive (homozygote), dominante (heterozygote), eller X-bundet(hemizygote) sykdommer med full penetrans forventet i ung alder In vitro / in vivo funksjonelle studier viser ingen ødeleggende effekt på m. RNA eller protein Manglende segregering i affiserte familiemedlemmer Støttende evidens BP 1 BP 2 BP 3 BP 4 BP 5 BP 6 BP 7 Missens-variant i et gen hvor primært nullvarianter er kjent årsak til sykdom Påvist i trans med en patogen variant i gener forbundet med dominant arvegang, eller uavhengig av arvegang, påvist i cis med en annen pathogen variant In-frame del/ins i en repetitiv region uten kjent funksjon In silico evidens støtter ikke pathogenisitet Variant funnet i en pasient med en annen molekylær forklaring på sykdommen Anerkjent kilde har nylig rapportert varianten som benign uten tilgjengelig bevis En synonym variant hvor spleiseprediksjon verken viser noen effekt på konsensus spleisesete eller dannelse av nytt spleisesete. Nukleotiden er heller ikke høyt konservert

Pathogen (Klasse 5) 1 veldig sterk + ≥ 1 sterk + ≥ 2 moderate + 1 moderat + 1 støttende + ≥ 2 støttende ≥ 2 sterk 1 sterk + ≥ 3 moderate + 2 moderate + ≥ 2 støttende + 1 moderat + ≥ 4 støttende Sannsynlig pathogen (klasse 4) 1 veldig sterk + 1 moderat 1 sterk + 1 -2 moderate + ≥ 2 støttende ≥ 3 moderate 2 moderat + ≥ 2 støttende 1 moderat + ≥ 4 støttende Sannsynlig normalvariant (klasse 2): 1 sterk + 1 støttende ≥ 2 støttende Normalvariant (klasse 1) 1 sterk nok alene ≥ 2 sterk Veldig sterk (PVS 1) Sterk (PS 1 -PS 4) Moderat (PM 1 -PM 6) Støttende (PP 1 -PP 5) Sterk nok alene (BA 1) Strek (BS 1 -BS 4) Støttende (BP 1 -BP 7)

Eksempler……

NM_001396. 3(DYRK 1 A): c. 1309 C>T, p. Arg 437* DYRK 1 A er forbundet med autosomal dominant nedarvet Mental retardation type 7 (OMIM#614104) Ingen frekvens i Ex. Ac Nonsens i ekson 11 (totalt 13) NMD LOF er kjent årsak til sykdom Rapportert i en pasient med syndromal ID (foreldre ikke testet) Fakta styrke Punkt Beskrivelse Ingen frekvens i Ex. Ac støttende PM 2* Ikke påvist i kontrollpopulasjon Nonsens med kjent LOF årsak til sykdom Veldig sterk PVS 1 Nullvariant Rapportert i en pasient med syndromal ID Moderat PS 4* Rapportert i pasienter 1 Veldig sterk + 1 moderat + 1 støttende = Klasse 5 (sykdomsgivende)

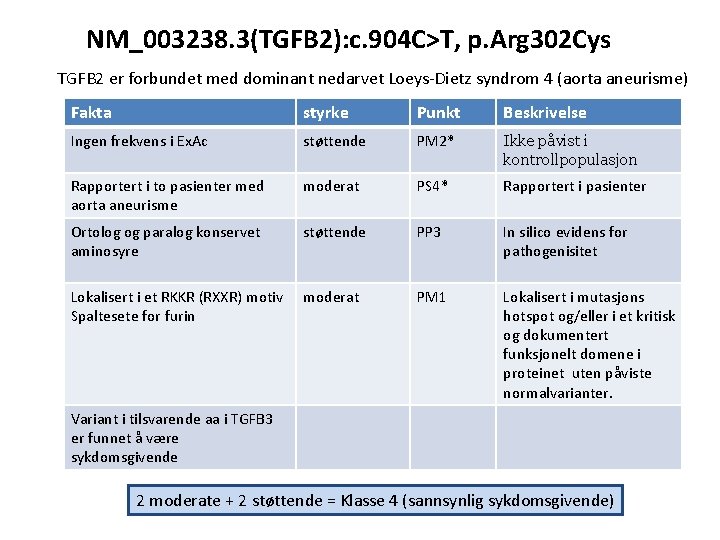
NM_003238. 3(TGFB 2): c. 904 C>T, p. Arg 302 Cys TGFB 2 er forbundet med dominant nedarvet Loeys-Dietz syndrom 4 (aorta aneurisme) Ingen frekvens i Ex. Ac Rapportert i to pasienter med aorta aneurisme Konservert aminosyre (ortholog) Menneske Chimpanse Ape Hund Storfe Mus Rotte Kylling Zebrafisk Frosk 301 301 273 301 270 270 KNSGKTPHLLLMLLPSYRLESQQTNRRKKRALDAAYCFRNVQDNCCLRPL KNSGKTPHLLLMLLPSYRLESQQSNRRKKRALDAAYCFRNVQDNCCLRPL KNSGKSPHLLLMLLPSYRLESQQSNRRKKRALDAAYCFRNVQDNCCLRPL KTSGKTPHLLLMLLPSYRLESQQSSRRKKRALDAAYCFRNVQDNCCLRPL KSSGKTPHLLLMLLPSYRLESQQSSRRRKRALDAAYCFRNVQDNCCLRPL KYSGKTPHLLLMLLPSYRLESQQPSRRKKRALDAAYCFRNVQDNCCLRPL RHSGQSPHLLLMLLPSYRLESQHKSHRQKRALDTAFCSRNVQDNRCLRSL KHTGRTPHLLLMLLPSYRLESQQSSRRKKRALDAAYCFRNVQDNCCLRPL 350 350 322 350 320 319

NM_003238. 3(TGFB 2): c. 904 C>T, p. Arg 302 Cys TGFB 2 er forbundet med dominant nedarvet Loeys-Dietz syndrom 4 (aorta aneurisme) Ingen frekvens i Ex. Ac Rapportert i to pasienter med aorta aneurisme Konservert aminosyre Konservert i TGFB 1 og TGFB 3 (paralog) TGFB 2 PSYRLESQ-QTNRRKKRALDAAYCFRNVQDNCCLRPLYIDFKRDLGWKWIHEPKGYNANF 345 TGFB 1 PLERAQH--LQSSRHRRALDTNYCFSSTEKNCCVRQLYIDFRKDLGWKWIHEPKGYHANF 321 TGFB 3 PPHRLDNPGQGGQRKKRALDTNYCFRNLEENCCVRPLYIDFRQDLGWKWVHEPKGYYANF 343 * * : *: : ****: ***. : . ***: * *****: : ****** ***

NM_003238. 3(TGFB 2): c. 904 C>T, p. Arg 302 Cys TGFB 2 er forbundet med dominant nedarvet Loeys-Dietz syndrom 4 (aorta aneurisme) Ingen frekvens i Ex. Ac Rapportert i en pasient med aorta aneurisme Konservert aminosyre Konservert i TGFB 1 og TGFB 2 Lokalisert i et RKKR (RXXR) motiv (spaltesete for furin, dannelse av moden TGFB) TGFB 2 PSYRLESQ-QTNRRKKRALDAAYCFRNVQDNCCLRPLYIDFKRDLGWKWIHEPKGYNANF 345 TGFB 1 PLERAQH--LQSSRHRRALDTNYCFSSTEKNCCVRQLYIDFRKDLGWKWIHEPKGYHANF 321 TGFB 3 PPHRLDNPGQGGQRKKRALDTNYCFRNLEENCCVRPLYIDFRQDLGWKWVHEPKGYYANF 343 * * : *: : ****: ***. : . ***: * *****: : ****** *** Ingen frekvensdata i Ex. Ac i RXXR motivet

NM_003238. 3(TGFB 2): c. 904 C>T, p. Arg 302 Cys TGFB 2 er forbundet med dominant nedarvet Loeys-Dietz syndrom 4 (aorta aneurisme) Fakta styrke Punkt Beskrivelse Ingen frekvens i Ex. Ac støttende PM 2* Ikke påvist i kontrollpopulasjon Rapportert i to pasienter med aorta aneurisme moderat PS 4* Rapportert i pasienter Ortolog og paralog konservet aminosyre støttende PP 3 In silico evidens for pathogenisitet Lokalisert i et RKKR (RXXR) motiv Spaltesete for furin moderat PM 1 Lokalisert i mutasjons hotspot og/eller i et kritisk og dokumentert funksjonelt domene i proteinet uten påviste normalvarianter. Variant i tilsvarende aa i TGFB 3 er funnet å være sykdomsgivende 2 moderate + 2 støttende = Klasse 4 (sannsynlig sykdomsgivende)


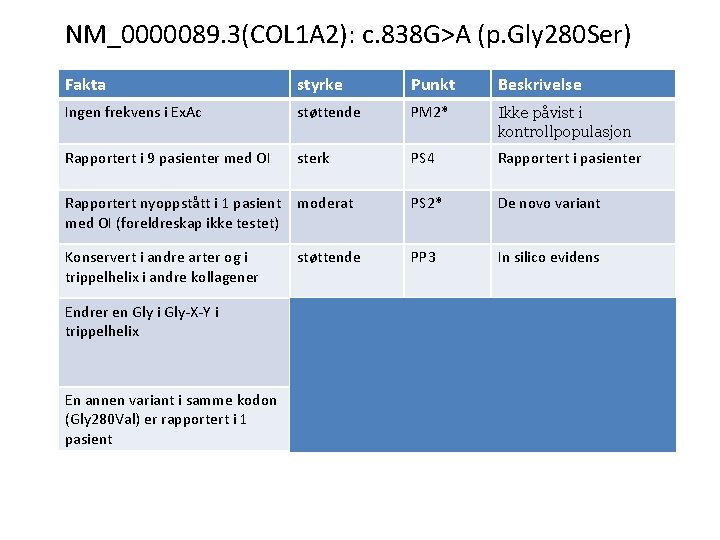
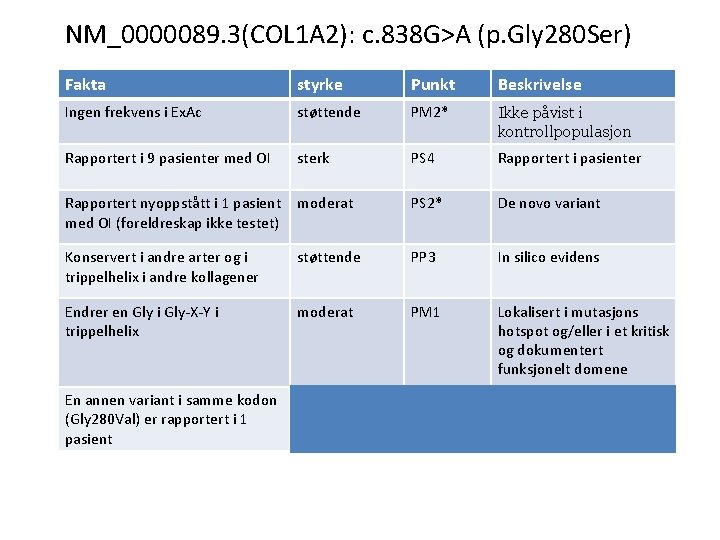
NM_0000089. 3(COL 1 A 2): c. 838 G>A (p. Gly 280 Ser) Col 1 A 2 er forbundet med autosomal dominant nedarvet osteogenesis imperfecta (OI) 1) Ingen frekvensdata i Ex. Ac 2) Rapportert i 7 pasienter med OI (Marini et al, 2007) Rapportert nyoppstått i 1 pasient med OI (Lindahl et al, 2015). Foreldreskap ikke testet. Rapportert i 2 pasienter med OI (Roschger et al. , 2008) 3) Konservert i alle arter og i trippelheliks i andre kollagener 4) Endrer en Glycin i Gly-X-Y repeat i trippelhelix av collagen type I. 80% av sykdomsgivende varianter i Col 1 A 1 og Col 1 A 2 endrer en Glycin i Gly-X-Y repeat 5) En annen variant i samme kodon (Gly 280 Val) er rapportert i en pasient med OI (Obafemi et al, 2008)

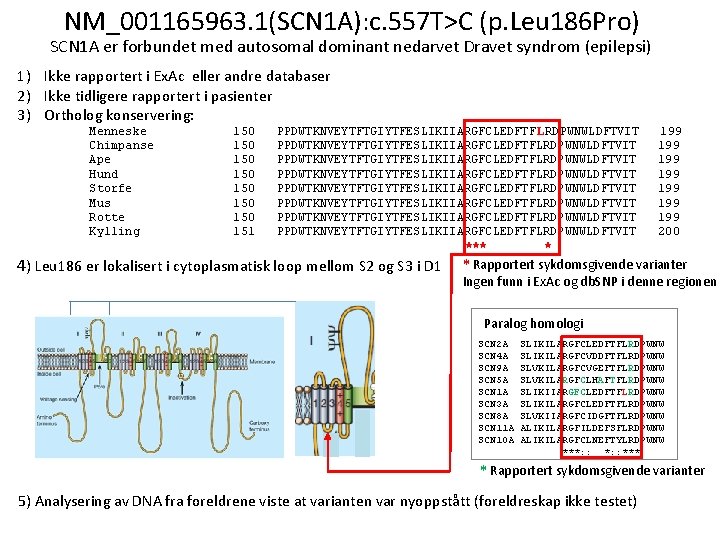
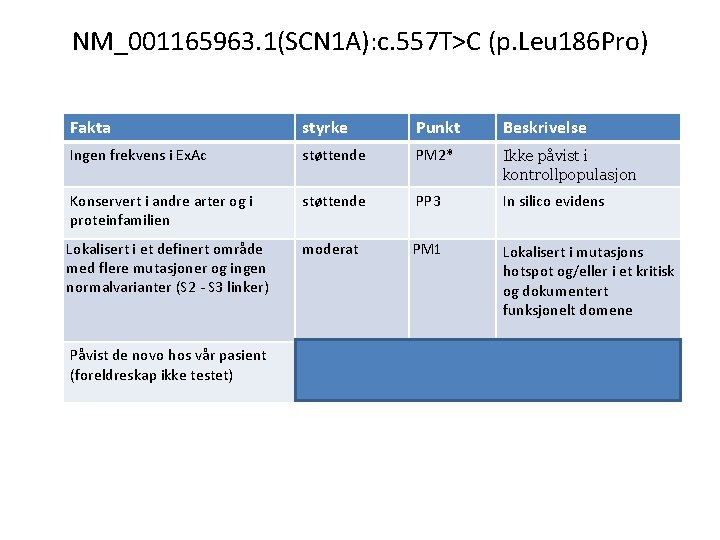
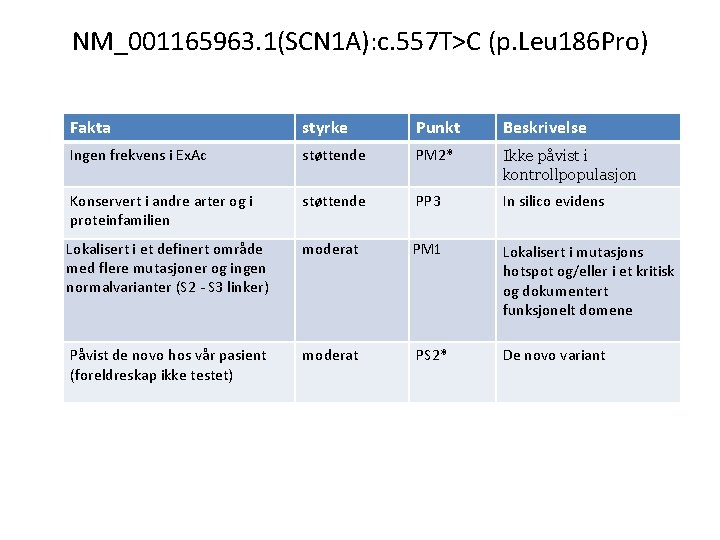
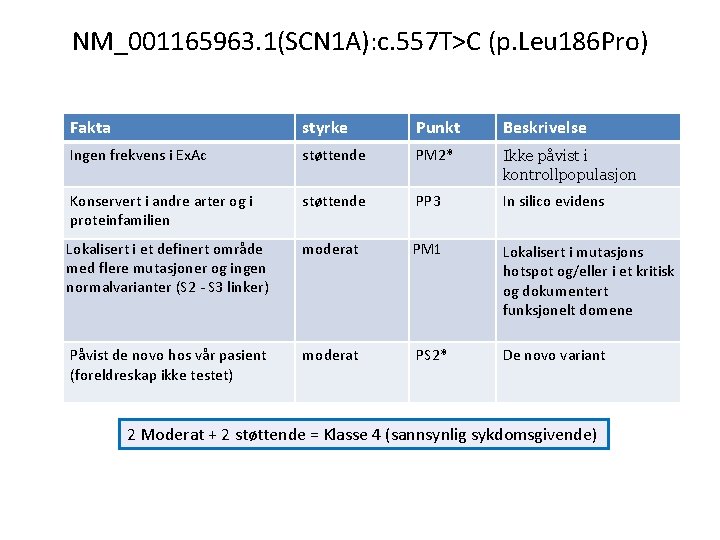
NM_001165963. 1(SCN 1 A): c. 557 T>C (p. Leu 186 Pro) SCN 1 A er forbundet med autosomal dominant nedarvet Dravet syndrom (epilepsi) 1) Ikke rapportert i Ex. Ac eller andre databaser 2) Ikke tidligere rapportert i pasienter 3) Ortholog konservering: Menneske Chimpanse Ape Hund Storfe Mus Rotte Kylling 150 150 151 PPDWTKNVEYTFTGIYTFESLIKIIARGFCLEDFTF LRDPWNWLDFTVIT PPDWTKNVEYTFTGIYTFESLIKIIARGFCLEDFTFLRDPWNWLDFTVIT PPDWTKNVEYTFTGIYTFESLIKIIARGFCLEDFTFLRDPWNWLDFTVIT *** * 4) Leu 186 er lokalisert i cytoplasmatisk loop mellom S 2 og S 3 i D 1 199 199 200 * Rapportert sykdomsgivende varianter Ingen funn i Ex. Ac og db. SNP i denne regionen Paralog homologi SCN 2 A SCN 4 A SCN 9 A SCN 5 A SCN 1 A SCN 3 A SCN 8 A SCN 11 A SCN 10 A SLIKILARGFCLEDFTFLRDPWNW SLIKILARGFCVDDFTFLRDPWNW SLVKILARGFCVGEFTFLRDPWNW SLVKILARGFCLHAFTFLRDPWNW SLIKIIARGFCLEDFTFLRDPWNW SLIKILARGFCLEDFTFLRDPWNW SLVKIIARGFCIDGFTFLRDPWNW ALIKILARGFILDEFSFLRDPWNW ALIKILARGFCLNEFTYLRDPWNW ***: : *** * Rapportert sykdomsgivende varianter 5) Analysering av DNA fra foreldrene viste at varianten var nyoppstått (foreldreskap ikke testet)

NM_001165963. 1(SCN 1 A): c. 557 T>C (p. Leu 186 Pro) SCN 1 A er forbundet med autosomal dominant nedarvet Dravet syndrom (epilepsi) 1) Ikke rapportert i Ex. Ac eller andre databaser 2) Ikke tidligere rapportert i pasienter 3) Ortholog konservering: Menneske Chimpanse Ape Hund Storfe Mus Rotte Kylling 150 150 151 PPDWTKNVEYTFTGIYTFESLIKIIARGFCLEDFTF LRDPWNWLDFTVIT PPDWTKNVEYTFTGIYTFESLIKIIARGFCLEDFTFLRDPWNWLDFTVIT PPDWTKNVEYTFTGIYTFESLIKIIARGFCLEDFTFLRDPWNWLDFTVIT *** * 4) Leu 186 er lokalisert i cytoplasmatisk loop mellom S 2 og S 3 i D 1 199 199 200 * Rapportert sykdomsgivende varianter Ingen funn i Ex. Ac og db. SNP i denne regionen Paralog homologi SCN 2 A SCN 4 A SCN 9 A SCN 5 A SCN 1 A SCN 3 A SCN 8 A SCN 11 A SCN 10 A SLIKILARGFCLEDFTFLRDPWNW SLIKILARGFCVDDFTFLRDPWNW SLVKILARGFCVGEFTFLRDPWNW SLVKILARGFCLHAFTFLRDPWNW SLIKIIARGFCLEDFTFLRDPWNW SLIKILARGFCLEDFTFLRDPWNW SLVKIIARGFCIDGFTFLRDPWNW ALIKILARGFILDEFSFLRDPWNW ALIKILARGFCLNEFTYLRDPWNW ***: : *** * Rapportert sykdomsgivende varianter 5) Analysering av DNA fra foreldrene viste at varianten var nyoppstått (foreldreskap ikke testet)

NM_0000089. 3(COL 1 A 2): c. 838 G>A (p. Gly 280 Ser) Fakta styrke Punkt Beskrivelse Ingen frekvens i Ex. Ac støttende PM 2* Ikke påvist i kontrollpopulasjon Rapportert i 9 pasienter med OI sterk PS 4 Rapportert i pasienter Rapportert nyoppstått i 1 pasient med OI (foreldreskap ikke testet) moderat PS 2* De novo variant Konservert i andre arter og i trippelhelix i andre kollagener støttende PP 3 In silico evidens Endrer en Gly i Gly-X-Y i trippelhelix moderat PM 1 Lokalisert i mutasjons hotspot og/eller i et kritisk og dokumentert funksjonelt domene En annen variant i samme kodon (Gly 280 Val) er rapportert i 1 pasient støttende PM 5* Annen variant i samme kodon

NM_0000089. 3(COL 1 A 2): c. 838 G>A (p. Gly 280 Ser) Fakta styrke Punkt Beskrivelse Ingen frekvens i Ex. Ac støttende PM 2* Ikke påvist i kontrollpopulasjon Rapportert i 9 pasienter med OI sterk PS 4 Rapportert i pasienter Rapportert nyoppstått i 1 pasient med OI (foreldreskap ikke testet) moderat PS 2* De novo variant Konservert i andre arter og i trippelhelix i andre kollagener støttende PP 3 In silico evidens Endrer en Gly i Gly-X-Y i trippelhelix moderat PM 1 Lokalisert i mutasjons hotspot og/eller i et kritisk og dokumentert funksjonelt domene En annen variant i samme kodon (Gly 280 Val) er rapportert i 1 pasient støttende PM 5* Annen variant i samme kodon

NM_0000089. 3(COL 1 A 2): c. 838 G>A (p. Gly 280 Ser) Fakta styrke Punkt Beskrivelse Ingen frekvens i Ex. Ac støttende PM 2* Ikke påvist i kontrollpopulasjon Rapportert i 9 pasienter med OI sterk PS 4 Rapportert i pasienter Rapportert nyoppstått i 1 pasient med OI (foreldreskap ikke testet) moderat PS 2* De novo variant Konservert i andre arter og i trippelhelix i andre kollagener støttende PP 3 In silico evidens Endrer en Gly i Gly-X-Y i trippelhelix moderat PM 1 Lokalisert i mutasjons hotspot og/eller i et kritisk og dokumentert funksjonelt domene En annen variant i samme kodon (Gly 280 Val) er rapportert i 1 pasient støttende PM 5* Annen variant i samme kodon

NM_0000089. 3(COL 1 A 2): c. 838 G>A (p. Gly 280 Ser) Fakta styrke Punkt Beskrivelse Ingen frekvens i Ex. Ac støttende PM 2* Ikke påvist i kontrollpopulasjon Rapportert i 9 pasienter med OI sterk PS 4 Rapportert i pasienter Rapportert nyoppstått i 1 pasient med OI (foreldreskap ikke testet) moderat PS 2* De novo variant Konservert i andre arter og i trippelhelix i andre kollagener støttende PP 3 In silico evidens Endrer en Gly i Gly-X-Y i trippelhelix moderat PM 1 Lokalisert i mutasjons hotspot og/eller i et kritisk og dokumentert funksjonelt domene En annen variant i samme kodon (Gly 280 Val) er rapportert i 1 pasient støttende PM 5* Annen variant i samme kodon

NM_0000089. 3(COL 1 A 2): c. 838 G>A (p. Gly 280 Ser) Fakta styrke Punkt Beskrivelse Ingen frekvens i Ex. Ac støttende PM 2* Ikke påvist i kontrollpopulasjon Rapportert i 9 pasienter med OI sterk PS 4 Rapportert i pasienter Rapportert nyoppstått i 1 pasient med OI (foreldreskap ikke testet) moderat PS 2* De novo variant Konservert i andre arter og i trippelhelix i andre kollagener støttende PP 3 In silico evidens Endrer en Gly i Gly-X-Y i trippelhelix moderat PM 1 Lokalisert i mutasjons hotspot og/eller i et kritisk og dokumentert funksjonelt domene En annen variant i samme kodon (Gly 280 Val) er rapportert i 1 pasient støttende PM 5* Annen variant i samme kodon

NM_0000089. 3(COL 1 A 2): c. 838 G>A (p. Gly 280 Ser) Fakta styrke Punkt Beskrivelse Ingen frekvens i Ex. Ac støttende PM 2* Ikke påvist i kontrollpopulasjon Rapportert i 9 pasienter med OI sterk PS 4 Rapportert i pasienter Rapportert nyoppstått i 1 pasient med OI (foreldreskap ikke testet) moderat PS 2* De novo variant Konservert i andre arter og i trippelhelix i andre kollagener støttende PP 3 In silico evidens Endrer en Gly i Gly-X-Y i trippelhelix moderat PM 1 Lokalisert i mutasjons hotspot og/eller i et kritisk og dokumentert funksjonelt domene En annen variant i samme kodon (Gly 280 Val) er rapportert i 1 pasient støttende PM 5* Annen variant i samme kodon

NM_0000089. 3(COL 1 A 2): c. 838 G>A (p. Gly 280 Ser) Fakta styrke Punkt Beskrivelse Ingen frekvens i Ex. Ac støttende PM 2* Ikke påvist i kontrollpopulasjon Rapportert i 9 pasienter med OI sterk PS 4 Rapportert i pasienter Rapportert nyoppstått i 1 pasient med OI (foreldreskap ikke testet) moderat PS 2* De novo variant Konservert i andre arter og i trippelhelix i andre kollagener støttende PP 3 In silico evidens Endrer en Gly i Gly-X-Y i trippelhelix moderat PM 1 Lokalisert i mutasjons hotspot og/eller i et kritisk og dokumentert funksjonelt domene En annen variant i samme kodon (Gly 280 Val) er rapportert i 1 pasient støttende PM 5* Annen variant i samme kodon 1 sterk +2 moderat + 3 støttende = Sykdomsgivende (Klasse 5)

NM_001165963. 1(SCN 1 A): c. 557 T>C (p. Leu 186 Pro) Fakta styrke Punkt Beskrivelse Ingen frekvens i Ex. Ac støttende PM 2* Ikke påvist i kontrollpopulasjon Konservert i andre arter og i proteinfamilien støttende PP 3 In silico evidens Lokalisert i et definert område med flere mutasjoner og ingen normalvarianter (S 2 - S 3 linker) moderat PM 1 Lokalisert i mutasjons hotspot og/eller i et kritisk og dokumentert funksjonelt domene Påvist de novo hos vår pasient (foreldreskap ikke testet) moderat PS 2* De novo variant

NM_001165963. 1(SCN 1 A): c. 557 T>C (p. Leu 186 Pro) Fakta styrke Punkt Beskrivelse Ingen frekvens i Ex. Ac støttende PM 2* Ikke påvist i kontrollpopulasjon Konservert i andre arter og i proteinfamilien støttende PP 3 In silico evidens Lokalisert i et definert område med flere mutasjoner og ingen normalvarianter (S 2 - S 3 linker) moderat PM 1 Lokalisert i mutasjons hotspot og/eller i et kritisk og dokumentert funksjonelt domene Påvist de novo hos vår pasient (foreldreskap ikke testet) moderat PS 2* De novo variant

NM_001165963. 1(SCN 1 A): c. 557 T>C (p. Leu 186 Pro) Fakta styrke Punkt Beskrivelse Ingen frekvens i Ex. Ac støttende PM 2* Ikke påvist i kontrollpopulasjon Konservert i andre arter og i proteinfamilien støttende PP 3 In silico evidens Lokalisert i et definert område med flere mutasjoner og ingen normalvarianter (S 2 - S 3 linker) moderat PM 1 Lokalisert i mutasjons hotspot og/eller i et kritisk og dokumentert funksjonelt domene Påvist de novo hos vår pasient (foreldreskap ikke testet) moderat PS 2* De novo variant

NM_001165963. 1(SCN 1 A): c. 557 T>C (p. Leu 186 Pro) Fakta styrke Punkt Beskrivelse Ingen frekvens i Ex. Ac støttende PM 2* Ikke påvist i kontrollpopulasjon Konservert i andre arter og i proteinfamilien støttende PP 3 In silico evidens Lokalisert i et definert område med flere mutasjoner og ingen normalvarianter (S 2 - S 3 linker) moderat PM 1 Lokalisert i mutasjons hotspot og/eller i et kritisk og dokumentert funksjonelt domene Påvist de novo hos vår pasient (foreldreskap ikke testet) moderat PS 2* De novo variant

NM_001165963. 1(SCN 1 A): c. 557 T>C (p. Leu 186 Pro) Fakta styrke Punkt Beskrivelse Ingen frekvens i Ex. Ac støttende PM 2* Ikke påvist i kontrollpopulasjon Konservert i andre arter og i proteinfamilien støttende PP 3 In silico evidens Lokalisert i et definert område med flere mutasjoner og ingen normalvarianter (S 2 - S 3 linker) moderat PM 1 Lokalisert i mutasjons hotspot og/eller i et kritisk og dokumentert funksjonelt domene Påvist de novo hos vår pasient (foreldreskap ikke testet) moderat PS 2* De novo variant

NM_001165963. 1(SCN 1 A): c. 557 T>C (p. Leu 186 Pro) Fakta styrke Punkt Beskrivelse Ingen frekvens i Ex. Ac støttende PM 2* Ikke påvist i kontrollpopulasjon Konservert i andre arter og i proteinfamilien støttende PP 3 In silico evidens Lokalisert i et definert område med flere mutasjoner og ingen normalvarianter (S 2 - S 3 linker) moderat PM 1 Lokalisert i mutasjons hotspot og/eller i et kritisk og dokumentert funksjonelt domene Påvist de novo hos vår pasient (foreldreskap ikke testet) moderat PS 2* De novo variant 2 Moderat + 2 støttende = Klasse 4 (sannsynlig sykdomsgivende)
 Rapportering och analys
Rapportering och analys Kundetælling
Kundetælling Site telemark
Site telemark Telemark marketing
Telemark marketing Telemark university college
Telemark university college Sio helse telefon
Sio helse telefon Kristian overgaard
Kristian overgaard Kristian vanderwaeren wikipedia
Kristian vanderwaeren wikipedia Kristian bolin
Kristian bolin Kristian krieger
Kristian krieger Kristian viekilde
Kristian viekilde Kristian stokke uio
Kristian stokke uio Kristian frich martínez de velasco
Kristian frich martínez de velasco Kristian zahrtmann
Kristian zahrtmann Islam vs kristian
Islam vs kristian Kristian sandahl
Kristian sandahl Kristian zarb adami
Kristian zarb adami Kristian stokke
Kristian stokke Kristian korunić
Kristian korunić Kristian hedegaard jensen
Kristian hedegaard jensen Eero parviainen
Eero parviainen Kristian jong høines
Kristian jong høines Kristian frich martínez de velasco
Kristian frich martínez de velasco Kristian filion
Kristian filion Kristian leko
Kristian leko Kristian ludlow
Kristian ludlow Combifoss
Combifoss Tabel univariat
Tabel univariat Mastercfa
Mastercfa Hggsp analyser les relations entre états et religions
Hggsp analyser les relations entre états et religions Stratification sociale
Stratification sociale