UNIVERSITE DE PICARDIE JULES VERNE Facult de Pharmacie




![Unités utilisés en DC [θ] (2, 303* ΔА *180) = 4π deg [θ] = Unités utilisés en DC [θ] (2, 303* ΔА *180) = 4π deg [θ] =](https://slidetodoc.com/presentation_image_h/79bc237f082038e1fbca3deddf01194e/image-9.jpg)









- Slides: 23

UNIVERSITE DE PICARDIE JULES VERNE Faculté de Pharmacie Chimie Organique - EA 3901 -DMAG-INERIS J. Pêcher, P. Sonnet, F. Y. Dupradeau LE DICHROISME CIRCULAIRE 25 novembre 2005

plan 1) Qu’est-ce que la lumière polarisée 2) Le dichroïsme circulaire 3) Conditions opératoires 4) Exemples 5) Avantages du dichroïsme circulaire

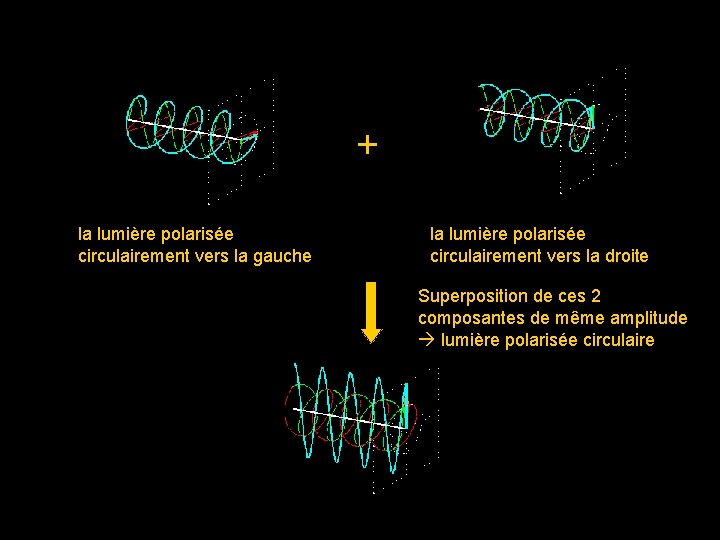
1) Qu’est-ce que la lumière polarisée Une source émet un très grand nombre de photons. Les différents vecteurs vibrations de ces photons peuvent prendre toutes les orientations possibles dans chaque section droite du faisceau émis : la lumière est dite naturelle La lumière naturelle possède une symétrie de révolution autour de la direction de propagation La réflexion de la lumière à l’aide d’un miroir a fait perdre à la lumière sa symétrie de révolution. Alors la lumière réfléchie n’est plus de la lumière naturelle : elle est dite polarisée La lumière polarisée peut se séparer en deux composante : la lumière polarisée circulairement vers la droite la lumière polarisée circulairement vers la gauche

+ la lumière polarisée circulairement vers la gauche la lumière polarisée circulairement vers la droite Superposition de ces 2 composantes de même amplitude lumière polarisée circulaire

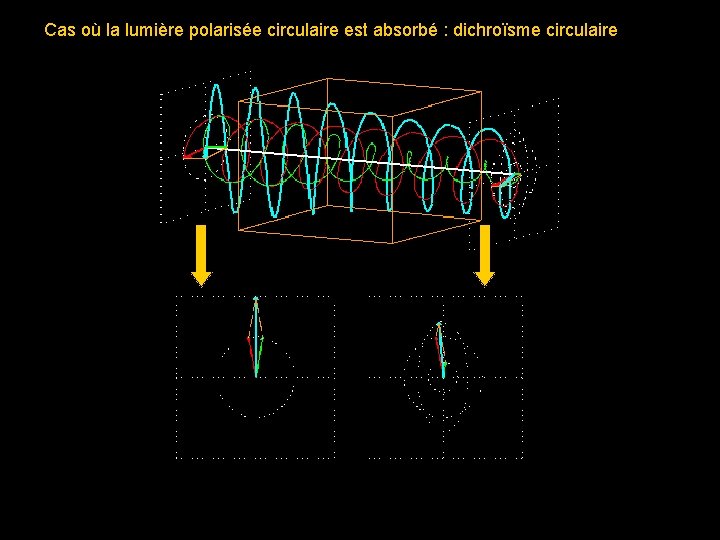
Cas où la lumière polarisée circulaire est absorbé : dichroïsme circulaire

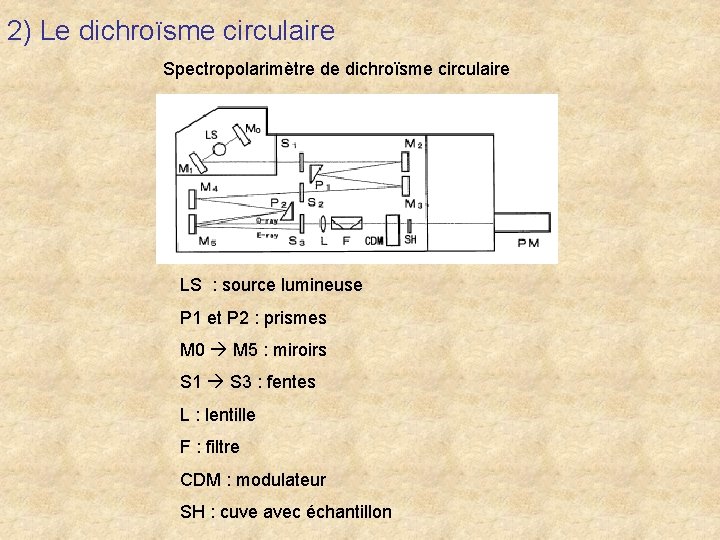
2) Le dichroïsme circulaire Spectropolarimètre de dichroïsme circulaire LS : source lumineuse P 1 et P 2 : prismes M 0 M 5 : miroirs S 1 S 3 : fentes L : lentille F : filtre CDM : modulateur SH : cuve avec échantillon

Les structures secondaires n’absorbent pas de façon égale la lumière polarisée circulaire gauche et la lumière polarisée circulaire droite. L’absorption préférentielle de l’une de ces polarisations résulte en une déviation de la résultante. La mesure de cette déviation est appelée DICHROISME CIRCULAIRE

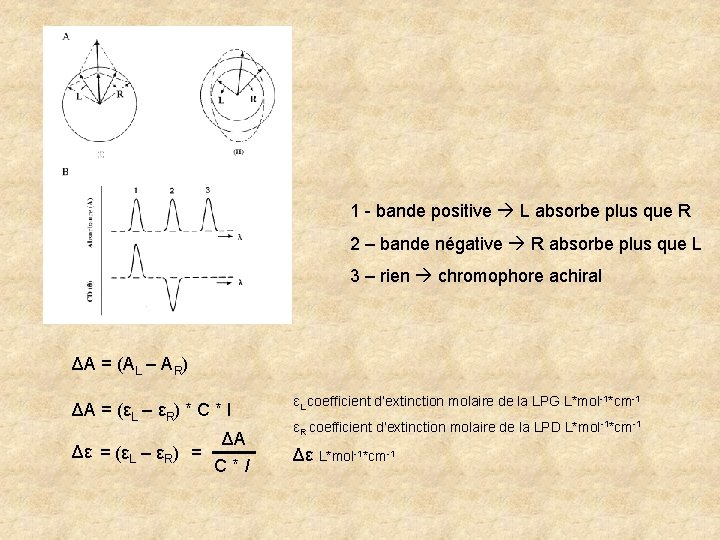
1 - bande positive L absorbe plus que R 2 – bande négative R absorbe plus que L 3 – rien chromophore achiral ΔА = (AL – AR) ΔА = (εL – εR) * C * l Δε = (εL – εR) = ΔА C*l εL coefficient d’extinction molaire de la LPG L*mol-1*cm-1 εR coefficient d’extinction molaire de la LPD L*mol-1*cm-1 Δε L*mol-1*cm-1
![Unités utilisés en DC θ 2 303 ΔА 180 4π deg θ Unités utilisés en DC [θ] (2, 303* ΔА *180) = 4π deg [θ] =](https://slidetodoc.com/presentation_image_h/79bc237f082038e1fbca3deddf01194e/image-9.jpg)
Unités utilisés en DC [θ] (2, 303* ΔА *180) = 4π deg [θ] = (2, 303* ΔА *180) 4π deg MRW = M N-1 * 100 C*l = 3298 * Δε C = concentration protéine en mol/L l = trajet optique dans la cuve en cm cm 2*dmol-1 * MRW * Ellipticité molaire deg*cm 2*dmol-1 1 10*C * l Ellipticité molaire par résidu deg*cm 2*dmol-1 M = masse de la protéine en Da N = nombre d’aminoacides C = concentration protéine en g/ml l = trajet optique dans la cuve en cm

Le dichroïsme circulaire est utilisé pour étudier la structure des : • Protéines • Sucres • ADN • Molécules chirales telles que cofacteur (pyridoxal-5’-phosphate), des flavines, des tri terpènes (acide ursolique), les hèmes, les flavocytochrome, des pigments photosynthètiques etc …

Deux régions spectrales intéressantes pour obtenir des informations structurales : 1) UV lointain 180 nm à 250 nm : - Effet de l’environnement sur la structure d’une molécule - Détermination du contenu en structure secondaire de protéine - Effet de ligands sur la structure - Etudes d’interactions protéine-protéine et acides nucléiques-protéines - Etudes dynamiques (dénaturation, renaturation) 2) UV proche 250 nm à 350 nm : - Empreinte digitale de la structure tertiaire - Effet de ligands sur la structure - Effet de l’environnement sur la structure d’une molécule

1) UV lointain 180 nm à 250 nm : chromophore liaison peptidique Structure Aléatoire - bande négative à 195 nm transition n->π* - bande positive à 212 nm transition π->π* Feuillet β - bande positive à 196 nm transition n->π* - bande négative à 218 nm transition π->π* Hélice α n ->π* électrons de non liaison de l’N du C du carbonyle π->π* électrons π du carbonyle - bande positive à 192 nm transition π->π* - bande négative à 209 nm transition π->π* - bande négative à 222 nm transition n->π* Hélice polyproline II - bande négative à 192 nm transition n->π - bande positive à 220 nm transition π->π* L’intensité et l’énergie de transition dépendent des angles Φ et ψ des structures secondaires

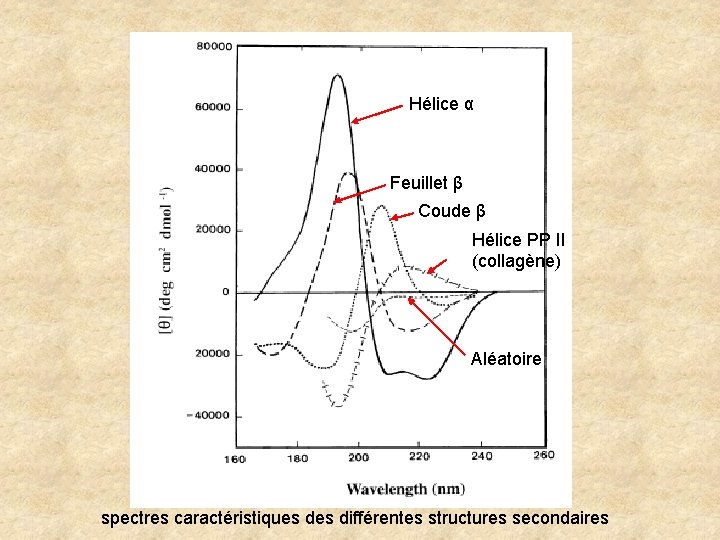
Hélice α Feuillet β Coude β Hélice PP II (collagène) Aléatoire spectres caractéristiques différentes structures secondaires

- Il est possible de déterminer, à partir d’un spectre DC en UV lointain, le pourcentage de structure secondaire présentes dans une protéine. - Il n’est pas possible de préciser la localisation de ces structures secondaires. - Pour déterminer le pourcentage de structures secondaires il fut considéré le signal DC comme étant la somme des contributions de différentes structures secondaires : θ = fα* θα + fβ* θβ + fp* θp θ = Σi fi* θi Où fi est la fraction molaire de la structure secondaire i dans la protéine et θi est le signal DC pour un échantillons 100% i à la longueur d’onde . Différents logiciels effectuent cette deconvolution : CCA, CONTIN, CDSSTR, K 2 D, LINCOMB, SELCON, VARSLC, XLSSTR

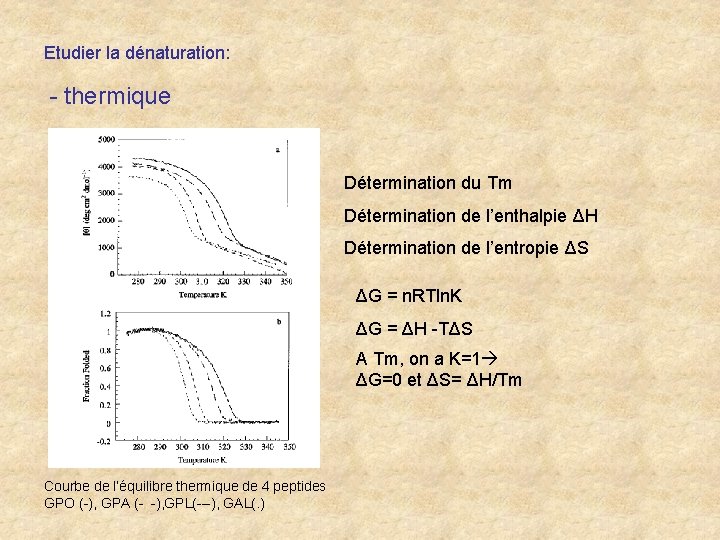
Etudier la dénaturation: - thermique Détermination du Tm Détermination de l’enthalpie ΔH Détermination de l’entropie ΔS ΔG = n. RTln. K ΔG = ΔH -TΔS A Tm, on a K=1 ΔG=0 et ΔS= ΔH/Tm Courbe de l’équilibre thermique de 4 peptides GPO (-), GPA (- -), GPL(---), GAL(. )

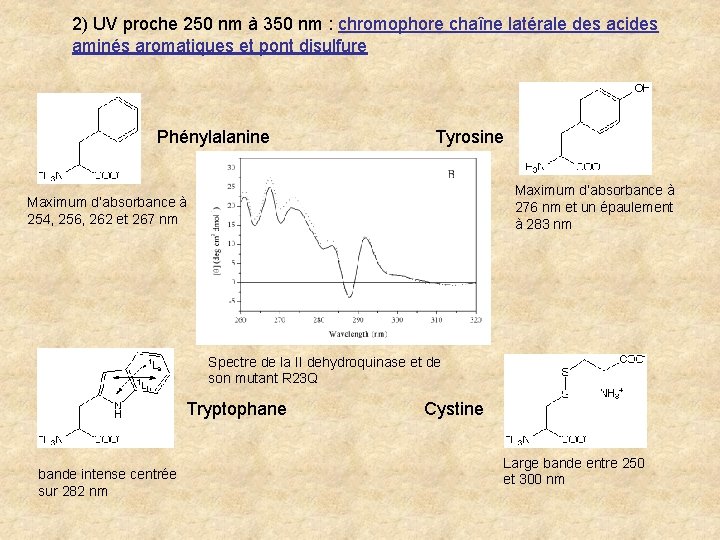
2) UV proche 250 nm à 350 nm : chromophore chaîne latérale des acides aminés aromatiques et pont disulfure Phénylalanine Tyrosine Maximum d’absorbance à 276 nm et un épaulement à 283 nm Maximum d’absorbance à 254, 256, 262 et 267 nm Spectre de la II dehydroquinase et de son mutant R 23 Q Tryptophane bande intense centrée sur 282 nm Cystine Large bande entre 250 et 300 nm

3) Conditions opératoire : Détermination de la concentration exacte de la molécule a étudié : méthode colorimétrique (biuret, Folin-lowry, Bradford …) A 280 nm lecture directe (avantage non destructif) Calibration du spectropolarimètre suivant la région spectrale étudiée Choix de la cuve (trajet optique) : 1 mm, 0. 5 mm, 0. 001 mm Choix de la région spectrale (UV lointain ou UV proche) Faible concentration µmol. L-1

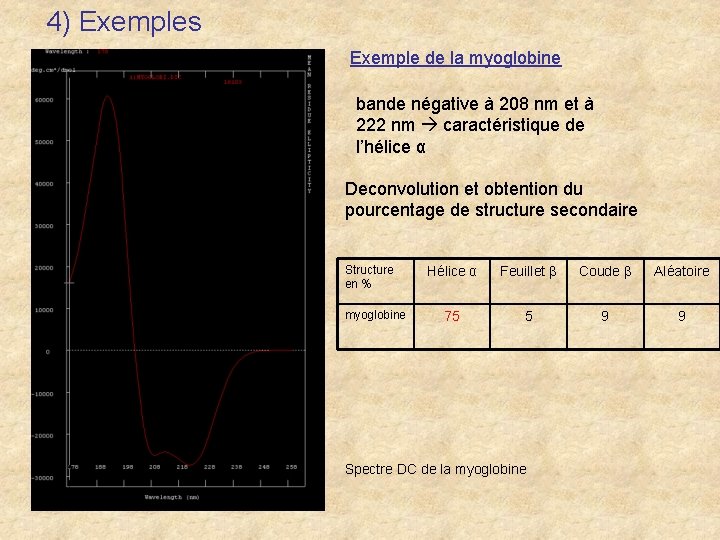
4) Exemples Exemple de la myoglobine bande négative à 208 nm et à 222 nm caractéristique de l’hélice α Deconvolution et obtention du pourcentage de structure secondaire Structure en % myoglobine Hélice α Feuillet β Coude β Aléatoire 75 5 9 9 Spectre DC de la myoglobine

Exemple de deux peptides TPRO bande négative 198 nm transition n->π* bande positive 228 nm transition π ->π* THYP bande négative 199 nm transition n->π* bande positive 220 nm transition π ->π* Spectres DC de 2 peptides (Tpro et Thyp) % hélice α % feuillet β % coude β % aléatoire TPRO 2 36, 5 25, 3 35, 6 THYP 1, 5 38 25, 3 34, 7

Exemple trois peptides gpp 3, gpp 5 et scramble Spectres DC de 3 peptides (gpp 3, gpp 5 et scramble) gpp 3 bande négative 198 nm transition n->π* bande positive 228 nm transition π ->π* gpp 5 bande négative 200 nm transition n->π* bande positive 227 nm transition π ->π* scramble bande négative 200 nm transition n->π* bande positive 226 nm transition π ->π* Structure caractéristique de l’hélice polyproline 2

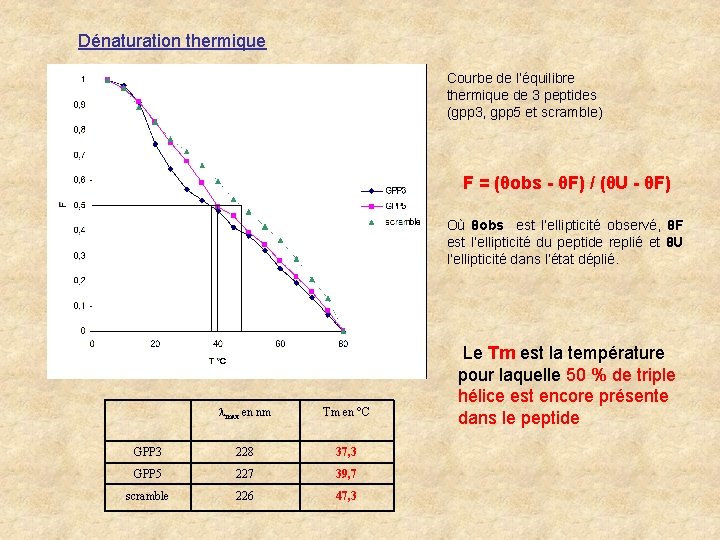
Dénaturation thermique Courbe de l’équilibre thermique de 3 peptides (gpp 3, gpp 5 et scramble) F = (θobs - θF) / (θU - θF) Où θobs est l’ellipticité observé, θF est l’ellipticité du peptide replié et θU l’ellipticité dans l’état déplié. max en nm Tm en °C GPP 3 228 37, 3 GPP 5 227 39, 7 scramble 226 47, 3 Le Tm est la température pour laquelle 50 % de triple hélice est encore présente dans le peptide

5) Les avantages du dichroïsme circulaire . On travaille en solution diluée. Des quantités de produit faibles (quelques dizaines de µg). Pas de limite de taille. Mesures rapides et simples. Mesures cinétiques à des temps très courts

Bibliographie S. M Kelly et Al, Biochimica et biophysica acta, 2005, 1751, 119 -139 N. K Shah et Al, Biochemistry, 1996, 35, 10262 -10268 http: //www. enzim. hu/~szia/cddemo/edemo 8. htm http: //www. food. rdg. ac. uk/online/fs 460/lecture 6. htm http: //www. callisto. si. usherb. ca/%7 Ebcm 514/6 a. html http: //www. huhaha. com/pages/cd. html
 Facult
Facult Jule bern
Jule bern Jules verne scifi
Jules verne scifi Organigramme collège public
Organigramme collège public Verne cesta na mesiac
Verne cesta na mesiac Planiquel
Planiquel Collège jules ferry mantes la jolie
Collège jules ferry mantes la jolie Picardie
Picardie Procedure chaine du froid pharmacie
Procedure chaine du froid pharmacie Scolarité pharmacie nantes
Scolarité pharmacie nantes Axe hypothalamo hypophysaire
Axe hypothalamo hypophysaire Pharmacie hippocrate sousse
Pharmacie hippocrate sousse Pharmacie
Pharmacie Manicol pharmacie
Manicol pharmacie Faculté de médecine constantine cours
Faculté de médecine constantine cours Esther kellenberger
Esther kellenberger Pharmacie beaujon
Pharmacie beaujon Organigramme de pharmacie
Organigramme de pharmacie Pharmacie gamma lausanne laboratoire
Pharmacie gamma lausanne laboratoire Pharmacie.univ-batna2
Pharmacie.univ-batna2 Pessaire cube remboursement
Pessaire cube remboursement Coursif
Coursif Faculté de pharmacie monastir
Faculté de pharmacie monastir Sanal sınıf programları
Sanal sınıf programları