UNA VISIN GLOBAL DEL HAPMAP PROJECT VARIACIN EN

UNA VISIÓN GLOBAL DEL HAPMAP PROJECT

VARIACIÓN EN LA SECUENCIA HUMANA DE DNA
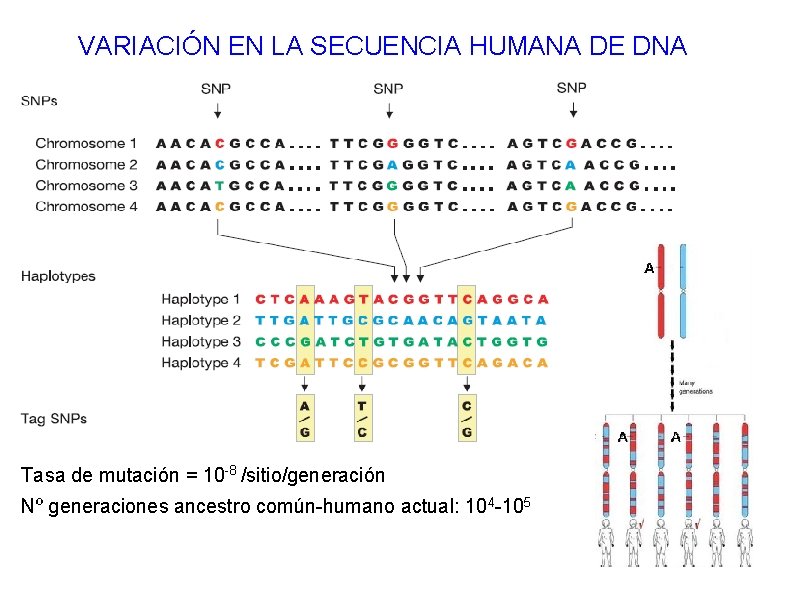
VARIACIÓN EN LA SECUENCIA HUMANA DE DNA Tasa de mutación = 10 -8 /sitio/generación Nº generaciones ancestro común-humano actual: 104 -105

CONSORCIO INTERNACIONAL HAPMAP ● Primer encuentro: Julio 2001 – Desarrollo del plan científico – Elección de las poblaciones – Discusión de las cuestiones éticas – Inicio formal del proyecto: Octubre 2002 ● ● ● Determinación grupos participantes Asignación de trabajo a cada grupo Fuentes de ayuda económica FASE I � Identificar patrones de variación genética y crear una base de datos pública y gratuita. � Descubrir factores genéticos de susceptibilidad a determinadas enfermedades. � No establece conexiones entre variantes particulares y enfermedades. � Aspectos básicos sobre hotspots de recombinación, bloques de DL y haplotipos. � Nuevos aspectos sobre la variación estructural y la recombinación e identifica loci que han estado sujetos a la selección natural durante la evolución humana.

HAPMAP PROJECT FASE II 2, 1 millones de nuevos SNPs �Mejora la selección de tag SNPs � Nuevos aspectos sobre las estructuras DL � Mejora la resolución del mapa genético y la localización de los hotspots � GRUPOS PARTICIPANTES
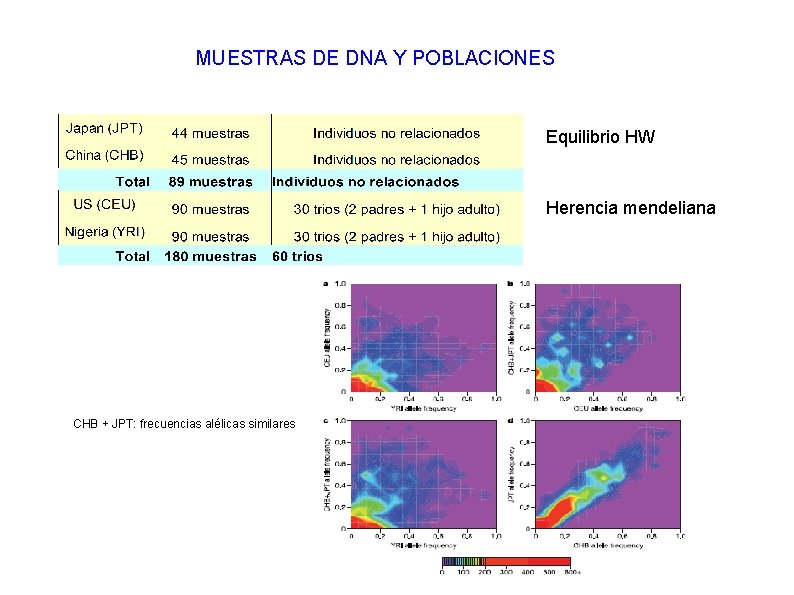
MUESTRAS DE DNA Y POBLACIONES Equilibrio HW Herencia mendeliana CHB + JPT: frecuencias alélicas similares

TÉCNICAS GENOTIPACIÓN Aproximación directa: variante individual ↔ enfermedad secuenciación de partes funcionales de genes candidatos Aproximación indirecta: marcador = conjunto de variaciones en la secuencia no requiere información sobre la funcional mayor eficiencia (90% de la variación) ● 5 técnicas de genotipación diferente: precisión, tasa éxito, coste ● SNPs obtenidos en cada centro, regenotipados en otros ● “random shotgun sequencing”: obtención de muchos SNPs ● Estratregia jerárquica: DL y haplotipos varían en el genoma
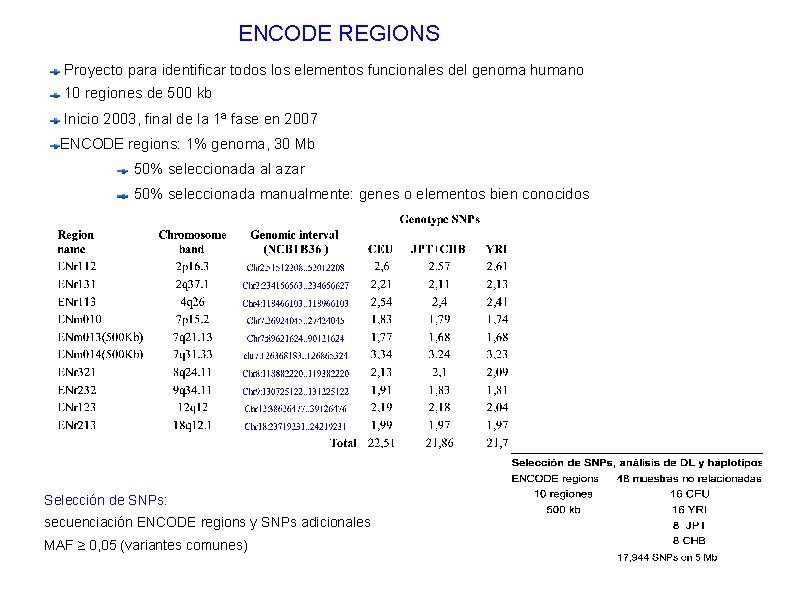
ENCODE REGIONS Proyecto para identificar todos los elementos funcionales del genoma humano 10 regiones de 500 kb Inicio 2003, final de la 1ª fase en 2007 ENCODE regions: 1% genoma, 30 Mb 50% seleccionada al azar 50% seleccionada manualmente: genes o elementos bien conocidos Selección de SNPs: secuenciación ENCODE regions y SNPs adicionales MAF ≥ 0, 05 (variantes comunes)
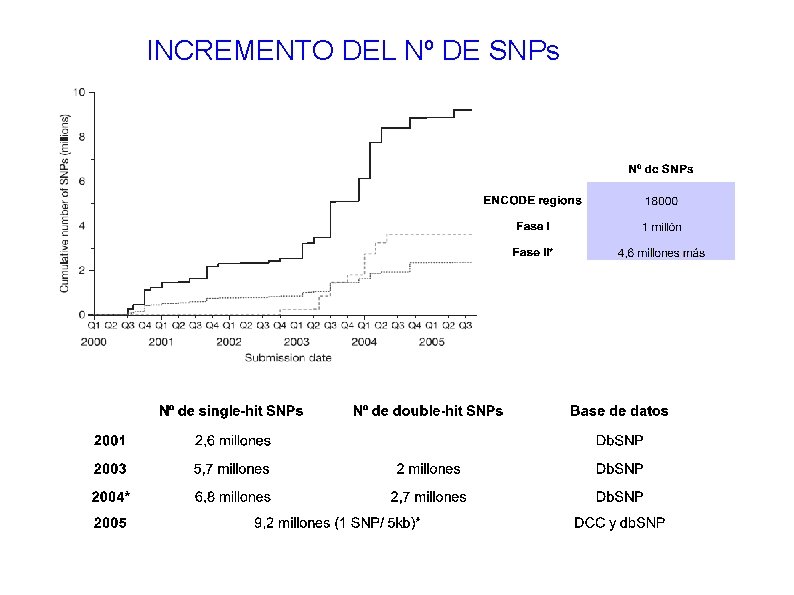
INCREMENTO DEL Nº DE SNPs

SNPs Espaciados regularmente CS mitocondrial: no recombinación CS Y: región no recombinante CS X: ↑ SNPs raros o monomórficos, ↓ comunes Precisión caracterización haplotipos: 99, 7%. Errores: 1 cada 8 Mb en CEU, 1 cada 3, 6 Mb en YRI CORRELACIÓN ENTRE SNPs Estudios de asociación Existe correlación entre dto. SNP y SNPs vecinos Proxy: SNP fuertemente correlacionado con uno o más SNPs Examinar uno es equivalente a examinar el otro.

DESEQUILIBRIO DE LIGAMIENTO ● Estructuras en bloque, hotspots ● Escala 1 -100 kb, discontínuas ● Determinadas por la tasa de recombinación ● Se han detectado 21, 617 hotspots (1 cada 122 kb) ● No todos los haplotipos interrumpidos por un hotspots ● CS X: haplotipos extensos ● Centrómero: no recombinación (100 SNPs / haplotipo) CS X: menor nº SNPs, menor tamaño población, recombinación restringida

RECOMBINACIÓN Y DL ● ↑ nº SNPs ↔ ↑ detección hotspots, precisión tasa recombinación ● Variación tasa recombinación => DL ● Exámen DL genoma : tasa rec. , composición sec. , caratc. génicas. ● ↓ telómeros ● ↑ centrómeros ● Correlación longitud CS (excepción CS X) ● Zonas + DL => + densidad génica ● Genes ir y neurofisiológicos: bajo DL (↑ diversidad génica, ambiente) ● DNA/RNA, daño, ciclo celular: alto DL.

TAG SNPS Son el conjunto de SNPs de un haplotipo genotipados para una enfermedad. Se seleccionan los SNPs con un alto grado de correlacion SNPs ENCODE reduccion 75 -90%, sin perdida de informaci'on Bajo numero de tag SNPs capta gran parte de la informacion MEJORA DE LA EFICACIA DEL ANALISIS Bajar el umbral de correlacion > menor cantidad de tag SNPs Elegir tags basados en el numero de SNPs que captan No claro si tag SNPs en una poblacion son utiles para otra Algunos autores dudan o estan en contra de este proyecto por este punto.
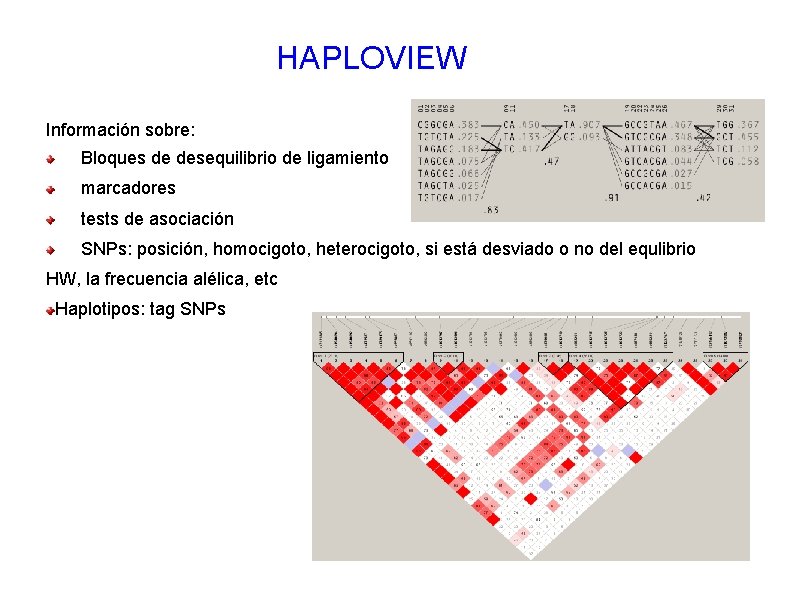
HAPLOVIEW Información sobre: Bloques de desequilibrio de ligamiento marcadores tests de asociación SNPs: posición, homocigoto, heterocigoto, si está desviado o no del equlibrio HW, la frecuencia alélica, etc Haplotipos: tag SNPs
- Slides: 14