Tecniche di estrazione Qualit del campione di partenza


































- Slides: 47

Tecniche di estrazione • Qualità del campione di partenza • Numero di campioni • Costo • Tempo • Semplicità e affidabilità • Automazione • Sicurezza

Materiale biologico di partenza Campionamento • Obiettivo del campionamento • Rappresentatività • Campioni singoli • Bulk di campioni

Matrici di partenza • Piante • Suolo • Matrici alimentari • Sangue • Feci

Matrici di partenza • Foglia: v giovane v sana • Suolo v polverizzato

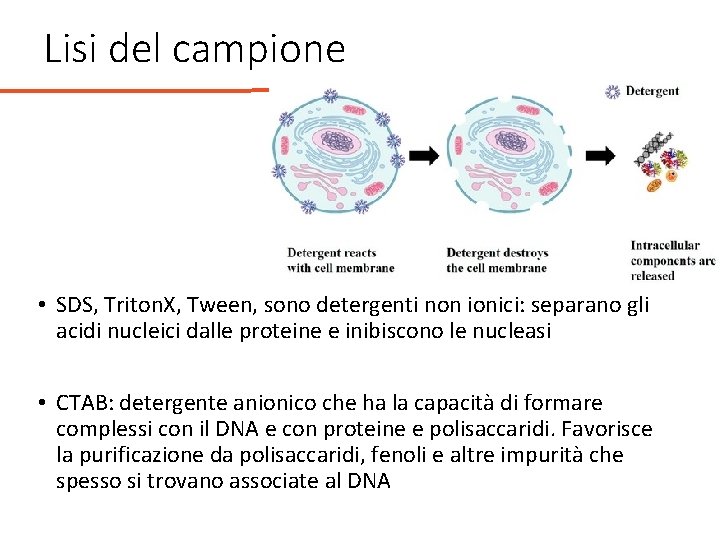
Lisi del campione • Buffer di lisi per rottura della parete e membrana cellulare • Sali caotropici • Detergenti • Lisi enzimatica • Lisi meccanica (sonicazione, elettroporazione, tissue lyser) • Bollitura • Raffreddamento

Lisi del campione • SDS, Triton. X, Tween, sono detergenti non ionici: separano gli acidi nucleici dalle proteine e inibiscono le nucleasi • CTAB: detergente anionico che ha la capacità di formare complessi con il DNA e con proteine e polisaccaridi. Favorisce la purificazione da polisaccaridi, fenoli e altre impurità che spesso si trovano associate al DNA

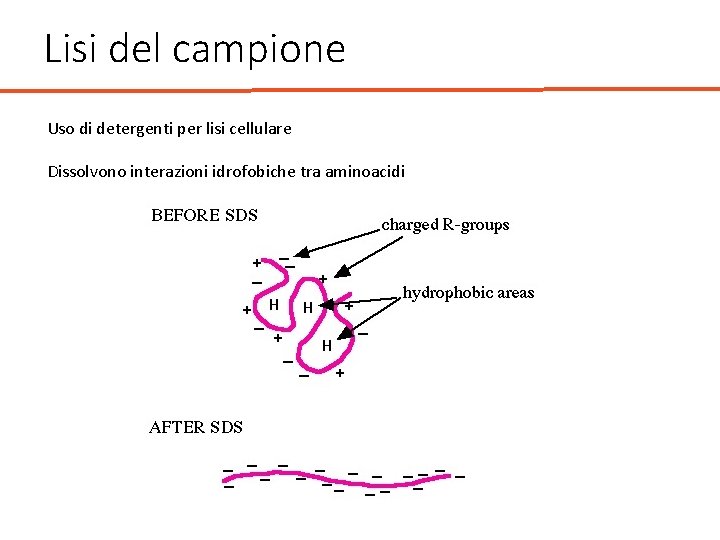
Lisi del campione Uso di detergenti per lisi cellulare Dissolvono interazioni idrofobiche tra aminoacidi

Lisi del campione Isotiocianato di guanidina e cloruro di guanidina • Sali caotropici • Provocano la rapida denaturazione delle proteine (dissoluzione della struttura terziaria e quaternaria). • Le interazioni idrofobiche vengono distrutte e aumenta la solubilità in acqua delle sostanze apolari. Questo destabilizza le membrane lipidiche e favorisce il rilascio di componenti cellulari

Lisi enzimatica • Enzimi per digerire la parete di peptidoglicani dei batteri: glicosidasi (lisozima), endopeptidasi e amidasi • Lisozima: distrugge la parete di batteri Gram + • Detergenti (Triton X 100) e agenti chelanti (EDTA) per distruggere la parete di Gram – (insensibili a lisozima) • Parete cellulare di lievito digerita da: β(1 -3) glucanasi, proteasi, β(1 -6) glucanasi, mannanasi and chitinasi • Funghi: Zymolase (ora abbandonato) e lyticase oppure rottura meccanica con biglie

Bollitura e Raffreddamento • Particolarmente utilizzata per estrarre DNA da matrici difficili quali batteri e funghi • Bollitura a circa 100°C per 15 min e successivo congelamento • Bagnetto • Microonde • Uso di azoto liquido • Abbattimento della temperatura a -80°C

Lisi meccanica Tissue lyser • La lisi del campione può essere effettuata a secco o con buffer di estrazione • La distruzione del campione avviene grazie ad una biglia di acciaio e a movimenti oscillatori ad alta frequenza • Tempo variabile a seconda della matrice di partenza • La lisi deve essere seguita da uno step di centrifugazione

Metodi più comuni di estrazione di DNA: CTAB • Per estrarre DNA da foglie, semi, alimenti trasformati, sangue • Procedura time-consuming • Utilizzo di prodotti chimici pericolosi (fenolo e cloroformio) • Uso di proteinasi K e mercaptoetanolo per rimuovere inibenti • Uso di RNase A e RNase T 1 per rimuovere l’RNA • Altri metodi più comuni si basano sull’uso di SDS e PVP, PEG, Na. Cl

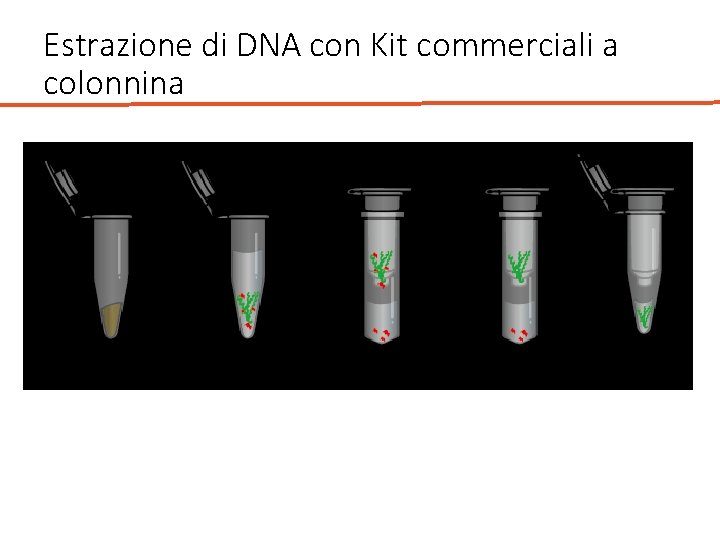
Estrazione di DNA con Kit commerciali a colonnina • Facilità d’uso, protocolli standardizzati e riproducibili • Matrici silicee (legame tra DNA e matrice in presenza di sali caotropici) • Possibilità di automatizzare l’estrazione • Uso di RNase A e Proteinasi K (no cloroformio) • Kit diversi a seconda della matrice di partenza

Estrazione di DNA con Kit commerciali a colonnina • 100 -200 mg di campione – lisi con tissue lyser o azoto liquido • Il buffer di lisi fa adsorbire il DNA alla membrana di silice • Il p. H acido e la concentrazione di sali del buffer assicurano che proteine e altri contaminanti non vengano trattenuti dalla membrana • In questo modo il DNA, legato alla membrana viene sottoposto a cicli di lavaggio • L’aggiunta del buffer di eluizione a p. H 9 fa staccare il DNA dalla membrana di silice

Estrazione di DNA con Kit commerciali a colonnina

Estrazione di DNA con Kit commerciali a colonnina Pro: • DNA altamente purificato • Resa di DNA ~ 100 -150 ng/µl • Lunghezza della catena di DNA Contro: • Molti trasferimenti dei campioni • Aumenta il rischio di contaminazioni • Time-consuming • Si possono processare un numero limitato di campioni

Estrazione di DNA con biglie magnetiche • Il DNA si lega alla superficie in silice presente nelle biglie magnetiche, in presenza di sali caotropici • Lavaggi con buffer contenti etanolo e eluizione in acqua distillata o low-salt buffer • Estrazione automatizzata con Biosprint 96 Qiagen o Kingfisher (Thermo Fisher )

Estrazione di DNA con biglie magnetiche • Quantità di campione di partenza: 30 -50 mg (tessuto vegetale), 200 mg (suolo o altra matrice) • Campione omogeneizzato in tissue lyser 5 min a 30 Hz, utilizzando una biglia da 5 mm in acciaio o una biglia da 3 mm in tungsteno, con buffer RLT (Guanidina tiocianato) o a secco • Estrazione avviene in piastre da 96 cosi composte: 1. Isopropanolo + campione + biglie magnetiche 2. Buffer RPW (cloruro di guanidina) 3. Etanolo al 96 -100% 4. Etanolo al 96 -100% 5. Tween 6. Acqua

Estrazione di DNA con biglie magnetiche Campione di partenza Lavaggio in Et. OH Biglie legate al DNA Separazione biglie+DNA da impurità Rilascio del DNA in acqua Campione finale purificato

Estrazione di DNA con biglie magnetiche Pro: • Procedura molto veloce e automatizzata • DNA altamente purificato • Bassa probabilità di contaminazione Contro: • Costoso • Resa piu bassa di DNA ~ 5 -20 ng/µl • DNA più frammentato

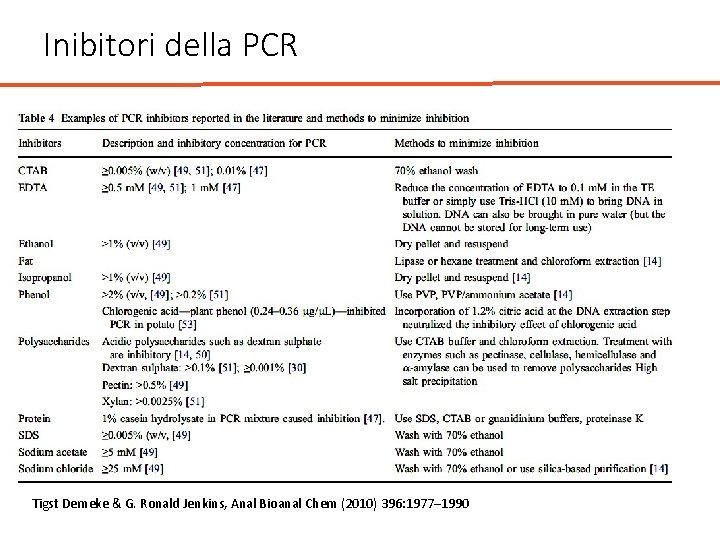
Inibitori della PCR • La presenza di inibitori possono ridurre l’efficienza e la riproducibilità della PCR • Gli inibitori possono interferire con la precipitazione e la denaturazione del DNA ma anche con la DNA polimerasi • Sequestrano gli ioni magnesio • Possono derivare dal tessuto dal quale si è estratto il DNA o dai reagenti stessi

Inibitori della PCR Tigst Demeke & G. Ronald Jenkins, Anal Bioanal Chem (2010) 396: 1977– 1990

Inibitori della PCR • Le sostanze umiche del suolo sono potenti inibitori della PCR • Detergenti ionici come SDS hanno una maggiore azione inibente rispetto ai detergenti non ionici come il Tween e il Triton X • Diluire il DNA aiuta a ridurre la concentrazione degli inibenti e il loro effetto negativo • L’aggiunta di BSA (Bovine serum albumin) riduce l’effetto degli inibenti e facilita la reazione di PCR

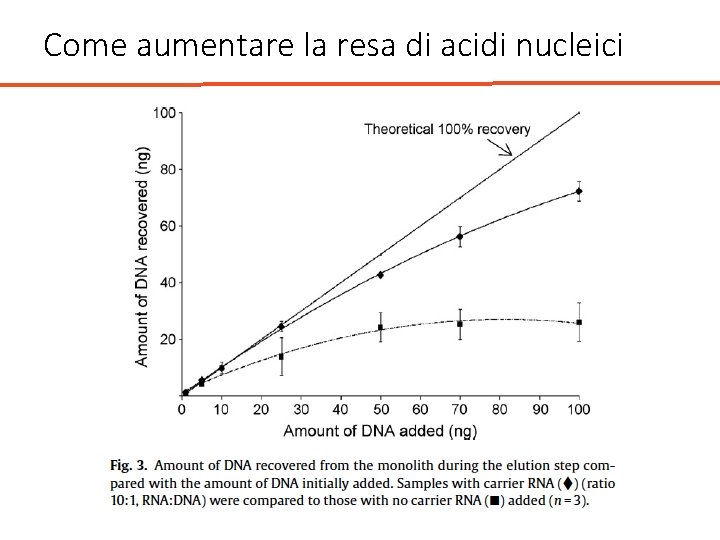
Come aumentare la resa di acidi nucleici Protocolli di clean-up (Genomic DNA Clean & Concentrator-10 Kit - Zymo Research) Uso di carrier inerti per migliorare l’efficienza di estrazione: - Glicogeno - RNA carrier (coda Poly-A) - Possono essere inclusi nelle soluzioni di lisi a base di sali caotropici - Aumentano la precipitazione del DNA o RNA - Riducono la degradazione dell’RNA virale da parte di RNase - Possono sovrastimare la quantità di acidi nucleici

Come aumentare la resa di acidi nucleici

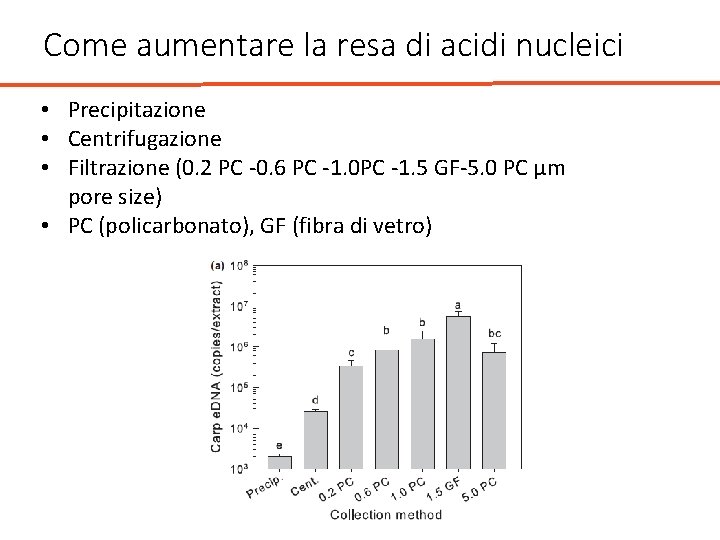
Come aumentare la resa di acidi nucleici • Precipitazione • Centrifugazione • Filtrazione (0. 2 PC -0. 6 PC -1. 0 PC -1. 5 GF-5. 0 PC µm pore size) • PC (policarbonato), GF (fibra di vetro)

Quantificazione del DNA • Elettroforesi su gel di agarosio • Real-Time PCR • Fluorimetro

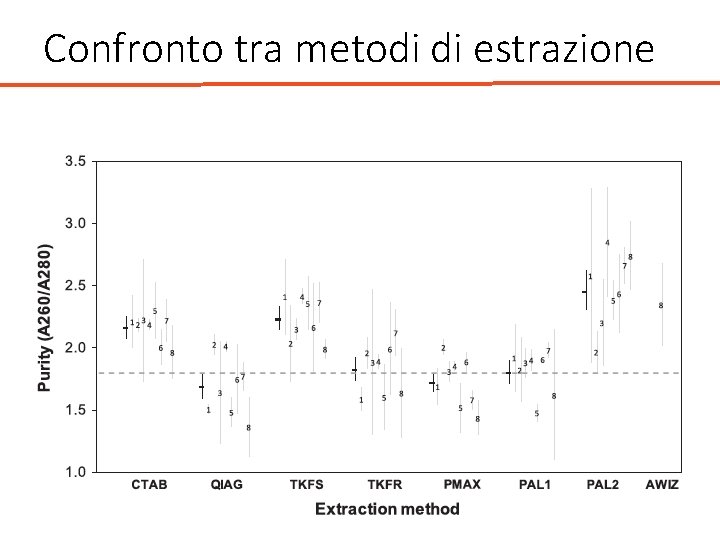
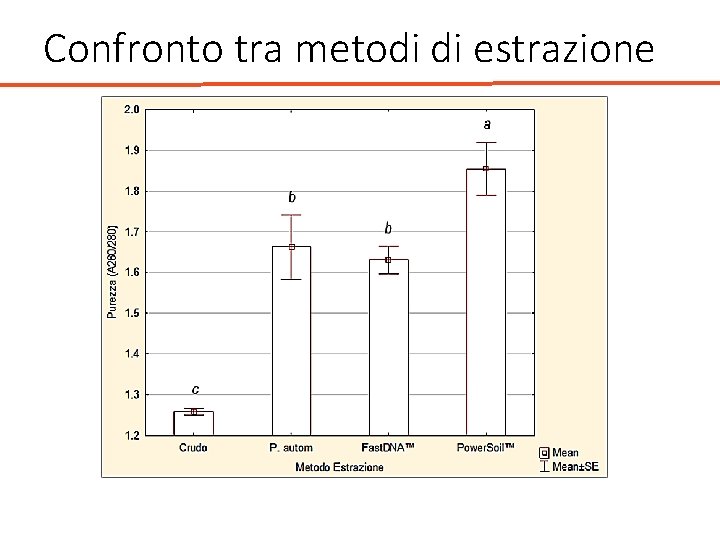
Quantificazione del DNA • Spettrofotometria (assorbanza a 260 nm) v. Purezza del DNA stimata dal rapporto 260/280 nm; il rapporto deve essere tra 1. 6 e 1. 8, se inferiore può esserci contaminazione proteica v. Rapporto 260/230 nm rileva possibili contaminanti organici che assorbono a una lunghezza d’onda di 230 nm v. Fenolo può assorbire tra 220 -240 nm v. Sali di guanidina assorbono a 230 nm

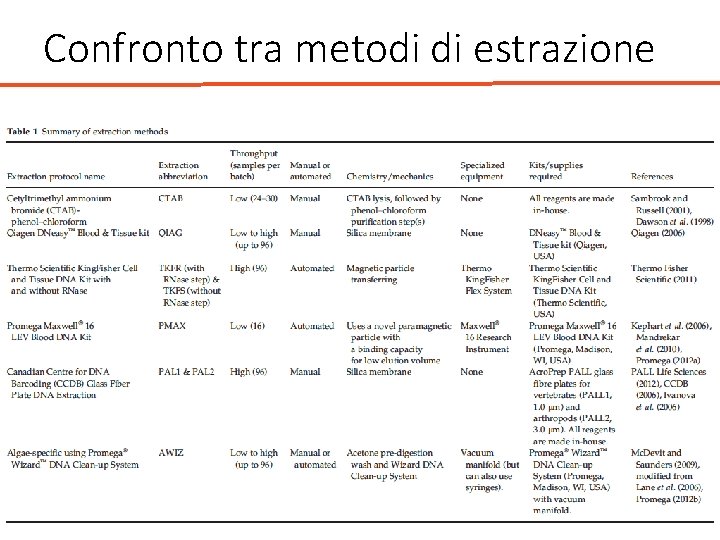
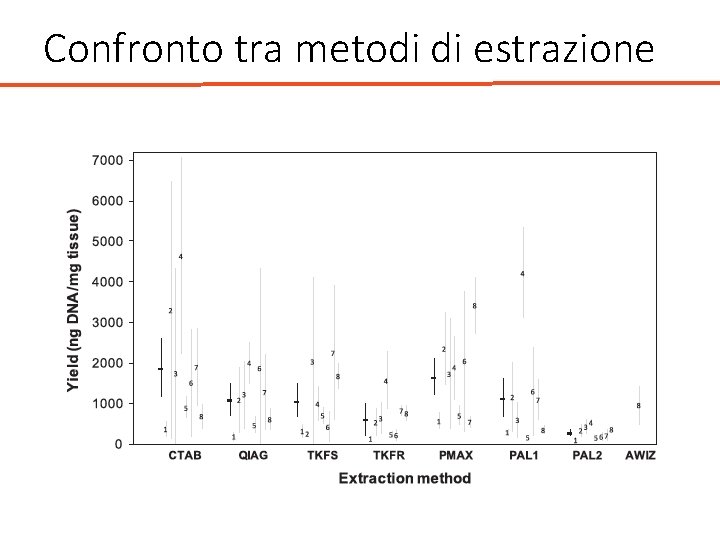
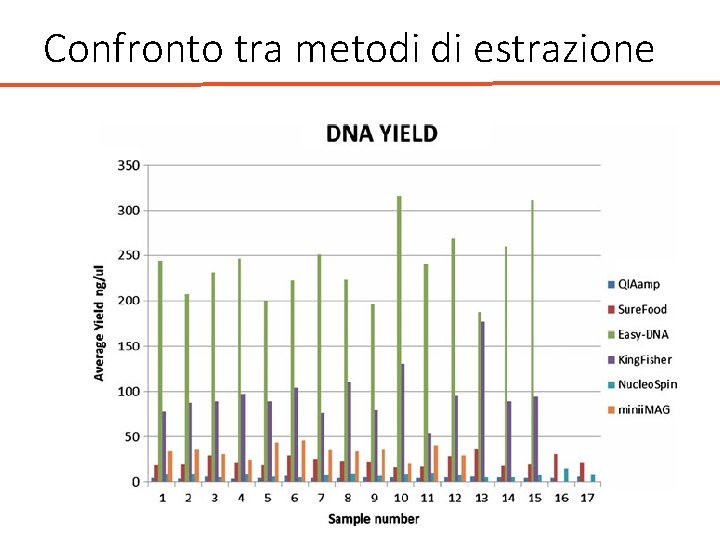
Confronto tra metodi di estrazione • Stabilire resa e purezza del DNA, efficacia del metodo, costo • 8 phyla: Annelida, Arthropoda, Cnidaria, Chordata, Echinodermata, Mollusca, Ochrophyta and Porifera.

Confronto tra metodi di estrazione

Confronto tra metodi di estrazione

Confronto tra metodi di estrazione

Confronto tra metodi di estrazione

Confronto tra metodi di estrazione

Confronto tra metodi di estrazione 1. Easy-DNA™Kit for genomic DNA Isolation (Invitrogen, Leek, The Netherlands). 2. Magne. Sil® KF, Genomic system(King. Fisher®) (Promega, Madison, WI, USA). 3. Sure. Food® PREP Campylobacter (Congen Biotechnologie Gmb. H, Berlin, Germany). 4. QIAamp Qiagen DNA stool mini kit (Qiagen, Hilden, Germany). 5. Nucli. SENS® Mini. MAG® (Bio. Mérieux SA, Lyon France). 6. Nucleo. Spin® Tissue (Macherey-Nagel Gmb. H & Co. KG, Du ren, Germany).

Confronto tra metodi di estrazione

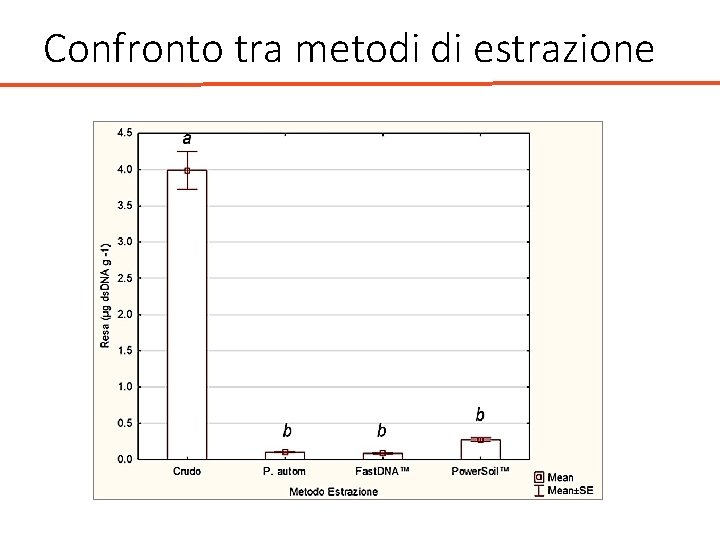
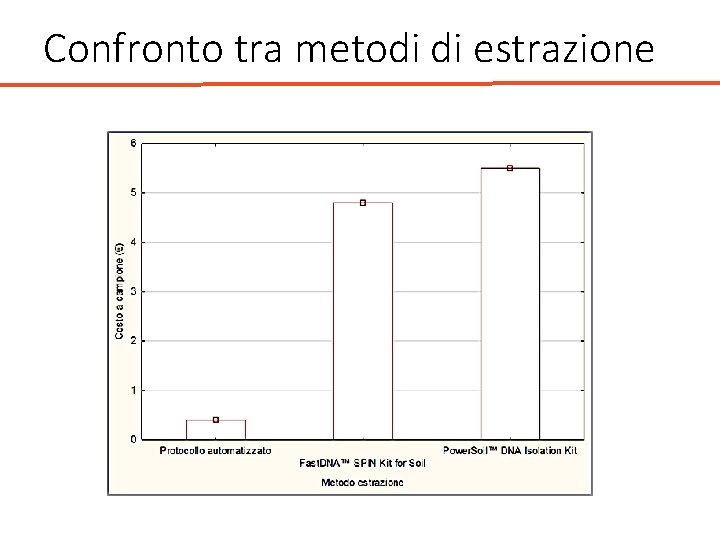
Confronto tra metodi di estrazione • Lavoro in corso di pubblicazione • Lo scopo del lavoro è stato quello di mettere a punto un protocollo automatizzato per l’estrazione e la purificazione del DNA genomico da suolo, al fine di ottenere un estratto idoneo per essere sottoposto ad analisi con metodi molecolari.

Confronto tra metodi di estrazione • Protocollo automatizzato • Fast. DNA™ SPIN Kit for Soil • Power. Soil™ DNA Isolation Kit

Confronto tra metodi di estrazione QUANTIFICAZIONE Fluorimetro Qubit™ 3. 0 ds. DNA GRADO DI PUREZZA Spettrofotometro UV/Vis Bio. Photometer™ Rapporto A 260/A 280

Confronto tra metodi di estrazione

Confronto tra metodi di estrazione

Confronto tra metodi di estrazione Arch_amo. A Nos. Z

Confronto tra metodi di estrazione

Estrazione di DNA da molluschi • Inibizione da polisaccaridi che vengono purificati e accumulati durante l’estrazione • Alcune specie sono piccole ed è difficile avere una resa di DNA sufficiente per sequenziamento • Kit più usati per analisi di PCR: 1. DNeasy Blood & Tissue Kit (Qiagen) 2. DNeasy Plant Mini Kit (Qiagen) 3. E. Z. N. A. Mollusc DNA Kit (Omega Bio-tek) • Tessuto omogeneizzato in buffer di lisi • Papanova et al. 2016 usa un protocollo con CTAB modificato (resa idonea per sequenziamento)

Estrazione di RNA da molluschi

Estrazione di RNA da molluschi

Estrazione di RNA • Estrazione automatizzata di RNA con biosprint 96: work in progress…
 Imballaggio economia aziendale
Imballaggio economia aziendale La qualit
La qualit Qualit eval
Qualit eval La qualit
La qualit Tavola dei numeri casuali
Tavola dei numeri casuali Fiche autocontrole qualit'enr
Fiche autocontrole qualit'enr Estrazione del lotto
Estrazione del lotto Metodi e tecniche del servizio sociale slide
Metodi e tecniche del servizio sociale slide Metodi e tecniche del lavoro di gruppo
Metodi e tecniche del lavoro di gruppo Tecniche di gestione del comportamento
Tecniche di gestione del comportamento Metodi di separazione dei miscugli estrazione
Metodi di separazione dei miscugli estrazione Estrazione solido-liquido - esercizi svolti
Estrazione solido-liquido - esercizi svolti Dov'è contenuta la clorofilla
Dov'è contenuta la clorofilla Probabilità estrazione contemporanea
Probabilità estrazione contemporanea Kiwi dna extraction
Kiwi dna extraction Imbuto separatore relazione
Imbuto separatore relazione Estrazione accelerata con solvente
Estrazione accelerata con solvente Estrazione con solvente zanichelli
Estrazione con solvente zanichelli Fenolo cloroformio alcool isoamilico
Fenolo cloroformio alcool isoamilico Estrazione solido liquido
Estrazione solido liquido Cortisone dopo estrazione dente
Cortisone dopo estrazione dente Tecniche di catalogazione
Tecniche di catalogazione Metodi elettrochimici
Metodi elettrochimici Tecniche di socializzazione
Tecniche di socializzazione Tecniche associate al pensiero computazionale
Tecniche associate al pensiero computazionale Tecniche analitiche di laboratorio
Tecniche analitiche di laboratorio Tecniche di separazione chimica
Tecniche di separazione chimica Animazione
Animazione Testo narrativo schema
Testo narrativo schema Jiu jitsu tecniche
Jiu jitsu tecniche Tecniche narrative svevo
Tecniche narrative svevo Tecniche investigative carabinieri
Tecniche investigative carabinieri Funzione mimetica dello spazio
Funzione mimetica dello spazio Struttura narrativa tipologie
Struttura narrativa tipologie Linguaggi e tecniche comunicative non verbali
Linguaggi e tecniche comunicative non verbali Tecniche di conduzione dei gruppi slide
Tecniche di conduzione dei gruppi slide Come e quando matura la conversione di verga il verismo
Come e quando matura la conversione di verga il verismo Presentazione dei personaggi
Presentazione dei personaggi Tecniche narrative schema
Tecniche narrative schema Teorie e tecniche della televisione
Teorie e tecniche della televisione Tecniche di purificazione
Tecniche di purificazione Testo narrativo letterario
Testo narrativo letterario Comunicazione organizzativa aziendale
Comunicazione organizzativa aziendale Tecniche di rappresentazione dello spazio
Tecniche di rappresentazione dello spazio Tecniche traduzione inglese
Tecniche traduzione inglese Tecniche pittoriche moderne
Tecniche pittoriche moderne Tecniche di comunicazione efficace slide
Tecniche di comunicazione efficace slide Misure tecniche, organizzative e procedurali
Misure tecniche, organizzative e procedurali