TALLER DE APLICACIONES PRCTIAS 1 Dra Mara Isabel

TALLER DE APLICACIONES PRÁCTIAS 1 Dra. María Isabel Fonseca
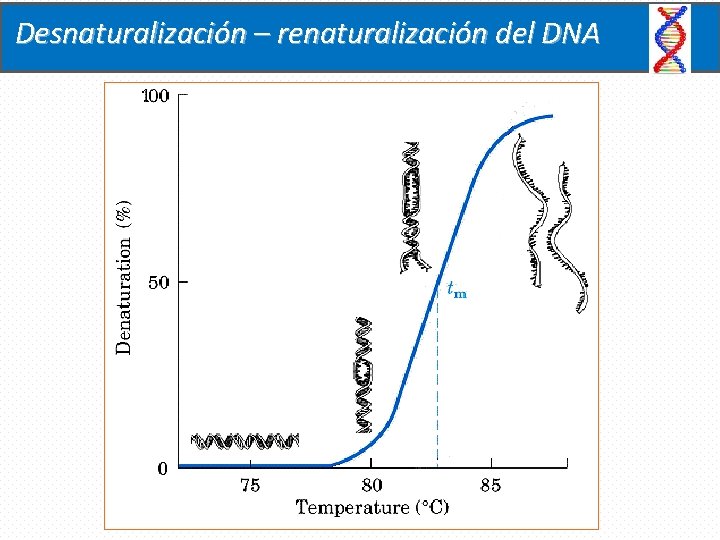
Desnaturalización – renaturalización del DNA

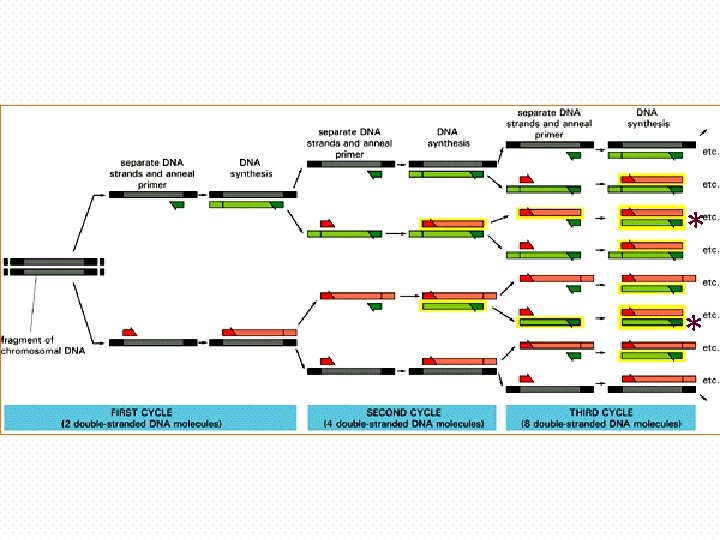
Polymerase Chain Reaction (PCR) Es objetivo de la PCR obtener un gran número de copias de un fragmento de ADN particular, partiendo de un mínimo; en teoría basta partir de una única copia de ese fragmento original, o molde. Se llama "cadena", porque los productos de la primera reacción se convierten en sustratos de la siguiente, y así sucesivamente. Kary Banks Mullis Premio novel de Química en 1993

Componentes necesarios para realizar PCR ADN target: que contiene la secuencia que se desea amplificar Dos de cebadores (S y AS) que delimitan la secuencia que se va a amplificar d. NTPs sustratos para la síntesis ADN polimerasa termoestable que cataliza la reacción Iones Mg++ Cofactor de la enzima Sn buffer mantiene el p. H y la fuerza iónica en la reacción necesario para una adecuada actividad del enzima agua

Controles utilizados en la PCR + -
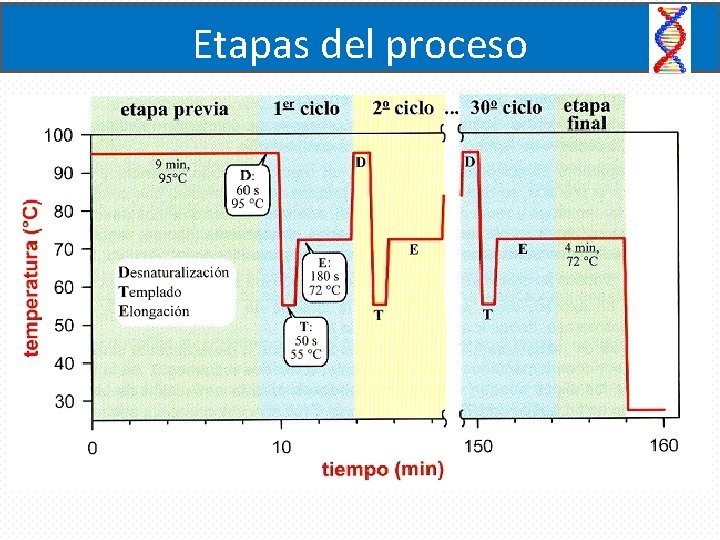
Etapas del proceso

El equipo utilizado para realizar la PCR Tubos con PCR TERMOCICLADOR
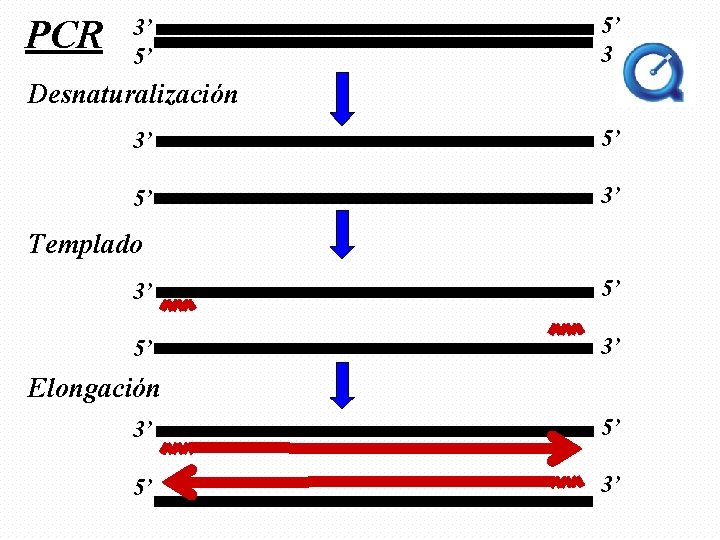
PCR 3’ 5’ 5’ 3’ Desnaturalización 3’ 5’ 5’ 3’ Templado 3’ 5’ 5’ 3’ Elongación 3’ 5’ 5’ 3’
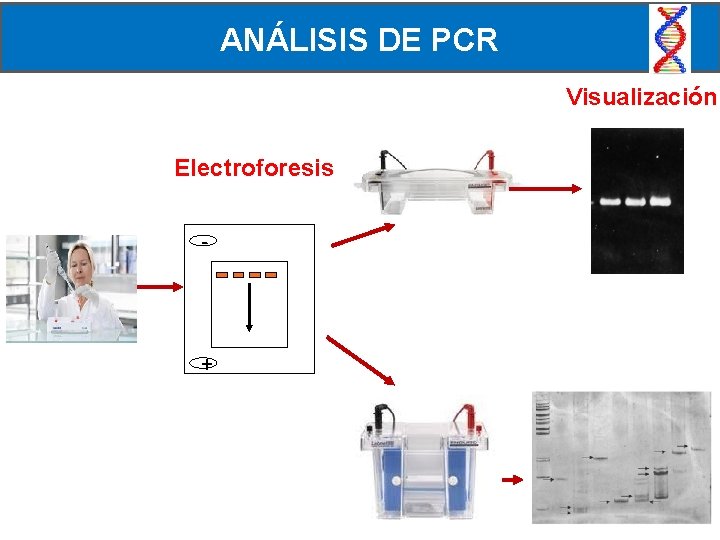
El equipo utilizado para realizar ANÁLISIS DE PCR la PCR Visualización Electroforesis - +

Marcadores Moleculares

MARCADORES MOLECULARES v LOS MARCADORES MOLECULARES SE DEFINEN COMO TODO O CUALQUIER FENOTIPO MOLECULAR ORIUNDO DE UN GEN QUE SE EXPRESA, COMO EN EL CASO DE LAS ISOENZIMAS, O DE UN SEGMENTO ESPECÍFICO DE ADN (CORRESPONDIENTE A REGIONES QUE SE EXPRESAN O NO DEL GENOMA). v PUEDEN SER MARCADORES MOLECULARES LAS PROTEÍNAS (Ag E ISOENZIMAS) Y EL ADN (REGIONES CODIFICANTES O NO CODIFICANTES)
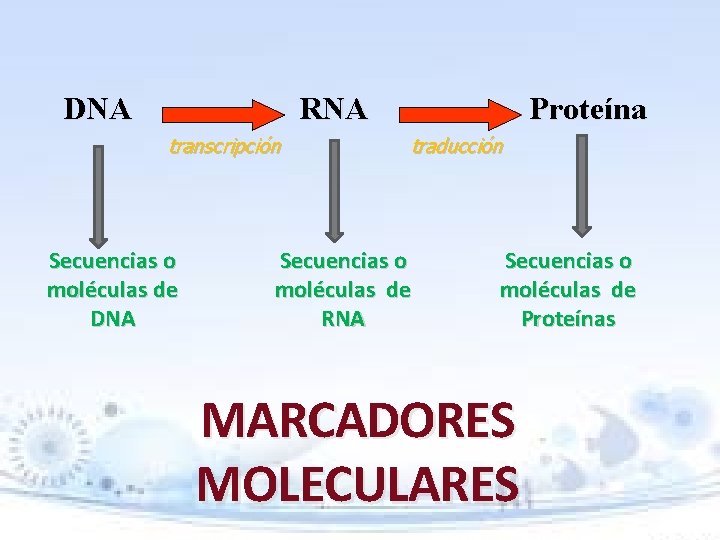
DNA RNA transcripción Secuencias o moléculas de DNA Proteína traducción Secuencias o moléculas de RNA Secuencias o moléculas de Proteínas MARCADORES MOLECULARES

Marcadores moleculares • Secuencias conocidas • Fáciles de detectar y seguir en la población • Codificantes o no codificantes • Secuencias conservadas • Secuencias polimórficas

Secuencias polimórficas • Variabilidad genómica poblacional • Análisis de diversidad • Identificación de individuos • El grado de polimorfismo permite distinguir la utilidad del marcador
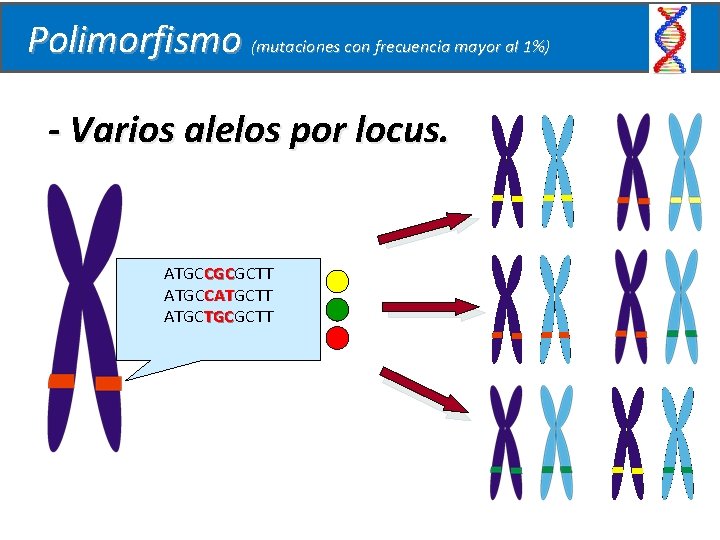
Polimorfismo (mutaciones con frecuencia mayor al 1%) - Varios alelos por locus. ATGCCGCGCTT CGC ATGCCATGCTT CAT ATGCTGCGCTT TGC
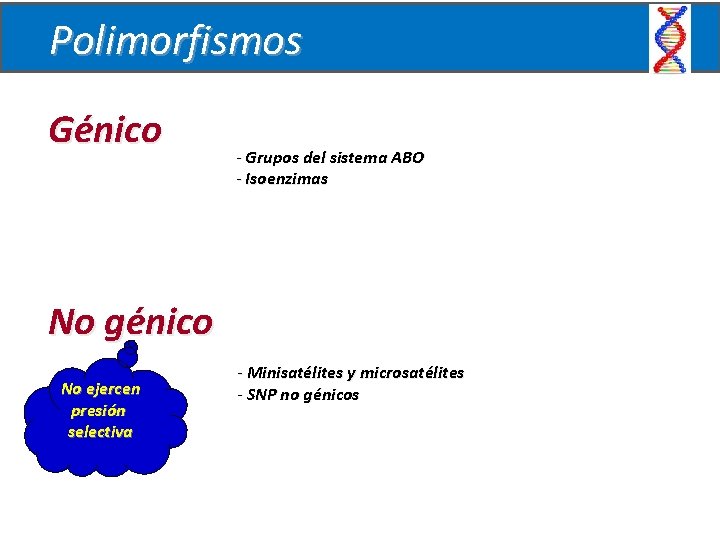
Polimorfismos Génico - Grupos del sistema ABO - Isoenzimas No génico No ejercen presión selectiva - Minisatélites y microsatélites - SNP no génicos

Secuencias relativamente conservadas • Grado de conservación y disponibilidad determina la utilidad (ej. Especies, variedades, formas especiales) • Regiones STS para mapear genomas • Regiones ETS para mapear transcriptomas • Familias génicas y genes homólogos
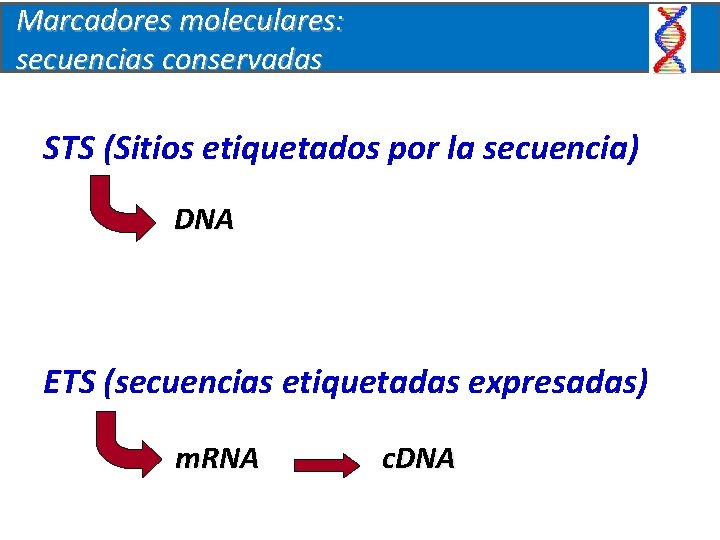
Marcadores moleculares: secuencias conservadas STS (Sitios etiquetados por la secuencia) DNA ETS (secuencias etiquetadas expresadas) m. RNA c. DNA

Marcadores moleculares: secuencias polimorficas SNP (polimorfismo de simple nucleótido) STR (microsatélites) VNTR (minisatélites) RFLP (sitios de cortes de enzimas de restricción) RAPD (fragmentos amplificados al azar)

MARCADORES MOLECULARES (MM) ¿Secuencias conocidas o desconocidas? Variabilidad - polimorfismo Factibilidad técnica de reconocerla
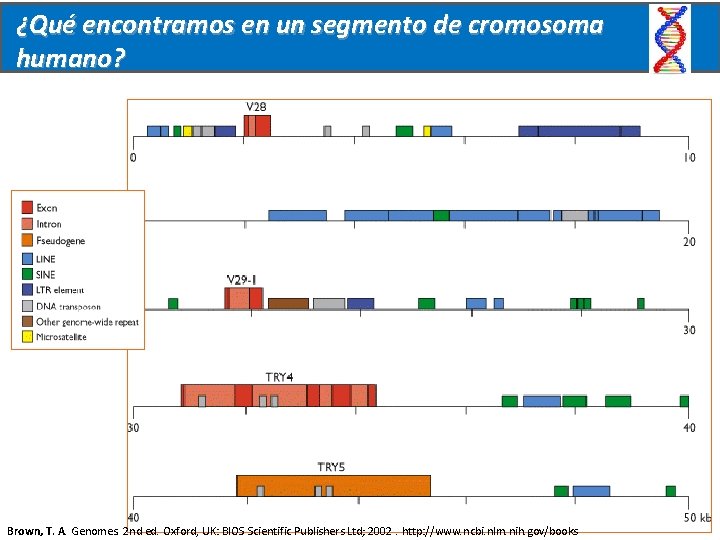
¿Qué encontramos en un segmento de cromosoma humano? Brown, T. A. Genomes. 2 nd ed. Oxford, UK: BIOS Scientific Publishers Ltd; 2002. http: //www. ncbi. nlm. nih. gov/books
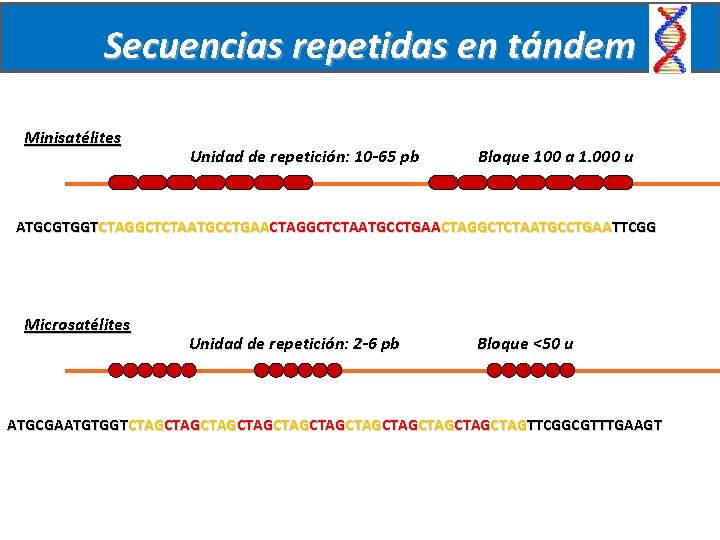
Secuencias repetidas en tándem Minisatélites Unidad de repetición: 10 -65 pb Bloque 100 a 1. 000 u ATGCGTGGTCTAGGCTCTAATGCCTGAACTAGGCTCTAATGCCTGAATTCGG Microsatélites Unidad de repetición: 2 -6 pb Bloque <50 u ATGCGAATGTGGTCTAGCTAGCTAGCTAGCTAGCTAGTTCGGCGTTTGAAGT
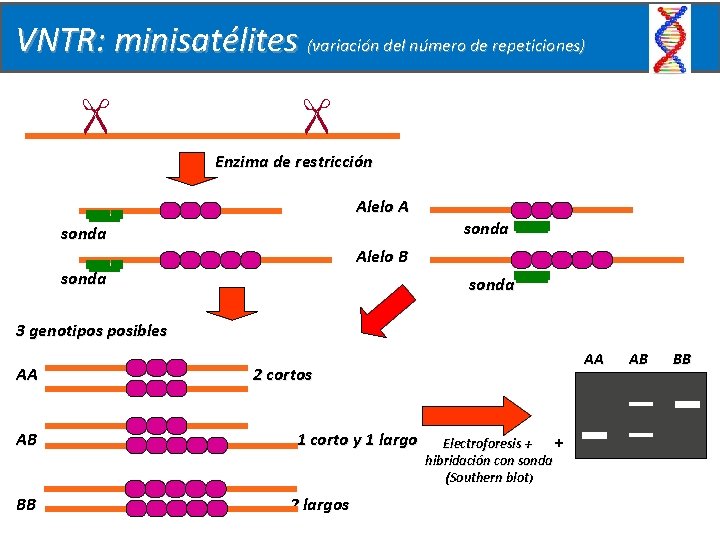
VNTR: minisatélites (variación del número de repeticiones) Enzima de restricción Alelo A sonda Alelo B sonda 3 genotipos posibles AA AB BB AA 2 cortos 1 corto y 1 largo 2 largos + Electroforesis + hibridación con sonda (Southern blot) AB BB
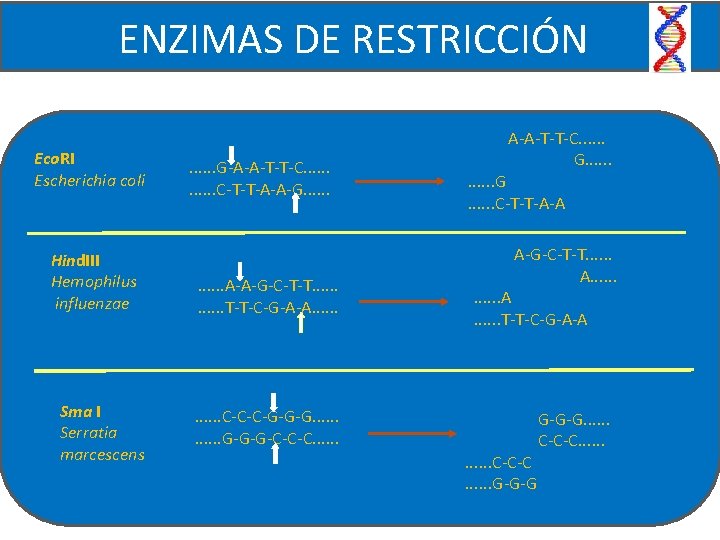
ENZIMAS DE RESTRICCIÓN Eco. RI Escherichia coli Hind. III Hemophilus influenzae Sma I Serratia marcescens . . . G-A-A-T-T-C. . . C-T-T-A-A-G. . . A-A-G-C-T-T. . . T-T-C-G-A-A. . . C-C-C-G-G-G. . . G-G-G-C-C-C. . . A-A-T-T-C. . . G. . . C-T-T-A-A A-G-C-T-T. . . A. . . T-T-C-G-A-A G-G-G. . . C-C-C. . . G-G-G
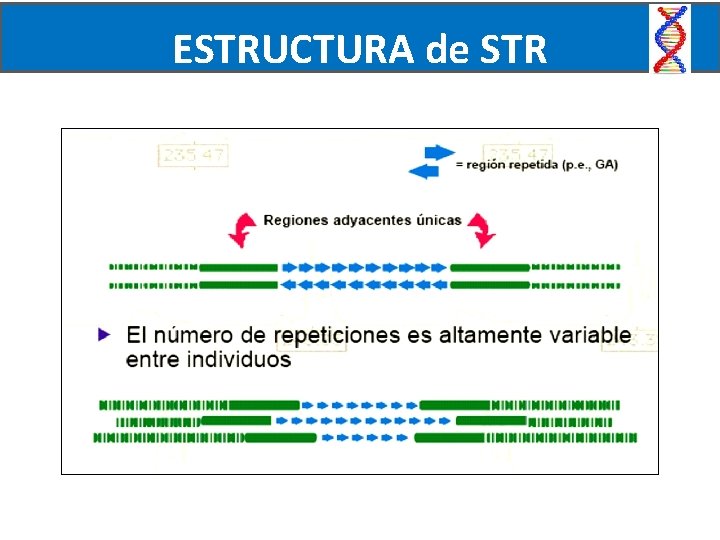
ESTRUCTURA de STR
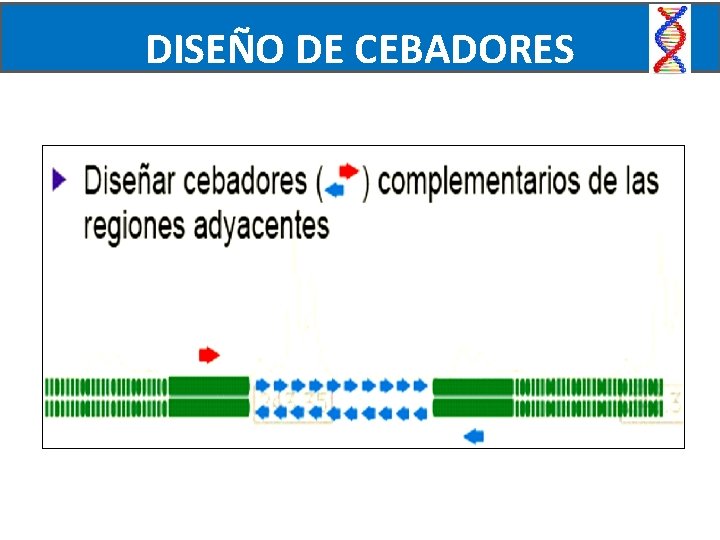
DISEÑO DE CEBADORES
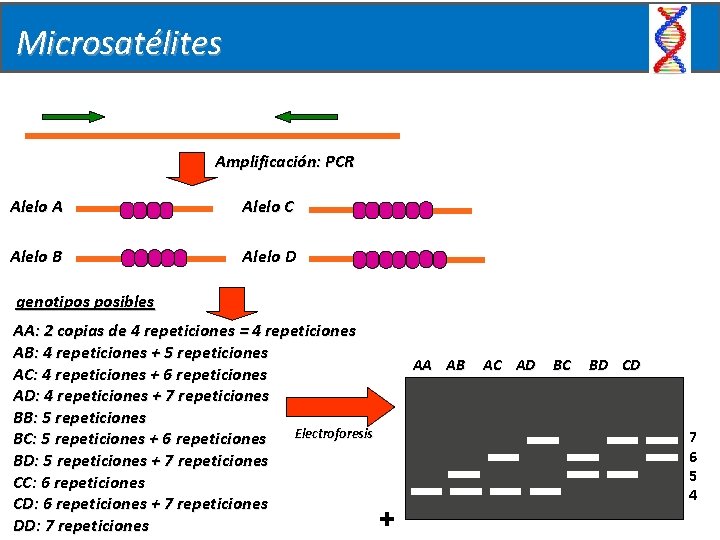
Microsatélites Amplificación: PCR Alelo A Alelo C Alelo B Alelo D genotipos posibles AA: 2 copias de 4 repeticiones = 4 repeticiones AB: 4 repeticiones + 5 repeticiones AC: 4 repeticiones + 6 repeticiones AD: 4 repeticiones + 7 repeticiones BB: 5 repeticiones Electroforesis BC: 5 repeticiones + 6 repeticiones BD: 5 repeticiones + 7 repeticiones CC: 6 repeticiones CD: 6 repeticiones + 7 repeticiones DD: 7 repeticiones AA AB + AC AD BC BD CD 7 6 5 4
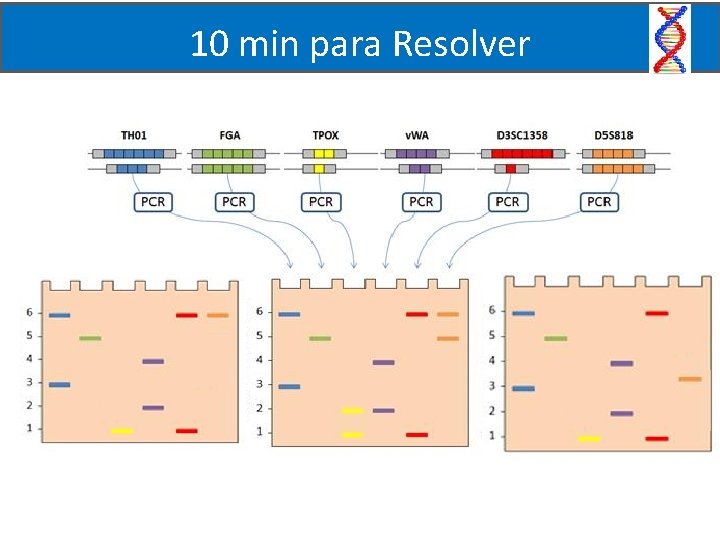
10 min para Resolver
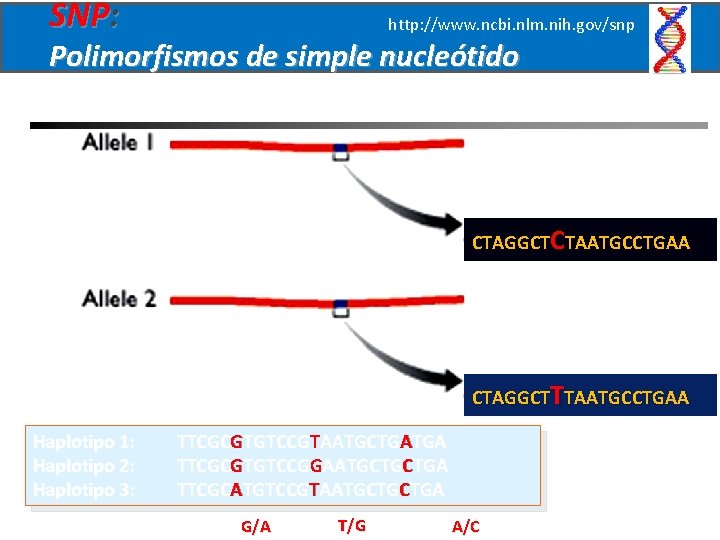
SNP: http: //www. ncbi. nlm. nih. gov/snp Polimorfismos de simple nucleótido CTAGGCTCTAATGCCTGAA CTAGGCTTTAATGCCTGAA Haplotipo 1: Haplotipo 2: Haplotipo 3: TTCGCGTGTCCGTAATGCTGATGA TTCGCGTGTCCGGAATGCTGCTGA TTCGCATGTCCGTAATGCTGCTGA G/A T/G A/C
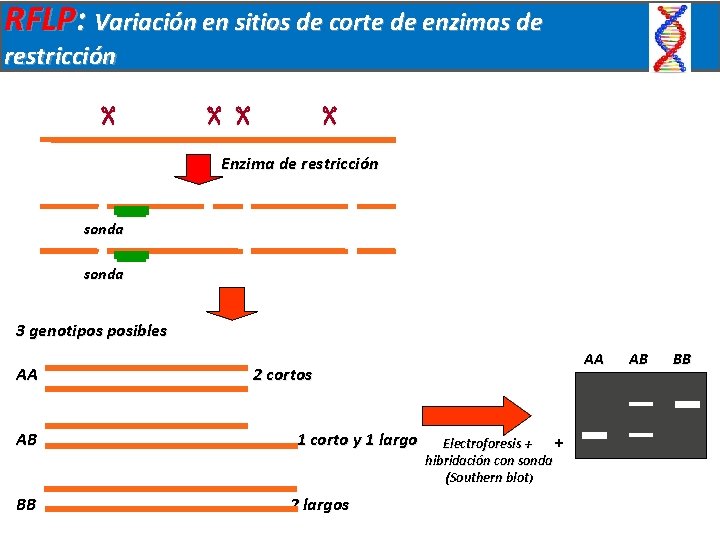
RFLP: Variación en sitios de corte de enzimas de restricción Enzima de restricción sonda 3 genotipos posibles AA AB BB AA 2 cortos 1 corto y 1 largo 2 largos + Electroforesis + hibridación con sonda (Southern blot) AB BB
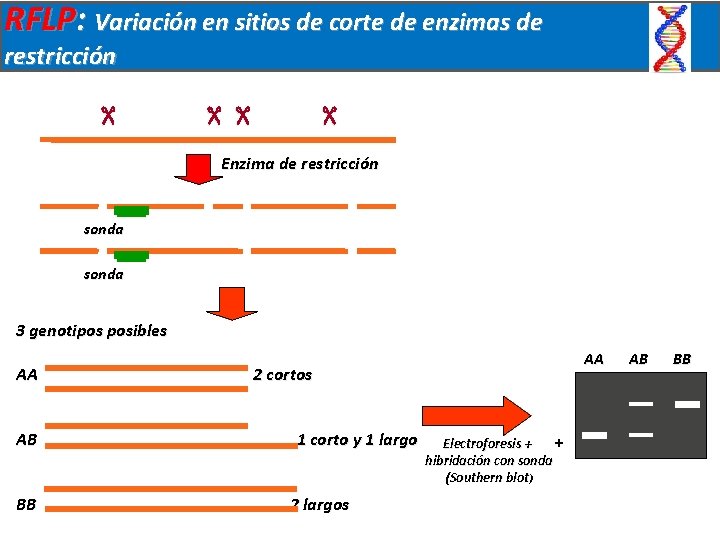
RFLP: Variación en sitios de corte de enzimas de restricción Enzima de restricción sonda 3 genotipos posibles AA AB BB AA 2 cortos 1 corto y 1 largo 2 largos + Electroforesis + hibridación con sonda (Southern blot) AB BB
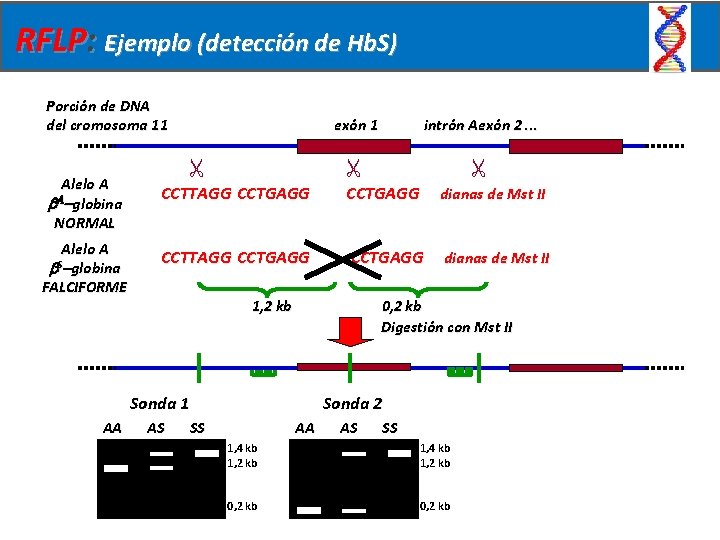
RFLP: Ejemplo (detección de Hb. S) Porción de DNA del cromosoma 11 intrón Aexón 2. . . Alelo A b. A-globina exón 1 CCTTAGG CCTGAGG dianas de Mst II NORMAL Alelo A b. S-globina FALCIFORME 1, 2 kb 0, 2 kb Digestión con Mst II Sonda 1 AA AS Sonda 2 SS AA AS SS 1, 4 kb 1, 2 kb 0, 2 kb
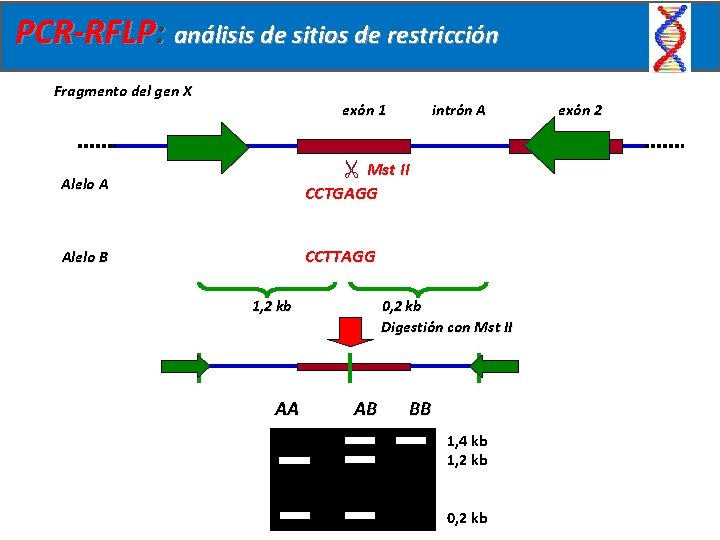
PCR-RFLP: análisis de sitios de restricción Fragmento del gen X exón 1 intrón A Alelo B CCTTAGG Alelo A Mst II CCTGAGG 1, 2 kb AA 0, 2 kb Digestión con Mst II AB BB 1, 4 kb 1, 2 kb 0, 2 kb exón 2

15 min para Resolver El polimorfismo A>G en el codón 655 del gen que codifica para la región transmenbrana del receptor HER 2 provoca un cambio aminoacídico de Ile>Val que impulsaría la actividad tirosina quinasa del receptor de manera constitutiva. Como HER 2 es el recepetor del factor de crecimiento, su actividad constitutiva implicaría que envié señales al nucleo activando genes involucrados en la proliferación celular. Las células con este polimorfismo crecen descontroladamente generando tumores que pueden derivar en un cáncer epitelial u otro dependiendo de donde se produce el crecimiento descontrolado. Producto de PCR: 300 pb En sitio de reconocimiento para la enzima se encuentra G originado un fragmento de 150 y otro de 200 pb. Esquematice todos los posibles resultados que obtendría al realizar la digestión con la enzima de restricción definiendo todos los genotipos posibles.

Marcadores para genomas desconocidos • Cuando no se disponen de datos genómicos. • Asociación fortuita con fenotipos, pero de base genómica. • Identificación de especies, variedades, grupos poblacionales acorde con el grado de polimorfismo
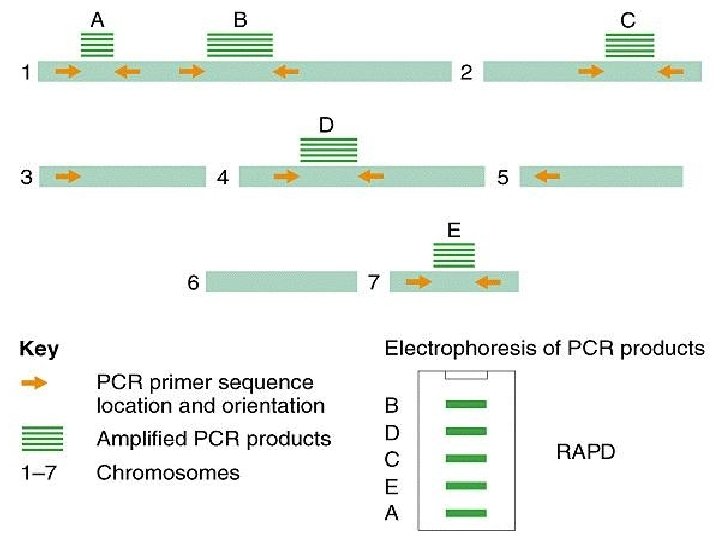
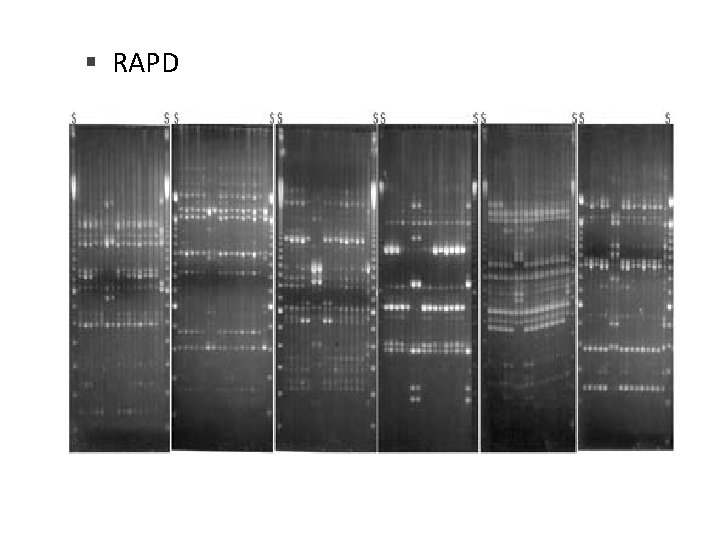
RAPD
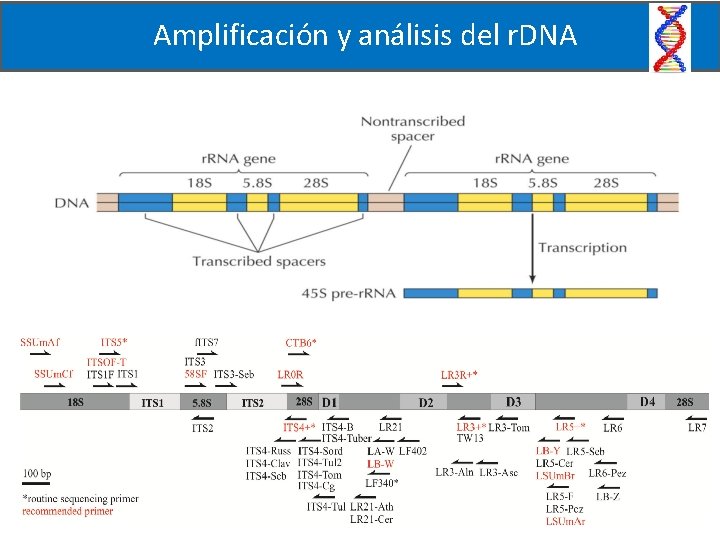
Amplificación y análisis del r. DNA
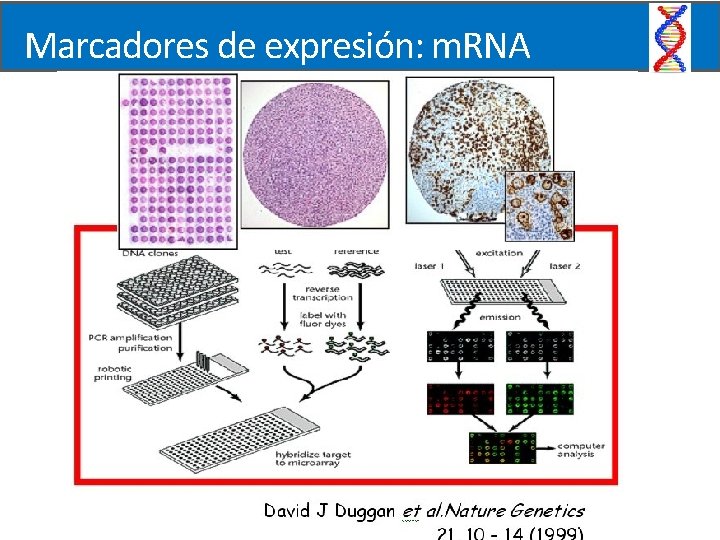
Marcadores de expresión: m. RNA

Aplicaciones: • análisis filogenético • selección asistida por marcador • pruebas de paternidad y trazabilidad de los alimentos • Estudios de linfomas, sarcomas y otros tumores. • Estudios de genes supresores de tumores y oncogenes. • Agentes infecciosos • Estudios de identidad • etc…

…en conclusión • Los marcadores moleculares constituyen una herramienta de la Biología Molecular que permiten el análisis del genoma a través de diferentes estrategias.

Enlaces que se utilizaron en el diseño Modern Genetic Analysis. Griffiths, Anthony J. F. ; Gelbart, William M. ; Miller, Jeffrey H. ; Lewontin, Richard C. New York: W. H. Freeman & Co. ; c 1999. Molecular Biology of the Cell. 3 rd ed. Alberts, Bruce; Bray, Dennis; Lewis, Julian; Raff, Martin; Roberts, Keith; Watson, James D. New York and London: Garland Publishing; c 1994. Molecular Cell Biology. 4 th ed. Lodish, Harvey; Berk, Arnold; Zipursky, S. Lawrence; Matsudaira, Paul; Baltimore, David; Darnell, James E. New York: W. H. Freeman & Co. ; c 1999. Human Molecular Genetics 2. 2 nd ed. Strachan, Tom and Read, Andrew P. Oxford, UK: BIOS Scientific Publishers Ltd; 1999. Genomes. 2 nd ed. Brown, T. A. Oxford, UK: BIOS Scientific Publishers Ltd; 2002
- Slides: 43