Summaries of of Affymetrix Gene Chip probe level

Summaries of of Affymetrix Gene. Chip probe level data 2002
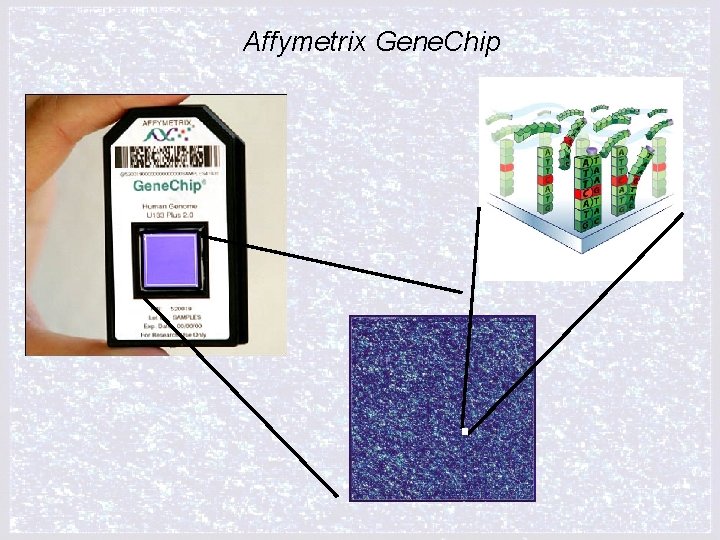
Affymetrix Gene. Chip
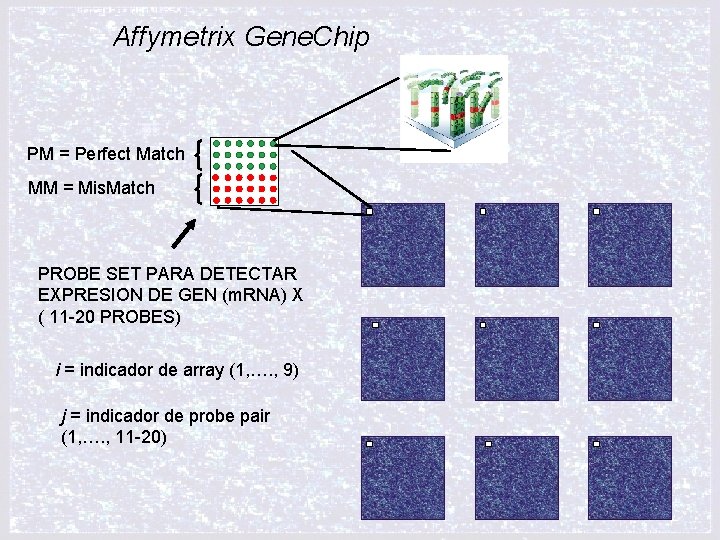
Affymetrix Gene. Chip PM = Perfect Match MM = Mis. Match PROBE SET PARA DETECTAR EXPRESION DE GEN (m. RNA) X ( 11 -20 PROBES) i = indicador de array (1, …. , 9) j = indicador de probe pair (1, …. , 11 -20)

4 MODELOS DE MEDICIÓN DE LA EXPRESIÓN GÉNICA EN Gene. Chip VERSIÓN VIEJA DEL SOFTWARE DE ANÁLISIS DE AFFYMETRIX MAS 5. 0 (ÚLTIMA VERSIÓN DEL SOFTWARE DE ANÁLISIS DE AFFYMETRIX) d. Chip RMA

4 MODELOS DE MEDICIÓN DE LA EXPRESIÓN GÉNICA EN Gene. Chip MODELOS COMPARADOS VERSIÓN VIEJA DEL SOFTWARE DE ANÁLISIS DE AFFYMETRIX MAS 5. 0 (ÚLTIMA VERSIÓN DEL SOFTWARE DE ANÁLISIS DE AFFYMETRIX) d. Chip RMA

VERSIÓN VIEJA DEL SOFTWARE DE ANÁLISIS DE AFFYMETRIX PROMEDIO DE LAS DIFERENCIAS ENTRE PARES DE PROBES PMij – MMij , j =1, …. . , J para cada arreglo i Se utiliza un promedio robusto para proteger contra outliers MODELO ESTADÍSTICO Diferencia de Promedios (AD): PMij – MMij = θi + εij , j =1, …. . , J θi = cantidad de expresión en el arreglo i εij = error PERO……………… AD es apropiada cuando la varianza del término de error εij es igual para j =1, …J εij ≠ para j =1, …J en datos a nivel probe de Gene. Chip mayor intensidad media de la probe mayor varianza!!!!

VERSIÓN NUEVA DEL SOFTWARE DE ANÁLISIS DE AFFYMETRIX (MAS 5. 0) TRANSFORMACIÓN LOGARÍTMICA : REDUCE LA DEPENDENCIA EN LA VARIANZA DE LA MEDIA SEÑAL: anti-log del promedio robusto (Tukey biweight) de los valores: log ( PMij – Ctij ) , j =1, …. . , J para cada arreglo i CT = MM , cuando MM < PM CT = CT < PM cuando MM ≥ PM (evita log de números < 0) MODELO ESTADÍSTICO log( PMij – CTij )= log( θi ) + εij , j =1, …. . , J PERO……………… varianza de una probe particular entre arreglos < < < varianza entre probes del probe set Efecto fuerte de afinidad de probe!!!!!

d. Chip SOFTWARE PACKAGE TIENE EN CUENTA EL EFECTO AFINIDAD DE PROBE ENTRE ARREGLOS CUANDO ESTAN DISPONIBLES MUCHOS ARREGLOS POR EXPERIMENTO MODELO MULTIPLICATIVO PMij – MMij = θiΦj + εij , i=1, …. . , I, j =1, …. . , J Φj = efecto de afinidad de probe

MEDICIÓN DE EXPRESIÓN RMA ( log scale Robust Multi-array Analysis) MODELO ADITIVO DE ESCALA LOGARÍTMICA T(PMij) = ei + aj + εij , i=1, …. . , I, j =1, …. . , J T = transformación de las intensidades PM por background (corrección, normalización, log. ) ei= escala log 2 del valor de expresión en arreglos i=1, …. , I aj= escala log efecto afinidad para probes j=1, …. , J ei se estima por un método robusto (median polish) Normalización y corrección de background según: Irizarry, R. et. al. (2003) Exploration, normalization, and summaries of high density oligonucleotide array probe level data, Biostatistics.

PROBLEMA DE MM SUBSTRACTION (background) Substracción de MM no siempre es apropiado: Irizarry, R. et. al. (2003) Exploration, normalization, and summaries of high density oligonucleotide array probe level data, Biostatistics. Naef, F. et. al. (2002) Empirical characterization oof the expression ratio noise structure in high-density oligonucleotide arrays, Genome Biology, 3, RESEARCH 0018. Hay información del binding no específico en la señal MM Pero. …. empíricamente la substración matemática no se traduce en la substracción biológica del background Hasta que no haya soluciones mejores……. . MEJOR IGNORAR MM !!

EXPERIMENTOS Comparación entre MAS 5. 0, d. Chip y RMA datos de experimentos de DILUCIÓN y SPIKE-IN previamente realizados CRITERIO DE COMPARACION: i) Precisión de la medida de expresión (desviación standard entre chips replica) ii) Consistencia de fold change (entre diferentes concentraciones de c. RNA hibridizadas al chip) iii) Especificidad y sensibilidad de la habilidad de la medición en la detección de expresión diferencial (curvas ROC, Receiver Operating Characteristic)

EXPERIMENTOS DE DILUCIÓN* Hibridizaciones por separado de c. RNA de higado humano c. RNA de línea celular proveniente del CNS X 6 concentraciones diferentes (1. 25, 2. 5, 5. 0, 7. 5, 10. 0 y 20. 0 μgrs. de c. RNA / vol. ) X 5 réplicas por clase de c. RNA n = 60 total * http: //qolotus 02. genelogic. com/datasets. nsf/
EXPERIMENTOS DE SPIKE-IN Affymetrix http: //www. affymetrix. com/analysis/download_center 2. affx DOS familias de datos Gene. Logic http: //qolotus 02. genelogic. com/datasets. nsf/

EXPERIMENTOS DE SPIKE-IN Affymetrix 14 spiking c. RNAs control humanos agregados a concentraciones conocidas Cuadrado latino de 14 grupos de arreglos x 14 grupos de concentraciones que rotan (0, 0. 25, 0. 5, 1, 2, 4, 8, 16, 32, 64, 128, 256, 512 y 1024 p. M), 3 replicas por condición, n=42 total

EXPERIMENTOS DE SPIKE-IN Gene. Logic 11 spiking c. RNAs control agregados a un background común de c. RNA de tejido humano AML Bio. B-5, Bio. B-M, Bio. B-3, Bio. C-5, Bio. C-3 y Bio. Dn-5 (E. coli) Cre. X-5, Cre. X-3 (fago P 1) Dap. X-5, Dap. X-M, y Dap. X-3 (B. subtilis) cuadrado latino cíclico de 12 x 12 concentraciones (0. 5, 1, 1. 5, 2, 3, 5, 12. 5, 25, 37. 5, 50, 75 y 100 p. M), 3 réplicas por condición, n=36 total
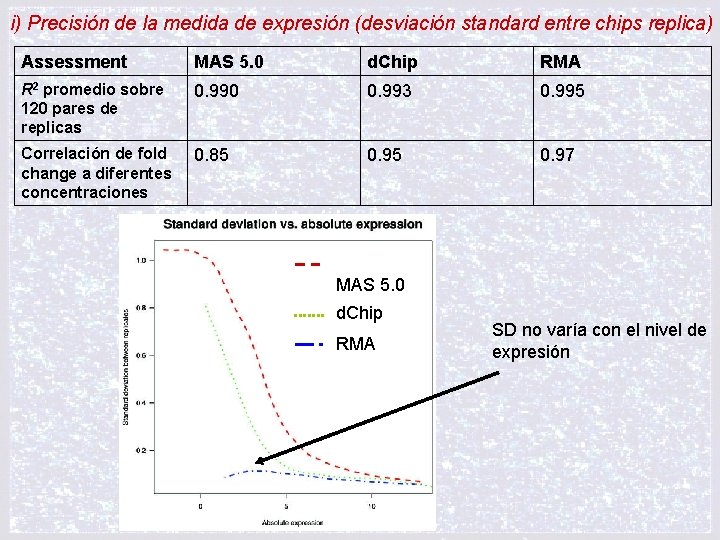
i) Precisión de la medida de expresión (desviación standard entre chips replica) Assessment MAS 5. 0 d. Chip RMA R 2 promedio sobre 120 pares de replicas 0. 990 0. 993 0. 995 Correlación de fold change a diferentes concentraciones 0. 85 0. 97 MAS 5. 0 d. Chip RMA SD no varía con el nivel de expresión
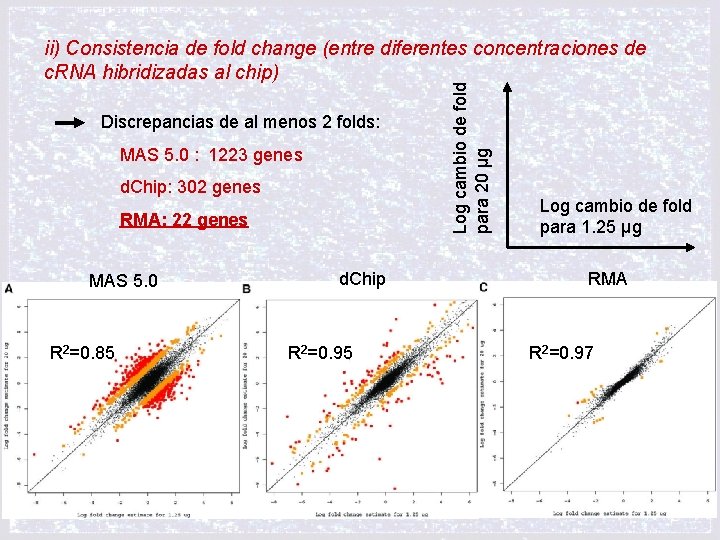
Discrepancias de al menos 2 folds: MAS 5. 0 : 1223 genes d. Chip: 302 genes RMA: 22 genes MAS 5. 0 R 2=0. 85 d. Chip R 2=0. 95 Log cambio de fold para 20 μg ii) Consistencia de fold change (entre diferentes concentraciones de c. RNA hibridizadas al chip) Log cambio de fold para 1. 25 μg RMA R 2=0. 97

iii) Especificidad y sensibilidad de la habilidad de la medición en la detección de expresión diferencial (curvas ROC, Receiver Operating Characteristic) 10 pares de arreglos elegidos random de las familias de datos de Affymetrix y Gene. Logic Estimaron fold change con MAS 5. 0, d. Chip y RMA Amplio rango de Cut-offs FALSOS POSITIVOS y VERDADEROS POSITIVOS genes NO spike in que presentan fold change mayor que el cut-off 1 - ESPECIFICIDAD genes spike in que presentan fold change mayor que el cut-off SENSIBILIDAD
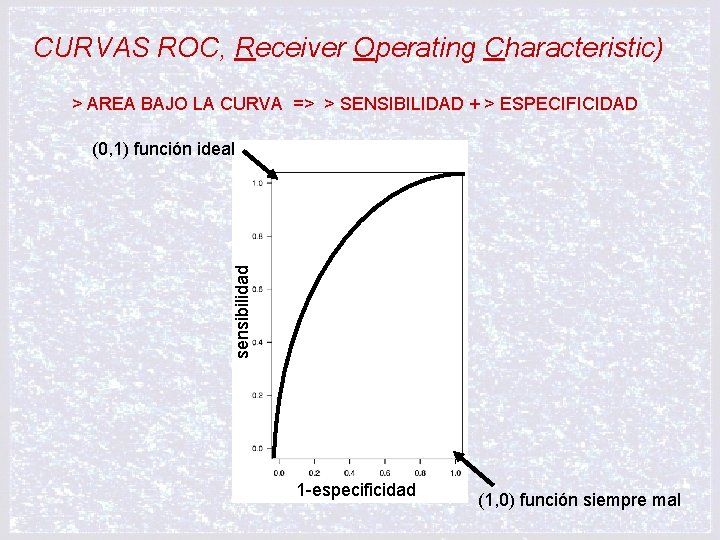
CURVAS ROC, Receiver Operating Characteristic) > AREA BAJO LA CURVA => > SENSIBILIDAD + > ESPECIFICIDAD sensibilidad (0, 1) función ideal 1 -especificidad (1, 0) función siempre mal
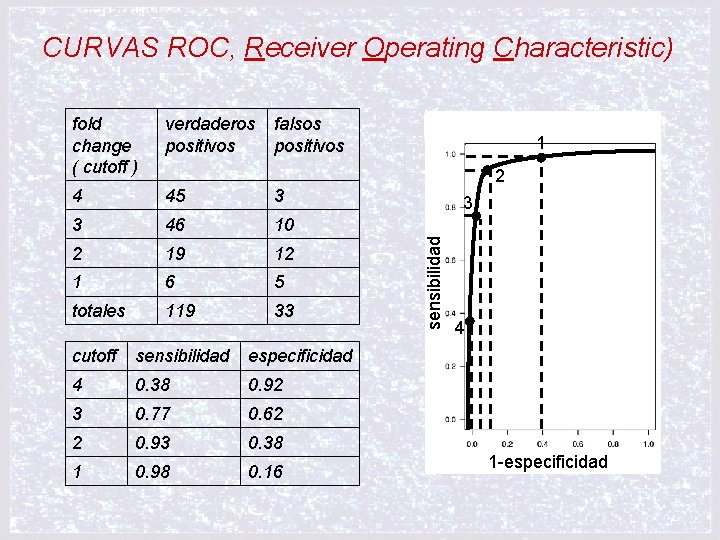
CURVAS ROC, Receiver Operating Characteristic) verdaderos positivos falsos positivos 4 45 3 3 46 10 2 19 12 1 6 5 totales 119 33 cutoff sensibilidad especificidad 4 0. 38 0. 92 3 0. 77 0. 62 2 0. 93 0. 38 1 0. 98 0. 16 1 2 3 sensibilidad fold change ( cutoff ) 4 1 -especificidad
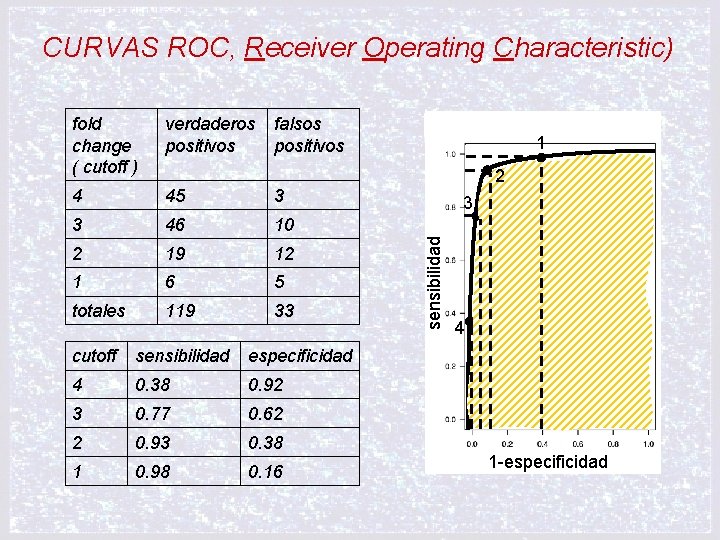
CURVAS ROC, Receiver Operating Characteristic) verdaderos positivos falsos positivos 4 45 3 3 46 10 2 19 12 1 6 5 totales 119 33 cutoff sensibilidad especificidad 4 0. 38 0. 92 3 0. 77 0. 62 2 0. 93 0. 38 1 0. 98 0. 16 1 2 3 sensibilidad fold change ( cutoff ) 4 1 -especificidad
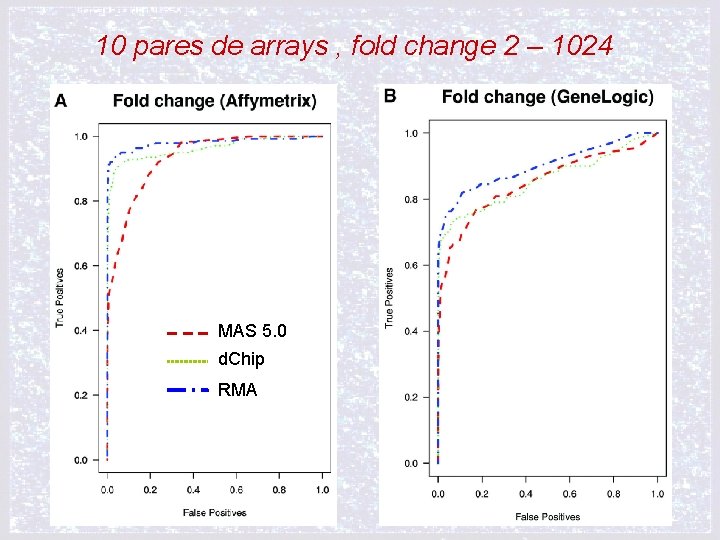
10 pares de arrays , fold change 2 – 1024 MAS 5. 0 d. Chip RMA
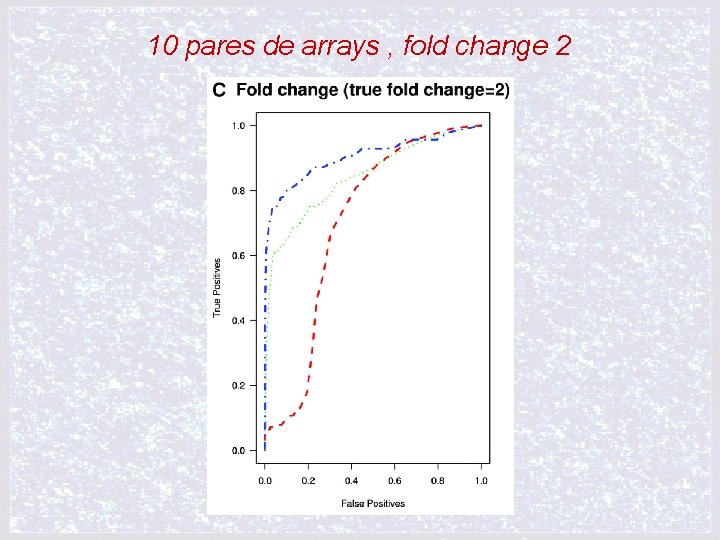
10 pares de arrays , fold change 2

Affymetrix ofrece una alternativa para calcular cambios en la expresión por fold change Valor P de estadística no paramétrica Crean un valor estadístico para RMA y d. Chip basado en estimados del error standard del probe level data COMPARAN EL P DE Affymetrix con los valores de RMA y d. Chip en curvas ROC
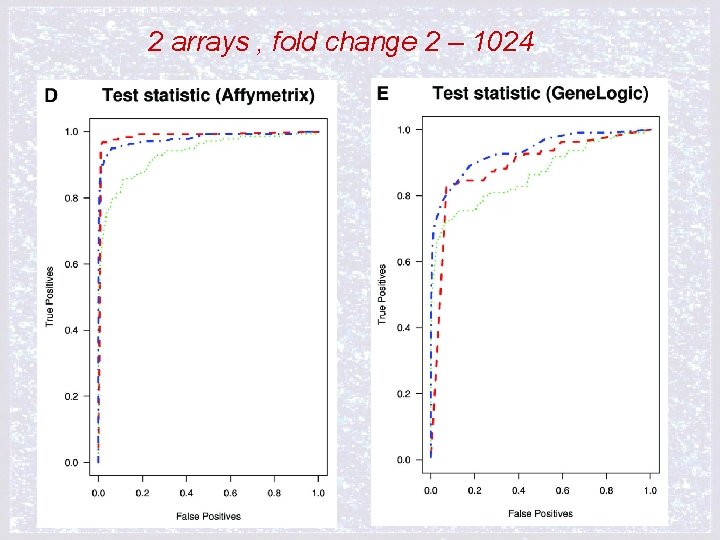
2 arrays , fold change 2 – 1024
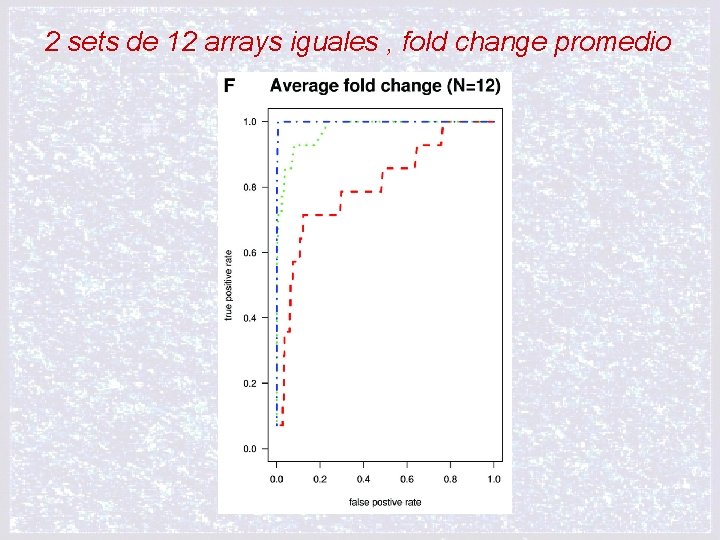
2 sets de 12 arrays iguales , fold change promedio
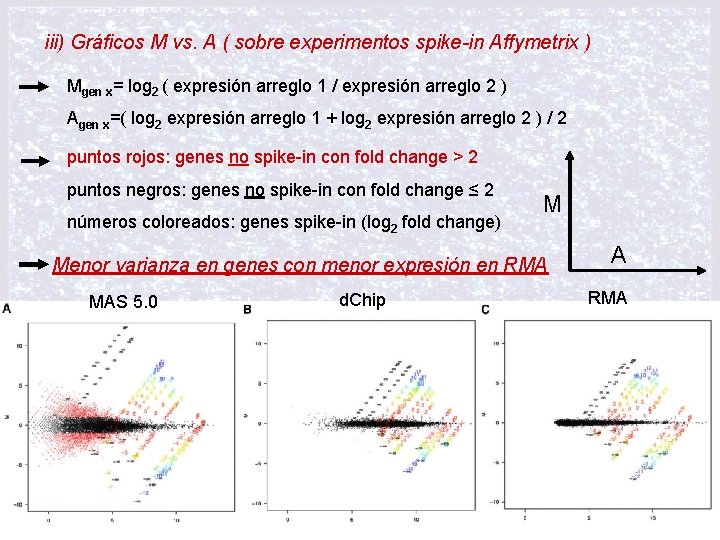
iii) Gráficos M vs. A ( sobre experimentos spike-in Affymetrix ) Mgen x= log 2 ( expresión arreglo 1 / expresión arreglo 2 ) Agen x=( log 2 expresión arreglo 1 + log 2 expresión arreglo 2 ) / 2 puntos rojos: genes no spike-in con fold change > 2 puntos negros: genes no spike-in con fold change ≤ 2 números coloreados: genes spike-in (log 2 fold change) M Menor varianza en genes con menor expresión en RMA MAS 5. 0 d. Chip A RMA
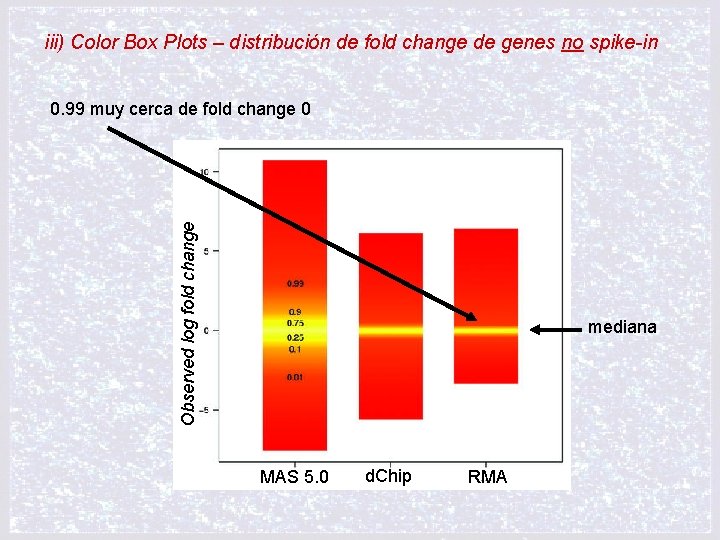
iii) Color Box Plots – distribución de fold change de genes no spike-in Observed log fold change 0. 99 muy cerca de fold change 0 mediana MAS 5. 0 d. Chip RMA

Conclusiones - Discusión RMA mide mejor que d. Chip y MAS 5. 0 respectivamente. RMA tiene mejor precisión particularmente para valores de expresión bajos. RMA estima mejor fold changes. RMA tiene mejor sensibilidad y especificidad para detectar diferencias de expresión por fold change.
- Slides: 29